家蚕热激蛋白HSP90的鉴定、进化与表达模式*
房守敏
(西华师范大学生命科学学院,南充 637002)
家蚕热激蛋白HSP90的鉴定、进化与表达模式*
房守敏
(西华师范大学生命科学学院,南充 637002)
热激蛋白90是一类广泛分布与各类生物体中、高度保守的分子伴侣。HSP90s具有帮助受损蛋白的转运、折叠,防止聚集并恢复其正常构象等功能。昆虫HSP90s与生长发育、滞育和杀虫剂抗性等均相关。本研究在全基因组水平对家蚕HSP90s进行了鉴定,共分析获得3个基因(BGIBMGA004612、BGIBMGA012753和BGIBMGA001241)。根据系统发生分析,对家蚕HSP90s分别命名为BmHSP90A1、BmHSP90B1和BmTrap1,它们分属于3个亚家族。家蚕与黑腹果蝇HSP90s直系同源基因的蛋白序列一致性均在60%以上。与其他物种HSP90s一样,家蚕HSP90s含有N-端ATPase结构域,4个特征性序列。基于组织和发育时期芯片及表达序列标签,发现家蚕HSP90s基因均有表达。基于芯片数据,BmHSP90A1在雌雄间具有非常相似的表达模式,而BmTrap1在5龄幼虫精巢中的信号值高达16 082.5,暗示着该基因可能与精巢发育相关。HSP90s作为一类功能广泛的分子伴侣,本研究将为其功能研究奠定基础。
家蚕;热激蛋白90;表达模式;序列分析;结构域
热激蛋白(又称热休克蛋白,Heat shock proteins,HSPs)是应激后细胞内优先合成的一组蛋白质,在正常状态下的细胞中也广泛存在,参与调控、维持细胞内多种蛋白的构象和功能[1-2]。热激蛋白是生物体耐受冷热应激的重要抗逆蛋白之一,生物体在应激过程中,热激蛋白能确保新合成的蛋白正确折叠,以及帮助变性蛋白的重折叠[3]。按照分子量大小可以分为不同的家族:15~30kDa之间的小热激蛋白small HSPs、HSP60s、HSP70s、HSP90s和HSP110s五大类[4-7]。HSP90在正常或胁迫条件下的各类细胞胞质中都有存在,作为陪伴蛋白与变性蛋白结合,维持它们的折叠状态。HSP90能与类固醇激素受体、酪氨酸激酶、丝氨酸-酪氨酸激酶等信号传导蛋白相互作用并形成复合体,使其具有生物活性[8-10]。在人体内,HSP90与多种癌症,如白血病(leukemia)、全身红斑性狼疮(systemic lupus erythematosus)及多药耐药性相关[11]。
HSP90s在分子进化中高度保守,根据物种和细胞器的分布特征,该家族可以分为5个亚家族HSP90A、HSP90B、HSP90C、TRAP(tumor necrosis factor receptor-associated protein)和HTPG(high temperature protein G)[3]。研究表明,HTPG仅分布于真细菌(Eubacterial),HSP90C则是特异分布于植物叶绿体的类型。而HSP90A、HSP90B和TRAP则是指分布位于胞质、内质网和线粒体的HSP90s。相对而言,HSP90A、HSP90B的分布较广,在原生生物、植物、真菌和动物中均有发现[3]。通常,胞质型HSP90A的C-端含有保守的特征性MEEVD基序[12]。大多数的内质网型HSP90B的C-端则含有KDEL四个氨基酸特征基序,其作为内质网定位信号,但并不是所有的HSP90B均含有此保守的基序[3]。
家蚕(Bombyxmori)是一种重要的经济昆虫,随着季节的改变,家蚕的生长和发育也将遭受温度变化的考验。HSP90s作为重要的环境适应性蛋白,其诱导表达可能帮助家蚕幼虫适应剧烈的环境温度变化及尽量克服其对家蚕带来的伤害[13]。然而,HSP90s作为重要的一类蛋白,其受温度和其他诱导因子的表达反应及其功能并未有报道。本研究主要对家蚕基因组中可能的HSP90s基因进行鉴定,并与其他昆虫的HSP90s比较分析,利用已有的芯片和表达序列标签(expressed sequence tags,ESTs)数据分析其表达模式,旨在为后续对HSP90s基因的功能分析奠定基础。
1 材料与方法
1.1 HSP90基因的鉴定
在Pfam(http://pfam.sanger.ac.uk/)下载HSP90s保守域蛋白序列PF00183.14,利用HMMER 3.0 软件(HMMER 3.0,http://hmmer.janelia.org/)在家蚕基因组预测的蛋白数据库(Silkdb, http://silkworm.swu.edu.cn/silkdb/)搜索潜在的HSP90s序列。并利用在线工具Pfam和SMART(http://smart.embl-heidelberg.de/)分析上述候选基因是否含有HSP90s保守域。
GenBank(http://www.ncbi.nlm.nih.gov/)数据库中下载黑腹果蝇(Drosophilamelanogaster)和蜜蜂(Apismellifera)[3]的HSP90s蛋白序列,与家蚕基因组序列作tBlastn同源性检索。提取含有候选基因的scaffold区段,利用FGENESH/FGENESH+(http://linux1.softberry.com/)软件预测基因的编码区(coding sequence,CDS)和推导的氨基酸序列。同样以Pfam和SMART在线工具分析预测的基因是否含有HSP90s保守域。我们对HSP90s编码的蛋白分子量MW/等电点pI在http://www.expasy.ch/tools/pi_tool.html工具网站进行了预测。
为了比较家蚕与其他鳞翅目昆虫HSP90s基因进化的关系,我们在数据库http://www.heliconius.org/和http://monarchbase.umassmed.edu/分别下载了诗神袖蝶(Heliconiusmelpomene)和帝王蝶(Danausplexippus)的基因组和预测的基因数据,利用家蚕HSP90s的鉴定方法,对2种蝴蝶的HSP90s进行了相似的分析。
1.2 系统发育分析
使用Clusta lx1.8 软件对黑腹果蝇、蜜蜂及本研究鉴定家蚕、诗神袖蝶和帝王蝶HSP90s蛋白序列进行了多序列比对。采用分子进化遗传分析软件MEGA6.0[14]构建HSP90s的邻近系统发生树(neighbor-joining phylogentic tree),选用Jones-Taylor-Thornton(JTT)模型,序列中的缺失采用成对删除(pair deletion)的方式计算两两序列间的遗传距离。另外,拓扑树各分支节点均进行重抽样检验( Bootstrap估算,重复1 000次),检验分子系统树各处的置信度。
2 结果与讨论
2.1 家蚕HSP90s的鉴定与进化分析
Pfam(http://pfam.sanger.ac.uk/)下载HSP90s隐马尔可科夫模型,利用HMMER搜索家蚕基因组预测的蛋白数据库,并通过保守域的分析,共鉴定出BGIBMGA004612、BGIBMGA012753和BGIBMGA001241三个基因。基于早期对黑腹果蝇和蜜蜂HSP90s的鉴定[3],我们在GenBank中下载黑腹果蝇和蜜蜂的HSP90s蛋白序列,并与家蚕基因组tBlastn比对搜索,提取与HSP90s同源的scaffold进行基因预测。Blast搜索与HMMER方法获得了一致的HSP90s基因序列。因此,家蚕基因组中共鉴定出3个HSP90s基因。利用相同的分析方法,获得了诗神袖蝶(HMEL006062、HMEL006610和HMEL017770)和帝王蝶(DPOGS213899、DPOGS206511和DPOGS211870)的HSP90s。
我们将鉴定的家蚕、诗神袖蝶和帝王蝶及已知的黑腹果蝇和蜜蜂HSP90s蛋白序列构建了邻近系统发生树(图1)。结果表明,这些昆虫HSP90s共聚为3簇,分别是HSP90A、HSP90B和TRAP,这些物种间的HSP90s表现出较明显的直系同源关系,表明这些热激蛋白在物种分化前已经产生,在物种形成后可能由于功能约束等原因保存着较低的进化速率。除蜜蜂HSP90A在物种内发生了一次基因扩增外,其他几个物种的HSP90s基因均只有3个基因。
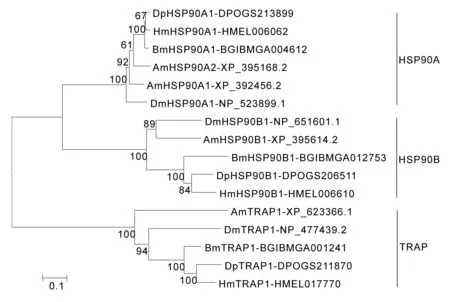
图1 家蚕和其他昆虫HSP90的邻近系统发生树
基于MEGA 6.0构建的邻近(Neighbor-Joining,NJ)树,选取的重要参数为:JTT模型、成对删除gaps及1 000重复;节点处仅显示了>50%的Bootstrap值;HSP90蛋白前面的Dp为帝王蝶(Danausplexippus)的简写;Hm为诗神袖蝶(Heliconiusmelpomene),Bm为家蚕(Bombyxmori);Dm为黑腹果蝇(Drosophilamelanogaster);Am蜜蜂(Apismellifera)。
2.2 HSP90s的保守结构域
HSP90s是一类高度保守、受ATP调节的二聚体分子伴侣。我们对家蚕与黑腹果蝇HSP90s序列相似性进行了计算(表1)。BmHSP90A1与DmHSP90A1的序列一致性高达84.5%,BmHSP90B1与DmHSP90B1为62.6%,BmTrap1与DmTrap1为62.8%。即使不同亚家族成员间的序列一致性也在30%以上。
HSP90s最重要的功能即是在ATP协助下帮助受损蛋白的转运、折叠,防止聚集并恢复其正常构象。HSP90s的N-端含有高度保守的ATPase结构域,是ATP /ADP结合位点,具有内源ATPase活性[15]。家蚕3个HSP90s蛋白在SMART(http://smart.embl-heidelberg.de/)在线预测发现N-端均含有ATPase结构域(图2)。另外,BmHSP90A的C-端含有保守的特征性MEEVD基序,是定位于胞质的特征序列。而KDEL四个氨基酸特征基序作为蛋白定位到内质网的信号,但BmHSP90B的C-端为RDEL,黑腹果蝇为HDEL。因此,HSP90B的C-端定位于内质网的特征基序发生了一定的分化。

表1 家蚕与黑腹果蝇HSP90s氨基酸序列一致性(%)矩阵

“▼”表示功能重要的残基。下划线对应组氨酸激酶样ATP酶区域。“*”标注的是特征性序列[3]。
HSP90s作为高度保守的基因,其编码蛋白除N-端的ATPase结构域外,还含有4个特征性序列(图2)。我们对图1中所有的HSP90s蛋白序列进行了特征性序列保守性分析(图3)。结果表明,特征性序列Signature I和III更为保守,在不同的亚家族间具有基本一致的序列。Signature II和IV在不同亚家族间部分位点发生了较大的分化,但其中功能重要的残基仍然保守(图2、图3)。

基于图1系统发生树中所有的序列进行特征性序列分析,logo图在http://weblogo.berkeley.edu/logo.cgi绘制。
2.3 家蚕HSP90s的表达模式
基于家蚕5龄3d各组织及发育时期的基因芯片数据[16],我们对家蚕的HSP90s基因的表达模式进行了分析。我们将HSP90s的CDS序列与家蚕芯片探针数据库进行Blastn比对,三个基因均找到了对应的探针序列,组织与发育时期芯片数据的表达模式如图4。BmHSP90B1基因在已有的芯片数据中未探测到其表达(信号值<400),但将其CDS与GenBank中家蚕表达序列标签(expressed sequence tags,ESTs)作Blastn搜索时,发现了大量来源不同组织的ESTs,因此,BmHSP90B1也是一个具有活性的热激蛋白。BmHSP90A1和BmTrap1基因在各发育时期和及5龄3d组织中均有表达信号(图4),它们在正常生理条件下也具有高水平表达,表明它们兼有持家基因的功能,而对细胞中的蛋白正确折叠等起保护的作用。BmHSP90A1在雌雄间具有非常相似的表达模式,而BmTrap1在5龄幼虫精巢中的信号值为16 082.5,而在雌性卵巢中的表达信号仅为2 647.83。BmTrap1在精巢呈现如此高的表达,是否与精巢发育、精子发生等相关有待进一步研究。
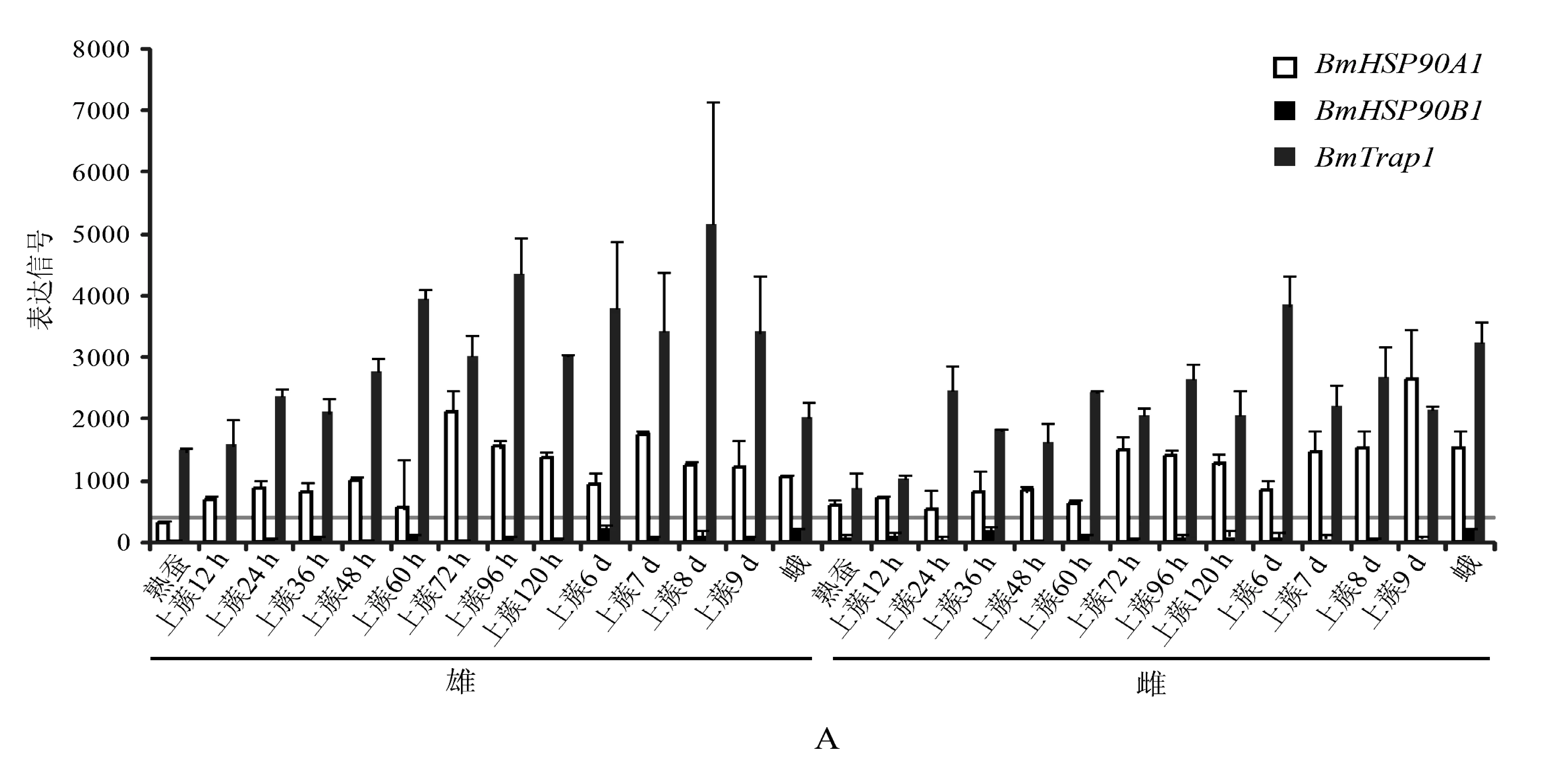
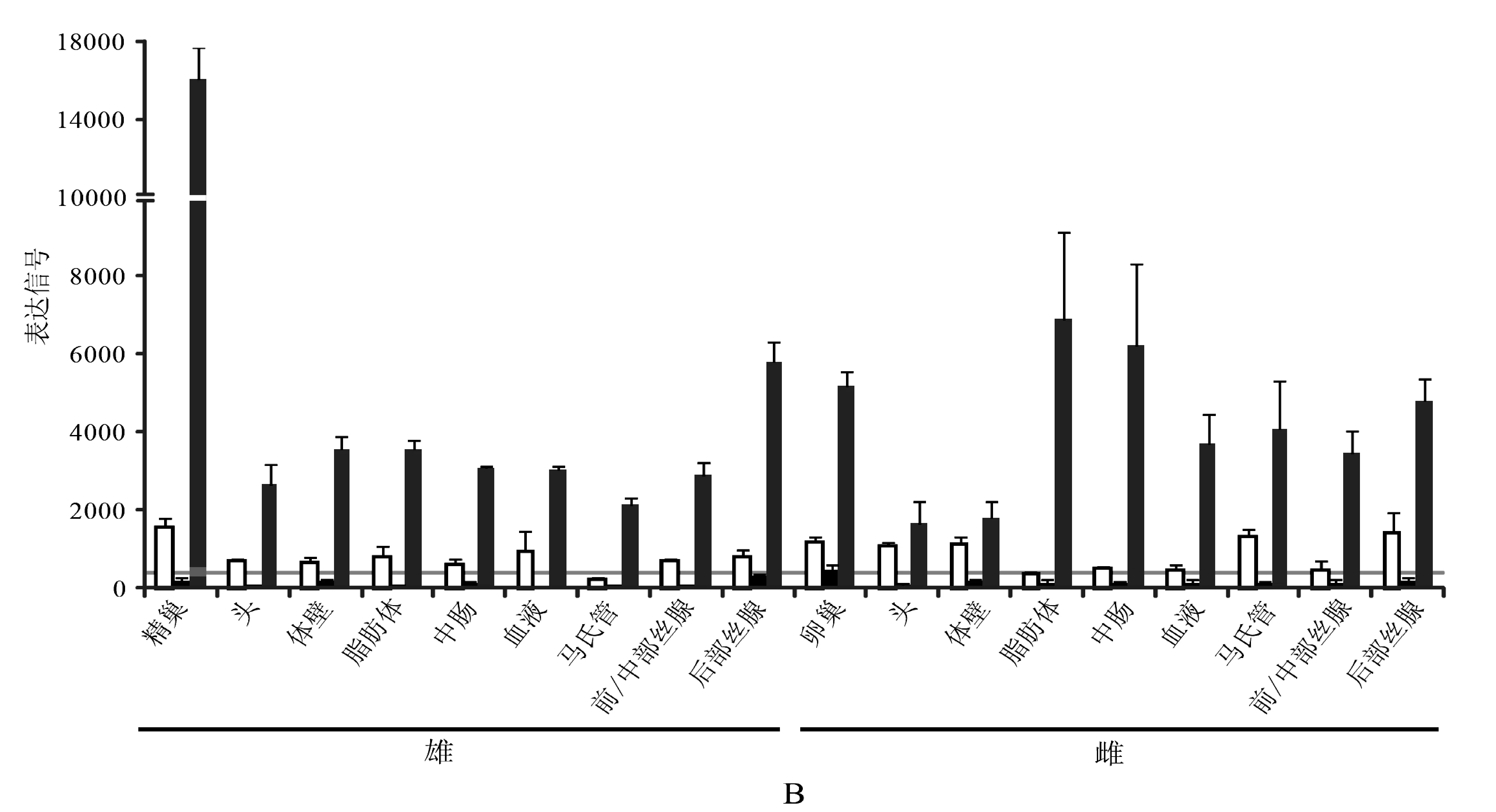
芯片数据下载自Silkdb。A)发育时期芯片数据;B)5龄3天幼虫各组织中的表达。表达信号值≥ 400即视为有表达[16]。
尽管昆虫HSP90s的生理功能研究起步较晚,但目前已经取得了一定的进展[17]。一方面,昆虫的热激蛋白基因可能在昆虫的生长发育、滞育期间、种群密度等方面起着一定的作用[17]。另一方面,昆虫HSP90s可能作用于维持蛋白的结构或活化蜕皮激素,与滞育是密切关联。另外,HSP90s蛋白可能与昆虫杀虫剂抗性相关,可能防止蛋白降解、抑制细胞膜中脂类的过氧化等。因此,HSP90s是保护细胞维持正常功能的重要蛋白之一,随着深入研究昆虫HSP90s在逆境条件下的功能,对农林害虫防治有着重要的指导意义和实际应用价值。
[1] Feder M E, Hofmann G E.Heat-shock proteins, molecular chaperones, and the stress response: evolutionary and ecological physiology[J]. Annu Rev Physiol, 1999,61:243-82.
[2] Frydenberg J, Hoffmann A A, Loeschcke V.DNA sequence variation and latitudinal associations in hsp23, hsp26 and hsp27 from natural populations ofDrosophilamelanogaster[J]. Mol Ecol, 2003,12(8):2025-32.
[3] Chen B, Zhong D, Monteiro A.Comparative genomics and evolution of the HSP90 family of genes across all kingdoms of organisms[J]. BMC Genomics, 2006,7:156.
[4] Craig E A, WeissmanJ S. Horwich A L.Heat shock proteins and molecular chaperones: mediators of protein conformation and turnover in the cell[J]. Cell, 1994,78(3):365-372.
[5] Tavaria M, Gabriele T, Kola I,etal. A hitch-hiker’s guide to the human Hsp70 family[J]. Cell Stress Chaperones, 1996,1:23-28.
[6] Gupta R S, Singh B.Phylogenetic analysis of 70 kD heat shock protein sequences suggests a chimeric origin for the eukaryotic cell nucleus[J]. Curr Biol., 1994,4:1104-1114.
[7] Becker J, Craig E A.Heat shock proteins as molecular chaperones[J]. Eur J Biochem, 1994,219:11-23.
[8] Wegele H, Muller L, Buchner J.Hsp70 and Hsp90-a relay team for protein folding[J]. Rev Physiol Biochem Pharmacol, 2004,151:1-44.
[9] Jackson S E, Queitsch C, Toft D.Hsp90:from structure to phenotype[J]. Nat Struct Mol Biol, 2004,11(12):1152-5.
[10]Zuehlke A, Johnson J L.Hsp90 and co-chaperones twist the functions of diverse client proteins[J]. Biopolymers, 2010,93(3):211-7.
[11]Csermely P, Schnaider T, Soti C,etal. The 90-kDa molecular chaperone family: structure, function, and clinical applications. A comprehensive review[J]. Pharmacol Ther, 1998,79(2):129-68.
[12]Gupta R S.Phylogenetic analysis of the 90 kD heat shock family of protein sequences and an examination of the relationship among animals, plants, and fungi species[J]. Mol Biol Evol, 1995,12(6):1063-73.
[13]Manjunatha H B, Rajesh R K, Aparna H S.Silkworm thermal biology:a review of heat shock response, heat shock proteins and heat acclimation in the domesticated silkworm,Bombyxmori[J]. J Insect Sci, 2010,10:204.
[14]Tamura K, Stecher G, Peterson D,etal. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol, 2013,30(12):2725-9.
[15]Nadeau K, Das A, Walsh C T.Hsp90 chaperonins possess ATPase activity and bind heat shock transcription factors and peptidyl prolyl isomerases[J]. J Biol Chem, 1993,268(2):1479-87.
[16]Xia Q, Cheng D, Duan J,etal.Microarray-based gene expression profiles in multiple tissues of the domesticated silkworm,Bombyxmori[J]. Genome Biol,2007, 8:R162.
[17]张珂, 翁群芳,付昊昊.昆虫热激蛋白90 的研究进展[J].生物技术通报, 2014:15-23.
Identification, evolution and expression patterns of HSP90 in the silkworm
FANG Shou-Min
(CollegeofLifeScience,ChinaWestNormalUniversity,Nanchong637002)
Heat shock protein 90 is a kind of highly conserved molecular chaperones which are widely distributed in a variety of organisms. HSP90s help the damaged proteins to transport, fold and prevent aggregation. Insects HSP90s are associated with growth and development, diapause and insecticide resistance, etc. In this study, HSP90s were identified in the whole genome of the silkworm. Three genes (BGIBMGA004612,BGIBMGA012753 andBGIBMGA001241) were characterized. Based on the phylogenetic analysis, the three silkworm HSP90s were namedBmHSP90A1,BmHSP90B1 andBmTrap1, which belong to three subfamilies. The orthologous HSP90s of silkworm andDrosophilamelanogastershared more than 60 percent of amino acid sequence identity. Silkworm HSP90s also contain N-terminal ATPase domain and four signature sequences. Based on tissue and developmental microarray and expression sequence tags, we found all silkworm HSP90s genes were expressed. In the microarray data,BmHSP90A1 showed similar expression pattern between male and female. Expression signal ofBmTrap1 reached to 16082.5, which suggested that it might be related to development of testis. HSP90s family is one kind of chaperone with wide range of functions, this research will provide the foundation for functional studies.
Bombyxmori; Heat shock protein 90; Expression pattern; Sequence analysis; Domain
*资助项目:西华师范大学基本科研业务费专项资金资助(13D001)。

