猪LTβR基因克隆、结构功能预测、组织表达谱分析及真核表达载体构建
吴正常,戴超辉,殷学梅,孙寿永,2,包文斌,2*,吴圣龙,2*
(1.扬州大学动物科学与技术学院 江苏省动物遗传繁育与分子设计重点实验室,扬州 225009;2.江苏省种猪繁育和健康养殖工程技术研究中心,扬州 225009)
猪LTβR基因克隆、结构功能预测、组织表达谱分析及真核表达载体构建
吴正常1,戴超辉1,殷学梅1,孙寿永1,2,包文斌1,2*,吴圣龙1,2*
(1.扬州大学动物科学与技术学院 江苏省动物遗传繁育与分子设计重点实验室,扬州 225009;2.江苏省种猪繁育和健康养殖工程技术研究中心,扬州 225009)
为了进一步探究猪LTβR基因的生物学功能,本研究以猪淋巴组织cDNA为模板扩增LTβR基因CDS区,进一步利用生物信息学对猪LTβR蛋白结构与功能以及参与的GO和Pathway通路进行分析,利用Real-time PCR检测LTβR基因在大白猪不同组织中的表达水平,同时将LTβR基因扩增产物克隆至真核表达载体pEGFP-C1中,并分别转染猪HEK293和IPEC-J2小肠上皮细胞系,通过显微镜观察其表达情况。结果表明,克隆获得猪LTβR基因CDS区全长为1 209 bp,编码402个氨基酸,发现LTβR为脂溶亲水性的不稳定蛋白,存在1个跨膜结构(205~227 aa),不存在信号肽,亚细胞定位表明LTβR为非分泌型蛋白。LTβR蛋白具有2个糖基化位点,18个潜在的磷酸化位点,其中包括PKC、CKI、CDC2、GSK3、CDK5、INSR等10种保守的特异性蛋白质激酶的结合位点。该蛋白保守区域为2个TNFR superfamily,分别位于32~125和128~192位氨基酸,并发现C422T>A141V突变位于该区域内。KEGG和GO分析发现,LTβR基因共参与12项GO功能分类,同时还参与NF-kappa B信号通路、IgA 合成的肠道免疫网络等5项调控通路。定量检测发现,LTβR基因在大白猪肺中的表达水平极显著高于其他组织(P<0.01),在肾和脾等免疫器官,十二指肠和空肠等肠道组织中也均有较高的表达水平。细胞水平转染试验及显微镜观察发现其在猪HEK293和IPEC-J2两种细胞中均表达。本研究为猪LTβR基因及其介导的信号通路的功能分析提供了材料和依据,今后有必要在细胞水平上进一步验证分析猪LTβR基因及其介导的信号通路在猪抵抗细菌感染过程中发挥的重要作用,同时将C422T突变作为猪抗病育种的一个潜在遗传标记进行系统分析。
猪;LTβR基因;克隆;真核表达载体;蛋白结构与功能
肠道是动物防止感染的第一道防线,肠道内各种免疫成分为其提供多方面、多层次的防护,构成了严密的免疫屏障,这就是肠道免疫系统,也叫黏膜免疫系统。肠道免疫系统是由免疫细胞、免疫球蛋白(IgA)及肠道微生物组成,其中猪肠道系统功能中起关键作用的是由B细胞转化为浆细胞后产生的IgA,它是肠道免疫屏障的一个重要组成部分,同时对肠道菌群也有调节作用。淋巴毒素(Lymphotoxin,LT)是淋巴细胞受抗原或有丝分裂原等刺激活化后及在某些肿瘤、自身免疫病的情况下分泌的一种细胞因子,分为LTα和LTβ两种亚基。学者利用LTα-/-的小鼠模型揭示,LTα基因在淋巴组织生成中发挥了重要作用[1],T.Shimazu等成功克隆出猪脾的LTβ基因,并初步认为LTβ基因在初生仔猪的小肠免疫系统发育中发挥了重要作用[2],因此分析LTβ基因受体的功能及作用机制显得尤为重要。
淋巴毒素β受体(LTβR),又名肿瘤坏死因子受体超家族成员(TNFRSF3),是一个富含半胱氨酸,具有类似肿瘤坏死因子受体结构域的膜蛋白。LTβR基因在淋巴器官的形成过程中,如肠系膜淋巴结的形成及次级淋巴结构和功能重建中,起着非常重要的作用[3-4]。研究报道,LTβR敲除小鼠体内血浆和肠道IgA显著减少,同时LTβR-/-小鼠缺乏所有的淋巴组织,包括结肠相关淋巴结,脾边缘区丢失,B/T细胞分离以及滤泡树突状细胞缺失等[5]。现有研究表明,可溶性的淋巴毒素α(s-LTα3)以及膜结合淋巴毒素β(LTαβ2)可以诱导IgA的生成,从而支持机体的免疫反应。LTβR是肠道IgA产生所必需的,W.Kang等发现,LTβR信号通路对肠道固有层细胞因子的适度表达以及对产生IgA的浆细胞的归巢起着重要的作用[6];此外,H.Guo等[7]利用疾病相关的eQTL定位结合贝叶斯定理筛选出6个免疫疾病调控相关基因,其中就包括LTBR基因。以上研究表明,LTβR基因对肠道免疫中IgA调控以及机体免疫疾病起关键作用。目前有关猪LTβR基因的序列、表达模式以及结构功能等研究相对较少,本研究根据GenBank数据库公布的猪LTβR基因序列(来源于电子克隆)进行mRNA分子克隆,在此基础上利用生物信息学分析猪LTβR蛋白序列特征,构建LTβR真核表达载体并进行细胞转染试验,同时进行LTβR基因的组织表达谱分析,旨在探讨猪LTβR基因的结构与功能,同时也为下一步细胞水平上的功能验证奠定基础。
1 材料与方法
1.1 试验材料
本试验选取的8头35日龄大白断奶仔猪来自常州康乐农牧有限公司(国家生猪核心育种场),屠宰后取11个组织样(心、肝、脾、肺、肾、胃、肌肉、胸腺、淋巴结、十二指肠和空肠组织),现场液氮保存,然后转移至-70 ℃冰箱备用。采用Trizol法提取总RNA,并利用NanoDrop 1000 Spectrophotometer(NanoDrop Technologies,Inc.,Wilmington,DE,USA)测定其OD值,并对OD260 nm/OD280 nm值为1.8~2.0的进行总RNA反转录,合成cDNA。
1.2 生化试剂
质粒小量快速提取试剂盒、切胶回收试剂盒和PCR纯化试剂盒均购自天根生化科技(北京)有限公司(TIANGEN);T4连接酶和限制性内切酶均购自赛默飞世尔科技(中国)有限公司;脂质体LipofectaInineTM2000购自Invitrogen公司(美国)。DL2000 marker、pMD19-T载体、Trizol和反转录试剂盒均购自宝生物工程(大连)有限公司;pEGFP-C1载体购自Clontech公司(Canada,Catalog#6084);JM110感受态细胞购自深圳市百恩维生物科技有限公司,DH5α感受态细胞、HEK293和IPEC-J2小肠上皮细胞由本实验室保存。
1.3 目的基因克隆及真核载体构建
根据GenBank数据库中猪LTβR序列(GenBank登录号:XM_005653161.1),采用Primer 5.0软件设计克隆引物,引物序列为上游:5′-CCTCCCATCTTCCTCCCAGG-3′,下游:5′-GGCTTC-TCCCAGACACCATC-3′,以猪淋巴组织cDNA为模板扩增LTβR基因,预期扩增片段大小为1 376 bp。PCR产物经1%琼脂糖凝胶电泳鉴定、回收后,克隆至pMD19-T载体上,连接产物转化为DH5α感受态细胞,琼脂平板培养菌落,37 ℃培养过夜。经PCR初步鉴定为阳性的菌液送往生工生物工程(上海)有限公司测序鉴定。
1.4 真核重组表达质粒的构建和鉴定
将测序正确的阳性克隆扩大培养,提取pMD19-T-LTβR重组质粒用xbaⅠ和AccⅠ双酶切克隆到pEGFP-C1载体,回收目的片段经纯化回收后,在T4 DNA连接酶作用下4 ℃连接过夜。连接产物转化至JM110感受态细胞中,涂布于含氨苄青霉素(100 μg·mL-1)LB平板,37 ℃倒置培养过夜。挑取单克隆摇菌提取质粒,进行双酶切鉴定并测序。
1.5 细胞培养及转染
利用普通质粒小抽试剂盒抽提重组质粒,猪HEK293和小肠上皮细胞系IPEC-J2分别在2 mL完全培养液(F12∶DMEM=1∶1,10%胎牛血清)进行培养,培养条件为37 ℃、5% CO2。按照1.0×106个·孔-1的密度接种于6孔板,培养至70%~80%时进行转染处理。参照脂质体LipofectaInineTM2000说明书,转染24 h后显微镜观察。
1.6 生物信息学分析
利用生物信息学手段分析LTβR蛋白的基本理化性质、基本结构、功能位点和区域,分别利用ProtParam(http://web.expasy.org/protparam/)进行LTβR蛋白的基本理化性质(氨基酸组成、分子量以及等电点等)分析; SignalP4.0(http://www.cbs.dtu.dk/services/SignalP/)进行蛋白的信号肽分析;TMHMM 2.0(http://www.cbs.dtu.dk/services/TMHMM-2.0/)进行跨膜分析;Anthepro蛋白分析软件基于Garnier方法进行蛋白的二级结构分析;在线软件NetNGlyc(http://www.cbs.dtu.dk/services/NetNGlyc/)分析LTβR蛋白糖基化位点;NetPhos(http://www.cbs.dtu.dk/services/NetPhos/)分析潜在磷酸化位点;NetPhosK 1.0(http://www.cbs.dtu.dk/services/NetPhosK/)分析保守的特异性蛋白激酶作用位点;利用PSORTⅡPrediction(http://psort.hgc.jp/)分析蛋白亚细胞定位;利用NCBI的CDD数据库[8]分析LTβR蛋白的功能结构域;利用KEGG和GO数据库分析LTβR基因所参与的GO功能和pathway通路。
1.7 猪LTβR基因组织表达谱检测
根据克隆的猪LTβR和已发表的GAPDH基因序列[9-10]设计荧光定量引物,采用Primer express 2.0 软件设计引物,LTβR基因上游引物:5′-GAAC-CACCTCTCCATCTGCC-3′,下游引物:5′-GTCCCAGAAGACGCAGAACA-3′,预期扩增片段长为133 bp;内参GAPDH基因上游引物:5′-ACATCATCCCTGCTTCTACTGG-3′,下游引物:5′-CT-CGGACGCCTGCTTCAC-3′,预期扩增片段长为187 bp,所有引物由生工生物工程(上海)有限公司合成。荧光定量PCR反应体系为20 μL:cDNA 1 μL,上下游引物各0.4 μL(10 μmol·L-1),ROX Reference Dye Ⅱ(50×) 0.4 μL,SYBR Green Real time PCR Master Mix(2×) 10 μL,ddH2O 7.8 μL。PCR反应条件:95 ℃ 15 s;95 ℃ 15 s;62 ℃ 34 s,40个循环;4 ℃保存,扩增结束后分析熔解曲线。运用熔解曲线上是否具有(85±0.8)℃ Tm峰来判断PCR扩增的单一性,采用相对定量的方法计算每个组织的表达水平,相对定量的结果采用2-ΔΔCt法[11]进行处理,2-ΔΔCt法计算公式:ΔΔCt =(待测组目的基因平均Ct值-待测组看家基因平均Ct值)-(对照组目的基因平均Ct值-对照组看家基因平均Ct 值),即每一个组织目标基因表达经内参均一化处理后相对于某个组织的倍数。每份样本进行3次实时PCR检测,取平均值。
2 结 果
2.1 猪LTβR基因CDS扩增及克隆测序
PCR扩增产物经1%琼脂糖凝胶电泳鉴定,获得单一目的条带,与预期扩增的片段大小(1 376 bp)一致,见图1A,将克隆产物与pMD19-T载体连接,送往上海生工生物工程有限公司进行测序。测序结果显示,该克隆的LTβR基因包含1 209 bp的CDS序列,编码402个氨基酸(图2);Blast分析显示,该克隆所包含的大白猪LTβR基因cDNA序列与GenBank提供的预测序列同源性达99.85%,并发现两处突变位点,分别为错义突变C422T(A141V)和沉默突变A432G,见图3。
2.2 重组质粒双酶切鉴定及细胞转染
用xbaI 和AccI双酶切pEGFP-C1-LTβR重组质粒及空载体,双酶切鉴定后测序,连接正确的即为阳性克隆(图1B)。pEGFP-C1-LTβR重组质粒分别转染猪HEK293和IPEC-J2小肠上皮细胞,在倒置荧光显微镜下观察可见靶细胞发出绿色荧光(图4),这表明通过重组表达质粒转导入细胞的LTβR过表达片段均可在两类细胞中有效表达,且HEK293细胞转染效率较高。
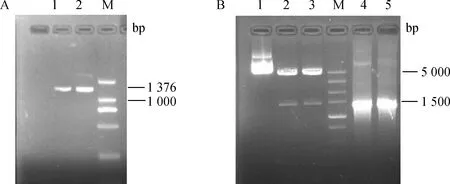
A.猪LTβR基因CDS区扩增图:M.DNA相对分子质量标准DL2000;1、2.LTβR基因编码区的扩增产物。B.PEGFP-C1-LTβR重组质粒的双酶切图:M.DNA相对分子质量标准DL5000;1.重组质粒的扩增产物;2、3.重组质粒PEGFP-C1-LTβR经酶切后的产物;4、5.LTβR基因编码区扩增产物A.The clone CDS result of pig LTβR gene:M.DL2000 marker;1 and 2.The amplification products of LTβR CDS sequence.B.The endonucleases digestion of PEGFP-C1-LTβR:M.DL5000 marker;1.The amplification product of PEGFP-C1-LTβR;2 and 3.The products after endonucleases digestion of PEGFP-C1-LTβR;4 and 5.The amplification products of LTβR CDS sequence图1 LTβR基因扩增产物及双酶切鉴定Fig.1 PCR products and endonucleases identification of LTβR gene
2.3 猪LTβR蛋白的基本理化性质和结构分析
利用在线软件分析发现,猪LTβR蛋白分子式为C1897H2952N534O586S32,分子质量约为43.64 ku,原子总数为6 001个,理论等电点pI为5.42,带正电荷和负电荷的氨基酸残基数分别为29和46个。蛋白质中脂肪链氨基酸含量表示脂溶指数(Aliphatic index),主要由Leu、Ile和Val组成,可以代表蛋白的稳定性,本研究发现LTβR蛋白脂溶指数为34.59,表明该蛋白是脂溶性蛋白。不稳定系数(Instability index)为49.92(>40),表明该蛋白不稳定。总平均亲水性(GRAVY)为-0.433,表明该蛋白为亲水性蛋白。猪LTβR蛋白最大疏水性位于第218位氨基酸,最大亲水性位于第29位氨基酸,整体来看亲水性区域大于疏水性(图5);猪LTβR蛋白中205~227位氨基酸之间含有一个跨膜结构,228~402位氨基酸属于胞内区,其余的为胞外区(图6);猪LTβR蛋白不存在信号肽,表明为非分泌蛋白;蛋白二级结构包括α螺旋(Alpha helix)73个,百分比为18.16%,β延伸链(Extended strand)66个,百分比为16.42%,无规则卷曲(Random coil)263个,百分比为65.42%。
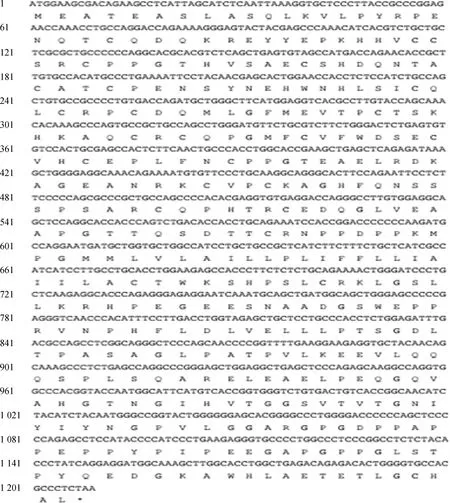
图2 猪LTβR基因编码序列及其编码氨基酸序列Fig.2 The coding sequence and amino acid sequence of pig LTβR

图3 猪LTβR基因编码区SNPs位点筛查Fig.3 Screening of SNP sites in pig LTβR CDS

A、B分别代表了pEGFP-C1-LTβR质粒侵染HEK293、IPEC-J2小肠上皮细胞荧光显微镜观察结果;左图表示荧光视野下的图片,右图表示可见光视野下的图片A,B represent fluorescence microscope observation of HEK293 and IPEC-J2 intestinal epithelial cells infected by pEGFP-C1-LTβR,respectively;left figures represent the photos under the view of fluorescence,right figures represent the photos under the view of visible light图4 LTβR重组质粒侵染HEK293和IPEC-J2小肠上皮细胞荧光显微镜观察 100×Fig.4 Fluorescence microscope observation of HEK293 and IPEC-J2 intestinal epithelial cells infected by LTβR recombinant plasmid 100×
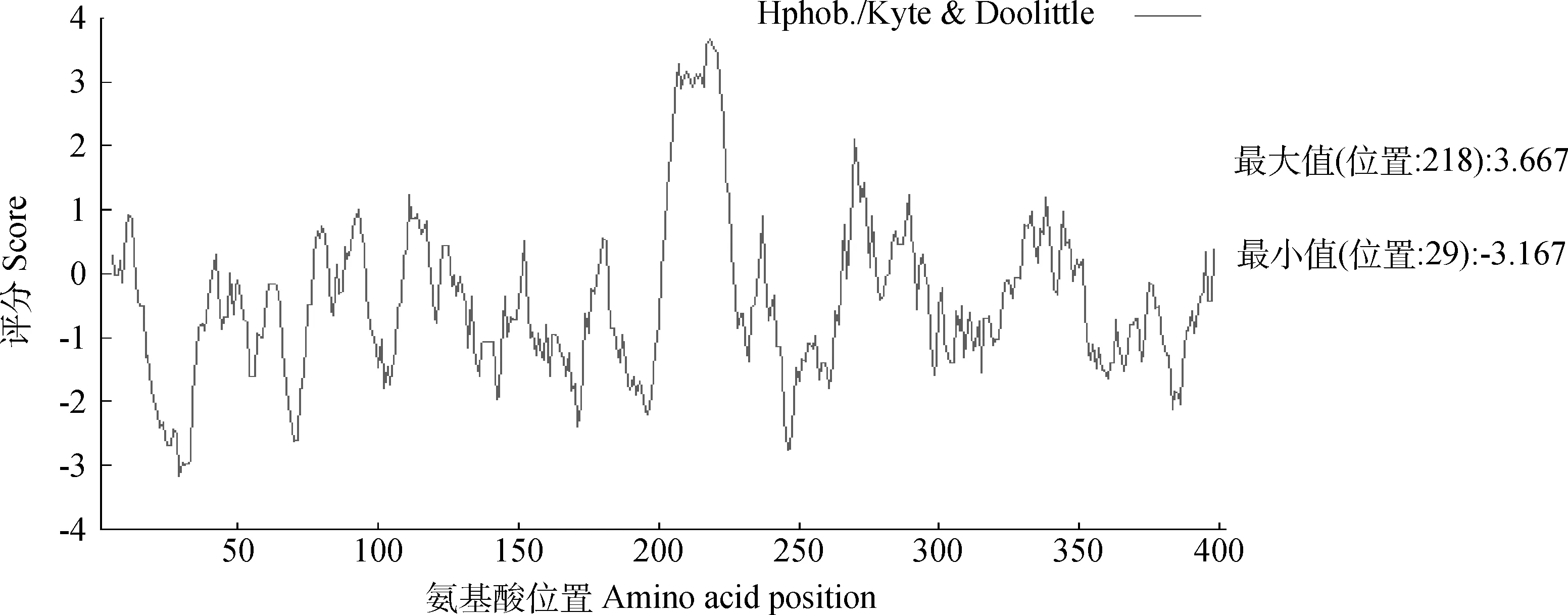
图5 猪LTβR蛋白的亲/疏水性分析Fig.5 Hydrophilic/hydrophobic analysis of pig LTβR protein
2.4 猪LTβR蛋白的功能分析
在线软件分析可知,猪LTβR蛋白存在2个糖基化位点,分别位于第21和158位氨基酸(图7);该蛋白存在18个潜在的磷酸化位点,包括5个Ser、9个Thr和4个Tyr(图8),上述磷酸化位点所对应的磷酸激酶预测结果显示,在LTβR蛋白上可能有PKC、CKI、CDC2、GSK3、CDK5、INSR等10种保守的特异性蛋白激酶的结合位点(表1),其中在226位处PKC分值最高为0.90。亚细胞定位结果显示,猪LTβR蛋白69.6%可能存在于细胞核内,13.0%可能存在质膜上,8.7%可能存在细胞质内,4.3%可能存在高尔基体内,由此进一步推测该蛋白可能为非分泌型蛋白。

图6 猪LTβR蛋白的跨膜结构分析Fig.6 Transmembrane structure analysis of pig LTβR protein
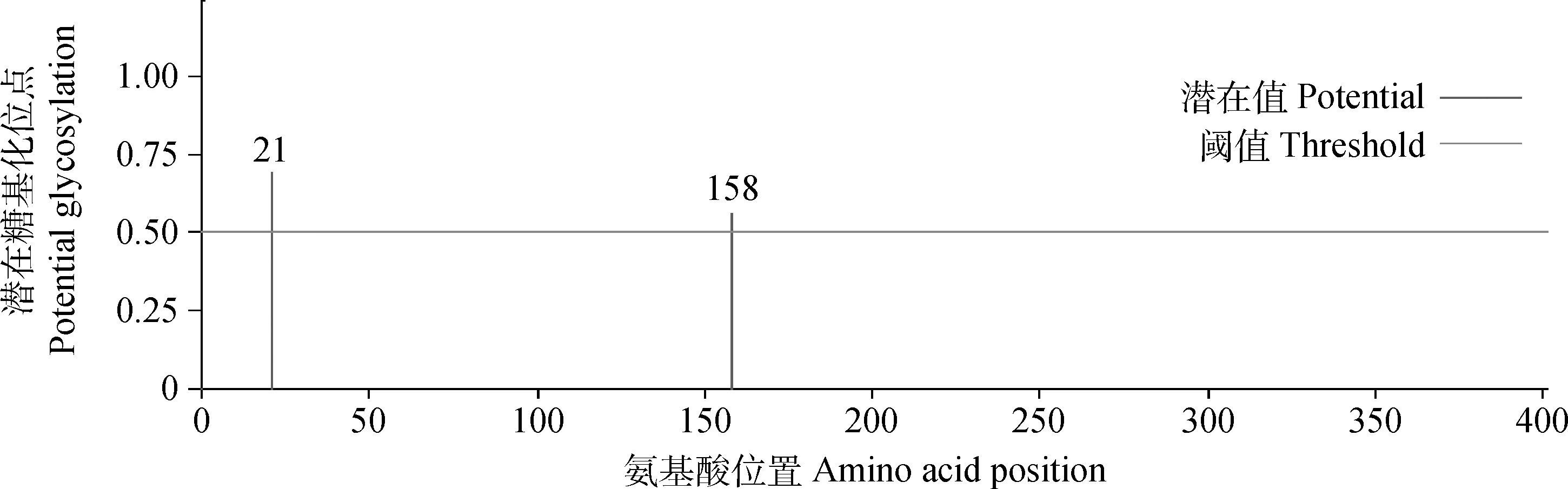
图内横线代表阈值;竖线代表潜在糖基化位点;数字代表氨基酸位置In the figure,abscissa indicate threshold;Vertical line indicate the potential glycosylation sites,number on vertical line indicate the position of amino acids图7 猪LTβR蛋白的糖基化位点预测Fig.7 Prediction of glycosylation sites of pig LTβR protein
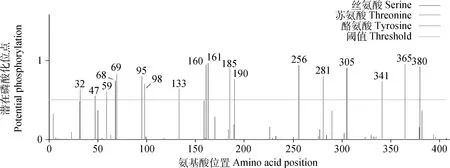
横线代表阈值;各条竖线分别代表潜在的酪氨酸、丝氨酸和苏氨酸磷酸化位点;丝氨酸位点.68、160、161、256、305;苏氨酸位点.47、59、95、98、133、185、190、281、380;酪氨酸.32、69、341、365Abscissa line indicate threshold;Vertical lines indicate potential tyrosine,serine and threonine phosphorylation sites,respectively;Serine sites.68,160,161,256,305;Threonine sites.47,59,95,98,133,185,190,281,380;Tyrosine sites.32,69,341,365图8 猪LTβR蛋白的磷酸化位点预测Fig.8 Prediction of phosphorylation sites of pig LTβR protein
猪LTβR蛋白含有2个TNFR(Tumor necrosis factor receptor)超级家族保守结构域,1个位于32~125位氨基酸,预测评估值E-value为6.16e-28,另一个位于128~192位氨基酸,预测评估值E-value为1.62e-07,该保守结构域由50’s环TNF结合位点(50’s loop TNF binding site)、90’s环TNF结合位点(90’s loop TNF binding site)和3个不同蛋白多肽结合位点5个部分组成。KEGG与GO分析发现,LTβR基因参与分子功能(3个)、细胞组成(1个)以及生物过程(8个)等共12项GO功能分类(表2),同时还参与细胞因子受体相关作用、NF-kappa B信号通路、IgA 合成的肠道免疫网络等5项重要的调控通路(表3)。
表1 猪LTβR保守的特异性蛋白激酶作用位点
Table 1 Conserved and specific phosphkinase binding sites in pig LTβR protein
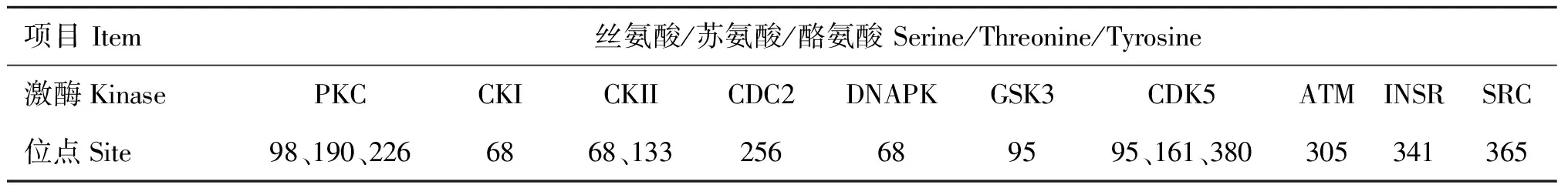
项目Item丝氨酸/苏氨酸/酪氨酸Serine/Threonine/Tyrosine激酶KinasePKCCKICKIICDC2DNAPKGSK3CDK5ATMINSRSRC位点Site98、190、2266868、133256689595、161、380305341365
表2LTβR基因参与的GO功能分析
Table 2 GO function analysis ofLTβRgene
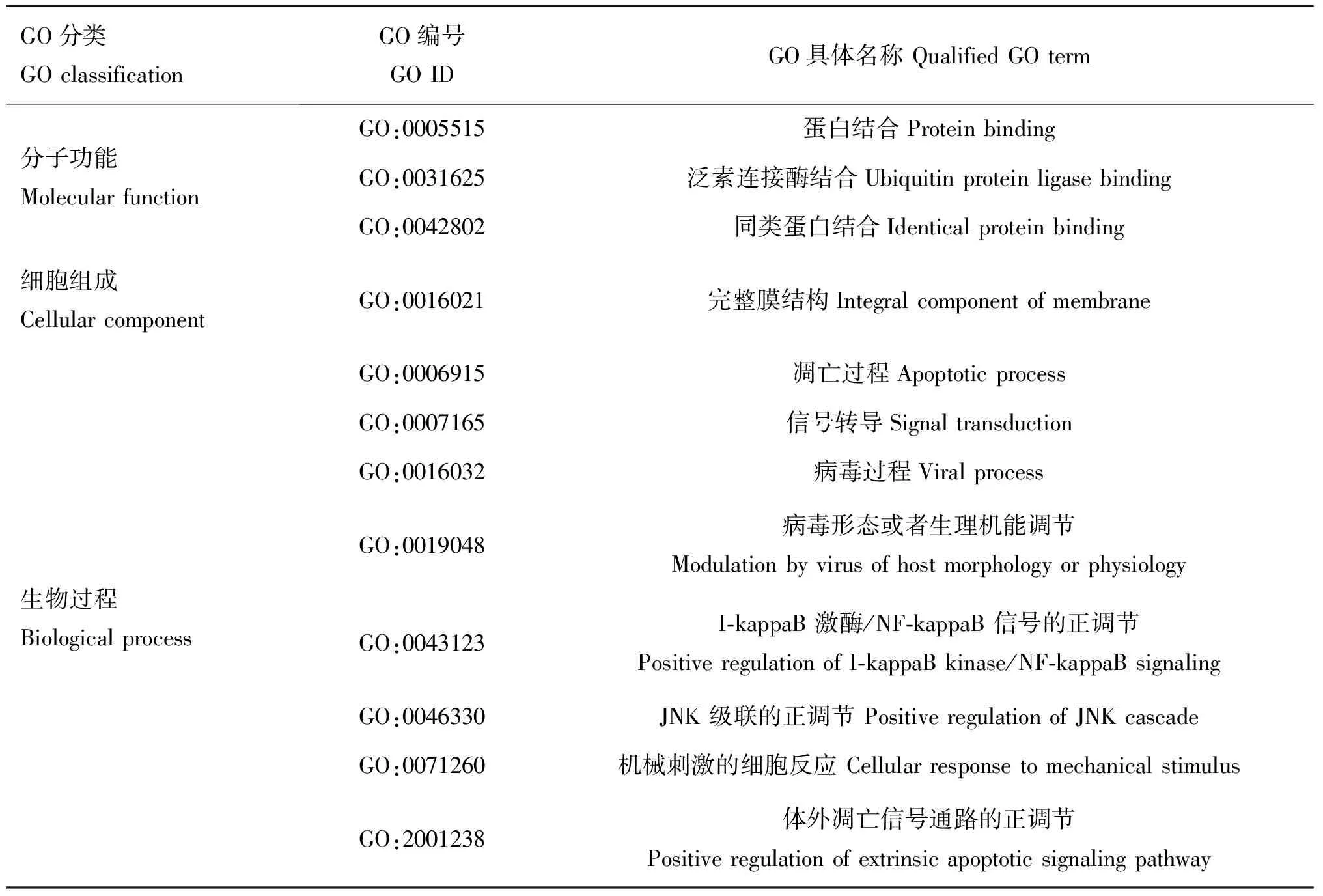
GO分类GOclassificationGO编号GOIDGO具体名称QualifiedGOterm分子功能MolecularfunctionGO:0005515蛋白结合ProteinbindingGO:0031625泛素连接酶结合UbiquitinproteinligasebindingGO:0042802同类蛋白结合Identicalproteinbinding细胞组成CellularcomponentGO:0016021完整膜结构Integralcomponentofmembrane生物过程BiologicalprocessGO:0006915凋亡过程ApoptoticprocessGO:0007165信号转导SignaltransductionGO:0016032病毒过程ViralprocessGO:0019048病毒形态或者生理机能调节ModulationbyvirusofhostmorphologyorphysiologyGO:0043123I⁃kappaB激酶/NF⁃kappaB信号的正调节PositiveregulationofI⁃kappaBkinase/NF⁃kappaBsignalingGO:0046330JNK级联的正调节PositiveregulationofJNKcascadeGO:0071260机械刺激的细胞反应CellularresponsetomechanicalstimulusGO:2001238体外凋亡信号通路的正调节Positiveregulationofextrinsicapoptoticsignalingpathway
表3LTβR基因参与的Pathway分析
Table 3 Pathway analysis ofLTβRgene

通路编号PathwayID通路名称Pathwaytermmap04060细胞因子受体相互作用Cytokine⁃cytokinereceptorinteractionmap04064NF⁃kappaB信号通路NF⁃kappaBsignalingpathwaymap04066HIF⁃1信号通路HIF⁃1signalingpathwaymap04672IgA合成的肠道免疫网络调控通路IntestinalimmunenetworkforIgAproductionmap05166HTLV⁃I感染HTLV⁃Iinfection
2.5 猪LTβRmRNA组织表达特异性
所提取组织的总RNA经2.2%甲醛变性凝胶电泳检测,无明显降解条带及DNA污染条带,ND-1000核酸/蛋白浓度测定仪测定RNA的A260 nm/ A280 nm为1.8~1.9,说明RNA提取质量较高可用于后续试验。组织表达谱检测结果发现(图9),LTβR基因在肺中表达水平极显著高于其他组织(P<0.01),其次为肝、肾和脾等免疫器官,十二指肠和空肠等肠道组织中也有一定的表达。
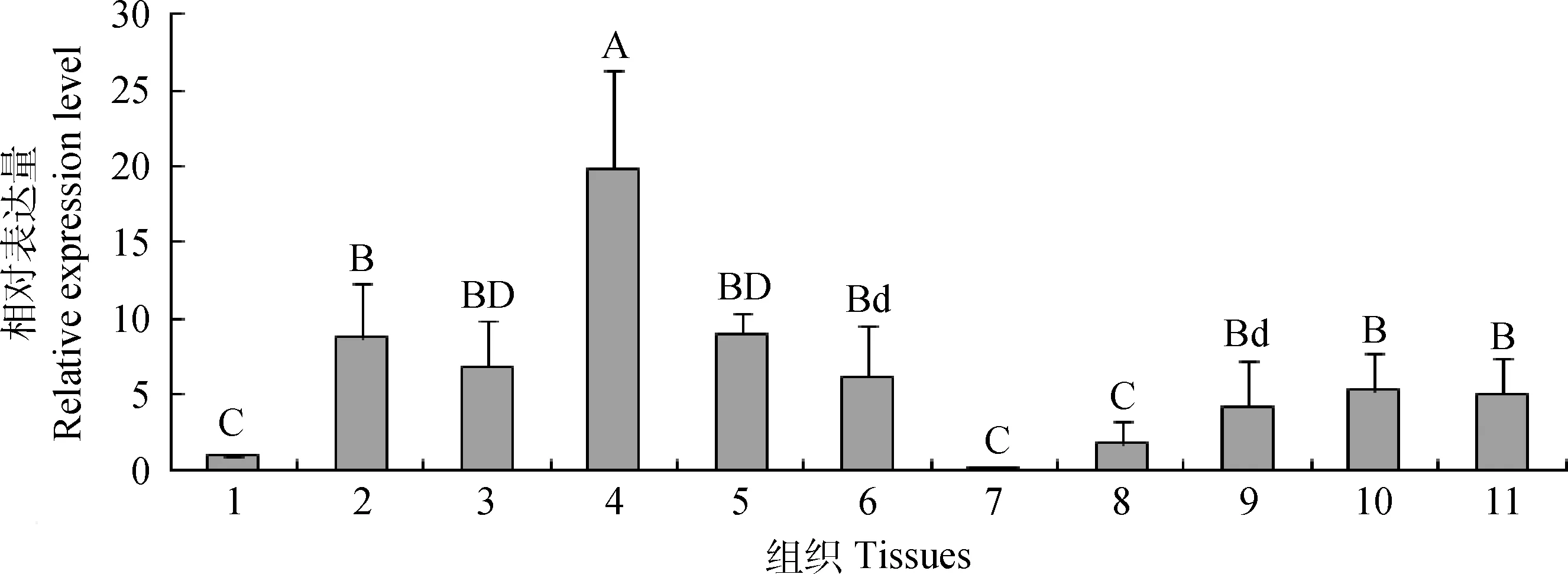
1~11分别表示心、肝、脾、肺、肾、胃、肌肉、胸腺、淋巴、十二指肠和空肠;不同组织间上标包含不同大写字母表示差异极显著(P<0.01),不同小写字母表示差异显著(P<0.05)1-11 represent heart,liver,spleen,lung,kidney,stomach,muscle,thymus,lymphatic,duodenum and jejunum;Superscript between different tissues containing different capital letters mean significant difference (P<0.01),different small letters mean significant difference(P<0.05)图9 LTβR在各组织中的表达分析Fig.9 Expression analysis of LTβR in different tissues
3 讨 论
本研究成功克隆得到大白猪LTβR基因全长CDS区(长度为1 209 bp),共编码402个氨基酸,没有检测到不同的可变剪接。与人、小鼠LTβR比对发现,氨基酸同源性分别为70.3%和66.9%。LTβR表达很广泛,主要表达在初级和次级淋巴样组织,以及肺、肾等内脏器官,外周血没有表达。很多贴壁细胞的原代细胞系(如HEK293、HT-29、U937等)中都能检测到它的表达,本研究成功构建了真核表达载体PEGFP-C1-LTβR,并通过转染荧光检测进一步证实了LTβR基因在猪HEK293细胞中具有较高的表达,同时也发现它在猪小肠上皮细胞IPEC-J2中也有表达。组织表达谱检测发现,LTβR基因在猪肺、肾以及脾等免疫器官中高表达,同时在十二指肠和空肠等肠道组织中也有较高水平的表达。LTβR能够分别特异性地结合LTα和LTβ形成异源三聚体LTα1β2和LIGHT两种配体,在生物体内发挥着不同的生物学功能[12-14]。LTα1β2主要表达在T淋巴细胞表面,通过与间质细胞表面的LTβR结合,参与次级淋巴器官和脾的发育。LTβR与LIGHT结合可诱导肿瘤细胞的凋亡,产生各种细胞因子,在肠系膜淋巴结的形成以及次级淋巴结结构和功能重建过程中起着重要作用,并且研究发现LIGHT是参与调节肠道炎症中不可缺少的物质[15]。鉴于LTβR在免疫器官形成和肠道免疫调控中发挥的作用,本研究从蛋白功能位点和保守区域进一步分析其作用机制,发现猪LTβR蛋白存在2个肿瘤坏死因子受体TNFR超家族保守域,分别位于第32~125和128~192位氨基酸,已有研究发现,TNFR家族蛋白在不同生物系统中细胞凋亡或者程序性细胞死亡中发挥重要的作用[16-18],TNFR区域作为LTβR蛋白主要的功能区域,而本研究发现了1处突变C422T>A141V位于TNFR功能区域,可能对LTβR蛋白功能造成一定的影响,因此C422T突变可能作为今后重点研究LTβR基因作为猪抗病育种的一个潜在遗传标记。
LTβR基因作为肿瘤坏死因子的家族成员,有关学者对其介导的信号通路在细菌微生物感染导致的宿主免疫应答中的重要作用做了大量的研究。目前,利用融合蛋白LTβR-IgG体内阻断LTβR介导的信号通路,是一种常用的研究其生物学功能的手段[19]。R.Lucas等利用牛分枝菌卡介苗(BCG)感染野生型小鼠和LTβR-IgG处理后的小鼠,发现LTβR信号通路的阻断抑制了处理组小鼠巨噬细胞的激活和iNOS合成的活性,从而导致小鼠脾的肉芽肿生成数目减少,这表明LTβR信号通路在牛分枝菌卡介苗感染中对Th1型免疫应答起着重要的促进作用[20]。LTβ-/-小鼠和野生型小鼠分别感染单核细胞增生李斯特菌(Listeria monocytogenes),发现处理小鼠对细菌呈现出了显著的易感性[21]。LTβR介导的信号通路在大肠杆菌等革兰阴性菌,啮齿枸橼酸杆菌(Citrobacter rodentium)引发的传染性结肠炎中也发挥了重要作用[22]。总的来说,阻断LTβR介导的信号通路会导致宿主在细菌感染后清除细菌的免疫机能受到严重损坏。此外,LTβR-IgG处理的小鼠对鼠巨细胞(MCMV)病毒的易感性增强,导致死亡率增高[23]。本研究分析发现,LTβR共参与5个信号通路即细胞因子受体相关作用、NF-kappa B信号通路、HIF-1信号通路、IgA 合成的肠道免疫网络调控通路和HTLV-I感染。LTβR与LTα1β2和LIGHT结合后可以激活NF-kappa B信号通路,并引起肠道淋巴形成等免疫调控[12-14],因此,LTβR介导的NF-kappa B信号通路和IgA 合成的肠道免疫网络调控通路显得尤为重要。本研究成功构建了LTβR基因真核表达载体,并在猪HEK293和IPEC-J2细胞中检测到其表达,今后可以进一步开展过表达以及RNA干扰试验,此外目前最流行的基因定点修饰技术-CRISPR/Cas9已经成功应用于细菌、人类细胞、斑马鱼、小鼠、猪、食蟹猴以及多种植物的基因组精确修饰[24],也可以结合CRISPR/Cas9基因敲除试验从细胞水平上更加全面地分析LTβR介导的信号通路在猪抗细菌感染中的作用机制,为今后从提高机体免疫能力的角度来进行分子选育与抗病育种提供指导和依据。
[1] BANKS T A,ROUSE B T,KERLEY M K,et al.Lymphotoxin-alpha-deficient mice.Effects on secondary lymphoid organ development and humoral immune responsiveness[J].JImmunol,1995,155(4):1685-1693.
[2] SHIMAZU T,TOHNO M,KATOH S,et al.Utilization of the porcine system to study Lymphotoxin-β regulation in intestinal lymphoid tissue[J].BiochemGenet,2009,47(1-2):126-136.
[3] LOCKSLEY R M,KILLEEN N,LENARDO M J.The TNF and TNF receptor superfamilies:integrating mammalian biology[J].Cell,2001,104(4):487-501.
[4] SHAKHOV A N,NEDOSPASOV S A.Expression profiling in knockout mice:lymphotoxin versus tumor necrosis factor in the maintenance of splenic microarchitecture[J].CytokineGrowthFactorRev,2001,12(1):107-119.
[5] FÜTTERER A,MINK K,LUZ A,et al.The lymphotoxin β receptor controls organogenesis and affinity maturation in peripheral lymphoid tissues[J].Immunity,1998,9(1):59-70.
[6] KANG W,KUDSK K A,SANO Y,et al.Effects of lymphotoxin β receptor blockade on intestinal mucosal immunity[J].JParenterEnteralNutr,2007,31(5):358-364.
[7] GUO H,FORTUNE M D,BURREN O S,et al.Integration of disease association and eQTL data using a Bayesian colocalisation approach highlights six candidate causal genes in immune-mediated diseases[J].HumMolGenet,2015,24(12):3305-3313.
[8] MARCHLER-BAUER A,LU S,ANDERSON J B,et al.CDD:a Conserved Domain Database for the functional annotation of proteins[J].NucleicAcidsRes,2011,39(suppl 1):D225-D229.
[9] BARBER R D,HARMER D W,COLEMAN R A,et al.GAPDH as a housekeeping gene:analysis of GAPDH mRNA expression in a panel of 72 human tissues[J].PhysiolGenomics,2005,21(3):389-395.
[10] 吴正常,殷学梅,夏日炜,等.猪杀菌/通透性增加蛋白基因siRNA载体构建及干扰效果评价[J].畜牧兽医学报,2015,46(3):491-496. WU Z C,YIN X M,XIA R W,et al.Construction and evaluation of porcine bactericidal/permeability-increasing protein gene(BPI) siRNA expression vector[J].ActaVeterinariaetZootechnicaSinica,2015,46(3):491-496.(in Chinese)
[11] LIVAK K J,SCHMITTGEN T D.Analysis of relative gene expression data using realtime quantitative PCR and the 2-ΔΔCtmethod[J].Methods,2001,25(4):402-408.
[12] CROWE P D,VANARSDALE T L,WALTER B N,et al.A lymphotoxin-beta-specific receptor[J].Science,1994, 264(5159):707-710.
[13] BROWNING J L,DOUGAS I,NGAM-EK A,et al.Characterization of surface lymphotoxin forms.Use of specific monoclonal antibodies and soluble receptors[J].JImmunol,1995, 154(1):33-46.
[14] MAURI D N,EBNER R,MONTGOMERY R I,et al.LIGHT,a new member of the TNF superfamily,and lymphotoxin alpha are ligands for herpesvirus entry mediator[J].Immunity,1998, 8(1):21-30.
[15] KRAUSE P,ZAHNER S P,KIM G,et al.The tumor necrosis factor family member TNFSF14(LIGHT) is required for resolution of intestinal inflammation in mice[J].Gastroenterology,2014,146(7):1752-1762.
[16] BAKER S J,REDDY E P.Transducers of life and death:TNF receptor superfamily and associated proteins[J].Oncogene,1996,12(1):1-9.
[17] GRUSS H J,DOWER S K.Tumor necrosis factor ligand superfamily:involvement in the pathology of malignant lymphomas[J].Blood,1995,85(12):3378-3404.
[18] SMITH C A,FARRAH T,GOODWIN R G.The TNF receptor superfamily of cellular and viral proteins:activation,costimulation,and death[J].Cell,1994,76(6):959-962.
[19] SPAHN T W,EUGSTER H P,FONTANA A,et al.Role of lymphotoxin in experimental models of infectious diseases:potential benefits and risks of a therapeutic inhibition of the lymphotoxin-β receptor pathway[J].InfectImmun,2005,73(11):7077-7088.
[20] LUCAS R,TACCHINI-COTTIER F,GULER R,et al.A role for lymphotoxin β receptor in host defense against Mycobacterium bovis BCG infection[J].EurJImmunol,1999,29(12):4002-4010
[21] EHLERS S,HÖLSCHER C,SCHEU S,et al.The lymphotoxin β receptor is critically involved in controlling infections with the intracellular pathogens Mycobacterium tuberculosis and Listeria monocytogenes[J].JImmunol,2003,170(10):5210-5218.
[22] SPAHN T W,MAASER C,ECKMANN L,et al.The lymphotoxin-β receptor is critical for control of murineCitrobacterrodentium-induced colitis[J].Gastroenterology,2004,127(5):1463-1473.
[23] BENEDICT C A,BANKS T A,SENDEROWICZ L,et al.Lymphotoxins and cytomegalovirus cooperatively induce interferon-β,establishing host-virus détente[J].Immunity,2001,15(4):617-626.
[24] 刘志国.CRISPR/Cas9系统介导基因组编辑的研究进展[J].畜牧兽医学报,2014,45(10):1567-1583. LIU Z G.Research progress on CRISPR/Cas9 mediated genome editing[J].ActaVeterinariaetZootechnicaSinica,2014,45(10):1567-1583.(in Chinese)
(编辑 郭云雁)
Gene Cloning,Prediction of Structure and Function,Analysis of Tissue Expression Profile and Construction of Eukaryotic Expression Vector of PigLTβRGene
WU Zheng-chang1,DAI Chao-hui1,YIN Xue-mei1,SUN Shou-yong1,2,BAO Wen-bin1,2*,WU Sheng-long1,2*
(1.KeyLaboratoryforAnimalGenetics,Breeding,ReproductionandMolecularDesignofJiangsuProvince,CollegeofAnimalScienceandTechnology,YangzhouUniversity,Yangzhou225009,China;2.JiangsuEngineeringResearchCenterfortheReproductionandHealthyBreedingofBoar,Yangzhou225009,China)
In order to further explore the biological function of pigLTβRgene,the coding sequence(CDS) ofLTβRgene was amplified from the cDNA of porcine lymphoid tissue and the protein structure and function,GO function,regulatory pathways of porcine LTβR were further analyzed by bioinformatics tools.Real-time PCR was used to detect the expression level of porcineLTβRgene in different tissues of Yorkshire.MeanwhileLTβRgene was cloned into the eukaryotic expression vector pEGFP-C1,then transfected into HEK293 cell and small intestinal epithelial cell IPEC-J2,respectively.The expression of recombinant plasmid was detected in both cells through microscopic observation.The results showed that the pigLTβRcDNA full length was 1 209 bp encoding 402 amino acids.LTβR was a fat-soluble,hydrophilic and unstable protein,containing a transmembrane structure between 205 and 227 amino acids,meanwhile no signal peptide and subcellular localization indicated that LTβR protein was non-secretory.LTβR protein had 2 glycosylation sites,18 potential phosphorylation sites including 10 conservative binding sites of specific protein kinase such as PKC,CKI,CDC2,GSK3,CDK5,INSR,etc.Pig LTβR protein had 2 conservative domains named TNFR superfamily,which were located in the region between 32 and 125 amino acid,between 128 and 192 amino acid,respectively.Besides the mutation C422T>A141V occurred in the conservative domain.KEGG and GO analysis showedLTβRgene participated in 12 GO function classifications,5 regulatory pathways such as NF-kappa B signaling pathway,intestinal immune network for IgA production,etc.Real-time PCR analysis showed the expression level ofLTβRgene in Yorkshire lung tissue was very significantly higher than other tissues(P<0.01).Moreover,LTβRgene was also highly expressed in both pig immune organs such as kidney,spleen and intestinal tissue such as duodenum,jejunum.Transfection experiments and microscopic observation showed the pEGFP-C1-LTβR recombinant plasmid was expressed in both HEK293 and IPEC-J2 cells.This study will provides materials and basis for studying the function ofLTβRgene and LTβR-mediated signaling pathways,therefore it is necessary to further verify and analyze the regulatory mechanism and important role ofLTβRgene and signaling pathways at cellular level,meanwhile we should systematically analyze the mutation C422T as a potential genetic marker for pig disease-resistant breeding.
pig;LTβRgene;cloning;eukaryotic expression vector;structure and function of protein
10.11843/j.issn.0366-6964.2015.12.003
2015-01-07
转基因生物新品种培育科技重大专项(2014ZX08006-001B);国家自然科学基金(31372285;31172183);江苏高校优势学科建设工程资助项目(PAPD)
吴正常(1987-),男,江苏南京人,博士生,主要从事猪遗传育种研究,E-mail:wuzhengchang@126.com
*通信作者:包文斌,研究员,博导,主要从事猪遗传育种研究,E-mail:wbbao@yzu.edu.cn;吴圣龙,研究员,博导,主要从事猪遗传育种研究,E-mail:slwu@yzu.edu.cn
S828;S813.3
A
0366-6964(2015)12-2135-11

