麦洼牦牛TLR1~10基因克隆及分子生物学特征分析
林宝山,兰道亮,陈亚冰,黄 偲,符 梅,李 解,李 键
(西南民族大学 生命科学与技术学院/西南民族大学 青藏高原研究院,成都 610041)
麦洼牦牛TLR1~10基因克隆及分子生物学特征分析
林宝山,兰道亮*,陈亚冰,黄 偲,符 梅,李 解,李 键*
(西南民族大学 生命科学与技术学院/西南民族大学 青藏高原研究院,成都 610041)
本研究克隆了麦洼牦牛天然免疫受体1~10基因的编码区,利用生物信息学工具分析基因特点,荧光定量PCR测定TLRs基因在不同组织中的表达量。序列比较分析结果表明,麦洼牦牛TLR基因与其他物种在核苷酸水平及氨基酸水平上均表现出很高的保守性。遗传进化方面,麦洼牦牛TLRs与牛和绵羊TLRs遗传进化距离最近,并与人、马、鼠TLRs等形成哺乳动物的一个分支,与鸡则形成遗传距离较远的一个分支。同时我们在进行系统发育分析时发现,TLR1、TLR6先聚为一小支,再与TLR10又聚为更紧密的一支,然后TLR1、2、6、10和TLR7、8、9分别聚集在两个单个的分支上,TLR其他成员各自成为一支。荧光定量结果表明,TLRs在麦洼牦牛各组织均有表达,但不同成员在不同组织的表达存在较大的差异。其中TLR2、TLR4和TLR6在脾表达量最高,在卵巢、小肠、肾、肝中有高表达,TLR1、TLR5、TLR7、TLR8、TLR9和TLR10在肾表达量最高,在肝、肾、脾等组织中高表达。综上所述,本研究的开展能为以后揭示TLRs在牦牛等高原模式动物分子免疫机制以及牦牛抗病育种奠定基础。
麦洼牦牛;TLR1-10;克隆;荧光定量PCR;组织表达分布
TLR如同天然免疫的眼睛,监视与识别各种不同疾病的相关分子模式 “PAMP”,引发一系列信号转导,进而导致炎性介质的释放,并最终激活获得性免疫系统,是机体抵抗感染性疾病的第一道屏障[1]。近期研究发现,TLRs参与并调控获得性免疫应答[2]。迄今,TLR家族在哺乳动物中至少有13个成员,其中,人类TLR1~10,小鼠TLR1~9、11~13,牛和羊TLR1~10,猪TLR1~10等。TLRs成员各自能够特异性地识别不同的PAMPs/DAMPs(病原体相关分子模式/损伤关联分子模式),并调节先天性与获得性免疫以限制病原微生物的入侵,同时清除宿主产生的机体残留物质。例如TLR2与TLR1或TLR6协同识别革兰阳性菌的肽聚糖、磷壁酸、脂蛋白等;TLR3识别病毒的双链RNA(dsRNA);TLR4识别革兰阴性菌的脂多糖、类脂A、热休克蛋白60/70等;TLR5识别细菌的鞭毛蛋白;TLR7和TLR8识别病毒单链RNA(ssRNA);TLR9识别细菌、病毒、真菌及一些原生动物非甲基化的CpG DNA等[1-3]。
青藏高原是中国最大、世界海拔最高的高原,平均海拔4 000~5 000 m,有“世界屋脊”和“第三极”之称。牦牛是生长在这里的一种特有古老物种,是世居青藏高原藏族人重要的生活和生产资源,也是当地畜牧经济发展的重要支柱。与平原动物相比,牦牛作为高原模式动物能够在青藏高原极度高寒、低氧等恶劣气候环境下生存,其抗病及免疫分子机制可能存在特殊性。同时近年来,牦牛疾病尤其是传染性疾病如犊牛腹泻病、乳房炎、寄生虫性疾病等疾病严重威胁着牦牛群体的健康,影响了牦牛产业的发展。因此研究牦牛分子免疫机制具有重要意义。
目前,小鼠和人类的 Toll样受体已经研究的比较深入了,关于家畜Toll样受体的鉴定和研究在最近几年也取得了较大的进展。K.MeGurie等将牛Toll样受体1~10完成了鉴定并绘制了物理图谱,发现其95%的核苷酸序列和人是一致的[4];M.Menzies等对羊TLR1~10基因进行了克隆与测序[5];A.Raja等对山羊TLR1~10基因进行了测序和分析[6];在猪和马上也都做了相关的研究[7-8],但是TLRs在牦牛上的研究基本空白。鉴于此,本研究克隆了麦洼牦牛TLR1~10基因并进行了相关的预测和分析研究,这为今后进一步揭示牦牛等高原模式动物免疫分子机制和高原动物重大疫病的防控提供依据,同时也为牦牛抗病育种提供重要的基础。
1 材料与方法
1.1 试验材料
本试验所有牦牛组织采自高原屠宰场(中国四川红原县,北纬31°51′~33°19′,东经 101°51′~103°23′,平均海拔 3 600 m),属于麦洼牦牛品种。分别采集雌性麦洼牦牛7头(年龄3~5岁,体重300~400 kg)11种组织样本(心、肝、脾、肺、肾、大肠、小肠、肌肉、胃、卵巢、乳腺),均放入液氮速冻,送回实验室-80 ℃保存。
1.2 方法
1.2.1 总RNA提取和cDNA合成 称取约1 g组织样本至液氮中研磨后,按照RNAprep pure 动物总RNA提取试剂盒(天根生化科技(北京)有限公司)提取总RNA。按照的RevertAidTMFirst Strand cDNA Synthesis Kit反转录试剂盒(Fermentas公司)进行反转录反应,采用20 μL体系:Oligo dT181 μL,5×Reaction Buffer 4 μL,RibolockTMRnase Inhibitor(20 U·μL-1) 1 μL,10 mmol·L-1dNTP Mix 2 μL,RevertAidTMM-MuLV Reverse Transcriptase(200 U·μL-1) 1 μL,模板1 μL,最后用RNase Free dH2O补足20 μL。反应条件:42 ℃ 60 min;70 ℃ 5 min。cDNA于-20 ℃保存备用。1.2.2 引物的设计与合成 麦洼牦牛TLR1~10克隆及荧光定量引物根据GenBank中登陆的牛TLR1~10基因序列,用Premier 5.0软件分别设计,内参引物根据牛β-actin序列设计。引物详细信息见表1、表2。以上所有引物由Invitrogen(上海)公司合成。
1.2.3 目的基因的克隆 每个TLR基因均以麦洼牦牛脾组织cDNA为模版,进行普通PCR扩增,扩增体系为50 μL。所有PCR产物经10 g·L-1琼脂糖凝胶电泳观察扩增结果,用Axygen的凝胶回收试剂盒回收纯化PCR产物,取适量已纯化PCR产物与克隆载体pMD19-T进行连接,转化DH5α感受态细胞并在Amp+琼脂平板上涂菌。挑取单个菌落接种于含Amp+的LB液体培养基中。经PCR鉴定后,将阳性重组质粒送往Invitrogen(上海)公司测序。
表1 基因TLR1~10 的克隆引物序列
Table 1 Primer sequences ofTLR1-10 genes

基因Gene引物名称Nameofprimers引物序列(5'-3')Sequenceofprimers产物长度/bpLengthoffragmentTLR1FRTTTAATTGACAGGTCAAAAAGAGGTTAATGTATTTCTGCTGCTTTTCC2287TLR2FRATGCCACGTGCTTTGTGGGAGTCAGTGATGCCATCCAACC2620TLR3FRGAAAACGAACTGGATTTGGACTAAGGGAGACGTATTTCCATAGAAGAGA2942TLR4FRAGGATGATGGCGCGTGCTCTAATTCAGTTTGATGGGGTAAG2807TLR5FRGGATCATGGGAGACTGCCTTAGGAGATGGTGGTTACATTTTGC2582TLR6FRAGAAGCTGTCATTTTGTTTACATAACCTTTCACATCATCATTTTC2364TLR7FRAGGTGTTTCCAATGTGGACATTCTTTGAACACCTGACTGTAGGT3142TLR8FRCCCTTTTCCTGCTCATCTCTGACGTGAATCGTTGGCTGTTAGGAC3033TLR9FRAGCTCAGACAGAAGACGATCAACGGCAGAAGTTCCGGTTATAG3303TLR10FRATTGTTACGTCTGTGCATAGTGGAACCTCCAAACCCTTCATTCAG2367
表2 基因TLR1~10和β-actin的荧光定量引物序列
Table 2 Real-time PCR primer sequences ofTLR1-10 andβ-actingenes
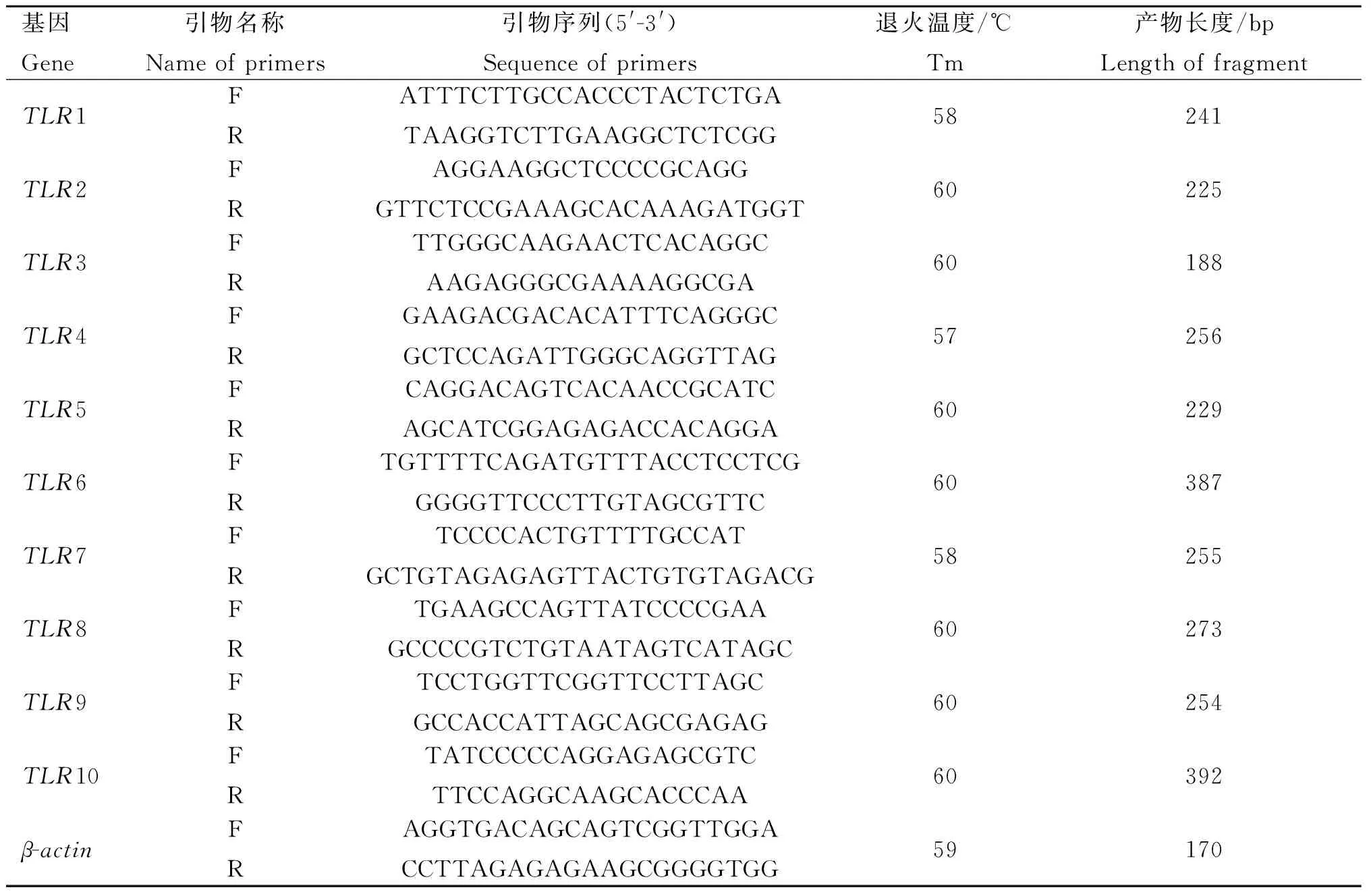
基因Gene引物名称Nameofprimers引物序列(5'-3')Sequenceofprimers退火温度/℃Tm产物长度/bpLengthoffragmentTLR1FRATTTCTTGCCACCCTACTCTGATAAGGTCTTGAAGGCTCTCGG58241TLR2FRAGGAAGGCTCCCCGCAGGGTTCTCCGAAAGCACAAAGATGGT60225TLR3FRTTGGGCAAGAACTCACAGGCAAGAGGGCGAAAAGGCGA60188TLR4FRGAAGACGACACATTTCAGGGCGCTCCAGATTGGGCAGGTTAG57256TLR5FRCAGGACAGTCACAACCGCATCAGCATCGGAGAGACCACAGGA60229TLR6FRTGTTTTCAGATGTTTACCTCCTCGGGGGTTCCCTTGTAGCGTTC60387TLR7FRTCCCCACTGTTTTGCCATGCTGTAGAGAGTTACTGTGTAGACG58255TLR8FRTGAAGCCAGTTATCCCCGAAGCCCCGTCTGTAATAGTCATAGC60273TLR9FRTCCTGGTTCGGTTCCTTAGCGCCACCATTAGCAGCGAGAG60254TLR10FRTATCCCCCAGGAGAGCGTCTTCCAGGCAAGCACCCAA60392β-actinFRAGGTGACAGCAGTCGGTTGGACCTTAGAGAGAAGCGGGGTGG59170
1.2.4 实时荧光定量PCR 实时荧光定量PCR参照β-actin对TLR基因组织表达进行定量检测。实时荧光定量采用20 μL反应体系:SYBR Premix ExTaqTMII(TaKaRa公司)10 μL,上、下游引物各0.8 μL,cDNA 2 μL,dH2O 6.4 μL。使用Bio-Rad公司荧光定量PCR仪进行荧光定量分析。优化处理后每个基因的最佳反应条件:95 ℃预变性30 s;95 ℃变性5 s,X ℃(表2)退火30 s,72 ℃延伸20 s,39个循环;添加一个熔解曲线,设置以dH2O为模板的阴性对照,每个组织进行3次重复。
1.2.5 分析与处理TLR克隆序列应用Lasergene软件包进行分析;蛋白质结构域由SMART在线软件预测分析(http://smart.embl-heidelberg.de/);TLRs同其他物种的多序列同源性比对利用在线软件ClustalW2(http://www.ebi.ac.uk/Tools/msa/clustalw2/);遗传进化关系树的建立应用Cltulax、MEGA5.1软件。荧光定量结果采用相对定量的2-△△CT法进行数据分析并用SpassV19.0软件进行数据处理,最后使用Excel制图功能绘制出麦洼牦牛TLRs各基因的组织相对表达柱状图。
2 结 果
2.1 目的基因的扩增
麦洼牦牛TLR1~10 基因的克隆扩增结果如图1。由图可知,扩增片段大小与预期扩增片段大小基本相符。
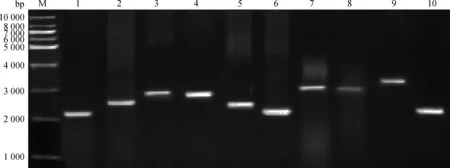
M.DNA相对分子质量标准;1~10.基因TLR1~10扩增的片段M.DNA marker 1kb;1-10.PCR products of TLR1-10 genes图1 基因TLR1~10的PCR扩增结果Fig.1 PCR products of TLR1-10 genes
2.2 蛋白质结构分析
采用SMART在线软件分析TLR1~10分子的结构特征(图2),结果显示,TLR1~10分子结构包含N末端信号肽结构、胞外区的LRRs结构域、跨膜区以及保内去TIR结构域,是典型的TLRs结构特征。但是TLR家族成员所含LRR及TIR结构域数量有很大的差异,其中,TLR3、TLR7、TLR8、TLR9含有数量较多的LRR区域,TLR1包含的LRR区域为最少。通过与牛、羊、人、鸡等不同物种TLRs蛋白功能结构与排布方式比较,发现麦洼牦牛TLR蛋白结构域与牛、羊数量与分布位置较为一致,与其他物种间TLRs蛋白大小相近,但蛋白功能结构域排布方式存在细微差别。
2.3 同源性分析
TLRs同其他物种的多序列同源性比对利用在线软件ClustalW2(http://www.ebi.ac.uk/Tools/msa/clustalw2/)(表3)实施。结果显示,麦洼牦牛TLR基因与其他物种均表现出很高的保守性。在核苷酸水平上,麦洼牦牛与牛和绵羊的同源性均大于96%,与人和鼠的同源性在75%~87%。在氨基酸水平上,麦洼牦牛与牛及绵羊的同源性为90%~99%,与人和鼠的同源性为64%~86%。在所有TLR家族成员中TLR7基因最为保守(氨基酸同源性达到80%~99.71%);而TLR2、TLR4相对较低,分别为66.58%~94.11%、64.07%~92.17%。
2.4 遗传进化树
麦洼牦牛TLR基因遗传进化树的建立利用了MEGA 5.0软件的Neighbor-Joining法,重复1 000次,将克隆所得麦洼牦牛TLR1~10分别与GenBank中登录的牛、羊、人、鼠、马、原鸡TLR1~10序列比对构建Bootstrap验证的系统发育树(图3)。结果显示,麦洼牦牛与牛和绵羊遗传进化距离最近,并与人、马、鼠等形成哺乳动物的一个分支。原鸡则形成与麦洼牦牛遗传距离较远的一个分支。值得一提的是,TLR1、TLR6先聚为一小支,再与TLR10又聚为更紧密的一支,然后TLR1、2、6、10和TLR7、8、9分别聚集在两个单个的分支上,TLR其他成员各自成为一支。

图2 TLR1~10基因蛋白结构域分析结果Fig.2 Protein domains prediction of TLR1-10 genes
2.5 标准曲线的建立
标准曲线以连续10倍倍比稀释(1×102~1×108copies·μL-1)的阳性质粒为模板,经定量PCR扩增后,计算Ct值得出最佳检测区域,并制作各基因的标准曲线。图4可见,TLR1~10和β-actin基因的相关系数(R2)大于0.990,说明Ct值与cDNA浓度间具有良好的线性关系;TLR1~10基因和β-actin基因扩增效率(E)在1.0左右,说明扩增效率基本相同,以上分析说明建立的荧光定量检测方法快速、敏感性高、特意性强等,可用于2-△△ct相对定量的PCR分析。
2.6 荧光定量PCR检测麦洼牦牛TLR1~10基因在各组织的表达分布
采用β-actin为参照基因,SYBR Green I为荧光染料,通过荧光定量PCR方法检测TLR1~10基因在麦洼牦牛各组织的表达分布。数据经分析处理后,绘制的麦洼牦牛TLR1~10基因在各组织相对表达柱状图(图5)。结果显示,麦洼牦牛TLRs在各主要器官组织均有表达,但不同成员在不同组织的表达存在较大的差异。其中TLR1、TLR2、TLR4和TLR6在脾表达量最高在卵巢、小肠、肾、肝中有较高表达,TLR5、TLR7、TLR8、TLR9和TLR10在肾表达量最高,在肝、脾、肺等组织中高表达。与其他组织相比较麦洼牦牛TLR1~10基因除TLR10外,其他成员均在心组织相对表达量最低。同时,通过比较发现麦洼牦牛TLR1、TLR6基因在各组织的表达比较相似,TLR2与TLR4、TLR7与TLR8、TLR9表达比较相似。
3 讨 论
Toll样受体作为连接动物机体天然免疫和获得性免疫的桥梁,通过识别病原体组织和细胞特异性表达模式进而使机体对各种致病挑战作出反应,抵御疾病。牦牛作为藏族人重要的生产生活资源,也是当地的畜牧经济发展不可或缺的重要畜种。其作为高原模式动物对青藏高原恶劣极端气候有较强的适应能力,然而近年来牦牛重大疫病的发生和不断加强的趋势所带来的影响和损失让人担忧。因此,研究牦牛分子免疫机制具有重要理论及实践意义。
表3TLR1~10基因的核苷酸序列同源性比较
Table 3 Alignment ofTLR1-10 gene sequences
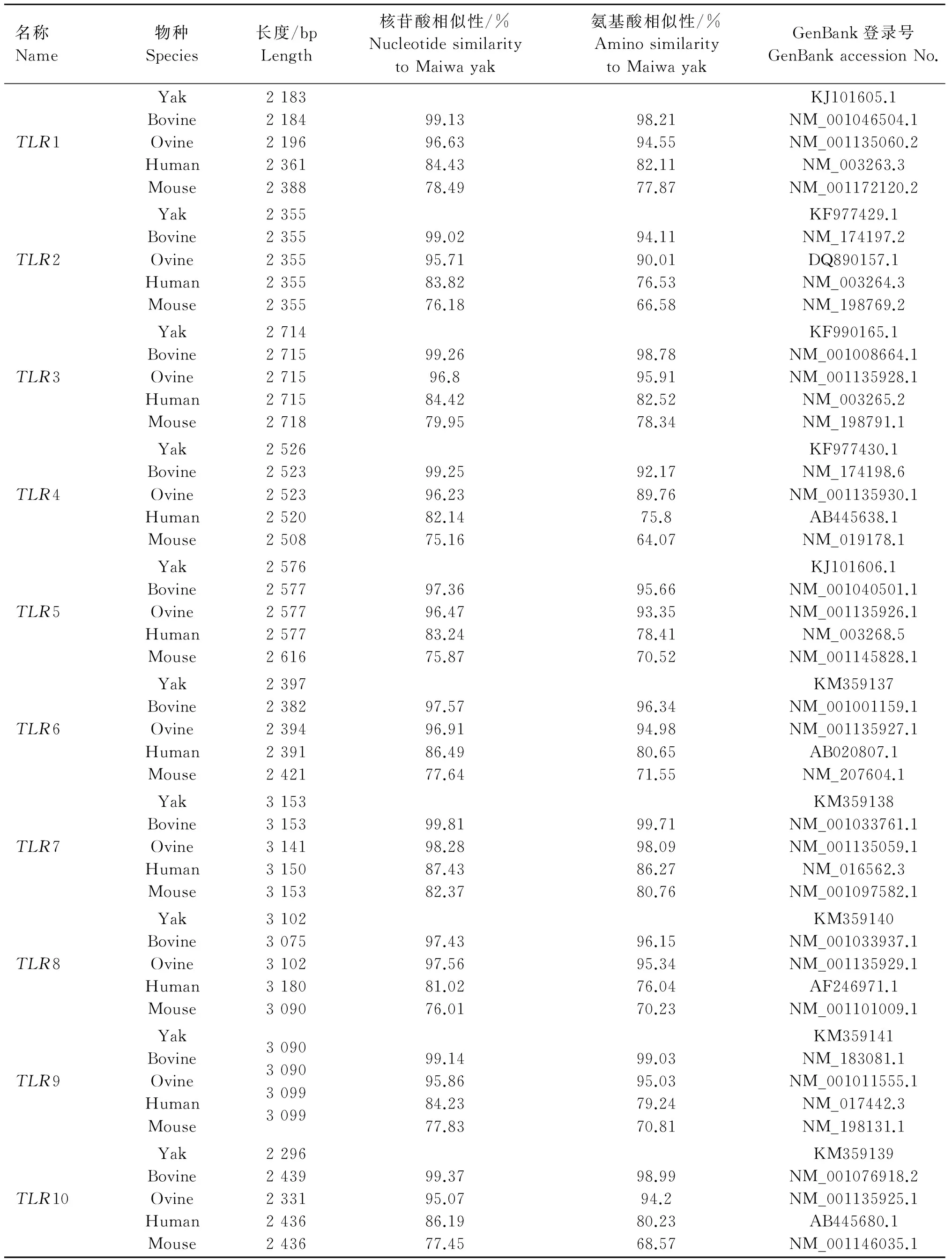
名称Name物种Species长度/bpLength核苷酸相似性/%NucleotidesimilaritytoMaiwayak氨基酸相似性/%AminosimilaritytoMaiwayakGenBank登录号GenBankaccessionNo.TLR1YakBovineOvineHumanMouse2183218421962361238899.1396.6384.4378.4998.2194.5582.1177.87KJ101605.1NM_001046504.1NM_001135060.2NM_003263.3NM_001172120.2TLR2YakBovineOvineHumanMouse2355235523552355235599.0295.7183.8276.1894.1190.0176.5366.58KF977429.1NM_174197.2DQ890157.1NM_003264.3NM_198769.2TLR3YakBovineOvineHumanMouse2714271527152715271899.2696.884.4279.9598.7895.9182.5278.34KF990165.1NM_001008664.1NM_001135928.1NM_003265.2NM_198791.1TLR4YakBovineOvineHumanMouse2526252325232520250899.2596.2382.1475.1692.1789.7675.864.07KF977430.1NM_174198.6NM_001135930.1AB445638.1NM_019178.1TLR5YakBovineOvineHumanMouse2576257725772577261697.3696.4783.2475.8795.6693.3578.4170.52KJ101606.1NM_001040501.1NM_001135926.1NM_003268.5NM_001145828.1TLR6YakBovineOvineHumanMouse2397238223942391242197.5796.9186.4977.6496.3494.9880.6571.55KM359137NM_001001159.1NM_001135927.1AB020807.1NM_207604.1TLR7YakBovineOvineHumanMouse3153315331413150315399.8198.2887.4382.3799.7198.0986.2780.76KM359138NM_001033761.1NM_001135059.1NM_016562.3NM_001097582.1TLR8YakBovineOvineHumanMouse3102307531023180309097.4397.5681.0276.0196.1595.3476.0470.23KM359140NM_001033937.1NM_001135929.1AF246971.1NM_001101009.1TLR9YakBovineOvineHumanMouse309030903099309999.1495.8684.2377.8399.0395.0379.2470.81KM359141NM_183081.1NM_001011555.1NM_017442.3NM_198131.1TLR10YakBovineOvineHumanMouse2296243923312436243699.3795.0786.1977.4598.9994.280.2368.57KM359139NM_001076918.2NM_001135925.1AB445680.1NM_001146035.1

图3 TLR1~10 基因的核苷酸序列系统进化树Fig.3 Phylogenetic tree of TLR1-10 gene sequences
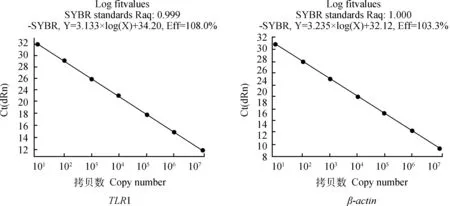
图4 Real-time PCR的标准曲线Fig.4 The standard curves of the real-time PCR

图5 TLR1~10基因组织表达分布Fig.5 Tissues expression and distribution of TLR1-10 genes
3.1 克隆及序列分析
本研究克隆了麦洼牦牛天然免疫受体TLR1~10家族基因,利用生物信息学相关软件对所得序列进行分析和预测。众所周知,TLRs分子中胞外区LRRs结构域的数量及排布是TLRs识别不同类别PAMPs配体的结构基础,TIR结构域是免疫信号向胞内传导,激活天然免疫的结构基础。麦洼牦牛TLR蛋白结构分析预测显示,TLR家族成员间结构及排布相似但各成员所含LRR及TIR结构域数量有较大的差异,提示,牦牛TLR不同成员可能有相似的PAMPs识别配体,但其信号传导通路可能不尽相同;与牛、羊、人、鸡等不同物种TLRs蛋白功能结构与排布方式比较,发现麦洼牦牛TLR蛋白结构域与牛、羊数量与分布位置较为一致,与其他物种间TLRs蛋白大小相近,但蛋白功能结构域排布方式存在差别。认为麦洼牦牛TLR1~10基因尽管与其他物种TLRs结构域存在细微差别,但由此可以推断不同物种间TLRs蛋白应该识别相类似的配体,发挥相同的功能。
牦牛与牛和绵羊等内地平原物种相比生活生长环境恶劣,同等条件下对某些传染性疾病的抵抗能力相对较强,本研究推测,麦洼牦牛免疫分子机制与其他物种相比可能存在特殊性,但同源性分析结果显示,麦洼牦牛TLR家族基因与其他物种表现出高同源性,其中同源性最高的是牛和绵羊,与人、马和鼠的同源性也相对较高。这一结果与K.MeGurie等在牛、羊及山羊上的研究结果基本一致[4-8]。推测,麦洼牦牛TLR家族基因可能在最初遗传进化时与其他物种的TLR一样起源于同一祖先,这也提示我们TLR基因在不同物种间依然保持有高度的同源性和保守型可能是因为其在机体天然免疫和获得性免疫过程中担任着非常重要的作用,其所识别的病原微生物结构模式要求其必须高度保守[9-11]。尽管TLRs基因有很高的保守性,然而在麦洼牦牛所有TLR家族成员中TLR2和TLR4却表现出相对较低的同源性,这说明在不断的遗传进化中部分TLRs成员会因为动物机体所处环境及所识别病原微生物的不同而发生特异性的改变[12-13]。虽然与其他物种TLR相比麦洼牦牛TLR家族基因编码区亦高度保守,但推测由于高原恶劣环境影响的差异性和麦洼牦牛物种本身的特殊性,麦洼牦牛TLR基因可能还是与其他物种相比存在差异,这种差异有可能存在于编码区之外的区域,如启动子区域或是由于甲基化等表观遗传学方面的差异造成[14-16]。
麦洼牦牛与其他物种的TLRs的遗传进化关系表明,麦洼牦牛进化距离最近的是牛和绵羊。在进行系统发育分析时发现,TLR1、2、6、10和TLR7、8、9分别聚集在两个单个的分支上,其中TLR1、TLR6、TLR10又聚为更紧密的一支,TLR其他成员各自成为一支。推测这种分组可能是基于配体结合特性,TLR7、8、9是作为内体与其配体结合,TLR1、6、10则作为异源二聚体与TLR的配体结合,进而传递细胞信号引起相关免疫反应。这也提示人们,保守的TIR在结合配体过程中通过相似的模式组合配体,进而在细胞信号传递中发挥重要的作用[17-19]。
3.2 荧光定量方法的建立及组织表达分布研究
本试验采用SYBR Green I荧光染料嵌合法检测麦洼牦牛天然免疫受体TLRs家族基因的荧光定量PCR方法。通过标准曲线分析,TLR1~10和β-actin基因的相关系数(R2)大于0.990,说明Ct值与cDNA浓度间具有良好的线性关系;TLR1~10和β-actin基因扩增效率(E)在1.0左右,说明扩增效率基本相同,以上分析说明建立的荧光定量检测方法快速、敏感性高、特意性强等,可用于2-△△Ct相对定量的PCR分析。数据分析上,本试验选择了相对定量法,利用内参基因β-actin消除mRNA的提取效率和反转录效率间的差异,对麦洼牦牛TLR1~10基因mRNA表达水平的检测结果进行校正,保证了研究结果的可靠性。试验证明了该方法具备很好的稳定性,是可行的。
组织表达结果显示,麦洼牦牛TLRs在各主要器官组织均有表达,但不同成员在不同组织的表达存在较大的差异。总体来看,麦洼牦牛TLR家族成员在免疫及与免疫相关的组织和器官例如脾、肝、肾、肺均有相对较高的表达量,这说明TLR作为天然免疫识别受体其与麦洼牦牛免疫相关的组织器官之间保持者相当紧密的联系,在机体面临病原入侵时参与其中并发挥重要作用。麦洼牦牛腹泻性疾病及麦洼牦牛生殖性疾病在饲养过程中特别常见,本研究中,麦洼牦牛TLR1、TLR2、TLR4及TLR6等成员在麦洼牦牛小肠和生殖相关的卵巢组织中高表达,提示我们TLRs很可能参与牦牛相关的黏膜免疫系统,在牦牛常见的肠道疾病以及生殖系统疾病的发生发展过程中扮演重要角色,这为人们进一步解决牦牛腹泻和生殖疾病导致的繁殖能力低下等难题提供新的思路。同时,通过比较发现,麦洼牦牛TLR1、TLR6基因在各组织的表达比较相似,TLR2与TLR4、TLR7与TLR8、TLR9表达比较相似。这与J.S.Chang等、K.A.Zarember等和A.O.Monica等的研究结果基本一致[20-22]。TLR2和TLR4除了能够各自识别革兰阳性菌的脂蛋白和脂多糖之外,TLR2、TLR4能够共同识别外分泌蛋白MD-2进而引起细胞反应,此外TLR2、TLR4在炎性反应中同时受一些炎性因子的调控[23]。TLR7、TLR8和TLR9在识别配体以及相关号传递过程中也均有很多相似和内在的联系,这提示人们麦洼牦牛这些基因之间表达相似可能是与其在识别病原微生物并参与动物机体抗病免疫过程中相互协调调控共同发挥作用有关。
综上所述,本研究成功克隆了麦洼牦牛TLR1~10基因,并利用相关生物学软件对其生物学特点进行分析,同时建立了检测麦洼牦牛TLR1~10 基因的荧光定量PCR 方法,并对天然免疫受体基因TLR在麦洼牦牛各组织中的表达进行了研究。这为进一步理解和研究天然免疫受体TLRs 在牦牛等高原动物的免疫分子机制以及抗病育种提供理论基础。
[1] 于莉莉,韩代书.Toll样受体(TLR)介导的天然免疫间的相互调节[J].中国组织化学与细胞化学杂志,2013,22(1):79-84. YU L L,HAN D S.Toll-like receptor(TLR) mediated between the natural immune adjustment[J].ChineseJournalofHistochemistryCytochemistry,2013,22(1):79-84.(in Chinese)
[2] SANG Y,YANG J,ROSS C R,et al.Molecular identification and functional expression of porcine Toll-like receptor TLR3 and TLR7[J].VetImmunolImmunopathol,2008,125:162-167.
[3] SHI Z A,KIYOSHI T,TSUNEYASU K.Toll-like receptors:critical proteins linking innate and acquired immunity[J].NatPublishGroup,2001,2(8):675-680.[4] MeGURIE K,JONES M,WERLING D,et al.Radiation hybrid mapping of all 10 characterized bovine Toll-like receptor[J].AnimGenet,2006,37:47-50.
[5] MENZIES M,INGHAM A.Identification and expression of Toll-like receptors 1-10 in selected bovine and ovine tissues[J].VetImmunolImmunopathol,2006,109:23-30.
[6] RAJA A,VIGNESH A R,MARY B A,et al.Sequence analysis of Toll-like receptor genes 1-10 of goat(Caprahircus)[J].VetImmunolImmunopathol,2011,140(3-4):252-258.
[7] MOROZUMI T,UENISHI H.Polymorphism distribution and structural conservation in RNA-sensing Toll-like receptors 3,7 and 8 in pigs[J].BiochimBiophysActa,2009,1790:267-274.
[8] 赵一萍,白东义,李 蓓,等.马Toll样受体表达水平SYBR GreenⅠ荧光定量RT-PCR检测方法的建立[J].畜牧兽医学报,2013,44(2):220-227. ZHAO Y P,BAI D Y,LI P,et al.Establishment of a real-time RT-PCR assay based on SYBR Green I for detection of the expression of horse toll-like receptor genes[J].ActaVeterinariaetZootechnicaSinica,2013,44(2):220-227.(in Chinese)
[9] 李根亮,穆淑梅,李彦芹,等.TLR—天然免疫中的特异性受体[J].生命的化学,2006,26(6):495-497. LI G L,MU S M,LI Y Q,et al.TLR specificity of innate immune receptors[J].ChemistryofLife,2006,26(6):495-497.(in Chinese)
[10] WERLING D,PIERCY J,COFFEY T J.Expression of Toll-like receptors(TLR) by bovine antigen-presenting cells-potential and ovine tissues[J].VetImmunolImmunopathol,2006,112:2-11.
[11] CAROLYN S J,NATHALIE A,JOSHUA M,et al.TLR signaling tailors innate immune responses in human microglia and astrocytes[J].JImmunol,2005,175(7):4320-4330.
[12] OSAMU T,KATSUAKL H,TARO K.et al.Differential roles of TLR2 and TLR4 in recognition of gram-negative and gram-positive bacterial cell wall components[J].Immunity,1999,10(11):443-451
[13] MOIRA M,AARON I.Identification and expression of Toll-like receptors 1-10 in selected bovine and ovine tissues[J].VetImmunolImmunopathol,2006,109(2):23-30.
[14] MARION J,FLORIAN H,JORG V,et al.Human TLR7 or TLR8 independently confer responsiveness to the antiviral compound R-848[J].NatImmunol,2002,3(499):602-606.
[15] JUNG S C,GEORGE C R,OLIVER J,et al.Molecular cloning and characterization of Toll-like receptors 1-10 in sheep[J].VetImmunolImmunopathol,2009,127(333):94-105.
[16] STANIS LM,WIERZBICKI P M.Decreased Toll-like receptor-5(TLR-5) expression in the mucosa of ulcerative colitis patients[J].JPhysiolPharmacol,2009,60(4):71-75.
[17] SANDRA S D,TSUNEYASU K,HIROAKI H,et al.Innate antiviral responses by means of TLR7-mediated recognition of single-stranded RNA[J].Science,2004,303(5663):1529-1531.
[18] 韩猛立,黄 新,何延华,等.牛Toll样受体实时荧光定量PCR检测方法的建立[J].农业生物技术学报,2011,19(3):521-529. HAN M L,HUANG X,HE Y H,et al.Establishment if a real-time fluorescent quantitative PCR assay for detection of bovine toll-like receptors genes[J].JournalofAgriculturalBiotechnology,2011,19(3):521-529.(in Chinese)
[19] VAHANAN M B,RAJ G D,PAWAR R M C,et al.Expression profile of toll like receptors in a range of water buffalo tissues(Bubalusbubalis)[J].VetImmunolImmunopathol,2008,116:149-155.
[20] CHANG J S,RUSSELL G C,JANN O,et al.Molecular cloning and characterization of Toll-like receptors1-10 in sheep[J].VetImmunolImmunopathol,2009,127:94-105.
[21] ZAREMBER K A,GODOWSKI P J.Tissue expression of human toll-like receptors and differential regulation of toll-like receptor mRNAs in leukocytes in response to microbes,their products,and cytokines[J].JImmunol,2002,168:554-561.
[22] MONICA A O,DAG I V,BEN H,et al.Genomic organization and transcript profiling of the bovine toll-like receptor gene cluster TLR6-TLR1-TLR10[J].Gene,2006,384:45-50.
[23] GOLDAMMER T.Mastitis increases mammary mRNA abundance of beta-defensin 5,toll-like-receptor 2(TLR2),and TLR4 but not TLR9 in cattle[J].Immunol,2004,11:174-185.
(编辑 郭云雁)
Molecular Cloning and Biological Characterization Analysis of Toll-like Receptors 1-10 in Maiwa Yak
LIN Bao-shan,LAN Dao-liang*,CHEN Ya-bing,HUANG Cai,FU Mei,LI Jie,LI Jian*
(CollegeofLifeScienceandTechnology,SouthwestUniversityforNationalities/CollegeofTibetanPlateauResearch,SouthwestUniversityforNationalities,Chengdu610041,China)
In this study,the coding region of Maiwa yak toll-like receptors 1-10 genes were cloned,and then the bioinformatics tool was used to analyze the characteristics ofTLRssequences,and real-time PCR was used to detect the expression pattern ofTLRs.The results showed that Maiwa yakTLRgenes exhibited high homologies at the nucleotide and the amino acid levels with other species.Phylogenetic relationships showed that Maiwa yak had a nearest relationship with cattle and sheep,Maiwa yak was clustered into a branch with human,horse and mouse.It was noteworthy that TLR1,TLR6 first clustered into a small branch,then gathered with TLR10 for a closer one,then TLR1,2,6,10 and TLR7,8,9 were gathered in the 2 individual branches,respectively.The other members of TLRs became respective one.Real-time PCR results showed thatTLRswere expressed in all tissues of yak,but different members had different expression patterns.TLR2,TLR4 andTLR6 had the highest expression in the spleen,followed with ovary,small intestine,kidney,liver.TLR1,TLR5,TLR7,TLR8,TLR9 andTLR10 had the highest expression in kidney,higher expression in liver,kidney,spleen.The study provide useful information for further study in immune molecular mechanisms and disease resistance breeding of yaks.
Maiwa yak;TLR1-10;cloning;real-time PCR;expression and distribution in tissues
10.11843/j.issn.0366-6964.2015.05.007
2014-08-01
西南民族大学中央高校青年教师基金项目(2014NZYQN37);西南民族大学研究生学位点建设项目(2014XWD-S0906)
林宝山(1987-),男,藏族,甘肃天祝藏族自治县人,硕士生,主要从事高原动物分子免疫机制方面的研究,E-mail:172407151@qq.com
*通信作者:兰道亮,副教授,博士,主要从事高原动物分子免疫机制方面的研究,E-mail:landaoliang@163.com;李 键,教授,博士,主要从事动物生殖调控研究,E-mail:lijian@swun.cn
S823.8+5;S813.3
A
0366-6964(2015)05-0728-10

