基于叶绿体psbA-trnH序列和trnL-F序列的牡丹野生种间关系
冯玉兰, 成 娟, 臧荣鑫, 王明明, 陈 红, 何丽霞
(1. 西北民族大学 生命科学与工程学院,兰州 730030; 2. 甘肃省林业科学技术推广总站,兰州 730046)
基于叶绿体psbA-trnH序列和trnL-F序列的牡丹野生种间关系
冯玉兰1, 成 娟2, 臧荣鑫1, 王明明1, 陈 红1, 何丽霞2
(1. 西北民族大学 生命科学与工程学院,兰州 730030; 2. 甘肃省林业科学技术推广总站,兰州 730046)
对牡丹组全部9个野生种15个居群及凤丹的叶绿体psbA-trnH和trnL-F序列进行测序,并采用邻位相连法分别构建了 psbA-trnH序列和trnL-F序列的系统发育树。结果显示:1)两段序列的基因树均支持牡丹组划分为两个亚组的分类方法,与前人的研究结果一致。2)psbA-trnH序列基因树表明大花黄牡丹与黄牡丹的亲缘关系最近,这与大花黄牡丹曾被确定为黄牡丹的一个变种的历史一致。3)革质花盘亚组内,基于psbA-trnH和trnL-F序列的研究结果大体一致,分歧主要在四川牡丹的地位问题。4)psbA-trnH序列和trnL-F序列基因树分别表明杨山牡丹与凤丹具有最近或较近的亲缘关系。
芍药属;牡丹组;系统发育;psbA-trnH序列;trnL-F序列
牡丹属芍药科(Paeoniaceae)芍药属(Paeonia)牡丹组(Sect. Moutan DC.),为多年生落叶灌木,所有种皆为我国特有[1]。牡丹组9 个种[2]分为两个亚组,革质花盘亚组(Subsect. Vaginatae)包括四川牡丹(Paeoniadecomposita)、卵叶牡丹(Paeoniaqiui)、稷山牡丹(Paeoniajishanensis)、杨山牡丹(Paeoniaostii)和紫斑牡丹(Paeoniarockii)5个种,肉质花盘亚组(Subsect. Delavayanae)包括大花黄牡丹(Paeonialudlowii)、紫牡丹(Paeoniadelavayi)、狭叶牡丹(Paeoniapotanini)、黄牡丹(Paeonialutea)4个种。牡丹广泛分布于中国四川、云南、西藏、陕西、甘肃等地[3]。
目前对于芍药属牡丹组各野生种亲缘关系研究已取得一定进展,但对牡丹野生种的起源、进化以及牡丹组的分类系统等问题还存在许多分歧[4]。这是由于依据经典分类学,单纯用表型性状去推测具有不同基因型的分类群之间的系统演化关系会遇到许多问题。因此,关于牡丹野生种间的亲缘关系还需进一步探讨与研究。
植物叶绿体基因组(cpDNA)具有单系遗传,分子水平上具有明显差异以及适用于不同分类阶元系统发育研究的特点[5],被认为是研究种间甚至种内材料系统进化和亲缘关系的理想工具[6]。叶绿体psbA-trnH 序列是位于psbA和trnH间的一段非编码序列,与编码区基因相比可提供更多的系统发育信息。在芍药属中,psbA-trnH 区间的进化速率要高于 matK 基因,稍慢于 ITS,同时序列中的 gap 编码还可提高物种的鉴定率[7-8]。叶绿体trnL-F序列是由 trnL内含子与trnL-F非编码间区以及被分隔的trnL3′端外显子区所组成,这段序列不受功能的限制,表现出进化速率大于功能编码区的特点,可用于属间及属下分类阶元的系统发育研究[9]。
目前尚未有利用叶绿体DNA这两个间隔子序列对牡丹组全部野生种种间亲缘关系的分析。因此,本研究使用牡丹组全部9个野生种15个居群及凤丹的叶绿体psbA-trnH和trnL-F序列进行分析,通过比较牡丹野生种的核苷酸序列组成和排列信息,以期对其进行分子系统学研究,为芍药属牡丹组植物野生种间亲缘关系提供直接的分子证据,并为牡丹组植物的进化和演化分析提供新的依据。

表1 材料来源
1 材料与方法
1.1 材料与试剂
1.1.1 材料
供试材料为牡丹9个野生种共15个居群及凤丹,均取自甘肃省林业科学技术推广总站牡丹种质资源圃。样品编号、名称及引种地见表1。
1.1.2 试剂
天根植物组DNA提取试剂盒、天根Taq PCR Kit、天根纯化试剂盒
1.2 方法
1.2.1 总DNA的提取
取新鲜的牡丹叶片,洗净晾干后,经液氮研磨,采用天根植物组DNA提取试剂盒(50次/盒)对样品进行总DNA的提取。共得到16个DNA样品(见图1-a)。

图1 总DNA提取结果(a); psbA-trnH引物PCR扩增及纯化结果(b,c); trnL-F引物PCR扩增结果(d,e); 以及克隆文库蓝白斑筛选(f); M 为DNA Marker 2000; 1-32为扩增序列编号;CK为对照。
Fig 1 Total DNA (a), PCR products amplified by psbA-trnH Primers (b) and purified products (c), PCR products amplified by trnL-F primers (d,e), and blue-white selection (f).
1.2.2 PCR反应
针对叶绿体不同区断分别采用2套引物(psbA-trnH 区( 引物为:psbA-trnH )、 trnL-F区(引物为: trnL-F )),按照天根Taq PCR Kit 说明书对所提DNA(5倍稀释)进行扩增。PCR总体系为25 uL, 其中:buffer 2.5 uL; dNTP 2 uL; Primer 1 uL; ddH2O 18.1 uL;DNA template 1 uL;Taq polymerase 0.4 uL。psbA-trnH PCR程序为:94℃ 预变性5min; 94℃变性30s, 54℃退火30s, 72℃延伸1min,共35个循环;72℃再延伸5min。trnL-F PCR程序为:94℃预变性5min;94℃变性30s, 50℃退火1min, 72℃延伸2min,共35个循环;72℃再延伸5min。PCR产物用1%凝胶电泳监测,其中, psbA-trnH引物扩增序列为1~16号(见图1-b,6′、10′为6、10号DNA样品的重复扩增);trnL-F引物扩增序列为17~32号(第一次扩增后23、26号可以用,其余重新扩增,见图1-d,e,22′、26′为6、10号DNA样品的重复扩增)。
1.2.3 纯化与连接
采用天根纯化试剂盒对psbA-trnH 引物扩增产物进行凝胶纯化(30uL洗脱,见图1-c),纯化后连接至 pGEM-T 载体 ( Promega, USA),连接体系为:buffer 5uL;vector 0.5uL;T4 ligase 0.5 uL;ddH2O 3uL;DNA template 1uL;total 10 uL。连接时间为14h(4°C冰箱中)。将连接产物转化 Escherichia coli DH5α感受态细胞构建克隆文库(见图1-f),每一克隆文库随机挑取 1个阳性克隆斑利用T7载体引物进行DNA序列测序(上海美吉生物技术公司)。
trnL-F 引物扩增产物无需纯化,直接连接至pGEM-T 载体,连接体系同上。后续实验步骤同psbA-trnH。在进行DNA序列测序时,采用T7/SP6载体引物双向测定。
1.2.4 测序和序列分析
最终获得32条DNA序列。利用MEGA 6.06软件,采用Neighbor-joining法构建系统进化树, 自展数( bootstrap) 为1000。
2 结果与分析
2.1 DNA 序列分析
2.1.1 psbA-trnH序列分析
本研究测得牡丹野生种15个居群及凤丹的psbA-trnH序列长为443~463bp,变化由少数种的碱基缺失所致,对位排列后长度为471bp,空位始终作为缺失处理,GC 含量变化范围为 33.0%~34.50%,保守位点437个,变异位点22 个,简约信息位点 16 个,单独位点6个,分别占序列总长度的92.8%,4.7%,3.4%和1.3%。
2.1.2 trnL-F序列分析
本研究测得牡丹野生种15个居群及凤丹的trnL-F区序列长为1054~1074bp,变化由少数种的碱基缺失所致,对位排列后长度为1081 bp,空位始终作为缺失处理,GC 含量变化范围为 35.5%~36.0%,保守位点1036个,变异位点37个,简约信息位点5个,单独位点32个,分别占序列总长度的95.8%,34.2%,0.5%和 3.0%。
2.2 分子系统树
基于叶绿体psbA-trnH和trnL-F序列的牡丹组全部9个野生种共15个居群及凤丹的系统发育树分别见图2、图3。系统树显示, 全部野生种在聚类关系上分为两大类: 狭叶牡丹、紫牡丹、黄牡丹及大花黄牡丹聚为第一大类;紫斑牡丹、杨山牡丹、凤丹、四川牡丹、稷山牡丹及卵叶牡丹聚为第二大类。
基于psbA-trnH序列的系统发育树(图2)表明:大花黄牡丹与黄牡丹亲缘关系最近;紫牡丹与狭叶牡丹的亲缘关系最近;稷山牡丹、卵叶牡丹以及紫斑牡丹的子午岭、保康和临洮3个居群聚为一支;四川牡丹、杨山牡丹和‘凤丹’及紫斑牡丹的舟曲、文县和党川3个居群聚为一支。
基于trnL-F序列的系统发育树(图3)表明:大花黄牡丹与狭叶牡丹亲缘关系最近;四川牡丹、稷山牡丹、卵叶牡丹以及紫斑牡丹的子午岭、保康和临洮3个居群聚为一支;杨山牡丹和凤丹及紫斑牡丹的舟曲、文县和党川3个居群聚为一支。
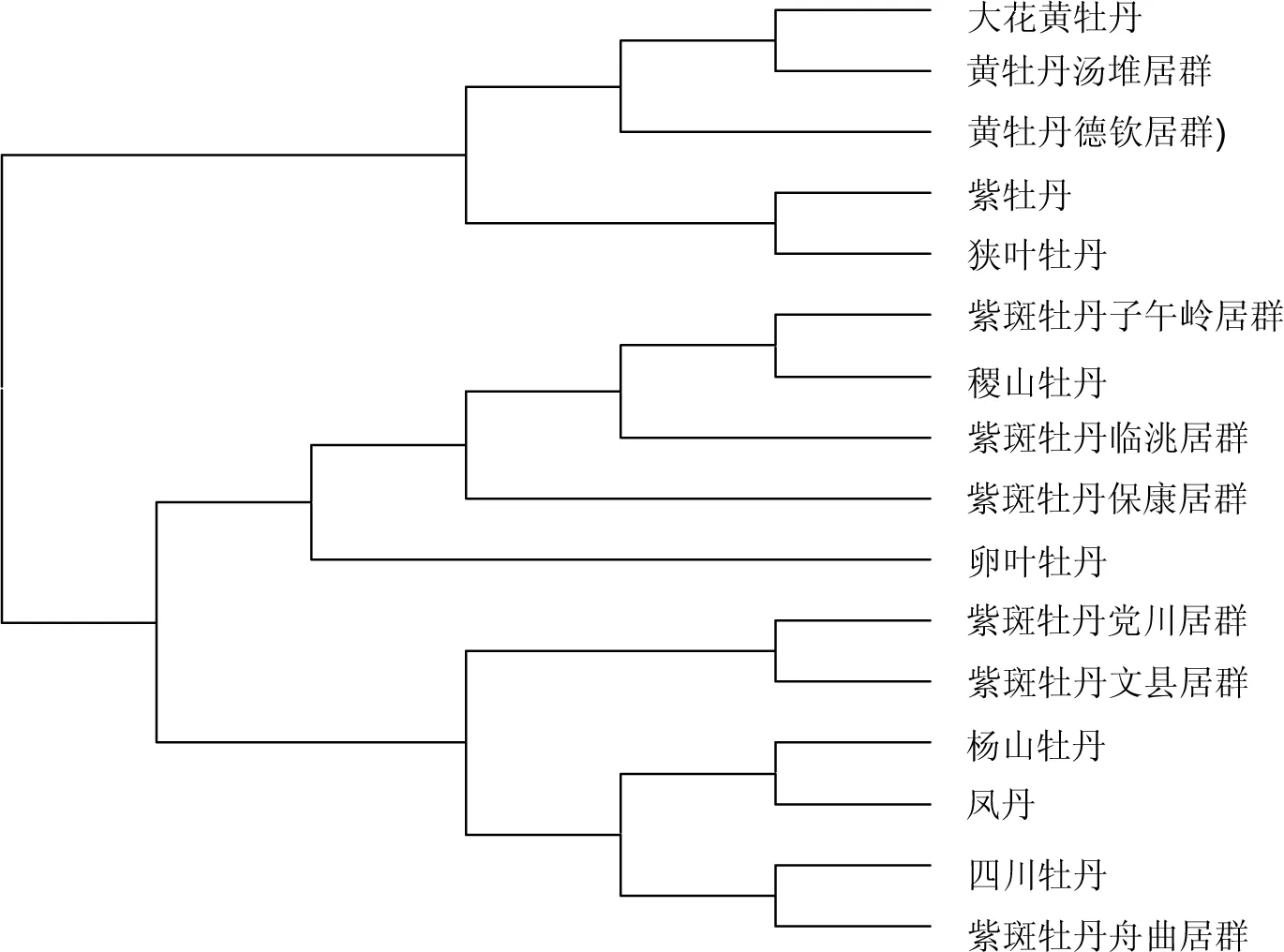
图2 用邻位相连法构建的 psbA-trnH序列系统发育树
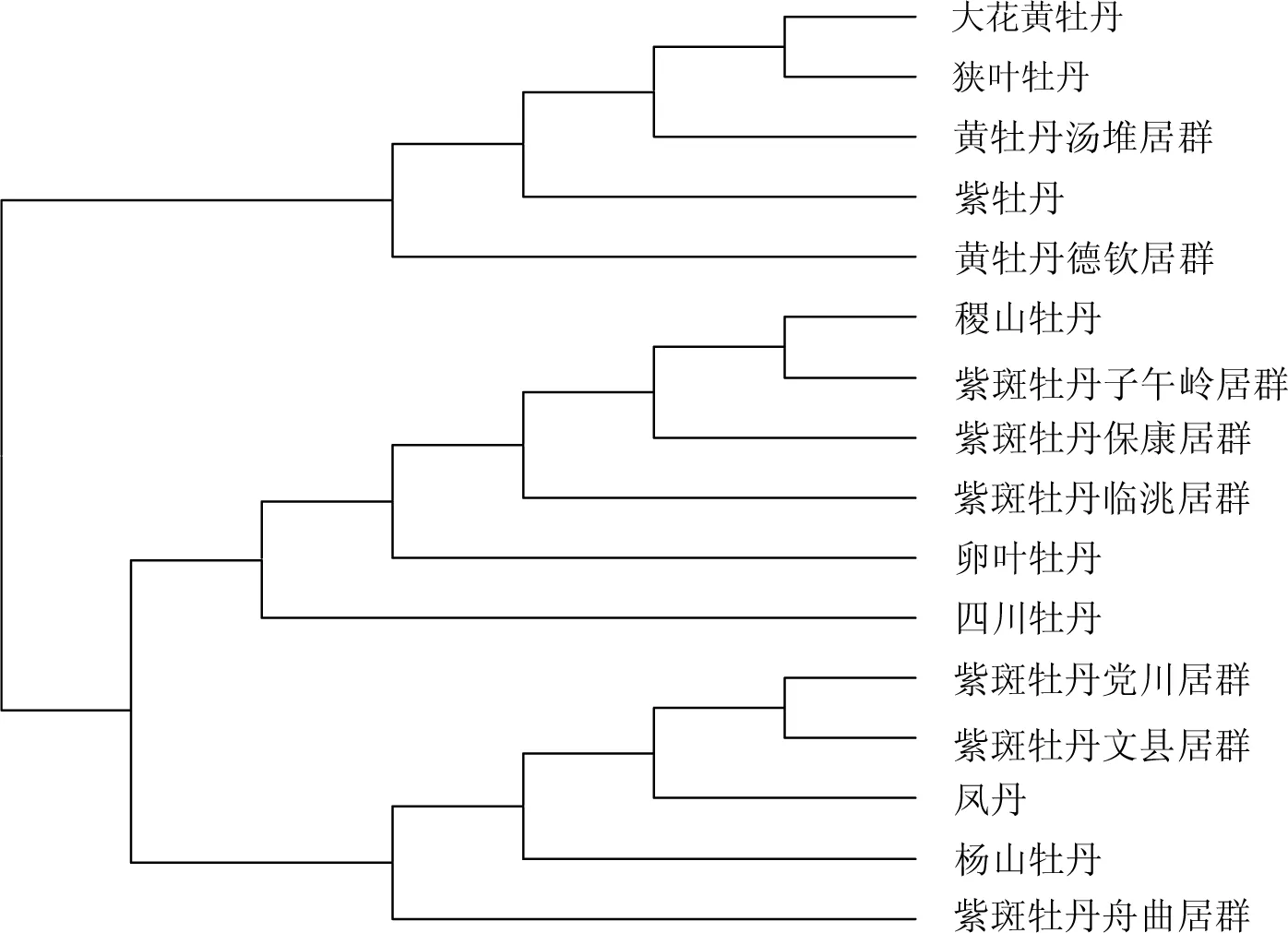
图3 用邻位相连法构建的trnL-F序列系统发育树
3 讨论
根据李嘉珏等的分类方法可将牡丹组分为 9 个野生种[11],其中肉质花盘亚组包括狭叶牡丹、紫牡丹、黄牡丹及大花黄牡丹共4个种;革质花盘亚组包括紫斑牡丹、杨山牡丹、四川牡丹、稷山牡丹及卵叶牡丹共5个种。本研究对9个野生种15个居群基于叶绿体psbA-trnH序列和trnL-F序列的基因树分析支持牡丹组划分为两个亚组的分类方法,与前人的研究结果一致(图2、图3)[4, 12-13]。
肉质花盘亚组内,本研究基于psbA-trnH序列的结果表明大花黄牡丹与黄牡丹的亲缘关系最近(图2),这与大花黄牡丹曾被确定为黄牡丹的一个变种的历史一致[14]。而基于trnL-F序列的结果表明,大花黄牡丹与黄牡丹的亲缘关系较近;黄牡丹和狭叶牡丹的亲缘关系也较近(图3)。
革质花盘亚组内,基于psbA-trnH和trnL-F序列的研究结果大体一致(图2、图3),其中稷山牡丹、卵叶牡丹以及紫斑牡丹的子午岭、保康和临洮3个居群聚为一支;杨山牡丹和凤丹及紫斑牡丹的舟曲、文县和党川3个居群聚为一支。分歧主要在四川牡丹的地位问题,基于psbA-trnH序列的结果表明,四川牡丹与紫斑牡丹的关系最近,与Tank等的gpat分析结果一致[15-16];而基于trnL-F序列的结果表明,四川牡丹与卵叶牡丹的关系较近。目前的报道中有关四川牡丹与其他野生种间的关系尚无一致的结论[16]。关于杨山牡丹和凤丹的关系一直有两种观点,其中一种观点认为凤丹是杨山牡丹的同种异名[17],另一种观点认为凤丹是杨山牡丹演化的栽培亚种[18]。本实验基于叶绿体psbA-trnH序列的基因树分析表明杨山牡丹与凤丹亲缘关系最近,而基于叶绿体trnL-F序列的基因树分析则显示凤丹不仅与杨山牡丹具有很近的亲缘关系,也与紫斑牡丹的党川和文县居群具有较近的亲缘关系。
综上所述,由于牡丹种质在长期的自然选择和人工选择下积累了丰富的遗传变异[19-20],仅凭某一水平或某一手段无法准确推测各分类群间的亲缘关系,必须根据植物学形态特征及多种分子水平上的分类方法对其进行更深入的研究,为牡丹种质资源利用及品种改良等提供科学依据。
[1]Hao Q, Liu, Z A, Shu Q Y, Zhang R E, de Rick J, Wang L S. Studies on Paeonia cultivars and hybrids identification based on SRAP analysis[J]. Hereditas, 2008, (1):38-47.
[2]李嘉珏,张西方,赵孝庆. 中国牡丹[M]. 北京:中国大百科全书出版社, 2011.
[3]Ji L J, Wang Q, Teixeira da Silva J A, Yu X N. The genetic diversity ofPaeoniaL[J]. Scientia Horticulturae, 2012. 62-74.
[4]林启冰,周志钦,赵 宣,等.基于Adh 基因家族序列的牡丹组( Sect. MoutanDC. )种间关系[J]. 园艺学报, 2004, (5):627-632.
[5]石开明,彭昌操,彭振坤,等.DNA序列在植物系统进化研究中的应用[J].湖北民族学院学报(自然科学版), 2002, (04):5-10.
[6]Rajora O P, Dancik B P. Chloroplast DNA variation in Populus. II. Interspecific restriction fragment polymorphisms and genetic relationships among Populus deltoides, P. nigra, P. maximowiczii, and P. x canadensis[J]. Theor Appl Genet, 1995, (3-4):324-30.
[7]Sang T, Crawford D J, Stuessy T F. Chloroplast DNA phylogeny, reticulate evolution, and biogeography ofPaeonia(Paeoniaceae)[J]. American journal of botany, 1997, 84(8):1120-1120.
[8]高利霞.基于trnL-F序列和psbA-trnH序列的10种棘豆属植物分子系统学研究[D]. 呼和浩特:内蒙古师范大学,2013.
[9]宫 霞,王焕冲,杨 勇,等.基于TrnL-F序列的葡萄科各属分子特征及系统发育学研究[J]. 果树学报, 2013, (01):28-36.
[10]Shaw J, Lickey E B, Beck J T, Farmer S B, Liu W, Miller J, Siripun K C, Winder C T, Schilling E E, Small R L. The tortoise and the hare II: relative utility of 21 noncoding chloroplast DNA sequences for phylogenetic analysis[J]. Am J Bot, 2005, (1):142-66.
[11]李嘉钰. 中国牡丹品种图志 西北、西南、江南卷[M].北京:中国林业出版社,2006.
[12]李莹莹. 基于CDDP标记的牡丹遗传多样性分析及分子身份证构建[D]. 泰安: 山东农业大学, 2013.
[13]赵 宣,周志钦,林启冰,等.芍药属牡丹组(Paeonia sect.Moutan)种间关系的分子证据:GPAT基因的PCR-RFLP和序列分析[J]. 植物分类学报, 2004, (03):236-244.
[14]于 玲,何丽霞,李嘉珏,等.牡丹野生种间蛋白质谱带的比较研究[J]. 园艺学报,1998, (1):99-101.
[15]Tank D C, Sang T. Phylogenetic Utility of the Glycerol-3-Phosphate Acyltransferase Gene: evolution and Implications inPaeonia(Paeoniaceae)[J]. Molecular Phylogenetics and Evolution, 2001, (3):421-429.
[16]李保印,周秀梅,蒋细旺,等.牡丹种及品种亲缘关系的分子生物学研究进展[J]. 河南农业大学学报, 2008, (01):121-126.
[17]洪德元,潘开玉. 芍药属牡丹组分类补注[J]. 植物分类学报, 2005, (03):284-287.
[18]李子峰,王 佳,胡永红,等.‘凤丹白’牡丹核型分析与减数分裂的细胞遗传学观察.园艺学报,2007, (2): 411-416.
[19]侯小改, 等. 部分牡丹品种遗传多样性的AFLP分析[J].中国农业科学,2006, (8): 1709 -1715.
[20]邹喻苹,蔡美琳,王子平.芍药属牡丹组的系统学研究―基于RAPD分析[J]. 植物分类学报, 1999, (03):13-20.
Phylogenetic Relationships ofPaeoniaSect. Moutan Based on Chloroplast psbA-trnH and trnL-F Sequences
FENG Yu-lan1, CHENG Juan2, ZANG Rong-xin1, WANG Ming-ming1, CHEN Hong1, HE Li-xia2
(1. Life Science and Engeneering College, Northwest University of Nationalities, Lanzhou 730030, China;2. Forestry Research Institution of Gansu Province, Lanzhou 730046, China)
The psbA-trnH sequences and trnL-F sequences of nine species in Sect. Moutan DC. and one cultivar Fengdan’ were sequenced, then the phylogenetic trees based on two sequences were constructed by Neighbor-joining (NJ) methods in MEGA 6.06, respectively. The results showed as follows: 1) Nine species in Sect. Moutan DC. were divided into two Subsects from two phylogenetic trees, consistent with previous research results. 2)Paeonialudlowii, that had been identified as a variation ofPaeonialutea, was most closely related toPaeonialuteabased on psbA-trnH sequences. 3) The results were largely consistent in Subsect. Vaginatae, except the relationship betweenPaeoniadecompositaand other four species bases on two sequences. 4)Paeoniaostiihad the closest or very closer relationship with the cultivar Fengdan’ based on psbA-trnH sequences and trnL-F sequences, respectively.
Paeonia; Sect. Moutan; phylogenetic relationships; psbA-trnH sequences; trnL-F sequences
2014-08-09;
2014-10-17
西北民族大学2012年度中央高校基本科研业务费项目(zyz2012068)
冯玉兰,硕士研究生,讲师,研究方向为生物技术,E-mail:fyl@xbmu.edu.cn。
S685.11;Q943.2
A
2095-1736(2015)01-0055-04
doi∶10.3969/j.issn.2095-1736.2015.01.055

