海洋玫瑰杆菌类群研究进展
陈正浩, 张永雨, 杨素萍
1 华侨大学, 生物工程与技术系, 厦门 361021 2 中国科学院城市环境研究所, 城市环境与健康重点实验室, 厦门 361021 3 厦门大学, 近海海洋环境科学国家重点实验室, 厦门 361005
海洋玫瑰杆菌类群研究进展
陈正浩1, 2, 张永雨2, 3, 杨素萍1,*
1 华侨大学, 生物工程与技术系, 厦门 361021 2 中国科学院城市环境研究所, 城市环境与健康重点实验室, 厦门 361021 3 厦门大学, 近海海洋环境科学国家重点实验室, 厦门 361005
海洋玫瑰杆菌类群(Roseobacterlineage)是属于α-变形菌纲中的一类系统发育相近,但生理代谢功能多样的细菌类群,包含40多个不同的细菌种属。它们在海洋中丰度较高,且分布极为广泛,尤其在近海与极地海洋中,其丰度约占整个浮游细菌群落的15%—25%。玫瑰杆菌类群通过其多样化的生理代谢功能(如好氧不产氧光合作用、一氧化碳氧化、硫化物降解等)在海洋碳、硫循环和全球气候调节中发挥着重要作用。此外,玫瑰杆菌类群还能产生多种具生物活性的次生代谢物质。简要综述了海洋玫瑰杆菌类群的生态分布特征、生存方式、生理代谢功能、基因组特征等的一些研究进展,并结合作者的工作对未来的研究进行了展望。
玫瑰杆菌; 好氧不产氧光合作用; 硫代谢; 次生代谢物质; 玫瑰杆菌噬菌体
玫瑰杆菌类群是一类隶属于α-变形菌纲(α-Proteobacteria)的一类系统发育相近,但生理代谢功能极其多样的细菌类群,包含40多个不同的细菌种属。它们在海洋中分布极为广泛,从近海到外海、从海水表层到海底、从热带到极地海洋,几乎无处不在。尤其是在近海与极地海洋中,玫瑰杆菌类群是重要的优势细菌类群,约占整个浮游细菌群落的15%—25%。另外,在一些高盐湖泊和土壤中也有发现玫瑰杆菌类群的存在[1- 2],表明对钠离子的高度依赖或需求是玫瑰杆菌类群的一大典型特征。
1991年日本学者Shiba首次通过分离培养获得两种玫瑰杆菌类群菌,分别是Roseobacterdenitrificans与Roseobacterlitoralis,并发现这两种细菌均具有好氧不产氧光合作用和多种其它代谢功能[3]。后来,人们又陆续在实验室分离培养获得多种不同的玫瑰杆菌菌株,同时利用分子生态学方法,如细菌16S rRNA基因的克隆文库测序、环境DNA高通量测序、FISH杂交技术等,证实了玫瑰杆菌类群菌在海洋中大量存在[2]。同时发现玫瑰杆菌类群菌的另一个显著特点,即较易在实验室获得分离培养,且实验室条件下培养的很多玫瑰杆菌能够维持其野生性状,因而玫瑰杆菌类群菌也成为对海洋细菌生理生态功能进行研究的模式生物[4]。
近年来,人们又陆续发现海洋玫瑰杆菌类群菌具有多种不同的生存方式和生理代谢功能。有的以单细胞形式生存,有的以多细胞小规模群居,有的常与海洋浮游植物、藻类、无脊椎动物和脊椎动物等共生。另外,很多玫瑰杆菌除了具有好氧不产氧光合异养作用外,还具有氧化温室气体CO、高效降解芘、萘和荧蒽等多环芳烃类化合物、产生多种次生代谢物质、以及可降解藻类渗透物二甲基巯基丙酸内盐(DMSP)从而产生与气候调节相关的气体二甲基硫(DMS)等[2, 4]。近年来,玫瑰杆菌类群在海洋生态系统中的重要作用和地位引起了人们的广泛关注,并围绕其开展了大量研究。本文着重从其生态分布、生存方式、生理代谢功能等方面分析并论述了玫瑰杆菌类群菌在海洋生态系统中的重要作用,综述了关于海洋玫瑰杆菌类群研究的近期进展。
1 玫瑰杆菌类群在海洋环境中的生态分布及其多样性
目前已知的玫瑰杆菌类群包括已在实验室通过分离培养获得的细菌纯系和一系列通过构建克隆文库等方法获得的不可培养的玫瑰杆菌序列,它们之间16S rRNA基因序列的相似性均大于89%。 截至目前,已获鉴定的玫瑰杆菌类群菌包括40多个不同的细菌种属[5]。表1所示即为隶属于玫瑰杆菌类群中的典型属种。从表1可以看出,玫瑰杆菌类群在海洋中的分布极为广泛,从近海到外海、从海洋表层到海底沉积物、从温带海洋到极地海洋几乎无处不在。另外,玫瑰杆菌类群还常与藻类(尤其是甲藻)、珊瑚、头足类动物(乌贼、鱿鱼)、贝类幼虫等共生。同时,玫瑰杆菌类群中的一些细菌还被发现存在于高盐湖泊或土壤环境[4,6]。
大量研究报道表明,玫瑰杆菌类群菌在近海与极地海洋中丰度最高,约占整个浮游细菌群落的15%—25%[4]。但关于玫瑰杆菌类群菌在全球海洋中的丰度,不同的报道往往存在着一定的差异,这可能是由于不同的测定方法以及所在海域不同所致。已报道的关于玫瑰杆菌类群菌丰度测定的方法主要有分离培养法、荧光探针杂交法、克隆文库法、定量PCR法、BAC文库与鸟枪文库法等[2]。 Eilers 等利用原位荧光杂交技术发现美国东南海岸近海中的微生物群落中玫瑰杆菌类群菌的比例高达20%[7]。 DeLong和Karl与 Suzuki 等利用BAC文库技术研究发现在美国加利福尼亚蒙特里近海表层和80 m深处水体中,玫瑰杆菌类群菌占整个微生物群落的比例分别高达21.1%与23.6%[8- 9]。然而Moran 与Venter等利用鸟枪克隆文库法研究发现玫瑰杆菌类群菌在Sargasso 海的中所占的比例仅为3%[10- 11]。
综合以往的大量研究,不难发现玫瑰杆菌类群对盐具有耐受性,除去一些特别的菌种如Ketogulonicigenium.和几种南非金矿中的细菌外,玫瑰杆菌只存在于海洋或者高盐度环境中,对盐的吸收或耐受能力是该类群细菌的一大典型特征。

表1 玫瑰杆菌类群所包含的典型属种Table 1 Type strains of genera within the Roseobacter clade
2 玫瑰杆菌类群的生理特征及其在海洋碳、硫循环中的作用
2.1 好氧不产氧光合特性
好氧不产氧光合细菌(Aerobic anoxygenic photosynthesis bacteria, AAPB)是在有氧条件下以有机物、硫化物或氨等作为供氢体,通过细菌叶绿素捕获光能进行光合作用但并不释放氧气的一类原核生物。AAPB在海洋中有着广阔的生存空间,广泛分布于全球海洋的真光层。它们在海洋碳循环和能量循环中起着不可忽略的重要作用[12]。
20多年前第1株被发现的AAPB菌即为海洋玫瑰杆菌R.denitrificans[3],不过直到最近,AAPB在海洋生态系统中的重要性才受到广泛关注。AAPB有着独特的生理生态特征,如专性好氧、可以进行光合作用生长但却不全依赖于光、含有细菌叶绿素a 以及丰富多样的类胡萝卜素[13]。 之后,陆续发现AAPB在玫瑰杆菌类群中大量存在,目前已发现10多个不同种属的玫瑰杆菌类群菌具有好氧不产氧光合异养作用的功能。绝大多数这类玫瑰杆菌是专性好氧的,在厌氧条件下不能进行光合生长。但也有部分玫瑰杆菌如Roseobacterdenitrificans和Dinoroseobactershiba等可利用亚硝酸盐或者三甲胺N-氧化物作为电子受体进行厌氧生长,属于兼性厌氧细菌,并与紫硫细菌在一些生理特征上具有高度的相似性[2]。
最初AAPB被认为是仅生存于特殊环境中的非常规海洋细菌,直到后来发现细菌叶绿素a以及光合反应中心蛋白基因(pufLM)与细菌叶绿素合成基因(bch)在海洋浮游细菌中广泛存在,人们才开始认识到AAPB在海洋生态环境中的重要生态意义[12]。在表层海水中海洋玫瑰杆菌类群通过好氧不产氧光合异养功能补充能量,可一定程度上降低对表层海水中有机底物的需求量,使有限的碳源可支持微生物更多更好地生长,因而这类具好氧不产氧光合异养功能的玫瑰杆菌类群在碳限制的海洋环境中往往会表现出更强的生存竞争优势,并影响海洋环境中有机碳源的储量和生态动力学变化[14]。
2.2 一氧化碳氧化作用
一氧化碳(CO)是大气对流层中的一种重要的化学反应物,它能通过清除大气中主要氧化因子-羟自由基,从而影响温室气体臭氧以及甲烷的分解。在海洋环境中,太阳光的照射可引起碳水化合物的氧化,从而释放CO气体。玫瑰杆菌类群中的很多成员被发现具有氧化CO的作用,实现CO的消耗[4]。
目前有2种证据表明海洋玫瑰杆菌类群菌参与CO的生物氧化:第1种证据是在实验室条件下获得了可以氧化CO的玫瑰杆菌纯系Ruegeriapomeroyi[15];另一个证据是在玫瑰杆菌(如Ruegeriapomeroyi, 原称为Silicibacterpomeroyi)基因组中发现了控制一氧化碳氧化的操纵子基因(cox)[10]。据报道,R.pomeroyi被证实在近海和远海表层水体中CO浓度分别为10 nmol/L和2 nmol/L的情况下进行CO的氧化。不过玫瑰杆菌并不像其它专性CO氧化菌那样,以CO作为唯一碳源和能源进行化能自养生长,而仅是利用CO作为补充能源进行异养生长[10]。
R.pomeroyi的基因组上包含2个与CO氧化有关的操纵子,分别属于2个不同的CO脱氢酶(coxL)基因簇BMS和OMP,它们对CO的亲和性存在一定差异。BMS簇对CO的亲和性相对较低,而OMP簇对CO较为敏感。这2种基因在同一个生物体中的同时存在是对环境的一种适应优势,使得该生物能在不同CO浓度范围条件下发挥出最佳的酶活力状态[15]。
人们通过设计CO脱氢酶基因coxL的特异引物,并利用分子生态学技术研究发现大多数CO氧化菌隶属于α-、β-和γ-变形菌纲,其中包含Ruegeria属与Roseobacter属等的一些海洋玫瑰杆菌[4]。 截止目前已有隶属于6个不同种属的海洋玫瑰杆菌类群菌被发现具有氧化CO的能力,对CO的氧化也被认为可能是在阳光照射的表层水体中玫瑰杆菌类群细菌提高自身新陈代谢能力的一种生存策略。
2.3 芳香族化合物降解
海洋中维管植物生长过程中产生的芳香类化合物是微生物碳库的重要组分,可促进那些能通过降解芳香族化合物获得代谢能量的细菌生长。人们发现维管植物的周围往往富集着大量的玫瑰杆菌类群菌,暗示玫瑰杆菌类群可能具有芳香族化合物代谢能力。玫瑰杆菌类群中的一些特征菌株,例如SagittulastellataE- 37,可以利用芳香族化合物作为主要生长基质进行生长[16]。Buchan 等[17- 18]在16种不同的玫瑰杆菌中均检测到β-己二酮代谢途径中的一种重要的裂环酶基因,该基因编码的蛋白酶可促进木质素的转化。另外,Buchan 等[18]在富含羟基、甲级和氨基取代环等芳香族化合物的盐泽地中检测到120种不同的裂环酶基因,其中一半以上的该基因可以追溯到海洋玫瑰杆菌类群,表明很多海洋玫瑰杆菌类群菌可能具有降解芳香类化合物能力,并为其生长提供能量补充。
近年来,关于玫瑰杆菌类群菌对芳香族化合物的降解能力不断有新的研究报道。例如,Brito 等[19]在富含芘、萘和荧蒽等物质的巴西红树林沉积物中分离得到8株玫瑰杆菌类群菌,它们均表现出了不同程度的芳香族化合物降解能力(10%—100%)。Petra 等[20]在受石油污染的海洋水体中亦发现存在着大量玫瑰杆菌类群菌。
目前在玫瑰杆菌类群中发现的芳香类化合物降解途径,除了上述的β-己二酮代谢途径外,还包括可降解酚醛塑料的龙胆酸盐途径与苯甲酸盐好氧降解途径等。其中龙胆酸盐途径亦广泛存在于土壤细菌[4]。
2.4 参与海洋硫循环
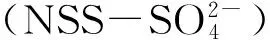
除此之外,海洋玫瑰杆菌类群菌还具有转化其它多种有机硫(如二甲基硫、巯基甲醇、甲基化硫和二甲基亚砜等)以及对无机硫进行代谢的能力,例如对单体硫、硫化物、亚硫化物、过硫化物等的转化[2, 6, 24]。
第1株在DMSP中生长并分离得到的好氧细菌即为玫瑰杆菌类群中的SilicibacterPomeroyi,它也是目前已知的唯一一种既能通过直接降解、又能通过去甲基化两种途径来降解DMSP的海洋细菌[2]。另外,Moran与Todd等[10, 25]亦在R.Pomeroyi的基因组中发现5个对甘氨酸甜菜碱和DMSP代谢相关的基因。因此,玫瑰杆菌类群菌在能大量释放DMSP的藻类附近往往生长最为旺盛[26]。
3 玫瑰杆菌类群的生活方式及其次生代谢产物
3.1 与藻类共生
很多玫瑰杆菌类群中的细菌与藻类(尤其是甲藻类)建立了互惠互利的共生关系。一方面玫瑰杆菌类群细菌可利用藻类释放的二甲基巯基丙酸(DMSP)作为碳源和能源满足自身生长的需要,同时由于玫瑰杆菌类群中很多为好氧不产氧光合细菌,藻类(如腰鞭毛藻)可携带与其共生的玫瑰杆菌类群菌游动到富含溶解有机质(dissolved organic carbon, DOM)和光照适宜的真光层,从而促进玫瑰杆菌类群菌的生长;另一方面,玫瑰杆菌类群菌可通过新陈代谢产生无机盐和维生素如B12等, 反过来也促进藻类的生长[26]。 图1所示即为浮游植物(含藻类)与玫瑰杆菌类群菌共生关系的模式图。玫瑰杆菌类群与藻类之间的物质交换或相互作用关系很可能是通过一种由vir基因调控的4型分泌系统(T4SS)或通过群体感应信号(QS)来进行调控[26]。Alavi等[27]曾在甲藻Pfiesteria的培养基中发现50%的细菌菌落都属于玫瑰杆菌类群,同时利用激光共聚焦扫描电镜观察到玫瑰杆菌类群菌与甲藻Pfiesteria细胞的表面紧密相连。除与甲藻共生外,近来Hube 等[28]发现在一种海洋蓝藻哈氏节球藻周围,玫瑰杆菌类群菌亦广泛存在。
然而,玫瑰杆菌类群与藻类的共生关系也不是一成不变,当藻类处于新陈代谢活性较弱或频临消亡的状态时,可能会发出某种信号,诱使玫瑰杆菌类群菌变为溶藻菌,分泌溶藻物质,加快藻类的死亡,两者之间由共生关系转变为敌对关系[29]。例如,Riclea等[30]研究发现玫瑰杆菌R.pomeroyi可分泌次生代谢物质-内酯,促进藻类的死亡;Phaeobactergallaeciensis可分泌新型抑藻物质roseobacticide促进海洋球石藻等赤潮藻的消亡[31]。
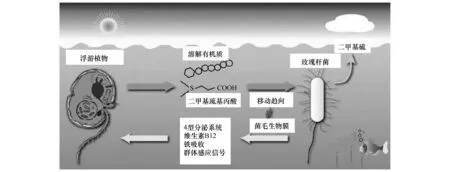
图1 浮游植物与玫瑰杆菌类群菌的共生关系模式图[26]Fig.1 A model of the symbiosis relationship between roseobacters and phytoplankton
3.2 与真核动物共生
除了与海洋浮游植物共生外,玫瑰杆菌类群的细菌还常与珊瑚、乌贼、鱿鱼、贝类幼虫、鱼类等无脊椎和脊椎动物共生,通过分泌次生代谢物质-如杀菌剂,保护动物免受病原菌的侵害,具有益生效果[2]。Hjelm等[32]发现玫瑰杆菌类群RGALL分支中的菌株所分泌的次生代谢物质对鱼幼体的致病菌(如弧菌)有抵抗作用。Ruiz-Ponte 等[33]发现R.galleaeciensis对扇贝的幼虫具有相似的益生效果。目前已发现的玫瑰杆菌类群菌所分泌的能抑制病原细菌的次生代谢物质主要有tropodithietic acid(TDA)、靛青素(Indigoidine)等[34- 35],绝大多数均为吲哚、吲哚衍生物、环状二肽和色胺酮等。
不过在玫瑰杆菌类群中也有一些细菌本身即为病原菌,例如玫瑰杆菌Roseovariuscrassostreae的感染是引起牡蛎幼虫疾病的主要致病菌,有时可导致牡蛎死亡率高达90%以上[36]。
3.3 群体感应
群体感应是指细菌能自发产生、释放一些被称为自诱导物质(autoinducer, AI)的信号分子,并通过感知其浓度变化,调节微生物群体行为的信息交流方式。胞外的AI浓度能随细菌密度的增加而增加,当达到一个临界浓度时,AI能启动菌体内相关基因的表达,调控细菌的生物行为,如产生毒素、形成生物膜、产生抗生素等,以适应环境的变化[37]。由于这一感应现象只有在细菌密度达到一定阈值后才会发生,因而群体感应也被称为细胞密度依赖的基因表达[38]。根据细菌合成的信号分子和感应机制不同,群体感应系统基本可分为3个代表性的类型。在革兰氏阴性细菌中最常见的即为LuxI/LuxR群体感应系统,该系统一般利用酰基-高丝氨酸内酯(AHL)类化合物分子作为信号分子[38]。据报道在玫瑰杆菌类群的有些细菌中亦存在这种LuxI/LuxR群体感应系统。LuxI的生物信息学同源性分析研究表明,在已知基因组的玫瑰杆菌类群菌中,80%以上都至少编码了1个LuxI的同系物[39],它们通过产生AHL进行群体感应,调节自身的生命活动[40]。尤其当玫瑰杆菌类群菌在静止培养时,这种群体感应现象会更加明显,同时诱发细菌产生抗生素、溶藻物质、毒素及生物膜等,抑制其它微生物或藻类的生长[2]。
4 玫瑰杆菌类群的基因组特征与基因转移因子(gene transfer agent, GTA)
据报道,截至目前玫瑰杆菌类群中已有40多种细菌完成了全基因组测序,其基因组大小在3.0—5.5 Mbp之间,平均约为4.4 Mbp, 具有极其多样的细胞代谢调控基因,参与多种新陈代谢活动,如CO氧化、硫代谢、合成次生代谢产物等[5]。目前已知玫瑰杆菌类群中基因组最小的是LoktanellavestfoldensisSKA 53, 全长仅为3.06 Mbp, 最大的是Roseovariussp. HTCC2601, 基因组全长为5.4 Mbp[2, 4]。
在玫瑰杆菌类群菌染色体外经常会存在着多个不同大小的质粒。例如:在玫瑰杆菌R.litoralis中,质粒约占整个基因组大小的5%,在S.pomeroyi中质粒所占比例更高,约为10%。质粒上常携带一些功能基因(如pca、puf和nir等)[2, 4],这些位于染色体外的遗传元件在玫瑰杆菌类群细菌多样化的生理代谢功能中发挥重要作用。
此外,近年来人们发现在玫瑰杆菌类群中,尤其是Roseobacter属的细菌中经常存在一种类似于噬菌体颗粒的结构,被称为基因转移因子(GTA)。GTA是一种由细菌释放的、形态和有尾病毒类似的生物颗粒,它所携带的遗传物质是宿主基因组的随机小片段而不包含编码GTA自身的基因, 其主要的功能即促进微生物间的水平基因转移[36]。 编码GTA的基因簇一般包含15个基因,分别命名为orfg1- 15[41]。Biers等[42]研究了玫瑰杆菌R.pomeroyiDSS- 3 的GTA基因簇结构,发现DSS- 3含有GTA基因组15个基因的13个,同时验证了DSS- 3可以借助GTA发生水平基因转移。由于编码GTA的部分结构基因(如衣壳蛋白基因g5)具有较强的保守性,Zhao等[43]以g5基因作为分子标记基因研究了含GTA基因的细菌在美国Chesapeake Bay表层海水的多样性,利用特异性引物对22株Roseobacer的g5基因进行扩增,结果显示所测试的玫瑰杆菌菌株都包含g5基因。另外,g5基因序列的进化分析表明Chespeake Bay 中总共有12个含g5基因的Roseobacter和Rhodobacter的亚群,其中11个亚群属于Roseobacter,暗示GTA基因在Roseobacter中可能广泛存在。
5 玫瑰杆菌类群与病毒(噬菌体)间的相互关系
海洋噬菌体是海洋生态系统中丰度最高的一种生物类群,它们在控制原核生物的死亡率、调节微生物群落结构、影响微食物网过程以及参与海洋碳、氮等元素的生物地球化学循环中发挥着重要作用[44]。海洋中的噬菌体无时无刻不在威胁着细菌的生存,在表层海水中每天约有高达30%的细菌会被噬菌体所裂解,在一些特殊的环境,如深海海底,由噬菌体所导致的细菌死亡率竟高达80%以上[44],鉴于玫瑰杆菌类群菌是近海环境中的重要优势细菌,约占整个近海浮游细菌群落的15%—25%,近海环境中可能含有大量能够感染玫瑰杆菌类群的噬菌体存在。近年来人们也的确发现海洋玫瑰杆菌类群的细菌较易受噬菌体感染,目前已有多篇关于海洋玫瑰杆菌噬菌体的研究报道。
Rohwer等[45]十几年前分离到1株能够感染RoseobacterSIO67 的玫瑰杆菌噬菌体RoseophageSIO1, 对其全基因组进行测序和注释后,发现RoseophageSIO1 的多个酶基因与大肠杆菌噬菌体T3 和T7 中相对应的蛋白基因序列非常相似,暗示着海洋病毒(主要指噬菌体)与陆地病毒在基因组成上具有一定程度的同源性。据我们所知,这也是关于玫瑰杆菌噬菌体的第一篇报道。然而,在之后的近10年时间里,关于玫瑰杆菌噬菌体的研究一直处于空白状态。直到近期,这一方面的研究才开始受到重视,并连续有多篇相关报道。
2009年Angly等[46]在加利福尼亚近海又分离到4株能够感染RoseobacterSIO67的噬菌体。本文作者[47]也以玫瑰杆菌RoseobacterdenitricansOCh114作为宿主菌,自中国南海分离到1株感染特异性较高的噬菌体RDJLΦ1, 该噬菌体具有较强的裂解能力:当其感染宿主细菌时,在宿主细胞内的潜伏期约为80 min,随后即引起宿主细胞的瓦解。后来,通过与 Huang等[48- 50]合作又深入细致地研究了该噬菌体侵染宿主的发生过程与机制、以及某些宿主突变体抵抗该病毒侵染的机理、并对该病毒基因组进行了测序与注释分析等。另外,我们还分离到了1株感染SilicibacterpomeroyiDSS- 3的噬菌体DSS- 3 Φ1[51]。
除此之外,还有多个玫瑰杆菌类群菌噬菌体的报道,其宿主主要包括RoseovariusnubinhibensISM[52]、Sulfitobactersp. EE- 36[53]、Celeribactersp. IMCC12053[54]等,由此可见玫瑰杆菌类群菌可能较易被噬菌体感染。由于噬菌体感染对细菌的生理生化过程具有十分重要的影响,因此对海洋中玫瑰杆菌类群的生态动力学过程及其生态意义进行综合考察与评价时,其周围环境中大量存在的噬菌体与它们之间的相互作用关系也是一个不可或缺的重要考察因素。
6 结语
综上所述,鉴于玫瑰杆菌类群菌的上述特点与重要性,围绕该类群细菌已开展了大量的研究工作,目前玫瑰杆菌类群已成为海洋细菌中被研究最多的细菌类群之一,已有40多个不同的细菌种属被鉴定,其中绝大多数都是近几年刚刚发表。另外已有40多株玫瑰杆菌类群菌完成了全基因组测序,这为今后人们围绕该类群细菌开展深入研究奠定了极为宝贵的遗传信息背景。还有以下几个方面的科学问题有待进行深入研究:(1)近年来随着我国城市化和工业化进程急剧加快,近海污染问题越来越重,重金属、有机污染物、以及抗生素等含量均出现不同程度的超标,玫瑰杆菌类群作为近海环境中的重要优势细菌类群,在污染物的化学行为以及生物转发与降解方面可能发挥着某种未知的作用,因此针对玫瑰杆菌类群菌在海洋污染物的生物降解、对重金属生物转化与修复及其生物地球化学循环方面可加强研究;(2)由于玫瑰杆菌类群菌可产生具有生物活性的次生代谢物质,如抗生素、溶藻物质、以及多种化学信号物质等,因而围绕玫瑰杆菌类群的细菌在海洋生物活性物质资源开发和利用方面可开展深入研究;(3)虽然玫瑰杆菌类群菌较易被病毒感染,然而目前仅在少数种属的玫瑰杆菌种群中分离得到其感染性病毒(噬菌体),不过即使通过对这少数玫瑰杆菌病毒的研究,也已凸显了玫瑰杆菌病毒类群具有较高的生物多样性,并可能以多种不同的方式与其宿主之间发生相互作用。由于海洋中大量存在的病毒对细菌宿主的生存及其生理生态过程有着重要的影响,因而在实验室建立更多隶属于不同种属玫瑰杆菌类群的宿主-病毒相互作用体系,并围绕其相互作用关系开展深入研究,将有助于对海洋玫瑰杆菌类群的生态动力学过程进行更全面系统的了解。
[1] Labrenz M, Collins M D, Lawson P A, Tindall B J, Braker G, Hirsch P.Antarctobacterheliothermusgen. nov., sp. nov., a budding bacterium from hypersaline and heliothermal Ekho Lake. International Journal of Systematic and Evolutionary Microbiology, 1998, 48(4):1363- 1372.
[2] Wagner-Dobler I, Biebl H. Environmental biology of the marineRoseobacterlineage. Annual Review of Microbiology, 2006, 60(1): 255- 280.
[3] Shiba T.Roseobacterlitoralisgen. nov., sp. nov., andRoseobacterdenitrificanssp. nov., aerobic pink-pigmented bacteria which contain bacteriochlorophylla. Systematic and Applied Microbiology, 1991, 14(2): 140- 145.
[4] Buchan A, Gonzalez J M, Moran M A. Overview of the marineRoseobacterlineage. Applied and Environmental Microbiology, 2005, 71(10): 5665- 5677.
[5] Brinkhoff T, Giebel H A, Simon M. Diversity, ecology, and genomics of theRoseobacterclade: a short overview. Archives of Microbiology, 2008, 189(6): 531- 539.
[6] Lenk S, Moraru C, Hahnke S, Arnds J, Richter M, Kube M, Reinhardt R, Brinkhoff T, Harder J, Amann R, Mußmann M.Roseobacterclade bacteria are abundant in coastal sediments and encode a novel combination of sulfur oxidation genes. The ISME Journal, 2012, 6(2): 2178- 2187.
[7] Eilers H, Pernthaler J, Peplies J, Glockner F O, Gerdts G, Amann R. Isolation of novel pelagic bacteria from the German bight and their seasonal contributions to surface picoplankton. Applied and Environmental Microbiology, 2001, 67(11): 5134- 5142.
[8] DeLong E F, Karl D M. Genomic perspectives in microbial oceanography. Nature, 2005, 437(7057): 336- 342.
[9] Suzuki M T, Preston C M, Béjà O, de la Torre J R, Steward G F, DeLong E F. Phylogenetic screening of ribosomal RNA gene-containing clones in bacterial artificial chromosome (BAC) libraries from different depths in Monterey Bay. Microbial Ecology, 2004, 48(4): 473- 488.
[10] Moran M A, Buchan A, Gonzalez J M, Heidelberg J F, Whitman W B, Kiene R P, Henriksen J R, King G M, Belas R, Fuqua C, Brinkac L, Lewis M, Johri S, Weaver B, Pai G, Eisen J A, Rahe E, Sheldon W M, Ye W Y, Miller T R, Carlton J, Rasko D A, Paulsen I T, Ren Q H, Daugherty S C, Deboy R T, Dodson R J, Durkin A S, Madupu R, Nelson W C, Sullivan S A, Rosovitz M J, Haft D H, Selengut J, Ward N. Genome sequence ofSilicibacterpomeroyireveals adaptations to the marine environment. Nature, 2004, 432(7019): 910- 913.
[11] Venter J C, Remington K, Heidelberg J F, Halpern A L, Rusch D, Eisen J A, Wu D Y, Paulsen L, Nelson K E, Nelson W, Fouts D E, Levy S, Knap A H, Lomas M W, Nealson K, White O, Peterson J, Hoffman J, Parsons R, Baden-Tillson H, Pfannkoch C, Rogers Y H, Smith H O. Environmental genome shotgun sequencing of the Sargasso Sea. Science, 2004, 304(5667): 66- 74.
[12] Jiao N Z, Zhang Y, Zeng Y H, Hong N, Liu R L, Chen F, Wang P X. Distinct distribution pattern of abundance and diversity of aerobic anoxygenic phototrophic bacteria in the global ocean. Environmental Microbiology, 2007, 9(12): 3091- 3099.
[13] Hauruseu D, Koblizek M. Influence of light on carbon utilization in aerobic anoxygenic phototrophs. Applied and Environmental Microbiology, 2012, 78(20): 7414- 7419.
[14] Oz A, Sabehi G, Koblizek M, Massana R, Beja O.Roseobacter-like bacteria in red and mediterranean sea aerobic anoxygenic photosynthetic populations. Applied and Environmental Microbiology, 2005, 71(1): 344- 353.
[15] King G M. Molecular and culture-based analyses of aerobic carbon monoxide oxidizer diversity. Applied and Environmental Microbiology, 2003, 69(12): 7257- 7265.
[16] Gulvik C A, Buchan A. Simultaneous catabolism of plant-derived aromatic compounds results in enhanced growth for members of theRoseobacterlineage. Applied and Environmental Microbiology, 2013, 79(12): 3716- 3723.
[17] Buchan A, Collier L S, Neidle E L, Moran M A. Key aromatic-ring-cleaving enzyme, protocatechuate 3, 4-dioxygenase, in the ecologically important marineRoseobacterlineage. Applied and Environmental Microbiology, 2000, 66(11): 4662- 4672.
[18] Buchan A, Neidle E L, Moran M A. Diversity of the ring cleaving dioxygenase genepcaHin a salt marsh bacterial community. Applied and Environmental Microbiology, 2001, 67(12): 5801- 5809.
[19] Brito E M S, Guyoneaud R, Goni-Urriza M, Ranchou-Peyruse A, Verbaere A, Crapez M A C, Wasserman J C A, Duran R. Characterization of hydrocarbonoclastic bacterial communities from mangrove sediments in Guanabara Bay, Brazil. Research in Microbiology, 2006, 157(8): 752- 762.
[20] Petra J S, Simon K L, Kadali K K, Patil S S, Ball A S. The importance of weathered crude oil as a source of hydrocarbonoclastic microorganisms in contaminated seawater. Journal of Microbiology & Biotechnology, 2012, 22(9): 1185- 1192.
[21] Rink B, Seeberger S, Martens T, Duerselen C D, Simon M, Brinkhoff T. Effects of phytoplankton bloom in a coastal ecosystem on the composition of bacterial communities. Aquatic Microbial Ecology, 2007, 48: 47- 60.
[22] Goecke F, Thiel V, Wiese J, Labes A, Imhoff J F. Algae as an important environment for bacteria-phylogenetic relationships among new bacterial species isolated from algae. Phycologia, 2013, 52(1): 14- 24.
[23] Dickschat J S, Zell C, Brock N L. Pathways and substrate specificity of DMSP catabolism in marine bacteria of theRoseobacterclade. Chembiochem, 2010, 11(3): 417- 425.
[24] Krishnani K K, Kathiravan V, Natarajan M, Kailasam M, Pillai S M. Diversity of sulfur-oxidizing bacteria in greenwater system of coastal aquaculture. Applied Biochemistry and Biotechnology, 2010, 162(5): 1225- 1237.
[25] Todd J D, Kirkwood M, Newton-Payne S, Johnston A W. DddW, a third DMSP lyase in a model Roseobacter marine bacterium,RuegeriapomeroyiDSS- 3. ISME Journal, 2011, 6(1): 223- 226.
[26] Geng H F, Belas R. Molecular mechanisms underlying roseobacter-phytoplankton symbioses. Current Opinion in Biotechnology, 2010, 21(3): 332- 338.
[27] Alavi M, Miller T, Erlandson K, Schneider R, Belas R. Bacterial community associated withPfiesteria-like dinoflagellate cultures. Environmental Microbiology, 2001, 3(6): 380- 396.
[28] Hube A E, Heyduck-soller B, Fischer U. Phylogenetic classification of heterotrophic bacteria associated with filamentous marine cyanobacteria in culture. Systematic and Applied Microbiology, 2009, 32(4): 256- 265.
[29] Amaro A M, Fuentes M S, Ogalde S R, Venegas J A, Suárez-isla B A. Identification and characterization of potentially algal-lytic marine bacteria strongly associated with the toxic dinoflagellateAlexandriumcatenella. Journal of Eukaryotic Microbiology, 2005, 52(3): 191- 200.
[30] Riclea R, Gleitzmann J, Bruns H, Junker C, Schulz B, Dickschat J S. Algicidal lactones from the marineRoseobacterclade bacteriumRuegeriapomeroyi. Beilstein Journal of Organic Chemistry, 2012, 8: 941- 950.
[31] Seyedsayamdost M R, Carr G, Kolter R, Clardy J. Roseobacticides: small molecule modulators of an algal-bacterial symbiosis. Journal of the American Chemical Society, 2011, 133(45): 18343- 18349.
[32] Hjelm M, Bergh Ø, Riaza A, Nielsen J, Melchiorsen J, Jensen S, Duncan H, Ahrens P, Birkbeck H, Gram L. Selection and identification of autochthonous potential probiotic bacteria from turbot larvae (Scophthalmusmaximus) rearing units. Systematic and Applied Microbiology, 2004, 27(3): 360- 371.
[33] Ruiz-Ponte C, Cilia V, Lambert C, Nicolas J L.Roseobactergallaeciensissp. nov., a new marine bacterium isolated from rearings and collectors of the scallopPectenmaximus. International Journal of Systematic and Evolutionary Microbiology, 1998, 48(2): 537- 542.
[34] D′Alvise P W, Melchiorsen J, Porsby C H, Nielsen K F, Gram L. Inactivation ofVibrioanguillarumby attached and planktonicRoseobactercells. Applied and Environmental Microbiology, 2010, 76(7): 2366- 2370.
[35] Cude W N, Mooney J, Tavanaei A A, Hadden M K, Frank A M, Gulvik C A, May A L, Buchan A. Production of the antimicrobial secondary metabolite indigoidine contributes to competitive surface colonization by the marine RoseobacterPhaeobactersp. strain Y4I. Applied and Environmental Microbiology, 2012, 78(14): 4771- 4780.
[36] Brinkhoff T, Bach G, Heidorn T, Liang L, Schlingloff A, Simon M. Antibiotic production by aRoseobacterclade-affiliated species from the German Wadden Sea and its antagonistic effects on indigenous isolates. Applied and Environmental Microbiology, 2004, 70(4): 2560- 2565.
[37] Waters C M, Bassler B L. Quorum sensing: cell-to-cell communication in bacteria. Annual Review of Cell and Developmental Biology, 2005, 21(1): 319- 346.
[38] Miller M B, Bassler B L. Quorum sensing in bacteria. Annual Review of Microbiology, 2001, 55(1): 165- 199.
[39] Zan J D, Liu Y, Fuqua C, Hill R. Acyl-Homoserine lactone quorum sensing in theRoseobacterclade. International Jounal of Molecular Sciences, 2014, 15(1): 654- 669.
[40] Zan J D, Cicirelli E M, Mohamed N M, Sibhatu H, Kroll S, Choi O, Uhlson C L, Wysoczynski C L, Murphy R C, Churchill M E A, Hill R T, Fuqua C. A complex LuxR-LuxI type quorum sensing network in a roseobacterial marine sponge symbiont activates flagellar motility and inhibits biofilm formation. Molecular Microbiology, 2012, 85(5): 916- 933.
[41] 蔡海元. 基因转移因子——一类在海洋中广泛存在的介导水平基因转移的新型生物因子. 微生物学报, 2012, 52(1): 12- 21.
[42] Biers E J, Wang K, Pennington C, Belas R, Chen F, Moran M A. Occurrence and expression of gene transfer agent genes in marine bacterioplankton. Applied and Environmental Microbiology, 2008, 74(10): 2933- 2939.
[43] Zhao Y L, Wang K, Budinoff C, Buchan A, Lang A, Jiao N Z, Chen F. Gene transfer agent (GTA) genes reveal diverse and dynamicRoseobacterandRhodobacterpopulations in the Chesapeake Bay. The ISME Journal, 2009, 3(3): 364- 373.
[44] 张永雨, 黄春晓, 杨军, 焦念志. 海洋微生物与噬菌体间的相互关系. 科学通报, 2011, 56(14): 1071- 1079.
[45] Rohwer F, Segall A, Steward G, Seguritan V, Breitbart M, Wolven F, Azam F. The complete genomic sequence of the marine phageRoseophageSIO1 shares homology with nonmarine phages. Limnology and Oceanography, 2000, 45(2): 408- 418.
[46] Angly F, Youle M, Nosrat B, Seguritan V, Breitbart M, Wolven F, Azam F. Genomic analysis of multiple Roseophage SIO1 strains. Environmental Microbiology, 2009, 11(11): 2863- 2873.
[47] Zhang Y Y, Jiao N Z. Roseophage RDJLΦ1, infecting the aerobic anoxygenic phototrophic bacteriumRoseobacterdenitrificansOCh114. Applied and Environmental Microbiology, 2009, 75(6): 1745- 1749.
[48] Zhang Y Y, Zhang F, Yang J, Jiao N. Host responses of a marine bacterium,RoseobacterdenitrificansOCh114, to phage infection. Archives of Microbiology, 2012, 194(5): 323- 330.
[49] Huang C X, Zhang Y Y, Jiao N Z. Phage resistance of a marine bacterium,RoseobacterdenitrificansOCh114, as revealed by comparative proteomics. Current Microbiology, 2010, 61(2): 141- 147.
[50] Huang S J, Zhang Y Y, Chen F, Jiao N Z. Complete genome sequence of a marine roseophage provides evidence into the evolution of gene transfer agents in alphaproteobacteria. Virology Journal, 2011, 8(1): 124- 124.
[51] Zhang Y Y, Jiao N Z, Colquhoun R D, Halden R U, Chen F. Protein modifications related to phage resistance in a marine roseobacter. Aquatic Microbial Ecology, 2009, 55: 203- 207.
[52] Zhao Y L, Wang K, Ackermann H W, Halden R U, Jiao N, Chen F. Searching for a “Hidden”prophage in a marine bacterium. Applied and Environmental Microbiology, 2010, 76(2): 589- 595.
[53] Zhao Y L, Wang K, Jiao N Z, Chen F. Genome sequences of two novel phages infecting marine roseobacters. Environmental Microbiology, 2009, 11(8): 2055- 2064.
[54] Kang I, Jiang H, Oh H M, Cho J C. Complete genome sequence ofCeleribacterbacteriophage Pl2053L. Journal of Virology, 2012, 86(15): 8339-8340.
Research progresses of marineRoseobacterlineage
CHEN Zhenghao1, 2, ZHANG Yongyu2,3, YANG Suping1,*
1DepartmentofBiologicalEngineeringandTechnology,HuaqiaoUniversity,Xiamen361021,China2KeyLaboratoryofUrbanEnvironmentandHealth,InstituteofUrbanEnvironment,ChineseAcademyofSciences,Xiamen361021,China3StateKeyLaboratoryofMarineEnvironmentalScience,XiamenUniversity,Xiamen361005,China
MarineRoseobacterclade is a phylogenetically coherent, but physiologically diverse group of α-Proteobacteria, with members sharing >89% identity of the 16S rRNA gene. It contains more than 40 different genera.Roseobacterwas found to be abundant in marine environment, especially in coastal and polar oceans, whereRoseobacterclade comprises 15%—25% of the total bacterioplankton communities. Roseobacters also exist ubiquitously in the ocean. Based on culture collections, 16S rRNA clone libraries, and single-cell analyses, roseobacters have been identified in most marine environment sampled, such as sea ice, sea floor, hypersaline microbial mats, sponges, sea grasses, and coastal biofilms, etc. whereas, this bacterial group is noticeable absent in analogous freshwater and terrestrial soil environment. Quantitative 16S rRNA gene analyses show thatRoseobacterpopulations fall off with depth in the ocean, and are often most abundant in bacterial communities associated with marine algae.Roseobacterlineage is the only abundant marine group, whose cultivated representatives are closely related to the not-yet-cultivated environmental clone sequences. Diverse life styles have been found inRoseobacterclade, such as free living, particle associated, or in commensal relationships with marine phytoplankton, invertebrates, and vertebrates. Also, diverse physiological metabolic traits were found in this bacterial group. For example, some roseobacters can generate metabolic energy from light using the ancient purple bacterial mechanism of anaerobic photosynthesis without production of oxygen (anaerobic anoxygenic photosynthesis), while some roseobacters have the physiological metabolic traits of degradation of sulfur compounds or aromatics, oxidation of carbon monoxide, or reduction of trace metals, etc. Thus, it is considered that roseobacters may play important roles in marine carbon and sulfur cycles, as well as the global climate regulation. There are more than forty roseobacters whose whole genome sequences are available in the current public databases. Analysis of these genomes also reflected the metabolic versatility of theRoseobacterlineage. The cellular requirements of roseobacters for nitrogen are largely satisfied by regenerated ammonium and organic compounds (polyamines, allophanate, and urea), while the requirement for carbon sources are satisfied by amino acids, glyoxylate, and aromatic metabolites. Also, a large number of genes are predicted to encode proteins involved in the production, degradation, and efflux of toxins and metabolites, suggesting the potential for interacting with neighboring cells and impacting the routing of organic matter into the microbial loop. Laboratory experiments found that the various bioactive secondary metabolites of roseobacters include antagonist against fish larval bacterial pathogens, antibiotic against marine bacteria and algae, shellfish poison, and the bioactive LuxR-activating acylated homoserine lactones (AHLs), which are a class of signaling molecules involved in bacterial quorum sensing, etc. Recently, the interactions between roseobacters and their phages caused widespread attention. Several host-phage interaction systems of the roseobacters were set up in the laboratory. Laboratory experiments revealed that roseophages play important roles in mediating the physiology and promoting the evolution of roseobacters in marine environment. This review described the recent research progresses ofRoseobacterlineage in terms of their ecological distribution, lifestyle, physiological functions, and genome features. Finally we suggested future research directions based on our understanding of the literature and our own work.
Roseobacterlineage; aerobic anoxygenic photosynthesis; sulfur metabolism; secondary metabolites; roseophage
国家重大科学研究计划(2013CB955700); 国家自然科学基金项目(41006087, 31070054); 厦门市科技计划项目(3502Z20132014); 厦门大学近海海洋环境科学国家重点实验室访问学者基金(MELRS1206)
2013- 10- 17;
日期:2014- 07- 14
10.5846/stxb201310172507
*通讯作者Corresponding author.E-mail: yangsuping@hqu.edu.cn
陈正浩, 张永雨, 杨素萍.海洋玫瑰杆菌类群研究进展.生态学报,2015,35(5):1620- 1629.
Chen Z H, Zhang Y Y, Yang S P.Research progresses of marineRoseobacterlineage.Acta Ecologica Sinica,2015,35(5):1620- 1629.

