中国“合成生物学”973项目研究进展
陈国强,王颖
“合成生物学”这门工程和科学结合的新学科在近年来得到各国的关注和大量的投资。带着未来对重塑生命和改造生命的憧憬,各个学科包括生命、工程、计算机甚至机械制造等,都投入了设计生命和合成生命的研究行列中。
合成生物学是指按照一定的规律和已有的知识,一方面设计和建造新的生物零件、装置和系统;另一方面重新设计已有的天然生物系统为人类的特殊目的服务。简单地说, 合成生物学就是通过人工设计和构建自然界中不存在的生物系统来解决能源、材料、健康和环保等问题。合成生物学强调“设计”和“重设计”。设计、模拟、实验是合成生物学的基础。合成生物学不仅仅是实验,利用已有的生物学知识,根据实际的需要进行设计和重设计,建立数学模型对人工的设计进行模拟从而指导实验的进行也是合成生物学的方法。合成生物学的核心问题之一是“新生命的合成”。通过新生命的合成扩大生命的能力,更好地为人类服务。
我国是制造业大国,所以,自2011年科技部开始对“合成生物学”进行支持,包括973项目10项和863项目一项。我国主要支持了与制造业“工业生物技术”相关的“合成生物学”研究 7项,主要以容易改造的微生物为研究对象。同时,也开始支持了一项以哺乳细胞为对象的合成生物学,以及一项植物的“合成生物学”。下面对上述科技部支持的项目的研究进展进行回顾。
1 “人工合成细胞工厂”研究的主要进展
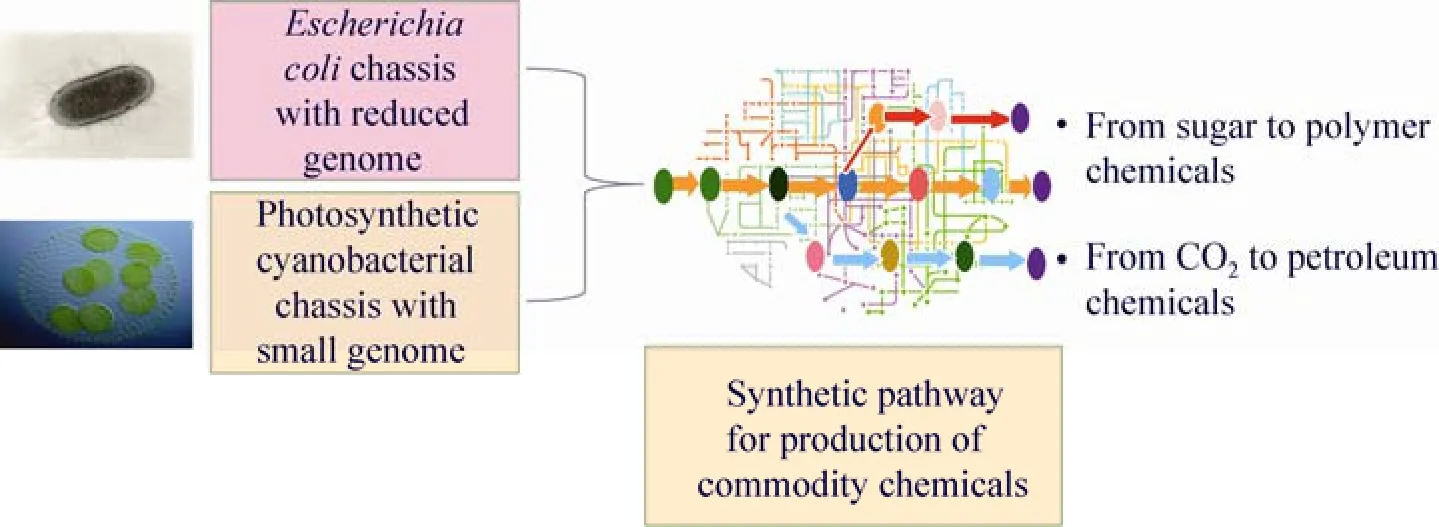
图1 人工合成细胞工厂973项目框架Fig. 1 Framework of 973 project on artificial cell factories. The project consists of constructions of E. coli chassis with simplified chromosome, phototrophic chassis cells with reduced chromosomes, and novel bulk chemicals biosynthesis pathways. The project should allow productions of bulk materials and chemicals from sugars or CO2.
以中国科学院天津工业生物技术研究所马延和为首席的“人工合成细胞工厂”973项目,以人工合成生物体的工业应用为目标,集中研究解决底盘细胞与化学品合成途径设计与构建等方面的关键科学问题,围绕大肠杆菌和光合蓝细菌两类代表性的底盘细胞构建,以及从 CO2与葡萄糖到化学品两类合成途径的创建,构建人工合成细胞工厂 (图1)。项目立项近4年来,在底盘细胞构建、化学品合成的元件设计合成、合成途径创建及合成细胞工厂构建,以及合成生物学基本技术开发方面取得了一系列重要进展。进一步发展和完善了基于芯片的基因合成技术,采用错配特异性内切酶和结合蛋白Muts降低寡核苷酸及组装的 DNA的错误率,将芯片合成的寡核苷酸错误率从1/500 bp降低到1/8 700 bp。同时实现了基因的原位扩增和组装,突破了目前需要先合成短片段DNA再拼接的传统DNA合成模式,大大降低了成本水平[1]。针对认识生命本质、突破细胞进化限制、实现人工生命体构建的需要,通过大肠杆菌基因组必需基因的分析,重构了模块化、成簇化“必需基因功能群”;在建立途径模拟计算设计技术的基础上[2],设计合成人工糖酵解途径,增加细胞生长10%,加快葡萄糖消耗速度达20%,为“糖酵解高速公路”构建奠定基础。建立了新的蛋白质设计方法,可成功实现给定目标结构的蛋白全序列从头设计[3],基于化学反应特征和胞内调控位点,计算设计与合成表征了满足30种重要化学品生物合成的功能元件,形成了约1 200个功能特性清晰的元件库,为化学品合成途径和高效人工合成细胞工厂创建奠定了物质基础功能元件[4]。利用DNA组装和精确表达调控技术[5],创建和优化了从葡萄糖到丁二酸、戊二胺、己二酸和 5-氨基乙酰丙酸等途径,达到预期原子经济性,其中5-氨基乙酰丙酸产量达到50 g/L,比国际水平高5倍,丁二酸产量达到125 g/L,产物对底物葡萄糖的转化率为105% (可部分固定 CO2)[6],技术指标处于国际领先水平。在对光合蓝细菌底盘细胞的生理调控机制进行系统研究和形成较为完整的理解和认识的基础上,构建了一系列获得抗逆性能提高的光合蓝细菌底盘细胞[7],通过光合模块、CO2固定和生物合成模块的重构和优化,实现了从 CO2生物合成酮、醇和酸等典型化学品,为发展和利用新的碳资源提供了可能,其中丙酮、D-乳酸和 3-羟基丁酸均为国际上首次报道[8-9]。
2 “新功能人造生物器件的构建及集成”研究的主要进展
以中国科学院上海生命科学院赵国屏为首席的 973项目围绕新功能人造生物器件的设计原理、合成组装、模块构建、标准化建库及在底盘细胞中集成与适配机制等关键科学问题和相关技术难点,以萜类化合物的人工异源合成为主要研究对象,取得了若干重要进展。尤其是在稀有人参皂苷CK[10](图2)、甜菊糖苷和丹参酮前体铁锈醇[11]等重要药用食用萜类化合物的器件挖掘、集成及异源合成方面取得重要突破。此外,在元件模块的挖掘[12-13]与合成技术创新 (MASTER ligation方法)[14]等方面,亦取得显著进展。
人参皂苷CK具有抗癌、保肝、抗炎症和治疗糖尿病等生理活性,但在人参中从未被检测到,历来依靠微生物来源的糖基水解酶体外制备。中国科学院上海生命科学研究院合成生物学重点实验室周志华课题组发现并鉴定了 1个来源于人参的UDP-糖基转移酶,构建了可以生产“非天然人参皂苷”的酵母细胞工厂,实现了从单糖到稀有人参皂苷 CK的从头全合成,发现人参体内可能合成CK的两条并行代谢途径 (图2)。
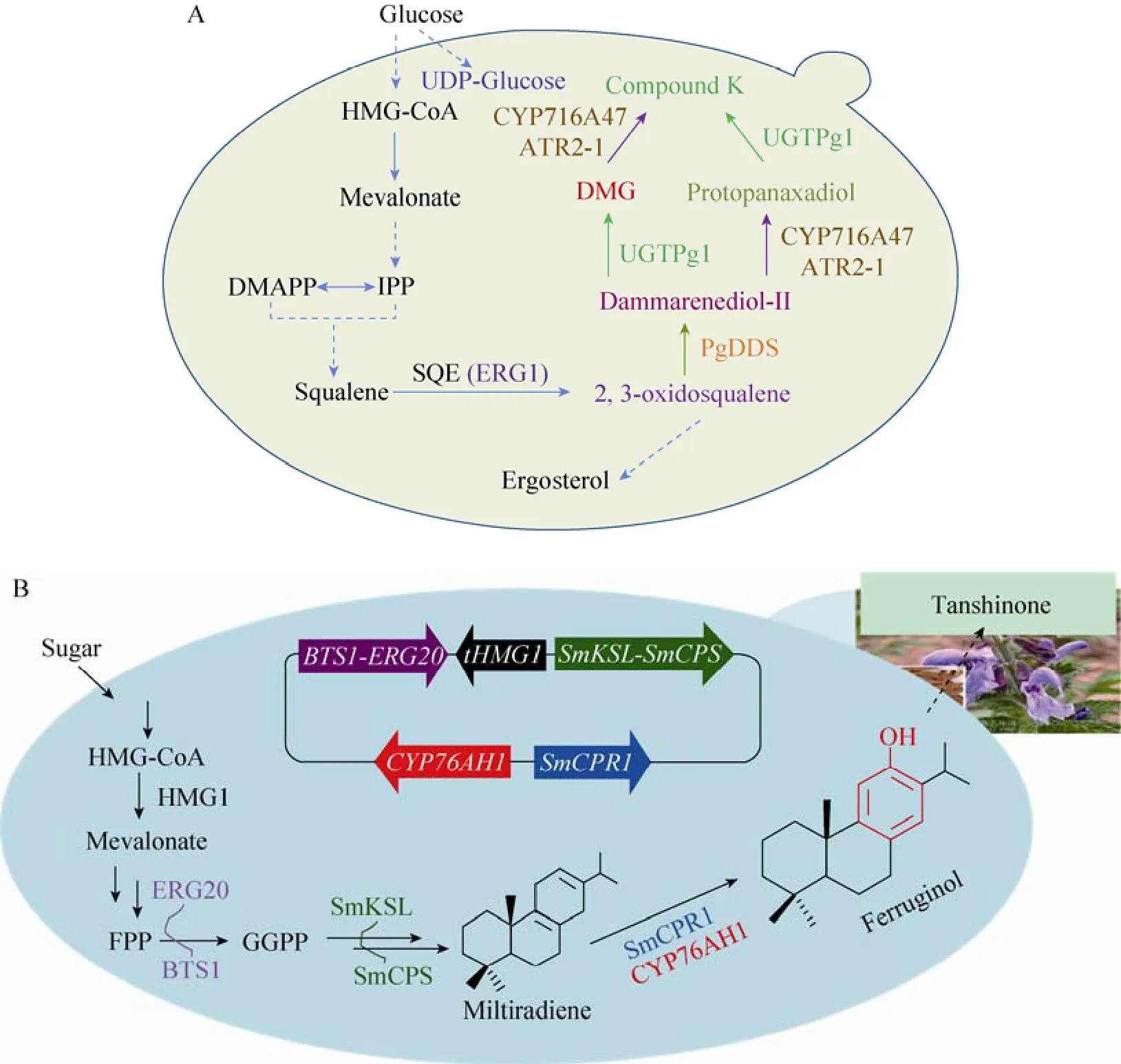
图2 重组酵母细胞工厂中人参皂苷CK (A) 和次丹参酮二烯及铁锈醇 (B) 的合成Fig. 2 Synthese of ginsenoside compound CK (A), miltiradiene and ferruginol (B) by recombinant yeast cell factories.
丹参酮为传统药用植物丹参的萜类活性产物。中国科学院大连化学物理研究所赵宗保课题组以酿酒酵母为宿主,采用模块途径工程策略,组装二萜前体合成、次丹参酮二烯合成和细胞色素P450氧化的模块 (CYP76AH1),优化模块内部各元件间的适配性,构建了次丹参酮二烯和铁锈醇人工合成细胞工厂,产量分别达到360 mg/L和40 mg/L,对丹参酮及其他植物源萜类化合物的微生物制造具有重要参考价值。
3 “微生物药物创新与优产的人工合成体系”研究的主要进展
上海交通大学的冯雁作为 973项目的首席科学家,通过工程化的系统设计、用标准化和模块化的元素来构建人工合成生物体系,高效地建立和完善微生物药物创新和优产的新模式、推动微生物药物产业的跨越式提升和发展。
武汉大学胡黔楠研究组构建了逆向设计微生物药物合成的平台。他们重组了具有化学转化意义和基于代谢途径的基因组规模代谢网络,从40 000多篇生物合成文献中有效整合了超过30 000多条的生物合成反应数据,实现了生物合成反应关键反应模式提取的技术rxnpatter;开发了基于生物合成反应结构的催化该反应类型的生物合成酶的在线预测服务器ECAssigner;构建了“生物合成反应数据库——生物合成转换规律——生物合成途径设计——生物合成实验验证”的技术体系Rxnip[15-17](图3)。该数据库受到国际上的广泛关注,被Elsevier公司的SciVerse收录。该工具不仅可用于微生物药物的合成,并且在平台化学品、生物材料等化合物的人工合成体系构建中也具有重要的应用潜力。
催化元件的系统匹配与优化是实现人工代谢途径高效运行的基础。由于传统代谢工程策略缺少相应途径的稳态动力学参数,以及对所研究代谢途径的认识不够充分,很难去建立一个通用、高效的系统,这也影响了构建人工合成生物体系的效率。应用“定向代谢工程”技术,即基于催化体系体外重建的合成途径快速优化的体系,通过系统的稳态动力学分析,快速获取了所研究代谢途径中各个组分的贡献,进而指导体内定向改造工作 (图 4)。应用这套体系对多种微生物药物的关键合成途径中的限速步骤进行了确定,在短时间内即极大提高了萜类化合物法尼烯[18]、番茄红素、胡萝卜素和虾青素等产量,体现了该方法的广阔应用潜力,其中番茄红素已经通过150 L中试发酵放大,发酵时间相比传统的霉菌发酵缩短为原来的1/5,目前正在进行进一步工业放大测试;而且这种方法可以拓展到聚酮类药物的优化和高产中,为聚酮合酶元件的适配机制的确定和元件的比例优化和筛选提供平台[19]。
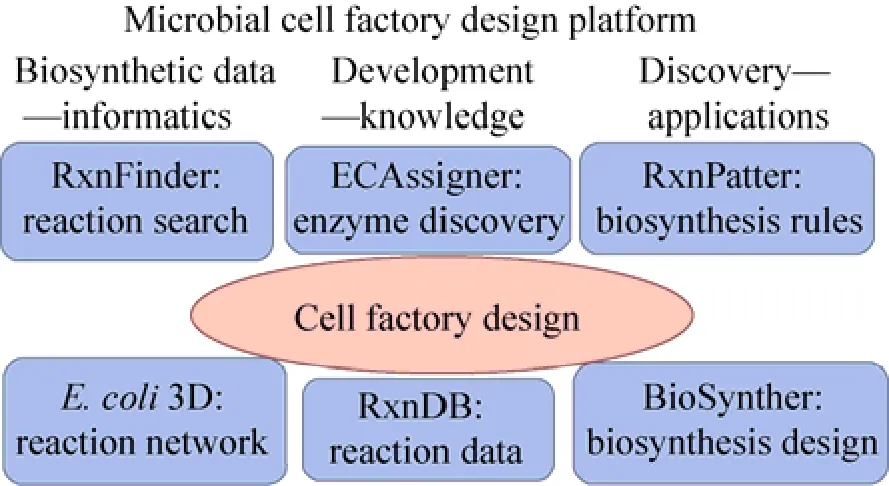
图3 微生物药物生物合成的逆向途径设计平台Fig. 3 Reverse biosynthesis pathway design platform for microbial pharmaceutics. It involves extraction of bioinformatics from antibiotics producing Streptomycetes,developments of models for biosynthesis and transformation, reverse design of biosynthesis pathways from secondary metabolic products et al.
4 “用合成生物学方法构建生物基材料的合成新途径”研究的主要进展
清华大学陈国强为首席的 973项目采用化学与生物结合的方法设计、合成和组装各种生物基材料聚羟基脂肪酸酯PHA的合成路径,通过合成生物学双稳态调控的方法改变了微生物的生长模式,并获得了有复合功能的微生物合成体系,实现细菌基因组减少10%的目标,使微生物达到生长快速、体积大、生物基材料结构丰富的新特点[20]。他们通过网络优化、最小基因组等设计了至少 3条新的多基因合成路径,并验证了其正确性。获得至少两个新的生物基材料[21]。在更大规模 (发酵罐上) 实现了微生物生长状态的变化,使下游处理简单化[22]。此外,他们建立了嗜盐单胞菌的遗传操作平台,并开发了其在人工模拟海水中、利用成分近似餐厨垃圾的混合底物实现连续无灭菌发酵的发酵工艺,降低了生产成本[23]。目前该项目已经建立
了嗜盐微生物的合成生物学研究体系。该体系通过合成生物学手段,构建了制造各种化学品的平台[24],包括 5-氨基乙酰丙酸、维生素 B12和 3-羟基丙酸等。该嗜盐细菌生物制造平台具有如下特点,克服了一般生物制造的缺点,使生物制造成本能大幅降低,包括:1) 节能:发酵过程无需高温高压灭菌;2) 节水:可以用海水替代淡水,而且过程产生的水可以多次循环利用;3) 连续发酵制造:由于染菌几率减少了,生产过程可以连续起来;4) 过程操作简单:由于无需灭菌和可连续的发酵,简化了操作过程;5) 设备投资大幅度减少:由于无需发酵过程无需高温高压灭菌,就不需要使用昂贵的不锈钢发酵罐和不锈钢管道系统,转而使用便宜的塑料、陶瓷甚至水泥罐体或管道等;6) 不与人争粮:筛选的嗜盐细菌可以利用淀粉、蛋白、脂肪、甚至纤维素和脂肪酸等生长,这些都是食物的组成。用餐厨废料也能使嗜盐细菌生长,制造我们所需的产品;7) 产物最终浓度大幅度提高:通过分子操作,使嗜盐细菌在高密度情况下仍然能继续生长,使产品最终浓度大幅提高;8) 产物纯化简单:通过表达细菌分离遏制基因,使细菌形态发生变化,能产生自凝絮作用,使菌体与发酵液自然分离;9) 一个菌种,多个产品:已经开发成功的分子操作技术,可以通过代谢工程使嗜盐细菌生产多种材料、化学品和燃料;10) 多个产品,相同的发酵条件:由于嗜盐细菌菌种一样,发酵工艺也能保持一致,使工艺开发简单化。
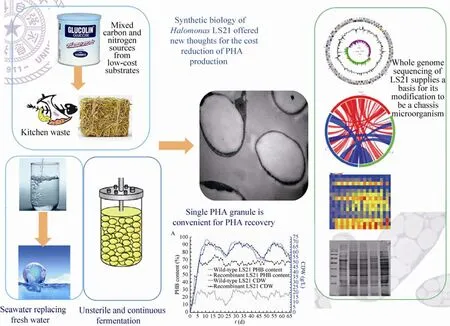
图5 嗜盐微生物的合成生物学研究体系 (克服了一般生物制造的缺点,使生物制造成本大幅降低)Fig. 5 Synthetic biology of Halomonas LS21 grown in seawater under unsterile and continuous processes(overcoming the disadvantages of bio-manufacturing including consumptions of fresh water, food based substrates as well as heavy demand on energy for sterilization et al). The whole genome of strain LS21 has been sequenced allowing for many DNA manipulations. The strain produces only a single PHA granule in each cell, making the PHA recovery convenient.
5 “抗逆元器件的构建和机理研究” 研究的主要进展
由清华大学林章凛主持的 973项目研究适配微生物和植物的合成生物学抗逆元器件 (即元件与模块),及其在工业和农业上的应用,以期提升我国庞大生物制造业的节能减排,如2 000亿元年产值的发酵产业菌种的抗酸和抗高温需求;并储备高效抗逆农业的战略技术,尤其是670多hm22%以及以下盐碱地的利用。由于产业和资源特点的不同,欧美国家尚未特别关注抗逆元器件研究,因此这也是我国参与国际合成生物学研究竞争的独特机遇。前两年项目围绕如下两个关键科学问题开展工作 (表 1):1)抗逆元器件构建的分子设计原理;2) 抗逆元器件在底盘生物的作用机理。建立了有特色的抗逆元件库、形成了抗逆器件设计和实验室评估能力;提出了利用极端微生物调控基因的重要科学思想[25];发展了一个系统的商用微生物抗酸器件逐级评估技术 (从实验室到发酵工厂),并在有重要产业应用价值的微生物抗酸器件和植物抗盐碱器件上取得重要突破,分别通过工业菌株工厂发酵的验证和田间试验的验证,大幅度地提前完成了相关实施效果的目标。
6 “合成微生物体系的适配性研究”研究的主要进展
以中国科学院微生物研究所张立新为首席的973项目,选择了微生物药物阿维菌素和polynikA为研究对象,开展基于生物底盘调控特性的生物元件表征和设计,解决底盘代谢网络与其加载的外源生物元件的兼容性问题,突出人工合成的调控元件对结构基因定时、定量表达的决定性作用[26]。以聚酮类药物和核苷肽类药物的合成途径作为代表性案例,在已建立的技术平台上高通量地分析和解决不同元件之间的适配性问题,构建大肠杆菌和链霉菌中的适配性研究体系。同时,对异源模块与生物底盘之间交互式作用的计算与预测、适配性研究与系统优化,深化对生物功能器件反应特性及加载生物元件的底盘代谢网络的认识,借鉴工程学原理在非生命系统中应用的成功经验,同时考虑生命系统的特殊性,实现对合成生物系统的各调控单元模块化、标准化,并最终在合成微生物体系中进行系统集成,揭示具有非天然新功能的生物元件、器件、模块,以及更高层次的合成生物系统构建过程的新原理和新途径[27](图 6)。

表1 抗逆元器件的构建和机理研究创新点Table 1 Construction of stress resistance biobricks and the novelties of the mechanism study
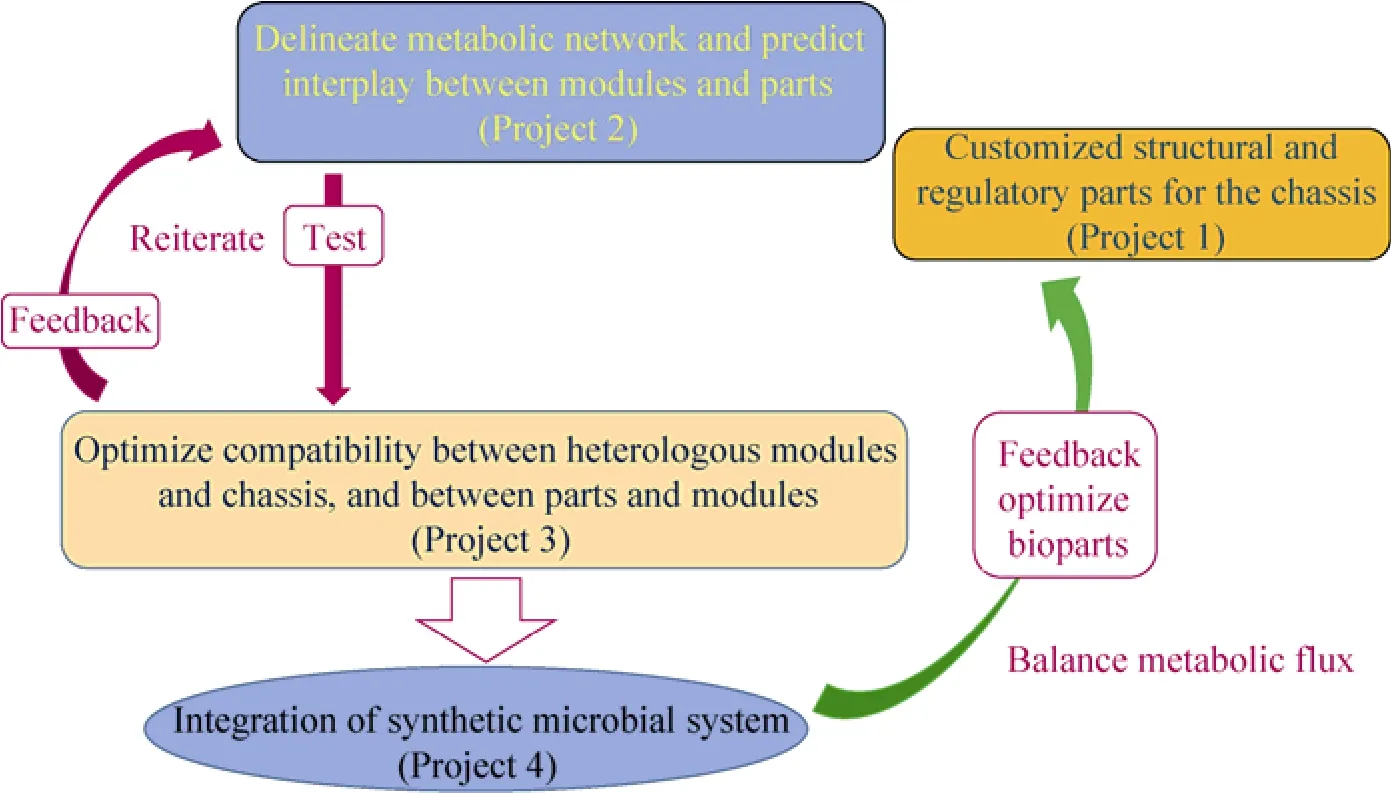
图6 “合成微生物体系的适配性研究”各课题相互之间的关系及课题与所需解决科学问题Fig. 6 Relationship among tasks and scientific problems to be solved by project of “Comaptibility study of synthetic microorganism systems”, including project 1 regulatory and structural parts based on chassis property;project 2 modules of chassis metabolic network and prediction on functions of parts; project 3 optimized compatibility of hetereogeous modules to chassis and optimization of modular compatibility; project 4 assembly of synthetic microorganism system.
本项目全面解析了模式链霉菌基因转录时序[28-30],得到了组成型和时序型启动子库,并改造获得了目前链霉菌中最强的启动子;建立基于流式细胞仪的链霉菌单细胞启动子表征平台;在链霉菌中建立 3套新型诱导表达系统;利用合成生物学遗传电路原理,建立新的抑制蛋白筛选方法;解析了抗生素介导的新调控机制,为适配性设计提供了思路和解决方案。同时,针对目标产物,首次考察了生物合成途径中基于自由能变化的原子经济性,构建了加载外源生物模块的大肠杆菌和链霉菌底盘代谢网络,并通过将FBA和MOMA结合来预测底盘细胞加载外源生物模块后的适配性,指导对链霉菌底盘中的前体供应、还原力、产物转运体系进行了改造,对大肠杆菌底盘细胞中建立糖酵解途径、磷酸戊糖途径和三羧酸循环途径代谢途径进行改造;对大肠杆菌底盘细胞中与polynikA及阿维菌素前体合成的代谢途径进行改造;设计强化NADPH等辅助因子的循环回补调控通路,保证大肠杆菌底盘细胞中的能量、辅助因子、还原力的平衡,保证合成途径的有效进行;对底盘细胞中与聚酮类药物合成竞争的脂肪酸合成途径进行改造;为大肠杆菌简约全基因组代谢网络模型提供实验数据。此外,在大肠杆菌底盘中建立初级代谢途径的检测手段,建立聚酮类药物、核苷肽类药物前体的高通量筛选方法。最后,还在链霉菌中开发了一种可精确设计和预测的模块化微调转录和翻译过程适配性的新方法,系统集成和自由拆分阿维菌素模块;综合组学分析解析阿维菌素高产机制,并进一步设计、合成、获得超高产菌株,并在工业生产上得到验证。
7 “微生物多细胞体系的设计与合成”研究的主要进展
以天津大学元英进为首席的973项目,围绕“合成微生物多细胞体系的设计与构建原理”和“合成微生物多细胞体系的适配与调控机制”两个关键科学问题展开研究[31-32],在合成微生物多细胞体系实现生物转化、复杂原料利用及低碳底物高碳转化等方面取得了初步进展:1) 在原有维生素C两步发酵混菌体系系统分析的基础上,设计和构建了由改造后的原一步菌Gluconobacter oxydans和改造后的原二步小菌Ketogulonigenium vulgare组成的一步发酵混菌体系,维生素 C前体 2-KGA的产量提高了12.8%,流程的简化使发酵综合成本可下降 30%以上 (图7);2) 根据代谢物互补原则,设计和构建了由酵母Yeast和希瓦氏菌属Shewanella组成的微生物产电人工多细胞体系,前者可利用木质纤维素、甘油甚至二氧化碳等产生乳酸,后者则利用乳酸产生电子 (图8)[33-35]。
8 “合成生物器件干预膀胱癌的基础研究”研究的主要进展
合成生物学技术应用于肿瘤研究在国内外是刚起步的探索性工作,以深圳大学蔡志明教授为首席的973项目“合成生物器件干预膀胱癌的基础研究”在系统生物学的基础上,将合成生物学技术应用于肿瘤治疗研究 (图9)。
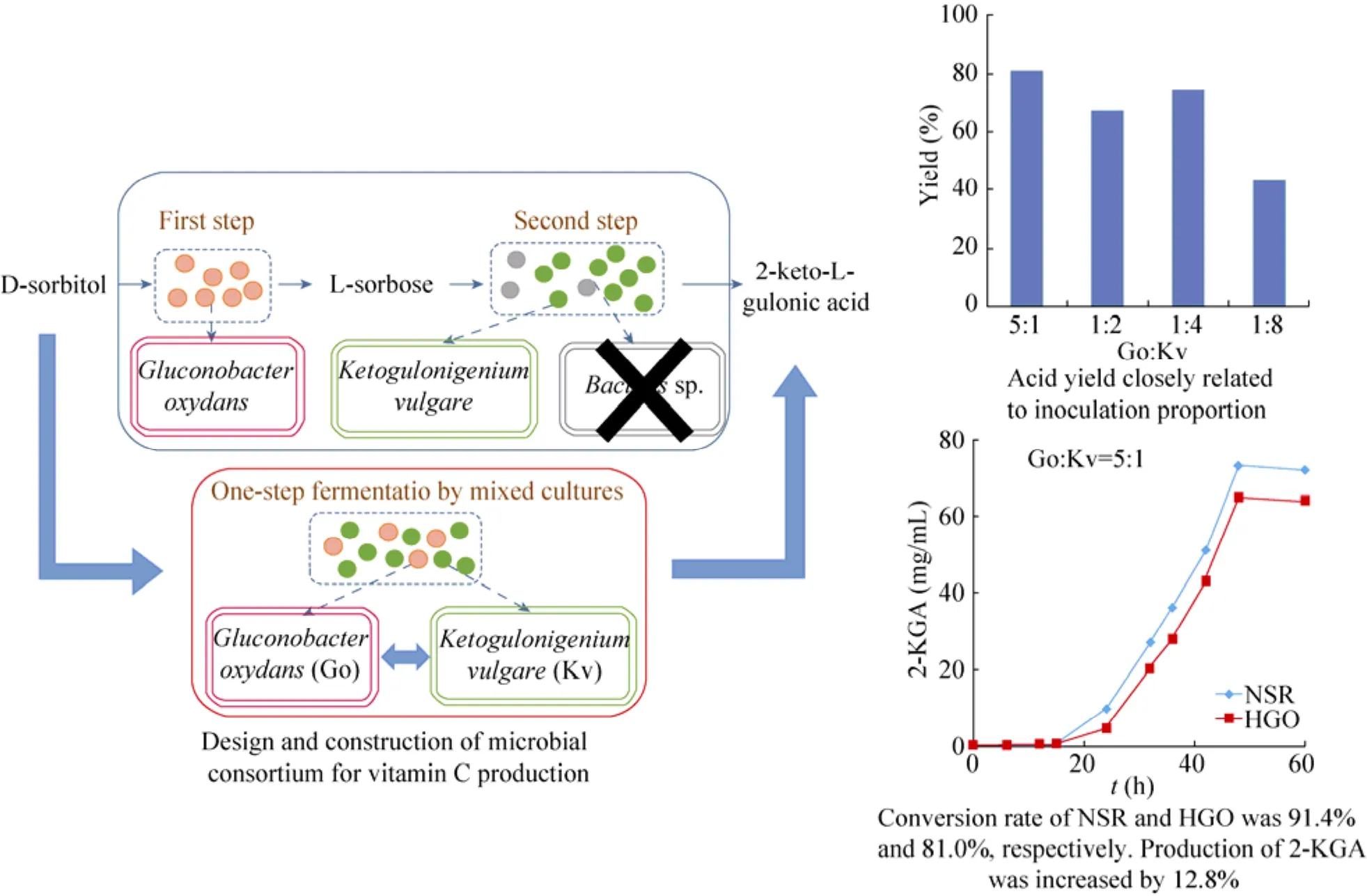
图7 维生素C一步发酵混菌体系设计与构建Fig. 7 Design and construction of one step mixed culture fermentation for vitamine C production.
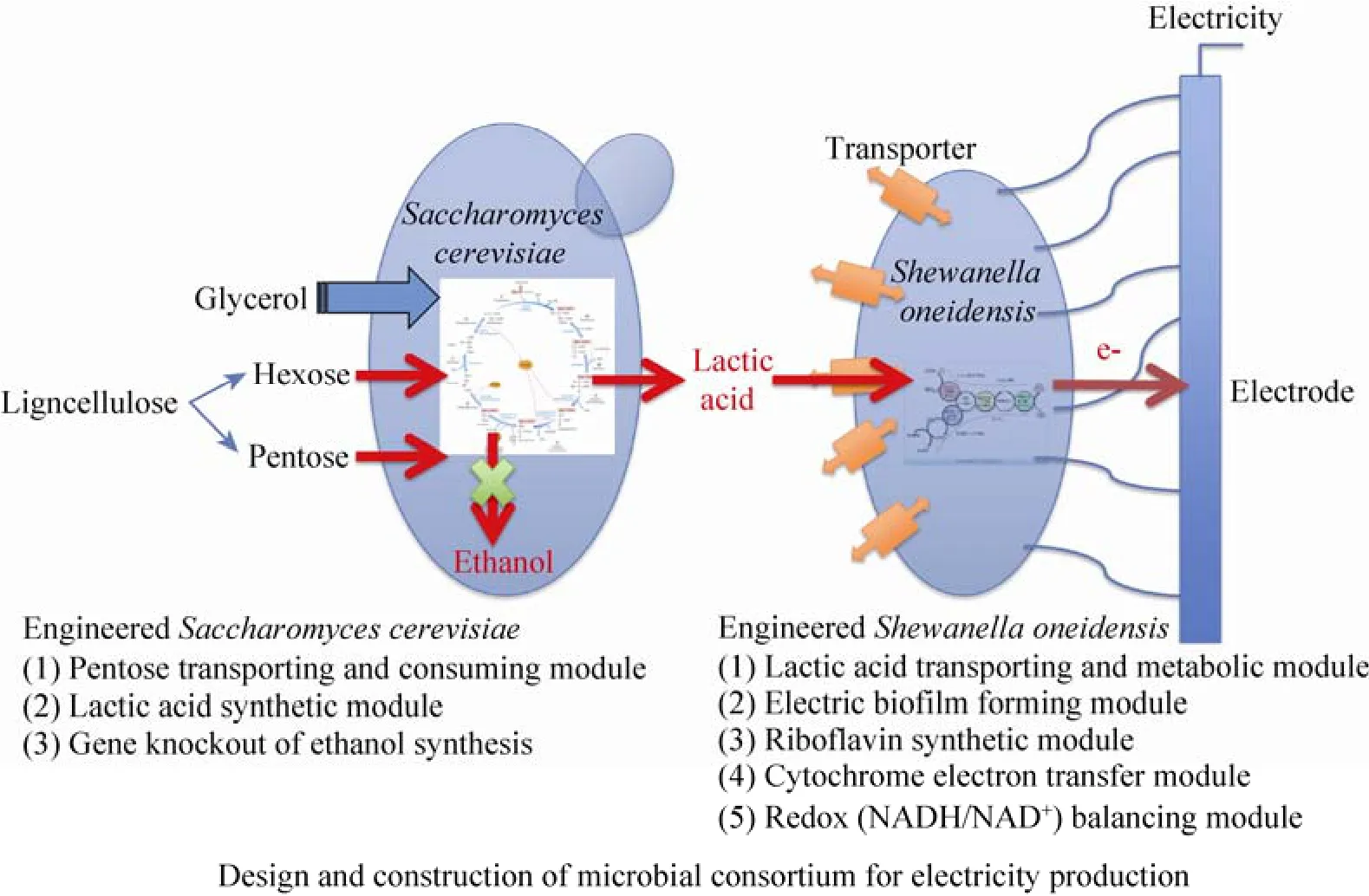
图8 微生物产电人工多细胞体系设计与构建Fig. 8 Design and construction of multiple microbial artificial cells for electricity generation.
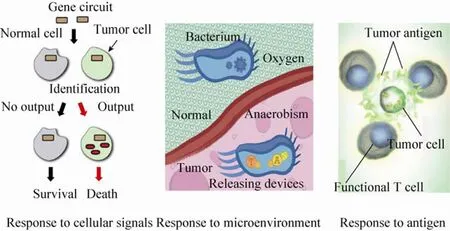
图9 合成生物器件干预肿瘤的基本策略Fig. 9 Basic strategy of interferences on cancer cells using synthetic biology parts.
项目发现了部分膀胱癌关键基因及通路,并在分析相关基因网络信息基础上,利用定量可控工程体系,表征膀胱癌发生发展过程中复杂事件的共性,挖掘出许多具有潜在利用价值的膀胱癌识别和治疗的复合靶标[36]。建立了一个完整的合成生物学靶点筛查与验证平台,用于确定这些潜在靶点是否为有效的靶点[37-38]。创建了正交、可逆的转录抑制子蛋白库,用于构建模块化的合成基因线路及在哺乳动物细胞中进行可编程的操作[39]。项目利用CRISPR-Cas9系统构建出了逻辑“与”门基因遗传线路[40]。该线路能利用膀胱癌细胞内的识别靶点特异区分膀胱癌细胞和其他类型细胞,并通过调控治疗靶点,有效减缓肿瘤细胞的生长速度,诱导其进入凋亡程序,或遏制其迁移运动能力。
项目利用实体肿瘤中低氧浓度的特点,改造出一株工程化的沙门氏菌,该菌以低氧为生存条件,可特异靶向到肿瘤内部,跨界表达多个转基因模块并破坏肿瘤的生长[41]。项目还筛选肿瘤特异性抗原,并通过构建遗传回路对 T细胞进行重编程,使其具备在人工控制条件下特异性识别和杀伤肿瘤细胞的能力。未来,项目将进一步将其优化得到的基因线路装载到细菌和 T细胞载体,合成具有个性化攻击力的治疗性产品,用于体内罹患膀胱肿瘤的小鼠的动物实验,并进一步测试和优化该器件对膀胱癌细胞的特异杀伤能力。
9 “新型微生物抗逆元件和功能模块研究”研究的主要进展
2014年启动的,以中国农业科学院生物技术研究所林敏为首席的 973项目,针对工农业微生物应用过程易受酸碱、高低温、高渗透压以及干旱等不利影响,根据微生物抗逆与转化分子机制,拟利用合成生物技术,设计优化具有抗高温、耐酸、耐盐以及抗干旱等抗逆标准基因元件与模块,以减少逆境环境的影响、提高工农业微生物的耐受性和相容性、改善生产工艺、提高生产效率和降低生产成本。项目拟解析和构建 3–5个耐高温模块,可使宿主菌耐受温度提高5–10 ℃;解析和构建1–2个耐酸功能模块;解析和构建3−5个耐渗透压功能模块,可使宿主菌耐盐浓度提高到5%–10%。
上述项目的成功实施,将使我国的生物制造,特别是微生物制造得到大幅度提升,使我国在国际合成生物学领域上占有一席之地。
致谢:上述材料由执行 973项目的各位首席科学家提供,在此感谢中国科学院天津工业生物技术研究所马延和研究员、中国科学院上海生命科学院赵国屏院士、上海交通大学冯雁教授、清华大学林章凛教授、中国科学院微生物研究所张立新研究员、天津大学元英进教授、深圳大学蔡志明教授和中国农业科学院生物技术研究所林敏研究员。
[1] Ma SY, Tang N, Tian JD. DNA synthesis,assembly and applications in synthetic biology.Curr Opin Chem Biol, 2012, 16(3/4): 260–267.
[2] Xu ZX, Zheng P, Sun JB, et al. ReacKnock:identifying reaction deletion strategies for microbial strain optimization based on genome-scale metabolic network. PLoS ONE,2013, 8(12): e72150.
[3] Xiong P, Wang M, Zhou XQ, et al. Protein design with a comprehensive statistical energy function and boosted by experimental selection for foldability. Nat Commun, 2014, 5: 5330 .
[4] Hu C, Chan SI, Sawyer EB, et al. Metalloprotein design using genetic code expansion. Chem Soc Rev, 2014, 43(18): 6498–6510.
[5] Yu XY, Liu TG, Zhu FY, et al. In vitro reconstitution and steady-state analysis of the fatty acid synthase from Escherichia coli. Proc Natl Acad Sci USA, 2011, 108(46): 18643–18648.
[6] Zhu XN, Tan ZG, Xu HT, et al. Metabolic evolution of two reducing equivalent-conserving pathways for high-yield succinate production in Escherichia coli. Metab Eng, 2014, 24: 87–96.
[7] Jin H, Chen L, Wang JX, et al. Engineering biofuel tolerance in non-native producing microorganisms.Biotechnol Adv, 2014, 32(2): 541–548.
[8] Zhou J, Zhang HF, Zhang YP, et al. Designing and creating a modularized synthetic pathway in Cyanobacterium synechocystis enables production of acetone from carbon dioxide. Metab Eng, 2012,14(4): 394–400.
[9] Wang B, Pugh S, Nielsen DR, et al. Engineering cyanobacteria for photosynthetic production of 3-hydroxybutyrate directly from CO2. Metab Eng,2013, 16: 68–77.
[10] Yan X, Fan Y, Wei W, et al. Production of bioactive ginsenoside compound K in metabolically engineered yeast. Cell Res, 2014, 24(6): 770–773.
[11] Guo J, Zhou YJ, Hillwig ML, et al. CYP76AH1 catalyzes turnover of miltiradiene in tanshinones biosynthesis and enables heterologous production of ferruginol in yeasts. Proc Natl Acad Sci USA,2013, 110(29): 12108–12113.
[12] Xu JB, Zhang H, Gan LS, et al. Logeracemin A, an anti-HIV Daphniphyllum alkaloid dimer with a new carbon skeleton from Daphniphyllum longeracemosum. J Am Chem Soc, 2014, 136(21):7631–7633.
[13] Guo H, Wang J, Li YM, et al. Insight into bicyclic thiopeptide biosynthesis benefited from development of a uniform approach for molecular engineering and production improvement. Chem Sci, 2014, 5(1): 240–246.
[14] Chen WH, Qin ZJ, Wang J, et al. The MASTER(methylation-assisted tailorable ends rational)ligation method for seamless DNA assembly. Nucl Acids Res, 2013, 41(8): e93.
[15] Hu QN, Deng Z, Hu HN, et al. RxnFinder:biochemical reaction search engines using molecular structures, molecular fragments, and reaction similarity. Bioinformatics, 2011, 27(17):2465–2467.
[16] Hu QN, Zhu H, Li XB, et al. Assignment of EC numbers to enzymatic reactions with reaction difference fingerprints. PLoS ONE, 2012, 7(12):e52901.
[17] Cao DS, Xu QS, Hu QN, et al. ChemoPy: freely available python package for computational biology and chemoinformatics. Bioinformatics,2013, 29(8): 1092–1094.
[18] Zhu FY, Zhong XF, Hu MZ, et al. In vitro reconstitution of mevalonate pathway and targeted engineering of farnesene overproduction in Escherichia coli. Biotechnol Bioeng, 2014, 111(7):1396–1405.
[19] Guo X, Liu TG, Deng ZX, et al. Essential role of the donor acyl carrier protein in stereoselective chain translocation to a fully reducing module of the nanchangmycin polyketide synthase.Biochemistry, 2012, 51(4): 879–887.
[20] Wang Y, Yin J, Chen GQ. Polyhydroxyalkanoates,challenges and opportunities. Curr Opin Biotechnol, 2014, 30: 59–65.
[21] Li SJ, Cai LW, Wu LP, et al. Microbial synthesis of functional homo-, random and block polyhydroxyalkanoates by β-oxidation deleted Pseudomonas entomophila. Biomacromolecules,2014, 15(6): 2310–2319.
[22] Tan D, Wu Q, Chen JC, et al. Engineering Halomonas TD01 for the low-cost production of polyhydroxyalkanoates. Metab Eng, 2014, 26:34–47.
[23] Yue HT, Chen XB, Ling C, et al. A seawater-based open and continuous process for polyhydroxyalkanoates production by recombinant Halomonas campaniensis LS21 grown in mixed substrates. Biotechnol Biof, 2014, 7: 108.
[24] Yin J, Chen JC, Wu Q, et al. Halophiles, coming stars for industrial biotechnology. Biotechnol Adv,2014, doi: 10.1016/j.biotechadv.2014.10.008.
[25] Lin ZL, Zhang Y, Wang JQ. Engineering of transcriptional regulators enhances microbial stress tolerance. Biotechnol Adv, 2013, 31(6): 986–991.
[26] Li J, Chen L, Du L, et al. Cage the firefly luciferin!-a strategy for developing bioluminescent probes.Chem Soc Rev, 2013, 42(2): 662–676.
[27] Liu G, Chater KF, Chandra G, et al. Molecular regulation of antibiotic biosynthesis in Streptomyces. Microbiol Mol Biol Rev, 2013,77(1): 112–143.
[28] Wang Q, Song FH, Xiao X, et al. Abyssomicins from the South China Sea deep-sea sediment Verrucosispora sp.: natural thioether michael addition adducts as antitubercular prodrugs. Angew Chem Int Ed , 2013, 52(4): 1231–1234.
[29] Cao B, Chen C, DeMott MS, et al. Genomic mapping of phosphorothioates reveals partial modification of short consensus sequences. Nat Commun, 2014, 5: 3951.
[30] Wang WS, Ji JJ, Li X, et al. Angucyclines as signals modulate the behaviors of Streptomyces coelicolor. Proc Natl Acad Sci USA, 2014,111(15): 5688–5693.
[31] Du J, Jia XQ, Yuan YJ. Research progress of synthetic microbial consortia. CIESC J, 2014,65(2): 390–395 (in Chinese).杜瑾, 贾晓强, 元英进. 人工合成微生物混菌体系的研究进展. 化工学报, 2014, 65(2): 390–395.
[32] Song H, Ding MZ, Jia XQ, et al. Synthetic microbial consortia: from systematic analysis to construction and applications. Chem Soc Rev,2014, 43(20): 6954–6981.
[33] Yuan YJ, Jia XQ, Cheng JS, et al. Design and construction of microbial consortia. A Sponsored Supplement to Science (Accepted).
[34] Yong YC, Yu YY, Zhang XH, et al. Highly active bidirectional electron transfer by a self-assembled electroactive reduced-graphene-oxide-hybridized biofilm. Angew Chem Int Ed, 2014, 53(17):4480–4483.
[35] Zhang CS, Su HJ, Baeyens J, et al. Reviewing the anaerobic digestion of food waste for biogas production. Renew Sustain Energy Rev, 2014, 38:383–392.
[36] Guo GW, Sun XJ, Chen C, et al. Whole-genome and whole-exome sequencing of bladder cancer identifies frequent alterations in genes involved in sister chromatid cohesion and segregation. Nat Genet, 2013, 45(12): 1459–1463.
[37] Liu YC, Huang WR, Zhou DX, et al. Synthesizing oncogenic signal-processing systems that function as both "signal counters" and "signal blockers" in cancer cells. Mol Biosyst, 2013, 9(7): 1909–1918.
[38] Liu YC, Han YH, Zhang H, et al. Synthetic miRNA-mowers targeting miR-183-96-182 cluster or miR-210 inhibit growth and migration and induce apoptosis in bladder cancer cells. PLoS ONE, 2012, 7(12): e52280.
[39] Li Y, Jiang Y, Chen H, et al. Modular construction of mammalian gene circuits using TALE transcriptional repressors. Nat Chem Biol, 2015,11(3): 207–213.
[40] Liu YC, Zeng YY, Liu L, et al. Synthesizing AND gate genetic circuits based on CRISPR-Cas9 for identification of bladder cancer cells. Nat Commun,2014, 5: 5393.
[41] Yu B, Yang M, Shi L, et al. Explicit hypoxia targeting with tumor suppression by creating an"obligate" anaerobic Salmonella typhimurium strain. Sci Rep, 2012, 2: 436.

