人工核酸内切酶介导的新一代基因组编辑技术进展
肖安,张博
遗传学的核心任务之一是阐释基因及其产物的功能,其发展经历了经典遗传学、现代遗传学和功能基因组学等三大重要阶段。遗传学最重要的研究策略之一为获取突变体以及对突变体进行基因型、表型分析,除此之外,转基因、RNAi以及其他特异性激活或抑制基因表达的方法也是遗传学研究中的常用技术。获取突变体的途径主要包括筛选天然突变体、人工随机诱变 (正向遗传学手段),以及针对基因组上特异位点进行靶向修饰 (反向遗传学手段) 等[1-2]。其中,能够实现对基因组进行定点诱变的反向遗传学方法是解读基因功能最直接、也是最具有挑战性的手段之一。这一研究思路一方面基于基因组、转录组高通量测序所提供的信息,另一方面则严重依赖于基因组编辑技术的发展。
在相当长的时期内,绝大多数生物体或体外培养细胞中缺乏有效的基因组靶向编辑技术手段,仅有的基于胚胎干细胞和同源重组等少数可行方案存在效率非常低、具有严格的物种局限性等缺点,这也在很大程度上制约了功能基因组研究的进程。随着DNA测序技术的飞速发展,完成全基因组序列测定的物种数量呈现爆发式增长,而基因组定点突变技术的瓶颈所带来的功能基因组研究的滞后效应也越发明显。科研工作者迫切需要一种简单、有效的基因打靶技术来解读记载在每一个基因组中的生命的奥秘。近几年,人们巧妙地利用细菌的一些特殊的基因表达调控机制,以及多年来对转录因子作用机制的研究成果,逐步创建出能够根据人们的意愿特异切割DNA靶序列的人工核酸内切酶 (Engineered endonuclease,简称EEN),并籍此在理论上实现了对任意物种/基因组的任意位点进行靶向修饰的梦想。该项技术的核心思路是针对目标位点 (基因组上特定的DNA序列) 设计并通过基因工程的方法构建特定的核酸内切酶,使其能够特异识别、结合并切割该靶序列,从而在基因组的特定位点造成DNA双链断裂 (Double strand break,简称DSB),最终利用细胞自身的DNA损伤修复的容错性造成靶位点序列产生突变[3]。目前EEN主要包括锌指核酸酶 (Zinc finger nuclease,ZFN)、转录激活因子样效应物核酸酶 (Transcription activator-like effector nuclease,TALEN)、规律性成簇间隔的短回文重复序列 (Clustered regularly interspaced short palindromic repeats,CRISPR)及相关蛋白 (CRISPR-associated,Cas) 系统(CRISPR/Cas系统) 等3种类型。这种基于人工核酸酶的新型基因组编辑技术原理简单、操作简便,在应用上对物种或细胞没有选择性,因此迅速成为拥有极其广泛发展前景和应用价值的热门研究领域之一。由于该项技术在思路上不同于传统的以胚胎干细胞为基础的基因打靶策略,并且在实用性和适用性上均产生了质的飞跃,因此笔者认为不妨借用对新型DNA测序技术的命名,将基于EEN的基因组靶向突变技术称为“新一代”基因组编辑技术。下面简单总结、比较EEN以及EEN介导的基因组靶向修饰的类型,并简单展望该项技术的发展前景。
1 人工核酸内切酶的类型与比较
1.1 ZFN
ZFN是3种人工核酸内切酶中最早被开发并投入应用的。人们很早就了解到,生物体中存在多种锌指蛋白 (Zinc finger protein,ZFP),它们的共同特点是包含数量不等 (通常为 3–7个) 的锌指基序 (Zinc finger motif),这些锌指能够介导蛋白质与核酸、小分子或其他蛋白质的特异相互作用。相当一部分ZFP能够特异识别并结合基因组 DNA上特定长度序列的靶位点,从而作为转录因子对基因表达进行调控。ZFN技术即利用了这一特性。通过人工改造锌指序列 (常用的是 C2H2类型的锌指) 中对识别并结合DNA靶位点产生重要作用的几个关键位置的氨基酸残基,通过基因工程的方法组装出识别特定靶位点的ZFP结构域,并和一个能够非特异切割 DNA的核酸酶结构域 (造成 DNA单链或双链断裂) 融合,便可得到有可能特异切割靶位点的 ZFN。通常选择的切割结构域来自细菌的FokⅠ核酸内切酶,它需要在靶序列的下游以二聚化的形式行使切割DNA的功能。因此,在具体应用中,通常需要构建一对ZFN单体,联合使用。通常每个ZFN含有3个锌指单元,可识别9 bp的靶序列 (每个锌指单元大致识别3个碱基)。两个靶序列分别位于DNA上相反的两条链上,并相距特定的间隔 (图 1A)。1996年,首个人工合成的ZFN被成功报道可在体外实现对靶序列的切割[4]。2002年,ZFN首次成功应用于个体水平,在果蝇中实现了基因的定点突变[5]。随后数年,这一技术的应用范围被扩充到多种生物体和培养细胞中,从而为很多模式生物开创了反向遗传学研究的新天地[6]。
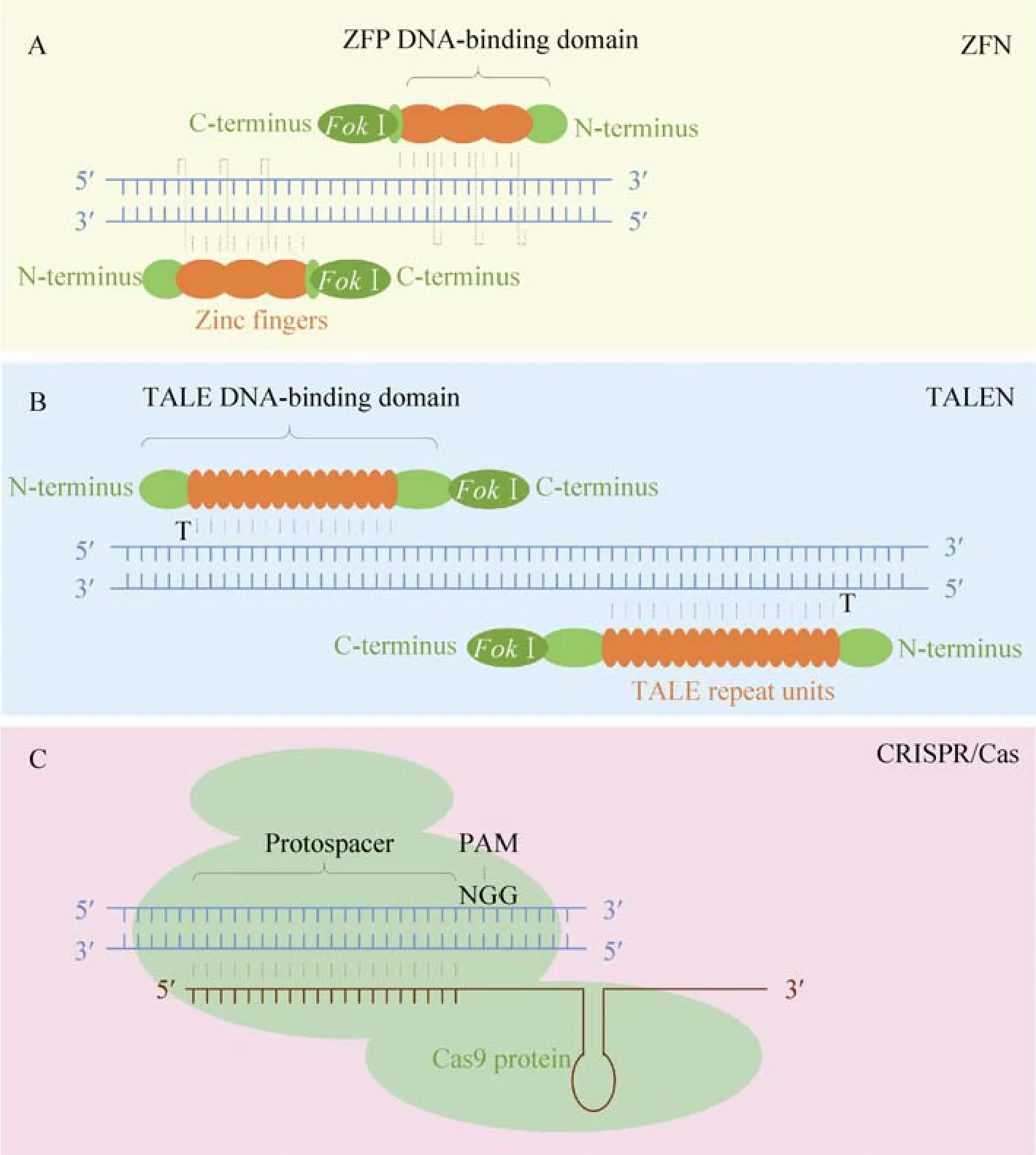
图1 三种人工核酸内切酶的基本结构与组成Fig. 1 The structure and components of the three types of EENs.
遗憾的是,ZFN技术在实际应用中存在很多问题。首先,对于ZFP与DNA的识别与结合规律人们了解得并不透彻。人工构建的ZFN和ZFP通常由多个锌指组装而成,但是每个锌指对其靶序列的识别与结合的能力和特异性受上下文 (周边的锌指单元) 的影响非常大,并且相邻的锌指之间存在着交错影响。经过多年研究,人们对于ZFN与其DNA靶序列的相互作用仅了解到非常有限的一些关联性,例如富含G的靶序列更容易获得有效的ZFN等,因而无法直接根据所需要识别的靶序列构建相应的ZFN[7]。为了获取一对能够有效工作的ZFN,往往需要测试或筛选数量非常可观的锌指序列组合库,不但需要消耗相当大的人力物力,而且最终的成功率也不高。同时,ZFN的诱变效率整体而言比较低 (在生物体中很多时候不超过10%)[8]。此外,ZFN还存在较高的脱靶效应(Off-targeting effect),即除了切割指定的靶位点,往往还会切割基因组上其他具有相似序列的位点[9-10]。人们尝试过多种方法改进 ZFN的特异性,例如,通过突变FokⅠ上特定的氨基酸残基得到多种FokⅠ变体,其中有些FokⅠ变体只有形成异源二聚体时才能组成有活性的内切酶复合体,这样就可以部分减少脱靶效应。但是,总体而言ZFN的特异性仍然不高。上述这些局限性无疑大大增加了ZFN使用的难度。不少研究组开发了各种ZFP/ZFN的构建与筛选方案,以期能够解决这些问题,但是这一技术的应用门槛始终相当高,一般只有少数商业公司才有一定的把握筛选出具有一定靶点识别与切割效率的锌指组合及其 ZFN。正在大家一筹莫展之际,TALEN和CRISPR/Cas系统相继“从天而降”,这才真正掀起了基因组编辑技术的一场革命。
1.2 TALEN
TALEN的基本原理和ZFN类似,也是由特异识别靶序列的DNA结合结构域和FokⅠ切割结构域融合而成,只是由特异性较强的 TALE结构域替代了ZFP。TALEN的DNA结合结构域来自于黄单胞菌属 (Xanthomonas) 植物病原菌表达的一种类转录激活因子 (Transcription activator-like effector, TALE)。TALE包含多个(通常为 12.5–35.5个) 串联的重复单元 (称为TALE repeat),各个单元的长度 (通常为33–35个氨基酸残基) 和序列骨架非常相似,仅第 12位和第13位的两个氨基酸残基存在着高度可变性,称为重复可变双残基 (Repeat variable di-residue,简称 RVD)。2009年,通过统计分析和实验测试,两个研究组同时揭示了 TALE重复单元的生物学功能——每个单元对应识别靶基因上的一个DNA碱基,是一种全新的DNA结合结构域[11-13]。此外,在识别并结合靶位点方面,TALE重复单元相互之间基本没有影响,而且所有能够识别 4种碱基的单元都已发现。仅仅1年后,人们就证明由TALE和FokⅠ融合得到的 TALEN能够对靶位点进行特异切割与高效突变[14](图1B)。TALE 重复单元与靶位点这种精确的一一对应关系极大方便了研究者的使用,从而免除了ZFN应用中大规模的筛选工作。TALEN对靶位点序列的要求也非常宽松,仅需要在靶序列5?端上游的第一个碱基为T即可。同时,TALEN的切割与诱变效率虽然也存在难以预测并且有时变动较大等问题,但平均而言相对于 ZFN要高很多。针对不同的位点,TALEN的活性/突变效率差异可以很大,在生物体中从无活性到突变效率接近100%都有可能[15]。例如,在斑马鱼中,有将近3/4的位点TALEN的突变效率可达1%以上,有1/4的位点的效率可高于 50% (本实验室未发表数据)。此外,目前的研究结果显示,TALEN的特异性非常好,脱靶效应要比ZFN低得多 (有可能与其识别区域长度很长有关)[16]。这些特性使得TALEN技术迅速发展起来,在很多领域取代了ZFN的应用。
TALEN技术应用的难点是TALEN表达载体的构建。TALE结构域比较大,可达 600–800个氨基酸残基 (1 800–2 400 bp编码序列),并且序列高度重复,难以使用常规的 PCR、酶切连接等分子克隆技术实现。多个研究组运用不同的思路和工具,开发了各种构建TALE和TALEN蛋白表达载体的方法,从而使得这一技术能够为更多的人使用[8]。
1.3 CRISPR/Cas系统
与ZFN和TALEN不同,CRISPR/Cas并非基于与DNA结合的转录因子改造而成,而是借鉴了多种原核生物和古核生物中天然存在的一套获得性免疫系统[17]。细菌能够捕获入侵的病毒、质粒等外来核酸序列片段,并将其嵌入到其基因组中一段高度串联重复并带有特定回文结构的区域 (CRISPR)。这一区域能够被转录、切割形成具有特定二级结构的小片段 RNA (称为CRISPR RNA,crRNA)。若细菌被带有同一序列的外来核酸感染,这些crRNA可在CRISPR相关蛋白 (Cas) 和其他 RNA (tracrRNA) 的辅助下,以碱基配对的方式特异性识别外源入侵序列,并通过各种不同的作用机制组装形成核酸酶切割复合物,降解入侵的核酸[2]。利用这一特性,2012年,通过改造研究得最为清楚、组成最为简单的Ⅱ型CRISPR/Cas系统 (仅由一个Cas9蛋白、一段带有20 nt识别序列的crRNA和一个通用的tracrRNA组成),人们在体外成功实现了对携带有同样的 20 bp序列称为Protospacer的DNA双链靶位点的特异切割[18]。同时,研究人员还进一步简化了这个系统,将上述两个 RNA分子通过一段连接序列合二为一,并且去除了不必要的区域,设计出一个长度为102 nt的RNA分子,称为向导RNA (Guide RNA, gRNA;也有文献称为单一向导 RNA,single guide RNA,sgRNA),并证明Cas9-gRNA的组合同样也能够实现对DNA的靶向切割 (图1C)。因此,CRISPR/Cas系统又称 Cas9/gRNA系统。半年后,两个研究小组使用人工构建的CRISPR/Cas系统成功地在包括iPS细胞在内的多种体外培养的人类细胞中实现了对基因组特定位点的切割与诱变[19-20],从而开创了这一新技术在活体中的应用。
与ZFN和TALEN相比较,CRISPR/Cas系统不是通过蛋白与核酸的相互作用,而是通过核酸与核酸之间的碱基互补配对作用来识别并结合靶序列,这使其原理和操作更为简单。CRISPR/Cas系统要求的靶位点序列规则也仅需要在识别区域的 3?端下游的 3个碱基为 NGG(称为 Protospacer adjacent motif,简称 PAM)[18]。在实际应用中,对于不同的靶位点,只需改变gRNA,Cas9蛋白则都是相同的,无需重新设计与构建;负责识别不同靶序列的 gRNA的长度非常短,可以通过多种方式在体外快速合成或快速构建体内表达系统。由此可见,CRISPR/Cas系统在应用的简便性上具有巨大的优势。此外,还可以构建多个gRNA,与Cas9共同导入生物体或细胞内,这样就有可能实现对多个靶位点的同时切割与突变。目前来看,CRISPR/Cas系统对靶位点的切割及诱变效率与 TALEN大致相当,不过,该系统对靶位点的选择性仍有待深入研究。
作为一种最新的基因组靶向诱变技术,CRISPR/Cas系统目前存在的一个最值得关注的问题应该是其潜在的脱靶效应。目前至少有两方面的因素会影响到该系统的特异性。一方面涉及到靶序列的长度。CRISPR/Cas系统以单一复合体的形式起作用,其识别的靶位点的长度固定为20 bp,短于一对TALEN通常所联合识别的 30–40 bp。更重要的影响因素涉及到其识别靶序列的机制——碱基互补配对往往会表现出一定的容错性。有报道表明,在人类细胞系中某些 CRISPR/Cas存在着相当可观的脱靶切割效率,这些脱靶效应很可能是由于碱基错配所导致的[21-24]。有研究组设法突变Cas9蛋白,将其改造为只能切割一条DNA单链的核酸缺刻酶 (Nickase) Cas9n,从而可以像ZFN和TALEN一样使用一对识别相反链的Cas9n/gRNA组合实现双链切割,以增加靶位点的序列长度,降低脱靶效应[19,23,25]。为了避免或尽量减少碱基错配造成的脱靶效应,部分提供靶位点设计的网络在线工具也增加了预测潜在脱靶位点、以供用户选择特异性较高的gRNA的辅助功能[25]。总体而言,关于CRISPR/Cas系统潜在脱靶效应的问题尚需进一步的研究和探索。
ZFN、TALEN、CRISPR/Cas系统的简要发展历程和比较如图2和表1所示。

图2 基于人工核酸内切酶的新一代基因组编辑技术的简要发展历程Fig. 2 A brief summary of the developmental progress of the EEN-mediated new-generation genome editing technology.

表1 三种人工核酸内切酶的特征比较Table 1 Comparison of the three types of EEN (engineered endonuclease)
2 借助人工核酸内切酶技术的基因组定点修饰方法
各种人工核酸内切酶所主要解决的问题都是针对染色体/基因组上所设计的靶位点实现特异切割、造成DSB。而借助于DSB对基因组进行靶向编辑主要包括以下3种策略。
2.1 单DSB导致的indel突变
非同源末端连接 (Non-homologous endjoining,NHEJ) 是细胞自发修复 DSB造成的DNA损伤的一种主要方式。细胞内的DNA发生DSB后,各种修复因子可将断裂产生的两个DNA末端直接连接起来。这一连接过程常常不够精确,往往可能发生小片段的插入和/或缺失(Small insertion/deletion,indel)。如果indel破坏了基因的读码框或提前引入翻译终止位点,那么就有可能破坏基因的功能和/或表达,造成该基因产生突变。在实际应用过程中,这一策略只需将人工核酸内切酶引入到生物体或培养细胞后,不需要进行其他操作,等待适当的时间后对indel诱变效率进行检测即可[6]。
大部分已报道的人工核酸内切酶的应用均使用的是NHEJ-indel诱变策略。这一方法所得到的突变等位基因的类型不可控制,因此绝大多数情况下只能对基因进行功能破坏性突变操作。在应用这一策略的时候,需要注意所选择的靶位点应该位于基因编码区比较靠前的外显子中 (但是要在翻译起始密码子ATG之后),并且最好在编码某个重要功能结构域的序列之前,这样突变后才能使整个蛋白失去正常作用。同时需要注意可变剪接、多重转录本,以及潜在的下游可选翻译起始位点等问题,尽量选在确定能够完全破坏整个基因功能的区域。
2.2 双DSB介导的基因组大片段删除
NHEJ-indel策略只能应用于单个蛋白编码基因,对于希望破坏非编码RNA基因、基因组顺式作用元件 (调控序列)、多个基因、多个转录本、多个剪接变体,或基因簇等情况,或是针对较大的或情况比较复杂的蛋白编码基因,包括希望去除某些特定的功能结构域等,indel往往难以达到预期目的。这时可以同时使用两组/两对人工核酸内切酶,使其分别识别基因组上两个不同的位点,在间隔一定距离的DNA双链上造成两个DSB,这样就有可能在 NHEJ介导的修复连接时,将两个位点两侧的序列直接连接起来,而丢掉两个DSB之间的区段。这样获得的突变类型属于大片段的缺失突变 (Large fragment deletion)。删除片段的长度可长可短,有的甚至可长达百万碱基对 (Mbp)。
这一策略最初在人类细胞中报道[26],随后在其他物种中也得到了应用,可顺利实现对miRNA簇的删除等[27]。不过,大片段删除的效率显著低于单点indel的突变效率,而且还会随着两个位点之间的距离增大而进一步下降。
2.3 同源重组介导的基因组精确修饰
同源重组 (Homologous recombination,HR)或同源介导的修复 (Homology directed repair,HDR) 是传统上在小鼠中应用的基因组定点修饰方法。通过引入两侧各带有一段和靶位点序列一致的同源臂 (Homologous arm) 的外源供体 (Donor) DNA,细胞染色体能够以很低的效率 (约10–6量级) 与供体DNA发生同源同组,从而将外源序列精确地置换到基因组中。由于天然发生的同源重组的效率太低,因此往往需要通过在供体DNA上添加正、负筛选标记并优化条件,并且筛选足够多的细胞,才有可能在众多细胞株中找到成功发生同源重组的细胞。因此,这一方法很难在缺乏有效筛选策略的生物体中实现。
曾有报道,在靶位点附近造成双链断裂或单链缺刻可极大地提高重组效率。因此,人工核酸内切酶技术出现后,人们便开始尝试用它提高HR的效率,以便在整体动物水平实现对基因组的精确修饰。结果表明,在同源重组的目标序列附近设计人工核酸内切酶造成DSB后,能将细胞中的重组效率提高 3–4个数量级,极大地方便了筛选工作[28]。同时,在很多传统上无法实现同源重组的物种中 (如斑马鱼),使用人工核酸内切酶定点切割也顺利实现了外源序列的精确整合[29]。
同源介导修复的最大特点是可以精确地操控和编辑基因组,借此可以实现精确的点突变(Point mutation)、基因修复 (Gene correction)、基因敲入 (Gene addition)、基因替换 (Gene replacement) 等,从而在疾病治疗、基因标记、特殊突变体构建等领域发挥重要作用。需要注意的是,人工核酸内切酶介导的同源重组效率仍然大大低于单点indel效率,同时两者之间还存在竞争关系,需要在重组供体上设计一些可供筛选的序列或其他分子标记。
3 人工核酸内切酶应用前景与展望
目前,对于这3种EEN,ZFN基本上已经完全被TALEN代替,TALEN则跟CRISPR/Cas系统并存,两者各有千秋。TALEN的优势是特异性高、脱靶效应较低;CRISPR/Cas系统的优势则是使用简便、成本低。不过,由于两者真正应用的时间都很短,TALEN技术建立迄今不足5年,CRISPR/Cas系统体系则仅两年多,因此都存在一些问题有待深入研究。例如,两种基因打靶体系都依然需要优化,应用范围与方式需要进一步拓展,作用机制仍有待深入研究;TALEN的成功率与靶点效率预测、CRISPR/Cas系统的成功率与特异性/脱靶效应、毒性与安全性等都有待进一步评估与优化。什么样的位点能够获得较高的突变效率?CRISPR/Cas系统的特异性/脱靶效应究竟如何?这些问题仍有待谨慎探索。
展望未来,这一技术至少会在以下几个领域中发挥越来越广泛而重要的作用:
1) 对模式生物基因组的操控。借助于EEN,对任意模式生物的基因组进行定点编辑已经由梦想逐渐变成了现实,反向遗传学研究方法也得到了飞跃式的发展。这一方法使得不少传统上缺乏反向遗传学工具的模式物种 (例如斑马鱼、大鼠) 的基因功能研究工作的难度大大降低,也节省了已有其他基因定点突变工具的模式物种的突变体构建时间 (例如小鼠、果蝇),同时还为很多以前研究不够充分的物种提供了几乎是唯一的可用工具 (包括很多灵长类和众多非传统模式物种在内)。
2) 在体外培养细胞中的应用。利用人工核酸内切酶,可同样有针对性地操作、编辑体外培养细胞的基因组。由于CRISPR/Cas工具极其容易应用的特性,特别是其 gRNA可以高通量构建,有研究人员已经开始着手构建人类细胞全基因组的靶向 gRNA库,相信在不久的将来就可以实现大规模的、全基因组范围的正向或负向选择性功能筛选。
实际上,这场革命不仅使模式生物和体外培养细胞的研究受益,而且必将产生更深远的影响。我们有理由乐观地憧憬,大众化的 EEN技术很有可能会像当年的分子克隆、PCR技术一样进入最普通的实验室,成为任何一个涉及到核酸研究的实验室必备的常规工具。
3) 在疾病基因治疗方面的应用。通过特异性修饰基因组的特定序列,人工核酸内切酶技术为基因治疗带来了新的希望。将这项技术跟iPS等干细胞技术相结合,其应用前景更是不可限量。这方面的工作目前主要集中在体外培养的人类细胞中,相关的尝试包括使用ZFN破坏T细胞内 HIV入侵至关重要的受体蛋白CCR5[30]、使用TALEN介导的HDR原位修复镰刀形红细胞贫血症、地中海贫血症、隐性营养不良性大疱性表皮松解 (Recessive dystrophic epidermolysis bullosa,RDEB) 等相关致病基因等[31-33]。这方面由于ZFN发展历史较长,对其细胞毒性、脱靶活性等方面的研究也较为充分,因此目前在这个领域暂时走在前面。笔者相信,在不久的将来,TALEN和CRISPR/Cas系统会奋起直追,在这一领域大显身手。同时,由于这一领域对于脱靶效应的要求会格外严格,因此,CRISPR/Cas系统更需要在这方面充分研究和分析论证。
4) 在经济物种中的应用。将人工核酸内切酶应用于经济动物 (家禽家畜) 或经济植物(农作物) 无疑也是一个非常令人期待的方向。通过特异性编辑和调控与家畜、作物相关性状有关联的基因,可实现对抗病、抗逆、产量、品质、生长条件等方面的性状改良,有可能大大缩短育种周期,并且实现精准育种。目前,在猪、牛等大型家畜[34-36]和家蚕、烟草、玉米等多种培养动植物中均已有相关的尝试和报道[37-38]。跟疾病治疗方面的应用类似,这方面的应用也要特别注意安全性、脱靶活性等问题。此外,由于涉及到基因操作,EEN技术为传统的转基因作物的安全性评估与管理带来了新的挑战,同时,也为此带来了新的思路和出路。
EEN的应用绝不仅限于上述4个领域。例如,EEN技术还可以用于改造宠物的形态、行为等特征,使其更加多样化、更贴近人类的需求等。总之,EEN技术既为生命科学基础理论研究,又为跟人类健康与生活密切相关的经济物种改良和人类疾病治疗带来了全新的方法和希望。正是由于预见到其强劲的发展势头,TALEN和CRISPR/Cas系统分别被美国《科学》杂志评为2012和 2013年度的十大科学进展之一,TALEN被誉为“基因组巡航导弹” (Genomic cruise missiles),CRISPR/Cas系统则被誉为“大众化的基因组微型手术刀” (Genetic microsurgery for the masses)。一个全新的技术出现仅仅两年之后,马上又被另一个更新的技术所超越,回首各种生命科学研究技术的发展,这样的例子可以说在历史上绝无仅有。最后,一个令人感兴趣的悬念是:CRISPR/Cas系统是反向遗传学技术的终极版本吗?还会有更简便的工具和/或方法出现吗?让我们拭目以待。
[1] Huang P, Xu LJ, Liang W, et al. Genomic deletion induced by Tol2 transposon excision in zebrafish.Nucleic Acids Res, 2012, 41(2): e36.
[2] Huang P, Zhu Z, Lin S, et al. Reverse genetic approaches in zebrafish. J Genet Genomics, 2012,39(9): 421–433.
[3] Nemudryi AA, Valetdinova KR, Medvedev SP, et al. TALEN and CRISPR/Cas genome editing systems: tools of discovery. Acta Naturae, 2014,6(3): 19–40.
[4] Kim YG, Cha J. Chandrasegaran S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci USA, 1996,93(3): 1156–1160.
[5] Bibikova M, Golic M, Golic KG, et al. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases. Genetics,2002, 161(3): 1169–1175.
[6] Xiao A, Hu YY, Wang WY, et al. Progress in zinc finger nuclease engineering for targeted genome modification. Hereditas (Beijing), 2011, 33(7):665–683 (in Chinese).
肖安, 胡莹莹, 王唯晔, 等. 人工锌指核酸酶介导的基因组定点修饰技术. 遗传, 2011, 33(7):665–683.
[7] Ramirez CL, Foley JE, Wright DA, et al.Unexpected failure rates for modular assembly of engineered zinc fingers. Nat Methods, 2008, 5(5):374–375.
[8] Xiao A, Wu YD, Yang ZP, et al. EENdb: a database and knowledge base of ZFNs and TALENs for endonuclease engineering. Nucleic Acids Res, 2013, 41(D1): D415–D422.
[9] Gabriel R, Lombardo A, Arens A, et al. An unbiased genome-wide analysis of zinc-finger nuclease specificity. Nat Biotechnol, 2011, 29(9):816–823.
[10] Pattanayak V, Ramirez CL, Joung JK, et al.Revealing off-target cleavage specificities of zinc-finger nucleases by in vitro selection. Nat Methods, 2011, 8(9): 765–770.
[11] Shen Y, Xiao A, Huang P, et al. TALE nuclease engineering and targeted genome modification.Hereditas (Beijing), 2013, 35(4): 395–409 (in Chinese).
沈延, 肖安, 黄鹏, 等. 类转录激活因子效应物核酸酶 (TALEN) 介导的基因组定点修饰技术.遗传, 2013, 35(4): 395–409.
[12] Boch J, Scholze H, Schornack S, et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science, 2009, 326(5959): 1509–1512.
[13] Moscou MJ, Bogdanove AJ. A simple cipher governs DNA recognition by TAL effectors.Science, 2009, 326(5959): 1501.
[14] Miller JC, Tan SY, Qiao GJ, et al. A TALE nuclease architecture for efficient genome editing.Nat Biotechnol, 2011, 29(2): 143–148.
[15] Moore FE, Reyon D, Sander JD, et al. Improved somatic mutagenesis in zebrafish using transcription activator-like effector nucleases(TALENs). PLoS ONE, 2012, 7(5): e37877.
[16] Mussolino C, Morbitzer R, Lütge F, et al. A novel TALE nuclease scaffold enables high genome editing activity in combination with low toxicity.Nucleic Acids Res, 2011, 39(21): 9283–9293.
[17] Li J, Zhang Y, Chen KL, et al. CRISPR/Cas: a novel way of RNA-guided genome editing.Hereditas (Beijing), 2013, 35(11): 1265–1273 (in Chinese).
李君, 张毅, 陈坤玲, 等. CRISPR/Cas系统: RNA靶向的基因组定向编辑新技术. 遗传, 2013,35(11): 1265–1273.
[18] Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity.Science, 2012, 337(6096): 816–821.
[19] Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science,2013, 339(6121): 819–823.
[20] Mali P, Yang LH, Esvelt KM, et al. RNA-guided human genome engineering via Cas9. Science,2013, 339(6121): 823–826.
[21] Fu YF, Foden JA, Khayter C, et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol, 2013,31(9): 822–826.
[22] Hsu PD, Scott DA, Weinstein JA, et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol, 2013, 31(9): 827–832.
[23] Mali P, Aach J, Stranges PB, et al. CAS9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering. Nat Biotechnol, 2013, 31(9):833–838.
[24] Pattanayak V, Lin S, Guilinger JP, et al.High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat Bitechnol, 2013, 31(9): 839–843.
[25] Ran FA, Hsu PD, Lin CY, et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell, 2013, 154(6): 1380–1389.
[26] Lee HJ, Kim E, Kim JS. Targeted chromosomal deletions in human cells using zinc finger nucleases. Genome Res, 2010, 20(1): 81–89.
[27] Xiao A, Wang ZX, Hu YY, et al. Chromosomal deletions and inversions mediated by TALENs and CRISPR/Cas in zebrafish. Nucleic Acids Res,2013, 41(14): e141.
[28] Urnov FD, Miller JC, Lee YL, et al. Highly efficient endogenous human gene correction using designed zinc-finger nucleases. Nature, 2005,435(7042): 646–651.
[29] Zu Y, Tong XJ, Wang ZX, et al. TALEN-mediated precise genome modification by homologous recombination in zebrafish. Nat Methods, 2013,10(4): 329–331.
[30] Perez EE, Wang JB, Miller JC, et al. Establishment of HIV-1 resistance in CD4+T cells by genome editing using zinc-finger nucleases. Nat Biotechnol,2008, 26(7): 808–816.
[31] Ma N, Liao BJ, Zhang H, et al. Transcription activator-like effector nuclease (TALEN)-mediated gene correction in integration-free β-thalassemia induced pluripotent stem cells. J Biol Chem, 2013,288(48): 34671–34679.
[32] Osborn MJ, Starker CG, McElroy AN, et al.TALEN-based gene correction for epidermolysis bullosa. Mol Ther, 2013, 21(6): 1151–1159.
[33] Sun N, Zhao HM. Seamless correction of the sickle cell disease mutation of the HBB gene in human induced pluripotent stem cells using TALENs.Biotechnol Bioeng, 2014, 111(5): 1048–1053.
[34] Lillico SG, Proudfoot C, Carlson DF, et al. Live pigs produced from genome edited zygotes. Sci Rep, 2013, 3: 2847.
[35] Proudfoot C, Carlson DF, Huddart R, et al. Genome edited sheep and cattle. Transgenic Res, 2015,24(1): 147–153.
[36] Tan WF, Carlson DF, Lancto CA, et al. Efficient nonmeiotic allele introgression in livestock using custom endonucleases. Proc Natl Acad Sci USA,2013, 110(41): 16526–16531.
[37] Belhaj K, Chaparro-Garcia A, Kamoun S, et al.Editing plant genomes with CRISPR/Cas9. Curr Opin Biotechnol, 2015, 32: 76–84.
[38] Daimon T, Kiuchi T, Takasu Y. Recent progress in genome engineering techniques in the silkworm,Bombyx mori. Dev Growth Differ, 2014, 56(1):14–25.

