香蕉Ran家族基因的全基因组分析
张雅玲,方智振,2,赖钟雄*
(1.福建农林大学园艺植物生物工程研究所,福建福州350002;2福建省农业科学院果树研究所,福建福州3500013)
香蕉Ran家族基因的全基因组分析
张雅玲1,方智振1,2,赖钟雄1*
(1.福建农林大学园艺植物生物工程研究所,福建福州350002;2福建省农业科学院果树研究所,福建福州3500013)
摘要:Ran是一类在多种基本细胞活动中扮演者重要角色的小GTP酶。从香蕉基因组中鉴定出9个MuRan家族基因成员,并对其染色体分布、基因结构、蛋白理化性质、结构域和系统进化等进行分析。结果表明:MuRan家族基因不均匀的分布在第1、4、5、6号等4条染色体上。MuRan蛋白与其它植物Ran蛋白的氨基酸序列都有很高的同源性,带有GTP水解结构域、RanGAP结合结构域和酸性尾巴等Ran蛋白的典型结构域,与拟南芥AtRan蛋白亲缘关系较近。为进一步研究香蕉MuRan家族基因的生物学功能提供参考。
关键词:香蕉;基因组;Ran;生物信息学
Ran(ras-like nuclear GTP-binding protein)是一种广泛存在于真核生物中且极为保守的小GTP酶,在多种基本细胞活动中扮演者重要的角色[1-3]。大量研究结果均表明植物Ran与动物Ran具有相似的功能,在核质转运和细胞分裂等过程中发挥着重要作用[4-10]。此外,Ran与其结合蛋白RanBP可能与植物激素敏感性、光信号转导、胁迫响应和抗病性有关。反义表达AtRanBP1c可提高转基因植株根系对生长素的敏感性[11]。茉莉酸可诱导水稻OsRAN2的表达[12]。Li等[13]研究发现低磷处理可影响玉米根系中Ran B1蛋白的表达。拟南芥根系Ran-1蛋白参与盐胁迫响应[14]。超表达OsRAN2可改变水稻
对盐胁迫的敏感性[4]。热胁迫和低温胁迫可影响植物Ran蛋白的表达[15-19]。Yoshimura等[20]发现2个野生西瓜根系Ran蛋白与干旱胁迫响应有关。Zhai等[21]发现长期积水可导致作用于Ran的miRNA的表达发生变化。不同光源可通过光敏色素介导的信号转导途径引起Ran基因的差异表达[5]。RanGAP2沉默可破坏Rx介导的抗性[22-23],Tameling等[24]进一步研究发现RanGAP2参与调控NB-LRR免疫受体Rx的核质分布,Rivas[25]提出RanGAP2通过调控Rx的核质分布参与植物防御的信号转导。这意味着Ran-GTP调控的核孔组分与植物免疫反应的控制有关。由此可见,Ran蛋白在植物的生长发育过程中扮演着极为重要的角色。
香蕉(Musaspp.)是全球广泛种植的热带果树,也是重要的经济作物和粮食作物。病虫、低温、干旱、光照不足和盐渍等胁迫严重影响香蕉的产量和品质。近年来,研究者在香蕉抗性和作用机理及其品种改良方面取得了一定的进展,但对Ran的研究较少,尚未见从全基因组水平对Ran家族基因进行分析和研究。香蕉基因组测序工作的完成为基因家族的分析奠定了基础。本研究基于香蕉基因组数据库和生物信息学手段对香蕉全基因组Ran家族基因进行分析,旨在为进一步研究Ran家族基因在香蕉生长发育过程中的作用与机制提供了有力的科学依据。
1材料与方法
1.1材料
试验基因组序列来自香蕉基因组数据库(http://banana-genome.cirad.fr)、拟南芥信息资源数据库TAIR(http://www.arabidopsis.org/)和Phytozome(http://www.phytozome.net/)。
1.2方法
根据香蕉基因组数据库中Ran家族基因cDNA序列和对应的全基因组序列,采用Gene Structure Display Server(http://gsds.cbi.pku.edu.cn)进行基因结构图的绘制。Ran蛋白的氨基酸数目、分子量和理论等电点采用ExPaSy的在线Protparam软件(http://web.expasy.org/protparam/)进行分析。香蕉与拟南芥Ran蛋白序列的比对分析采用DNAMAN8软件进行。香蕉Ran蛋白与拟南芥、毛果杨、葡萄和水稻Ran蛋白序列的比对用MEGA5软件自带的ClustalW进行,并采用邻接算法(Neighbor-Joining)进行系统进化树构建,Bootstrap值设为1000。
2结果与分析
2.1香蕉基因组Ran家族基因鉴定及在染色体上的分布
以拟南芥AtRan1的氨基酸序列为种子序列,在香蕉全基因组数据库中进行Blast检索,结果显示香蕉基因组中Ran家族基因注释为GTP-binding nuclear protein Ran。故以GTP-binding nuclear protein Ran为检索词进行检索,共检索到9个Ran基因,并依次命名为MuRan1-MuRan9(表1)。采用Protparam软件对9个Ran基因编码的蛋白的长度、分子量和等电点进行分析发现,MuRan7编码的蛋白最长,由476个氨基酸组成;MuRan6编码的蛋白最短,由246个氨基酸组成;根据等电点分析结果可以看出,MuRan1、MuRan7和MuRan9编码的蛋白为酸性蛋白,其余的均为碱性蛋白。根据香蕉基因组数据库的定位结果,MuRan9的定位未知,其余8个MuRan基因分别位于1号、4号、5号和6号染色体上,其中1号染色体分布最多,有3个Ran基因;4号染色体分布最少,仅有1个Ran基因;5号和6号染色体均分布了2个Ran基因(表1)。根据全基因组信息及相关文献表明,拟南芥、毛果样和葡萄基因组中Ran家族均为4个成员,水稻基因组中Ran家族仅有2个成员。香蕉基因组中Ran家族成员明显多于其他植物。这意味着香蕉Ran家族的成员及其功能可能较其他植物复杂,Ran家族基因可能在香蕉的生长发育过程中具有十分重要的作用。
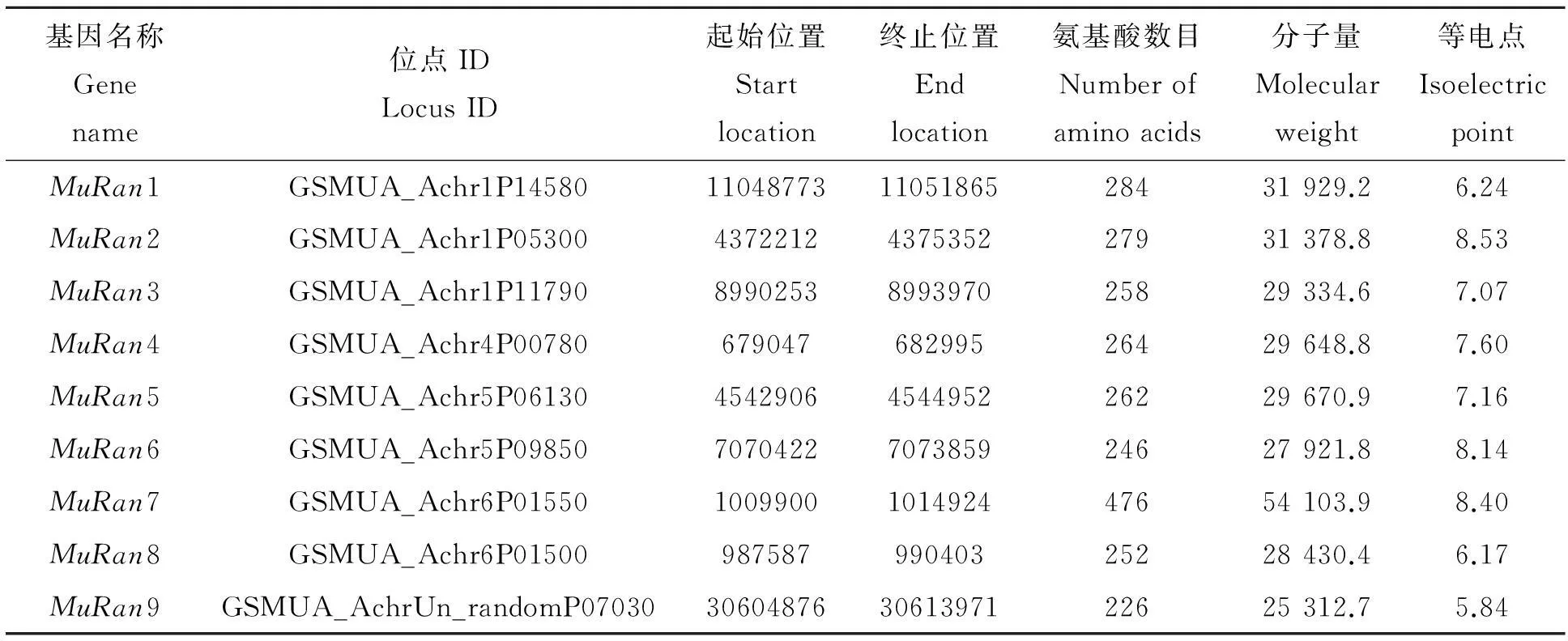
表1 香蕉基因组Ran家族成员
2.2香蕉基因组Ran家族基因的结构
采用Gene Structure Display Server(http://gsds.cbi.pku.edu.cn/)绘制出香蕉、拟南芥、水稻、毛果样和葡萄Ran家族基因的结构图。香蕉Ran家族9个基因MuRan1-MuRan9的外显子数目依次分别为7、9、8、9、9、7、11、7和9(图1)。拟南芥Ran家族4个基因AtRan1、AtRan2、AtRan3和AtRan4的外显子数目均为5、5、8和6;水稻Ran家族2个基因Os06g39875和Os05g49890的外显子数目均为7和8;葡萄Ran家族4个基因GSVIVT01015482001、GSVIVT01017234001、GSVIVT01035804001和GSVIVT01035805001的外显子数目均为8、8、9和9;毛果杨Ran家族4个基因Potri.018G030700.1、Potri.006G250300、Potri.006G250400.1和Potri.018G030900.1的外显子数目均为7、7、8和8。香蕉Ran家族基因的外显子数与拟南芥AtRan3与水稻、葡萄和毛果杨Ran家族基因的外显子数较为接近。根据香蕉基因组数据库的功能注释结果,MuRan1、MuRan2、MuRan3、MuRan4、MuRan5、MuRan6、MuRan7和MuRan8编码的蛋白均注释为Ran3。以上结果表明,香蕉Ran家族基因可能与拟南芥AtRan3的亲缘关系较近。
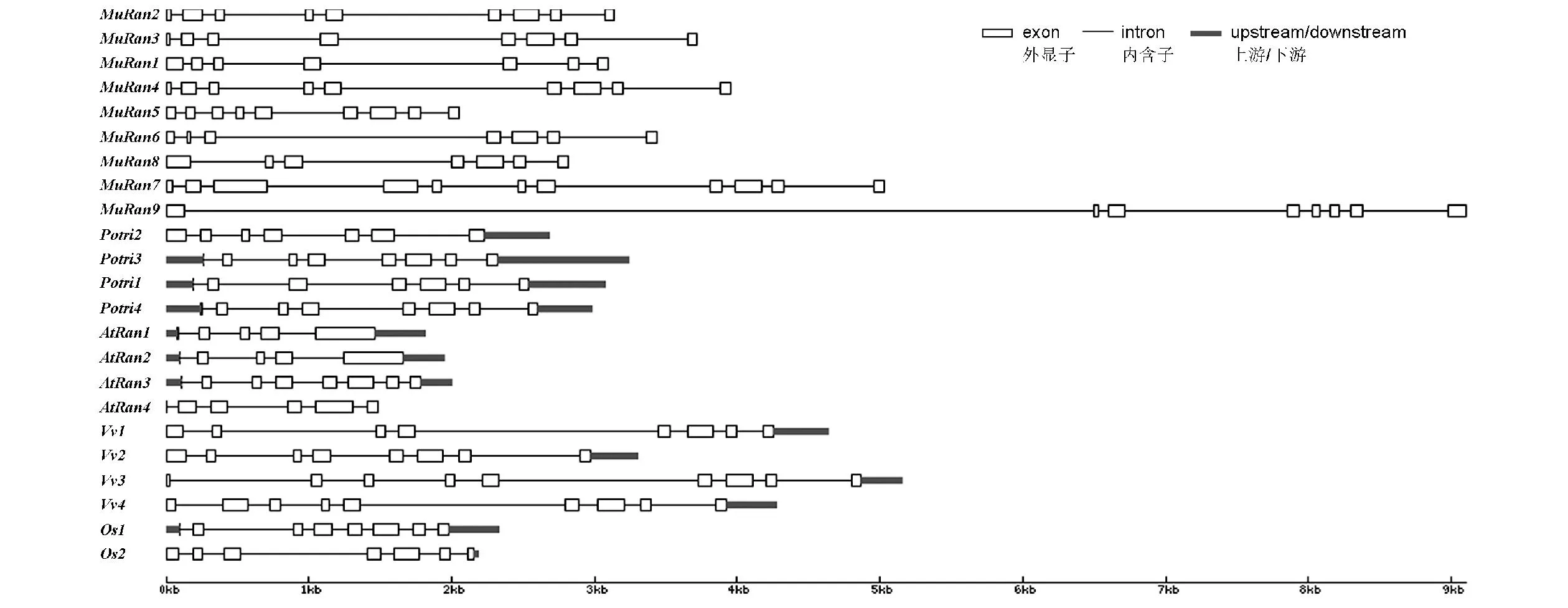
拟南芥、毛果杨、葡萄和水稻的Ran家族基因位点名称如下:AtRan1(AT5G20010),AtRan2(AT5G20020), AtRan3(AT5G55190),AtRan4(AT5G55080),Potri1(Potri.018G030700.1),Potri2(Potri.006G250300),Potri3(Potri.006G250400.1),Potri4(Potri.018G030900.1),Vv1(GSVIVT01015482001),Vv2(GSVIVT01017234001), Vv3(GSVIVT01035804001),Vv4(GSVIVT01035805001),Os1(Os05g49890),Os2(Os06g39875)Locus names of Ran family genes in Arabidopsis thaliana,Populus trichocarpa,Vitis vinifera and Oryza sativa are as follows:AtRan1(AT5G20010),AtRan2(AT5G20020), AtRan3(AT5G55190),AtRan4(AT5G55080),Potri1(Potri.018G030700.1),Potri2(Potri.006G250300),Potri3(Potri.006G250400.1),Potri4(Potri.018G030900.1),Vv1(GSVIVT01015482001),Vv2(GSVIVT01017234001), Vv3(GSVIVT01035804001),Vv4(GSVIVT01035805001),Os1(Os05g49890),Os2(Os06g39875)图1 香蕉、拟南芥、毛果杨、葡萄和水稻的Ran家族基因结构示意图Fig.1 Schematics of the structures of Ran family genes in Musa acuminata,Arabidopsis thaliana,Populus trichocarpa,Vitis vinifera and Oryza sativa
2.3香蕉基因组Ran蛋白的序列比对和系统进化分析

GTP结合与水解结构域(G1-G5)以粗线表示。RanGAP结合结构域和酸性C末端区域分别以星号和三角形表示。Conserved GTP binding and hydrolysis domains(G1-G5) were indicated by bold lines.The effector-binding domain(RanGAP-binding) and the acidic C-terminal region(acidic tail) are indicated with asterisks and triangles,respectively.图2 香蕉与拟南芥Ran蛋白序列比对分析Fig.2 Multiple alignments of Ran sequences from banana and Arabidopsis

图3 香蕉、拟南芥、毛果杨、葡萄和水稻Ran蛋白系统进化树Fig.3 Phylogenic tree of Ran proteins in Musa acuminata,Arabidopsis thaliana,Populus trichocarpa,Vitis vinifera and Oryza sativa
采用DNAMAN软件对香蕉与拟南芥Ran蛋白序列进行比对分析发现,香蕉基因组中预测的Ran蛋白与拟南芥Ran蛋白高度同源(图2)。除MuRan9外,其余香蕉MuRan蛋白均具有完整的GTP结合与水解结构域、RanGAP结合结构域和酸性C末端区域等典型的Ran蛋白功能结构域,且极为保守(图2)。此外,从图2中可以看出香蕉基因组中预测的Ran蛋白N端较拟南芥Ran蛋白多出一段长度不等的多肽。采用NCBI的Blastp对这些蛋白的N端部分序列进行分析发现,除了MuRan7与其他植物的钙调素结合蛋白有较高的同源性外,其余MuRan蛋白N端部分序列均为找到同源序列。
采用Mega 5的临位相连法对香蕉Ran蛋白与拟南芥、毛果杨、葡萄和水稻Ran蛋白的进化关系进行分析,结果表明除MuRan5外,其余MuRan蛋白与拟南芥AtRan3、3个毛果杨Ran蛋白(Potri.006G250400、Potri.018G030700、Potri.018G030900)以及4个葡萄Ran蛋白的亲缘关系较近(图3)。
3结论与讨论
小GTP酶Ran与动物和酵母的细胞分裂调控密切相关[26-27],并且参与微管动态、染色体排列以及核膜组装等过程[1]。研究表明Ran在细胞周期调控中的功能可能相当保守[10,28-30]。大量研究证据表明Ran在植物生长发育和胁迫响应过程中发挥着重要作用。目前,植物Ran的研究较少,相关的研究主要集中在动物上。大量植物基因组测序的完成,为从全基因组水平上研究植物Ran家族基因提供了条件。
本研究从香蕉全基因组中鉴定得到了9个MuRan基因,数量明显多于其他物种。与其他小G蛋白不同的是,Ran家族基因的成员数较少,人类和裂殖酵母中均只有1个Ran基因[31],拟南芥基因组中存在4个Ran基因[32]。香蕉中Ran家族基因的数量可能与其经历了3次全基因组复制有关[33]。同时,这也暗示着Ran基因在香蕉中具有重要的作用。香蕉Ran蛋白的序列与拟南芥、水稻、毛果杨和葡萄进行序列比对和系统进化分析结果表明Ran家族高度保守。但值得注意的是,与其他植物的Ran蛋白相比,香蕉Ran蛋白的N端多出一段长度不等的多肽,且这些多肽序列在NCBI数据库中未能找到同源序列。这可能与在香蕉基因组基因预测过程中将Ran基因的上游序列纳入编码区序列有关或者香蕉Ran家族基因进化出了新的功能。导致这一结果的真正原因还有待进一步研究。
Chen等[18]分析了不同胁迫下水稻OsRAN2的表达模式,发现低温可显著影响OsRAN2的表达。Paul and Kumar[19]研究发现低温可导致茶叶休眠组织和旺盛生长的组织中CsRan2表达量上升,而室温条件下休眠组织中CsRan2表达量下降。Chen等[18]研究发现超表达OsRAN2可显著提高水稻转基因植株的抗寒性。这些研究表明,Ran家族基因与植物对低温胁迫的响应有关。香蕉全基因组中MuRan基因MuRan1-MuRan8编码的蛋白均与其他植物的Ran蛋白高度同源,具有典型的结构域。因此,香蕉Ran蛋白的功能可能与其他植物Ran蛋白的功能相似,参与低温胁迫的响应。MuRan9编码的蛋白与其他香蕉Ran蛋白和植物的Ran蛋白存在较大的差异,具体功能还有待进一步研究。
参考文献:
[1]Clarke P R,Zhang C.Spatial and temporal coordination of mitosis by Ran GTPase[J].Nature Reviews Molecular Cell Biology,2008,9(6):464-477.
[2]Hetzer M,Gruss O J,Mattaj I W.The Ran GTPase as a marker of chromosome position in spindle formation and nuclear envelope assembly[J].Nature Cell Biology,2002,4(7):E177-E184.
[3]Joseph J.Ran at a glance[J].Journal of Cell Science,2006,119(17):3481-3484.
[4]Zang A,Xu X,Neill S,et al.Overexpression ofOsRAN2 in rice andArabidopsisrenders transgenic plants hypersensitive to salinity and osmotic stress[J].Journal of Experimental Botany,2010,61(3):777-789.
[5]Lee Y,Kim M H,Kim S K,et al.Phytochrome-mediated differential gene expression of plant Ran/TC4 small G-proteins[J].Planta,2008,228(1):215-224.
[6]Lü S,Fan Y,Jin C.Overexpression of a Ran GTPase homologous gene,FaRanfrom tall fescue,in transgenicArabidopsis[J].Biologia Plantarum,2011,55(2):331-334.
[7]Wang X,Xu Y,Han Y,et al.Overexpression ofRAN1 in rice and arabidopsis alters primordial meristem,mitotic progress,and sensitivity to auxin[J].Plant Physiology,2006,140(1):91-101.
[8]Wang X,Han Y,Chen C B,et al.Wheat RAN1 affects microtubules integrity and nucleocytoplasmic transport in fission yeast system[J].Acta Botanica Sinica,2004,46(8):940-947.
[9]Yano A,Kodama Y,Koike A,et al.Interaction between Methyl CpG-Binding protein and Ran GTPase during cell division in tobacco cultured cells[J].Annals of Botany,2006,98(6):1179-1187.
[10]Ach R A,Gruissem W.A small nuclear GTP-binding protein from tomato suppresses aSchizosaccharomycespombecell-cycle mutant[J].Proceedings of the National Academy of Sciences of the United States of America,1994,91(13):5863-5867.
[11]Kim S H,Arnold D,Lloyd A,et al.Antisense expression of an Arabidopsis ran binding protein renders transgenic roots hypersensitive to auxin and alters auxin-induced root growth and development by arresting mitotic progress[J].Plant Cell,2001,13(12):2619-30.
[12]Miché L,Battistoni F,Gemmer S,et al.Upregulation of jasmonate-inducible defense proteins and differential colonization of roots ofOryzasativacultivars with the endophyteAzoarcussp.[J].Molecular Plant-Microbe Interactions,2006,19(5):502-511.
[13]Li K,Xu C,Zhang K,et al.Proteomic analysis of roots growth and metabolic changes under phosphorus deficit in maize(ZeamaysL.) plants[J].Proteomics,2007,7(9):1501-1512.
[14]Jiang Y,Yang B,Harris N S,et al.Comparative proteomic analysis of NaCl stress-responsive proteins inArabidopsisroots[J].Journal of Experimental Botany,2007,58(13):3591-3607.
[15]Ferreira S,Hjern K,Larsen M,et al.Proteome profiling ofPopuluseuphraticaOliv.upon heat stress[J].Annals of Botany,2006,98(2):361-377.
[16]Xu C,Huang B.Root proteomic responses to heat stress in twoAgrostisgrass species contrasting in heat tolerance[J].Journal of Experimental Botany,2008,59(15):4183-4194.
[17]Xu C,Huang B.Differential proteomic response to heat stress in thermalAgrostisscabraand heat-sensitiveAgrostisstolonifera[J].Physiologia Plantarum,2010,139(2):192-204.
[18]Chen N,Xu Y,Wang X,et al.OsRAN2,essential for mitosis,enhances cold tolerance in rice by promoting export of intranuclear tubulin and maintaining cell division under cold stress[J].Plant,Cell & Environment,2011,34(1):52-64.
[19]Paul A,Kumar S.Responses to winter dormancy,temperature,and plant hormones share gene networks[J].Functional & Integrative Genomics,2011,11(4):659-664.
[20]Yoshimura K,Masuda A,Kuwano M,et al.Programmed proteome response for drought avoidance/tolerance in the root of a C3 Xerophyte(wild watermelon) under water deficits[J].Plant and Cell Physiology,2008,49(2):226-241.
[21]Zhai L,Liu Z,Zou X,et al.Genome-wide identification and analysis of microRNA responding to long-term waterlogging in crown roots of maize seedlings[J].Physiologia Plantarum,2012,147(2):181-193.
[22]Sacco M A,Mansoor S,Moffett P.A RanGAP protein physically interacts with the NB-LRR protein Rx,and is required for Rx-mediated viral resistance[J].The Plant Journal,2007,52(1):82-93.
[23]Tameling W I L,Baulcombe D C.Physical association of the NB-LRR resistance protein Rx with a Ran GTPase-activating protein is required for extreme resistance toPotatovirusX.[J].The Plant Cell Online,2007,19(5):1682-1694.
[24]Tameling W I L,Nooijen C,Ludwig N,et al.RanGAP2 mediates nucleocytoplasmic partitioning of the NB-LRR immune receptor Rx in the Solanaceae,thereby dictating Rx function[J].The Plant Cell Online,2010,22(12):4176-4194.
[25]Rivas S.Nuclear dynamics during plant innate immunity[J].Plant Physiology,2012,158(1):87-94.
[26]Drivas G T,Shih A,Coutavas E,et al.Characterization of four novel ras-like genes expressed in a human teratocarcinoma cell line[J].Molecular and Cellular Biology,1990,10(4):1793-1798.
[27]Bischoff F R,Ponstingl H.Catalysis of guanine nucleotide exchange on Ran by the mitotic regulator RCC1[J].Nature,1991,354(6348):80-82.
[28]Merkle T,Haizel T,Matsumoto T,et al.Phenotype of the fission yeast cell cycle regulatory mutant pim1-46 is suppressed by a tobacco cDNA encoding a small,Ran-like GTP-binding protein[J].The Plant Journal,1994,6(4):555-565.
[29]Matsumoto T,Beach D.Premature initiation of mitosis in yeast lacking RCC1 or an interacting GTPase[J].Cell,1991,66(2):347-60.
[30]Belhumeur P,Lee A,Tam R,et al.GSP1 and GSP2,genetic suppressors of the prp20-1 mutant in Saccharomyces cerevisiae:GTP-binding proteins involved in the maintenance of nuclear organization[J].Molecular and Cellular Biology,1993,13(4):2152-61.
[31]Jiang S Y,Ramachandran S.Comparative and evolutionary analysis of genes encoding small GTPases and their activating proteins in eukaryotic genomes[J].Physiological Genomics,2006,24(3):235-251.
[32]Haizel T,Merkle T,Pay A,et al.Characterization of proteins that interact with the GTP-bound form of the regulatory GTPase Ran inArabidopsis[J].The Plant Journal,1997,11(1):93-103.
[33]Dhont A,Denoeud F,Aury J M,et al.The banana(Musaacuminata) genome and the evolution of monocotyledonous plants[J].Nature,2012,488(7410):213-7.

许小向,蒋艳,洪艳平,等.红巧梅花色苷树脂静态纯化工艺研究[J].江西农业大学学报,2015,37(1):163-168.
Genome-Wide Analysis of TheRanGene Family
in Banana(Musaacuminata)
ZHANG Ya-ling1,FANG Zhi-zhen1,2,LAI Zhong-xiong1*
(1.Institute of Horticultural Biotechnology,Fujian Agriculture and Forestry University,Fuzhou 350002,China;2.Fruit Research Institute,Fujian Academy of Agricultural Sciences,Fuzhou 350013,China)
Abstract:Ran is a kind of small GTPases that plays an important role in fundmental cellular activities.In the present study,9 MuRan family genes were identified in banana genome and their chromosome distribution,gene structure,physical and chemical characteristics,function domain and evolutionary relationship were analyzed.The results indicated that MuRan family genes were evenly distributed in 4 chromosomes.MuRan proteins showed high homology with Ran proteins from other plants and contained characteristic domains of Ran proteins,including GTP Hydrolysis domain,RanGAP-binding domain and acidic tail and were relatively closer with AtRan in phylogenetic relationship.
Key words:banana;genome;Ran;bioinformatics
作者简介:张雅玲(1987—),女,硕士生,主要从事果树生物技术研究,E-mail:451476856@qq.com;*通信作者:赖钟雄,研究员,博士,E-mail:laizx01@163.com。
基金项目:国家香蕉产业体系专项资金(CARS-32-11)和福建省重大农业科技平台项目(2008N2001)
收稿日期:2014-04-14修回日期:2014-06-21
中图分类号:S668.1
文献标志码:A
文章编号:1000-2286(2015)01-0157-06

