活血丹小尺度空间遗传结构
左 威, 宋文静, 金则新, 李钧敏,*
1 上海师范大学生命与环境科学学院, 上海 200234 2 浙江省植物进化生态学与保护重点实验室, 台州 318000 3 台州学院生态研究所, 台州 318000
活血丹小尺度空间遗传结构
左 威1,2,3, 宋文静3, 金则新2,3, 李钧敏2,3,*
1 上海师范大学生命与环境科学学院, 上海 200234 2 浙江省植物进化生态学与保护重点实验室, 台州 318000 3 台州学院生态研究所, 台州 318000
活血丹(Glechomalonituba)是唇形科活血丹属的多年生克隆草本植物。采用简单重复序列区(ISSR)分子标记技术,比较分析了3个不同斑块活血丹的遗传多样性、克隆多样性以及小尺度空间遗传结构,并探讨其与生境异质性、繁殖体传播和人为干扰的相关性。结果表明:1)活血丹物种水平的遗传多样性很低,各斑块的遗传多样性较低,以水渠边斑块最高,平葛村斑块次之,竹林下斑块最低。2)活血丹物种水平的克隆多样性较高,各斑块活血丹的克隆多样性以水渠边斑块最大,平葛村斑块次之,竹林下斑块最低。3)遗传分化系数Gst为0.7129,表明活血丹的遗传变异大部分存在于斑块间;斑块间的基因流较小,仅为0.2004。4)空间自相关分析表明活血丹一定的空间距离下存在显著的空间遗传结构,竹林下斑块在100 cm 时存在显著性正相关,其X轴截矩为205.994cm;平葛村斑块在200 cm 时存在显著性正相关,其X轴截矩为235. 388cm;水渠边斑块在150 cm时存在显著性正相关,其X轴截矩为240.336cm。应用软件SPAGeDi 1.2软件对各斑块的空间遗传结构进行量化,表明平葛村斑块具有最强的空间遗传结构,水渠边和竹林下斑块的空间遗传结构较弱。活血丹的遗传结构、克隆结构及空间分布格局受到其繁殖体传播特征、人为干扰和繁殖权衡的影响,是其对生境异质性的适应结果。
活血丹; 克隆植物; 小尺度空间遗传结构; 简单重复序列区间(ISSR)
克隆植物是指在定居前期通过与母株相连的芽、分蘖或枝条等繁殖体产生无性繁殖的植物[1]。克隆植物常通过克隆繁殖形成与亲本基因型相同的后代,形成居群的遗传结构与遗传多样性。小尺度空间遗传结构是指植物基因型或遗传多样性的非随机的空间分布格局[2]。研究克隆植物的小尺度空间遗传结构,阐明克隆植物不同斑块的遗传格局、种间关系以及环境因子的作用[3],对研究克隆植物种群的形成、维持和衰退机制及植物定居、侵殖和演替的机理具有重要意义[4]。
早期研究克隆植物的遗传多样性主要是在表型和染色体水平进行的,但确定形态学性状存在一定的局限,如表型可塑性或克隆繁殖组织的老龄化等影响形态学性状的判断。随着分子生物学技术的发展和应用,基于PCR的DNA分子标记的发展为准确鉴定克隆基株提供了可靠技术保障,如简单重复序列区间(inter- simple sequence repeat, ISSR)[5]、随机扩增多态DNA (random amplified polymorphic DNA, RAPD)[6]、扩增片段长度多态性(amplified fragments length polymorphic, AFLP)[7]、简单重复序列(Simple sequence repeat, SSR)[8]已被广泛地用于克隆植物的克隆多样性及居群遗传结构的研究。其中,ISSR分子标记具有较大的优势,如相对于SSR,ISSR引物不需要目标序列的相关信息,引物设计较为简单;相对于AFLP,ISSR的操作较为简单[17];而相对RAPDs,ISSR实验结果更可靠,实验的可重复性更高[9]。已有较多学者应于ISSR分子标记技术来研究克隆植物的克隆多样性与空间遗传结构,如沙鞭(Psammochloavillosa)[10]、蛇莓(Duchesneaindica)[4]、竹叶眼子菜(Potamogetonmalaianus)[5]。
克隆植物在较小的空间尺度上就可形成不同的空间遗传结构,从而形成不同斑块的克隆多样性。如李钧敏和金则新发现蛇莓3个不同斑块的小尺度克隆结构存在差异明显[4];van Rossum 和Triest发现牛舌樱草(Primulaelatior)具有很强的小尺度空间遗传结构差异[11]; Bergf和Hamrick发现土耳其橡树林(Quercuscerris)在5—10 m的距离表现出明显的小尺度基因结构差异[12]。克隆植物的空间结构受生境的异质性、植物繁殖体传播、植物间相互作用、生物环境作用、外界干扰等影响,且不同物种的克隆植物其空间遗传结构受到的影响因素是不同的。如牛舌樱草的空间遗传结构的形成主要受较短的花粉扩散距离与近交的繁育系统的影响[11];Berg和Hamrick认为土耳其橡树的小尺度空间遗传结构是由于小的果实扩散距离及克隆生长造成的[12];李钧敏和金则新认为蛇莓的小尺度空间遗传结构是由于低的种子萌发率及强的克隆生长能力造成的,并受到环境因素的强烈影响[4]。因此,有必要对一些典型物种开展典型生境的小尺度空间遗传结构研究,以阐明克隆植物的空间遗传结构的形成及机制。
活血丹(Glechomalongituba)又名佛耳草、金钱草,为唇形科(Lamiaceae)活血丹属(Glechoma)多年生草本植物。活血丹以全草入药,茎中含有挥发油和多种药用成分,对于治疗肝胆及尿路等多种炎症有一定疗效[13]。活血丹为匍匐茎型克隆植物,广泛分布于全国各地,常常生长在海拔50—2000 m范围内的林缘、疏林下、草地中、溪边等阴湿生境中[14]。活血丹的生长受生境条件的限制及人为干扰较大,对生境条件具有较强的可塑性[15]。本文通过ISSR分子标记技术分析3个不同典型斑块的活血丹的克隆多样性,采用空间自相关分析研究活血丹的克隆空间结构,探讨其与生境异质性、繁殖体传播和人为干扰的相关性,其结果不仅可以丰富克隆植物空间遗传结构研究,而且可以进一步阐明克隆植物对环境的适应机理。
1 材料与方法
1.1 实验材料
于2008年4月分别在浙江省临安市昌化镇采集3个不同斑块中的活血丹,各斑块的基本情况如表1。将各斑块被划分成50cm×50cm的正方形方格,通过记录空间坐标(x,y) 对每个点上的个体进行定位[2](图1)。每株取其健康幼嫩叶片放入装有硅胶(干燥剂)的密封袋中干燥保存备用。

表1 不同活血丹斑块的生境概况
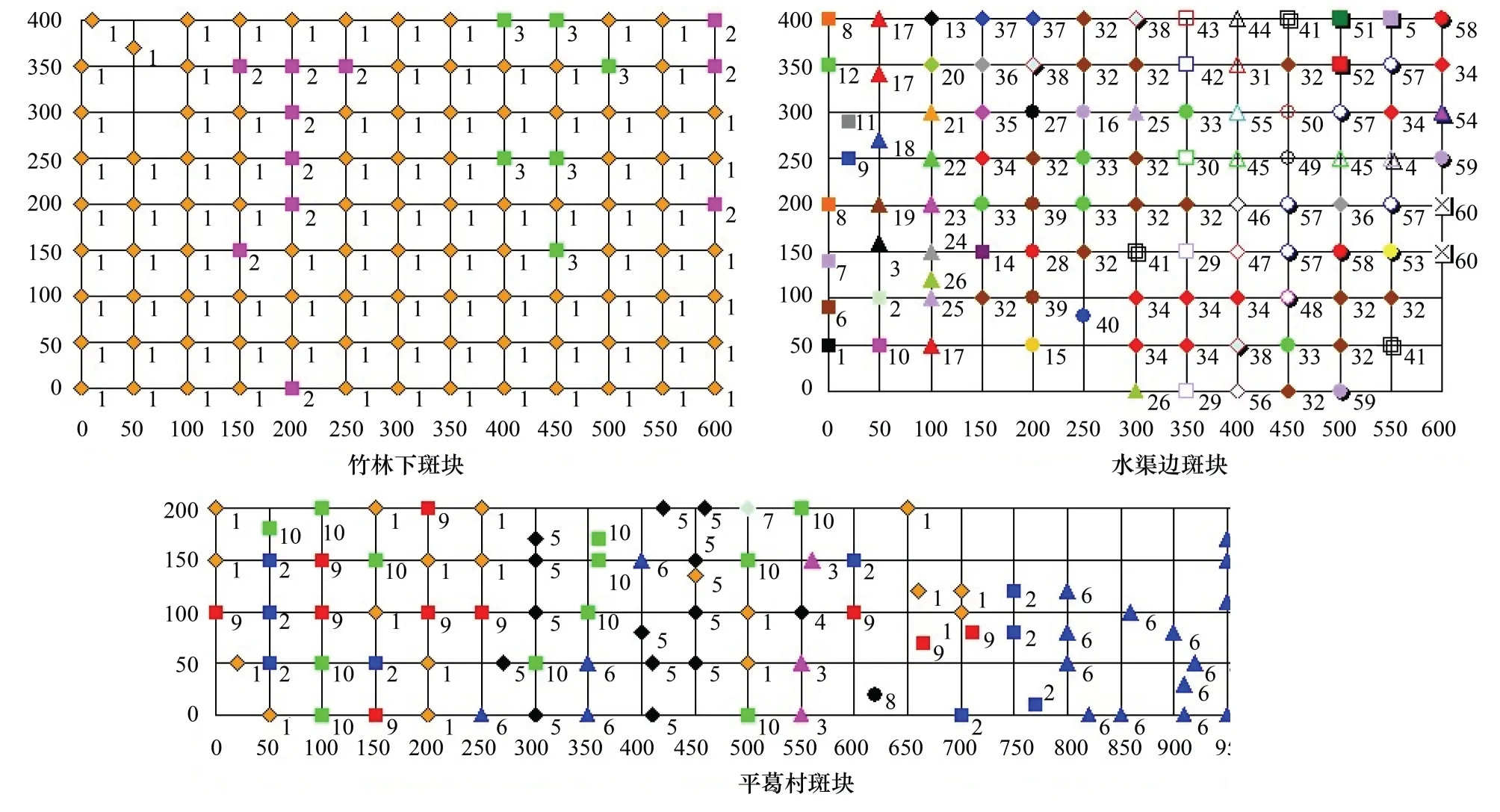
图1 不同斑块的个体及基因型分布图Fig.1 Distribution of the individuals and genotypes of Glechoma lonituba in different patches不同数字和颜色表示不同基因型,同一颜色和数字表示同一基因型
1.2 实验方法
1.2.1 DNA提取和定量
DNA提取采用改进的SDS法[16]提取总DNA。然后DNA经0.8%琼脂糖凝胶电泳分析,用伯乐公司的Gel Doc XR凝胶成像分析系统拍照定量,并最终稀释成浓度为10 ng /μL,然后放于-20℃冰箱保存备用。
1.2.2 ISSR扩增与产物的鉴定
ISSR引物是根据加拿大哥伦比亚大学(University of British Columbia, Set No9, No.801- 900)公布的序列,由上海生工生物工程公司合成。在PTC- 220热循环仪(美国伯乐公司)中进行扩增反应。经过测试镁离子浓度、模板DNA含量、dNTP浓度、DNA聚合酶用量、引物浓度、牛血清白蛋白(bovine serum albumin, BSA)对ISSR反应结果的影响,通过优化实验确定了最适的ISSR扩增反应条件为:10μL PCR反应体积,1μL Taq酶配套缓冲液(10mmol/L Tris-HCL pH 9.0,50mmol/L KCl,0 1% TritonX- 100),0.4 mmol/L MgCl2,10 pmo1引物,8 ng模板DNA,2mg/mL BSA,dNTP 0. 5mmol/L,1 U Taq酶(上海鼎国公司)。采用touch-down PCR扩增程序:94℃预变性5 m in,94℃变性30s,57℃退火1 m in,72℃延伸1.5min,每个循环下降0 5℃,共10个循环;94℃变性30s,52℃退火1 min,72℃延伸1.5min共25个循环;72℃完全延伸5min。DNA扩增产物在2%的凉脂糖凝胶(含0.5 μg/mL溴化乙锭)中电泳,电泳缓冲液为0. 5 ×TBE,用Gel Doc XR凝胶成像分析系统(美国伯乐公司)进行拍照并保存备用。用200 bp DNA梯度(上海华美公司)做为标准分子量参照物。阴性对照用双蒸水代替总DNA,其它物质不变。用100个ISSR引物使用上述体系进行PCR而后电泳、拍照,选择在3个活血丹居群中均可扩增出清晰条带,且条带不弥散、不模糊、重复性好,同时选择阴性对照中无条带的引物作为正式扩增的ISSR扩增引物。
1.2.3 数据统计与分析
对应反应产物在凝胶上的条带的分子量,有条带记为“1”,无条带记为“0”,得到ISSR分析的原始数据。然后通过POPGEN 32软件[17]计算多态位点百分率(P),Shannon信息指数(1)和Nei指数(h)。根据基因频率矩阵用POPGEN 32软件计算遗传分化系数(Gst),并估算基因流。
采用以下4个参数[4]估算克隆多样性:
(1) 种群中基因型(基株)总数(G),将全部位点基因型相同的植株视为来自同一基株,居群中基株总数即为G。
(2) 基因型比率(G/N),其中N是居群中所有个体(分株)总数。
(3) Simpson多样性指数(D):
D=1- ∑ [Ni(Ni-1) /N(N-1) ]
式中,Ni为居群内第i种基因型的总数。
(4) 基因型分布的均匀度 (E):
E=(D-Dmin)/(Dmax-Dmin)
其中,Dmin=[ (G-1) (2N-G) ]/[N(N-1)],Dmax=[(G-1)N]/[G(N-1)]。
对每个基株的空间分布进行定位,同时采用GenAIEx 6软件计算每个基因型与空间位置的空间自相关系数(r),以分析斑块内克隆的小尺度空间分布格局。采用软件SPAGeDi 1.2计算空间自相关系数与空间距离的自然对数值间线性回归方程的斜率(b)和第一距离等及内所有个体间空间自相关系数的平均值(F1),根据公式计算Sp=-b/(1-F1)来量化空间遗传结构[18]。
2 结果与分析
2.1 遗传多样性与遗传分化
对活血丹个体进行ISSR引物分析,发现采用6个ISSR引物即可分辨出活血丹不同克隆(表2)。采用此6个引物对3个活血丹斑块共313个个体的DNA样品进行了ISSR分析。结果如表3所示,活血丹物种水平上的遗传多样性比较低;而斑块间的遗传多样性很低,尤其是竹林下斑块。3个斑块间的遗传多样性差距较大,水渠边斑块最高,平葛村斑块次之,而竹林下斑块最低。遗传分化系数Gst高达71.29%,表明在大部分的遗传变异存在于斑块间,而仅有28.71%的遗传变异存在于斑块内。由Gst估算的活血丹的基因流较小,仅为0.2004。

表2 1SSR分析用的6个引物序列

表3 活血丹各斑块的遗传多样性
2.2 克隆多样性
不同斑块的克隆多样性指数(表4)表明活血丹物种水平的克隆多样性比较高,3个斑块间的克隆多样性以水渠边斑块最大,平葛村斑块次之,竹林下斑块最低。由图1可知,3个活血丹斑块均由多克隆组成,水渠边斑块的克隆数目较多,但组成克隆的分株数目较少,最大的克隆在所取样品中有15个分株;平葛村斑块的克隆大小次之,最大的克隆在所取样品中有24个分株;竹林下斑块的克隆最大,最大的克隆在所取样品中有98个分株;表明活血丹斑块中存在优势克隆。

表4 不同活血丹斑块的克隆多样性
2.3 小尺度空间遗传结构
3个不同斑块活血丹克隆分布在12个不同距离等级时的空间自相关分析结果如图2—图4所示。竹林下斑块在100 cm 时存在显著性正相关,在350 cm 时存在显著性负相关,其X轴截矩为205.994cm;平葛村斑块在200 cm 时存在显著性正相关,在450 cm 时存在显著性负相关,其X轴截矩为235. 388cm;水渠边斑块在150 cm时存在显著性正相关,在450 cm 时存在显著性负相关,其X轴截矩最大,高达240.336cm。 应用软件SPAGeDi 1.2软件对各斑块的空间遗传结构进行量化,表明平葛村斑块具有最强的空间遗传结构(Sp=0.0944),水渠边和竹林下斑块的空间遗传结构较弱(Sp=0.0558,Sp=0.0556)。

图2 水渠边斑块空间自相关曲线图 Fig.2 Correlograms showing the spatial autocorrelation coefficient r of Glechoma lonituba in Shuiqubian patchU和L分别是95%置信区间的上限和下限
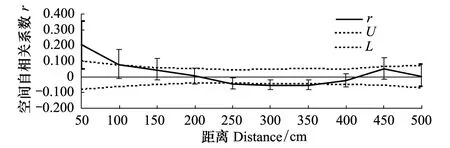
图3 竹林下斑块空间自相关曲线图 Fig.3 Correlograms showing the spatial autocorrelation coefficient r of Glechoma lonituba in Zhulinxia patchU和L分别是95%置信区间的上限和下限
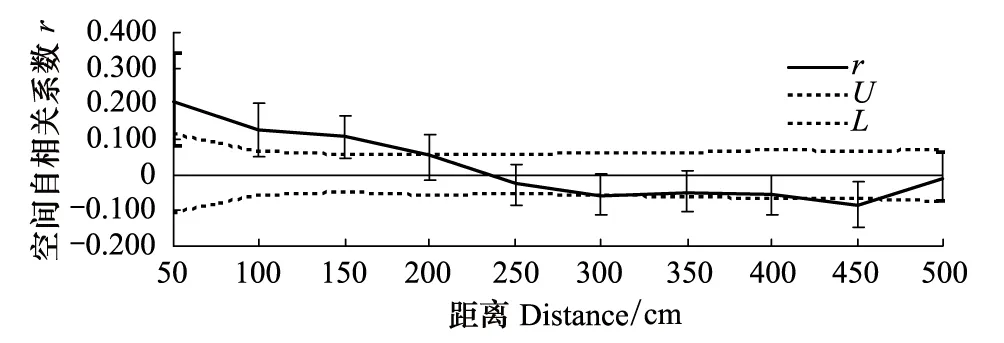
图4 平葛村斑块空间自相关曲线图 Fig.4 Correlograms showing the spatial autocorrelation coefficient r of Glechoma lonituba in Pinggecun patchU和L分别是95%置信区间的上限和下限
3 讨论
本研究显示克隆植物活血丹在较小距离范围内具有显著的空间遗传结构,如竹林下斑块、平葛村斑块和水渠边分别在100cm、200cm和150cm时存在显著性正相关。这可能与活血丹通过匍匐茎营无性繁殖的克隆习性有关,如李钧敏和金则新同样采用ISSR分析了匍匐茎克隆植物蛇莓的小尺度空间遗传结构,发现其三个不同斑块在空间距离为20cm时存在显著性正相关[4]。活血丹为多年生草本植物,可营无性繁殖与有性繁殖,但其无性繁殖较为容易,匍匐茎逐节生根,蔓延能力极强,在自然界中常通过产生较长的地上匍匐茎而表现出克隆生长习性[19]。另外,活血丹为虫媒传粉,种子千粒重为0.35g,落地可繁殖,虽然其种子数量不多,但萌发率较高,可达79.3%,这可能是造成活血丹空间遗传结构形成距离要略高于蛇莓的主要原因。
基于6个ISSR上物鉴定的活血丹的基因型,结果表明3个活血丹斑块均由多克隆组成,基株数目以水渠边斑块最多,平葛村次之,竹林下最低;每个基因型平均拥有的分株数目以水渠边斑块最少,平葛村斑块次之,竹林下斑块最多。水渠边斑块的克隆数目较多,但组成克隆的分株数目较少,最大的克隆由15个分株组成;平葛村斑块的克隆大小次之,最大的克隆由24个分株组成;竹林下斑块的克隆最大,最大的克隆由98个分株组成;表明竹林下的活血丹存在优势克隆。应用软件SPAGeDi 1.2软件对各斑块的空间遗传结构进行量化,表明平葛村斑块具有最强的空间遗传结构,水渠边和竹林下斑块的空间遗传结构较弱。空间自相关曲线图中X轴截矩标志着斑块中同一克隆所占据的最小平均长度[20]。空间自相关分析显示平葛村斑块活血丹基因型与空间距离的正相关的距离最大,可达200 cm,其X轴截矩为235.388cm,表明在平葛村优越的生境条件下,活血丹选择资源投入较小的克隆繁殖实现扩张,形成竞争压力小的密集克隆型[21],进行点面扩张,斑块左边表现为强势克隆的相互叠加,斑块右边表现出强烈的密集克隆。而水渠边的截距即克隆能到达的距离却最大,为240.336 cm,表明激烈的竞争环境下,活血丹表现出强烈的觅食反应,导致同一基株克隆表现为游击型克隆[21],进行线性扩张,而非点面扩张,同时水渠边斑块为逃避竞争和密度制约机制进行种子繁殖赋予此斑块最丰富的基因型,种子繁殖和克隆繁殖各尽所长逃离生境。而竹林下斑块活血丹基因型与空间距离的正相关的距离最小,为150 cm,其克隆能到达的距离也最小,为205.994 cm,这主要是因为竹林下条件对种子繁殖的强烈抑制,导致此斑块出现非常显著的克隆繁殖特征,形成密集克隆型[21],表现为基因型最集中的点面扩张,这种聚集式点面扩张对资源的消耗必然导致同一基因型的植物很难在更大的范围实现扩张,单个克隆所占据的空间最小。
克隆植物的空间结构受生境的异质性、植物繁殖体传播、植物间相互作用、生物环境作用、外界干扰等影响[22]。竹林下斑块与水渠边、平葛村斑块的生境差异较大,如竹林下斑块由于竹叶致密而具有较弱的光强,其土壤受到竹子生长的影响而缺水、板结。由于有性繁殖与无性繁殖两种方式在风险分摊、散布距离、斑块化生境中的资源获取能力、遗传多样性的维持作用等方面各有贡献,因此,克隆植物在不同生境下会采取繁殖权衡策略[23],如林下生境不利于种子萌发及幼苗生长,克隆植物通过克隆整合作用克服幼苗在林下的劣势来繁衍后代;在有限空间内,当种内竞争激烈而受到密度制约时[24],克隆植物可以通过自身的激素调节使更多的资源投资于种子繁殖,避免种内竞争[25]。竹林下斑块中限制活血丹开花、种子萌发、幼苗生长等限制性因素较多,如竹林致密的竹叶有很强的遮光作用可以影响开花和种子形成[26],竹林下缺水、板结的土壤可以导致自然条件下种子很难在其上萌发,这些限制因素直接影响种子繁殖以及幼苗生长,导致该斑块中的活血丹具有较高比例的克隆繁殖和较低的遗传多样性,以最广泛的克隆整合来确保后代的存活。而水渠边(SQB)和平葛村(PGC)不存在这些限制性因素的影响或受影响较小,因此具有较高的遗传多样性。水渠边由于水分充足,植物种类繁多,种间竞争激烈,密度制约机制强烈,活血丹趋向于种子繁殖,以逃避恶劣的生存环境,结果导致水渠边斑块比平葛村斑块具有更高的克隆多样性和遗传多样性。另外,平葛村存在显著的人畜干扰可能是导致平葛村斑块的克隆多样性和遗传多样性较水渠边明显降低的主要原因:如人为铲除土地上的灌木减少水肥竞争和光源争夺,农村施肥、灌溉使水肥相对充裕,植物选择性进行更低投资的克隆繁殖[22];牲畜的啃食和践踏使本应该产生种子的枝条被吃掉或采折,促进了分枝和分蘖的形成,同时减少了种子产生的数量,使得克隆生殖占优势。
活血丹的空间遗传结构可能是活血丹长期适应不同生境和不同人为干扰的结果,尤其是对光照、水分、养分等环境异质性因子的长期适应结果。面对不同生境,活血丹通过采取繁殖权衡来应对;面对小尺度的环境异质性,活血丹又采取不同的克隆型来应对。活血丹在适应的过程中进化出的优势克隆,其克隆型和非优势克隆的克隆型有很大不同,给我们研究克隆植物提供参考。本研究结果揭示了活血丹通过不同的繁殖方式、不同的克隆生长型来适应不同生境,有助于了解克隆植物的适应机制。
[1] Alpert P. Nutrient sharing in natural clonal fragments ofFragariachiloensis. Journal of Ecology, 1996, 84(3): 395- 406.
[2] Jin Z X, Li J M, Liu L L. Fine-scale spatial genetic structure within age classes of the two fragmented populations ofSinocalycanthuschinensisCheng et S. Y. Chang, an endangered plant species endemic to China. Biochemical Systematics and Ecology, 2012, 43: 117- 124.
[3] Stowe L G, Wade M J. The detection of small-scale patterns in vegetation. Journal of Ecology, 1979, 67(3): 1047- 1064.
[4] 李钧敏, 金则新. 匍匐茎草本植物蛇莓小尺度克隆结构. 生态学报, 2009, 29(7): 3540- 3548.
[5] 陈媛媛, 栗琪, 吴文颖, 李伟. 竹叶眼子菜居群遗传多样性和克隆结构. 应用生态学报, 2006, 17(11): 2034- 2040.
[6] Pluess A R, Stöcklin J. Population genetic diversity of the clonal plantGeumreptans(Rosaceae) in the Swiss Alps. American Journal of Botany, 2004, 91(12): 2013- 2021.
[7] Honnay O, Jacquemyn H, Roldán-Ruiz I, Hermy M. Consequences of prolonged clonal growth on local and regional genetic structure and fruiting success of the forest perennialMaianthemumbifolium. Oikos, 2006, 112(1): 21- 30.
[8] Hämmerli A, Reusch T B H. Genetic neighbourhood of clone structures in eelgrass meadows quantified by spatial autocorrelation of microsatellite markers. Heredity, 2003, 91(5): 448- 455.
[9] Kojima T, Nagaoka T, Noda K, Ogihara Y. Genetic linkage map of ISSR and RAPD markers in Einkorn wheat in relation to that of RFLP markers. Theoretical and Applied Genetics, 1998, 96(1): 37- 45.
[10] Li A, Ge S. Genetic variation and clonal diversity ofPsammochloavillosa(Poaceae) detected by ISSR markers. Annals of Botany, 2001, 87(5): 585- 590.
[11] Van Rossum F, Triest L. Fine-scale genetic structure of the commonPrimulaelatior(Primulaceae) at an early stage of population fragmentation. American Journal of Botany, 2006, 93(9): 1281- 1288.
[12] Berg E E, Hamrick J L. Fine-scale genetic structure of a turkey oak forest. Evolution, 1995, 49(1): 110- 120.
[13] 张前军, 杨小生, 朱海燕, 郝小江. 活血丹属植物的化学成分及药理研究进展. 中草药, 2006, 37(6): 950- 952.
[14] 陈尚, 李自珍, 王刚. 克隆植物生长型的研究进展. 生态学杂志, 1997, 16(4): 59- 63.
[15] 陶建平, 钟章成. 匍匐茎草本活血丹(Glechomalongituba)在不同养分条件下的克隆形态. 生态学报, 2000, 20(2): 207- 211.
[16] 李钧敏. 分子生物学实验. 杭州: 浙江大学出版社, 2010: 5- 5.
[17] Yeh F C, Boyle T J B. Population genetic analysis of co-dominant and dominant markers and quantitative traits. Belgian Journal of Botany, 1997, 129: 157- 163.
[18] Vekemans X, Hardy O J. New insights from fine-scale spatial genetic structure analyses in plant populations. Molecular Ecology, 2004, 13(4): 921- 935.
[19] 张丽丽, 董鸣, 李仁强, 王艳红, 崔清国, 何维明. 土壤养分斑块对比度改变活血丹克隆整合强度和方向. 植物生态学报, 2007, 31(4): 619- 624.
[20] Peakall R, Ruibal M, Lindenmayer D B. Spatial autocorrelation analysis offers new insights into gene flow in the Australian bush rat,Rattusfuscipes. Evolution, 2003, 57(5): 1182- 1195.
[21] 董鸣. 资源异质性环境中的植物克隆生长: 觅食行为. 植物学报, 1996, 38(10): 828- 835.
[22] 刘振国, 李镇清. 植物群落中物种小尺度空间结构研究. 植物生态学报, 2005, 29(6): 1020- 1028.
[23] Widén B, Cronberg N, Widén M. Genotypic diversity, molecular markers and spatial distribution of genets in clonal plants, a literature survey. Folia Geobotanica, 1994, 29(2): 245- 263.
[24] Gillman M, Bullock J, Silvertown J, Hill B C. A density-dependent model ofCirsiumvulgarepopulation dynamics using field-estimated parameter values. Oecologia, 1993, 96(2): 282- 289.
[25] Nishitani S, Takada T, Kachi N. Optimal resource allocation to seeds and vegetative propagules under density-dependent regulation inSyneilesispalmata(Compositae). Plant Ecology, 1999, 141(1/2): 179- 189.
[26] 王洪义, 王正文, 李凌浩, 陈雅君, 任丽昀. 不同生境中克隆植物的繁殖倾向. 生态学杂志, 2005, 24(6): 670- 676.
Fine-scale spatial genetic structure ofGlechomalongituba
ZUO Wei1,2,3, SONG Wenjing3, JIN Zexin2,3, LI Junmin2,3,*
1SchoolofLifeandEnvironmentScience,ShanghaiNormalUniversity,Shanghai200234,China2ZhejiangProvincialKeyLaboratoryofPlantEvolutionaryEcologyandConservation,Taizhou318000,China3InstituteofEcology,TaizhouUniversity,Taizhou318000,China
Fine-scale spatial genetic structure, which indicates nonrandom spatial distribution of genotypes or genetic diversity, has important consequences for population biology. The study of fine-scale spatial genetic structure can provide an understanding of the key processes and mechanisms involved in the maintenance of plant populations.Glechomalongitubais a perennial herbaceous clonal plant species that belongs to the Labiatae family.Glechomalongitubais a herb of medicinal importance that is widely distributed in China, and its phenotypic characteristics vary among different habitats. The genetic diversity, clonal diversity, and fine-scale spatial genetic structure ofGlechomalongitubaplants collected from three different patches (Shuiqubian, Pinggecun, and Zhulinxia) with different habitats were analyzed using inter-simple sequence repeat (ISSR) molecular markers. In addition, the correlation with habitat heterogeneity, propagule propagation, and human disturbance were also examined in the study. The results indicated the following: 1) Genetic diversity ofGlechomalongitubaat the species level was relatively low (percentage of polymorphic loci,P=31.15%; Shannon informative index,I=0.1601; Nei′s index,h=0.1096). Genetic diversity ofGlechomalongitubawas highest in Shuiqubian patch (P=21.31%,I=0.0965,h=0.0627), followed by Pinggecun patch (P=8.20%,I=0.0354,h=0.0226), and Zhulinxia patch (P=3.28%,I=0.0120,h=0.0073). 2) Clonal diversity ofGlechomalongitubaat the species level was relatively high (number of genets,G=73; ratio of genets to ramets,G/N=0.2332; Simpson′s diversity index,D=0.8843; genotypic evenness,E=0.8192). The clonal diversity ofGlechomalongitubawas highest in Shuiqubian patch (G=60,G/N=0.5660,D=0.9693,E=0.8747), followed by Pinggecun patch (G=10,G/N=0.1087,D=0.8430,E=0.9075), and Zhulinxia patch (G=3,G/N=0.0260,D=0.2642,E=0.3599). 3) Genetic differentiation coefficient (Gst) was 0.7129, which indicated that most of the genetic variation existed among patches, whereas little genetic variation existed within patches. The estimated gene flow was as low as 0.2004. 4) Spatial autocorrelation analysis showed that the autocorrelation coefficient ofGlechomalongitubain Zhulinxia patch was significantly positive at a distance of 100 cm with anX-intercept of 205.994 cm but significantly negative at a distance of 350 cm. The autocorrelation coefficient in Pinggecun patch was significantly positive at a distance of 200 cm with anX-intercept of 235.388 cm but significantly negative at a distance of 450 cm. The autocorrelation coefficient in Shuiqubian patch was significantly positive at a distance of 150 cm with anX-intercept of 240.336 cm but significantly negative at a distance of 350 cm. Analysis with SPAGeDi 1.2 software showed that the strength of spatial genetic structure in Pinggecun patch was greater than those in Shuiqubian and Zhulinxia patches. TheSpratio (used to compare the extent of spatial genetic structure among populations) for Pinggecun, Shuiqubian, and Zhulinxia patches was 0.0944, 0.0558, and 0.0556, respectively. The genetic diversity, clonal diversity, and fine-scale spatial genetic structure ofGlechomalongitubaare affected by propagule dispersal characteristics, human disturbance, and trade-off between investment in sexual reproduction and clonal propagation and might be a consequence of adaptation to habitat heterogeneity.
Glechomalongituba;clonal plant; fine-scale spatial genetic structure; inter-simple sequence repeat (ISSR)
国家自然科学基金(30870392)
2013- 12- 22;
日期:2014- 11- 03
10.5846/stxb201312223001
*通讯作者Corresponding author.E-mail: lijm@tzc.edu.cn
左威, 宋文静, 金则新, 李钧敏.活血丹小尺度空间遗传结构.生态学报,2015,35(17):5761- 5768.
Zuo W, Song W J, Jin Z X, Li J M.Fine-scale spatial genetic structure ofGlechomalongituba.Acta Ecologica Sinica,2015,35(17):5761- 5768.

