谷氨酸棒杆菌中苯酚羟化酶的鉴定及功能研究
杨志方,肖 潇,司美茹,成娟丽,2,沈锡辉,王 瑶
(1 西北农林科技大学 生命科学学院,陕西 杨凌 712100;2 运城学院 生命科学系,山西 运城 044000)
谷氨酸棒杆菌中苯酚羟化酶的鉴定及功能研究
杨志方1,肖 潇1,司美茹1,成娟丽1,2,沈锡辉1,王 瑶1
(1 西北农林科技大学 生命科学学院,陕西 杨凌 712100;2 运城学院 生命科学系,山西 运城 044000)
【目的】 鉴定谷氨酸棒状杆菌中ncgl2588基因编码的苯酚羟化酶,并验证其在苯酚降解中的作用。【方法】 构建谷氨酸棒状杆菌ncgl2588基因缺失突变株,比较突变株和野生型菌株利用苯酚能力的差异;同时将ncgl2588 基因克隆到表达质粒pET28a中并转化入大肠杆菌BL21(DE3),利用IPTG诱导重组菌株原核表达,纯化并测定重组蛋白对苯酚的羟化作用。【结果】 成功构建了谷氨酸棒状杆菌ncgl2588基因缺失突变株,其在以苯酚为惟一碳源和能源的培养基中不能生长,而野生型谷氨酸棒状杆菌能正常生长;pET28a-ncgl2588重组质粒在大肠杆菌BL21(DE3)中成功表达分子质量为73 ku的目的蛋白,此蛋白酶活为0.37 mmol/(min·mg);在以苯酚为惟一碳源和能源培养谷氨酸棒状杆菌时,苯酚羟化酶得到最高表达,用其他芳香族化合物培养则苯酚羟化酶表达较少;进化树分析发现,谷氨酸棒状杆菌中的苯酚羟化酶与酵母Trichosporoncutaneum中的苯酚羟化酶序列同源性高达43%,远远高于与细菌中苯酚羟化酶的同源性。【结论】 谷氨酸棒状杆菌中的ncgl2588基因编码苯酚羟化酶,且该酶为可诱导酶,在苯酚降解中发挥着重要作用。
谷氨酸棒状杆菌;基因敲除;苯酚羟化酶
苯酚等芳香族化合物在环境污染物中占有很大比例,并且可以在高等动物和人体内富集,当富集到一定浓度时会对机体造成严重伤害,而且芳香族化合物由于结构非常稳定,因此很难用物理化学方法降解[1-2]。目前已经发现了很多微生物有降解苯酚的能力,并且其降解机制已经基本阐明,在降解苯酚的各种酶中,苯酚羟化酶可催化苯酚生成邻苯二酚,是降解苯酚过程的首要步骤,因此受到关注。根据不同菌株中苯酚羟化酶的特性可大致将其分为3个类别:一是ComamonastestosteroniR5、PseudomonasputidaCF600和BH[3-5]等革兰氏阴性细菌中的苯酚羟化酶,这是一种由6个亚基组成的蛋白复合酶体; 二是BacillusthermoglucosidasiusA7 和BacillusstearothermophilusBR219[6-7]等革兰氏阳性细菌中的苯酚羟化酶,是由亚基PheA和PheB组成的异源四聚体(PheA2PheB2),这2个亚基单独存在时并无转化苯酚的活性,只有共同组成四聚体时才有降解苯酚的能力;三是丝孢酵母Trichosporoncutaneum和PseudomonaspickettiiPKO1[8-9]中的苯酚羟化酶,是由单一蛋白组成的同源二聚体。
谷氨酸棒状杆菌(Corynebacteriumglutamicum)属于高GC含量的革兰氏阳性细菌,广泛分布于土壤、沉淀物等自然环境中,能降解包括苯酚在内的多种芳香族化合物,但其苯酚降解机制尚未阐明[10-14]。本研究通过同源序列比对分析谷氨酸棒状杆菌CorynebacteriumglutamicumRES167中ncgl2588基因与酵母Trichosporoncutaneum中苯酚羟化酶之间的相似性,并对该基因进行突变体表型分析和酶学鉴定,以期阐明该基因与苯酚羟化酶的关系,为了解谷氨酸棒状杆菌降解苯酚的机制提供参考。
1 材料与方法
1.1 试验菌株、质粒及其培养
本研究所使用的菌株谷氨酸棒状杆菌C.glutamicumRES167、大肠杆菌E.coliDH5α、BL21(DE3)及质粒pET28a、pK18mobsacB均由西北农林科技大学沈锡辉教授实验室保存。E.coli、C.glutamicum用LB培养基分别在37和30 ℃下培养,LB培养基配方为:酵母提取物5.0 g/L,胰蛋白胨10.0 g/L,NaCl 10.0 g/L。测定谷氨酸棒状杆菌野生型和突变株对苯酚的利用能力时采用无机盐培养基,该培养基配方为:Na2HPO4·12H2O 2.0 g/L,KH2PO40.5 g/L,MgSO4·7H2O 0.03 g/L,NH4Cl 0.53 g/L,微量元素溶液1 mmol/L,酵母粉0.05 g/L,调节pH值至8.4。
根据需要,确定培养大肠杆菌所用抗生素终质量浓度为:卡那霉素50 μg/mL,氨苄青霉素100 μg/mL,氯霉素20 μg/mL; 确定培养谷氨酸棒状杆菌所用抗生素终质量浓度为:卡那霉素25 μg/mL,氯霉素10 μg/mL。
1.2 基因操作
谷氨酸棒状杆菌基因组DNA按Tauch等[15]的方法制备,DNA酶切、连接、质粒提取、凝胶电泳等按Sambrook等[16]的方法进行,谷氨酸棒状杆菌的电转化参照文献[17]的方法。试验过程中所用限制性内切酶和T4DNA连接酶均为NEB公司(美国)生产,pfuDNA聚合酶和TaqDNA聚合酶为全式金公司(中国北京)生产。
1.3 ncgl 2588缺失突变株的构建
利用引物Up F (5′-CGCAGGATCCCGCTTC-GGCCAGGTCAGGCC-3′)和Up R (5′-TCCATTGGGTCACCGGTTGC-3′)扩增ncgl2588基因上游(894 bp)片段,利用引物Down F (5′-GCAAC-CGGTGACCCAATGGATCCCCAAAATGAAAG-GCGCAC-3′)和Down R(5′-CCGACTCGAGTCG-CCGGGTTTCCACTCAGG-3′) 扩增ncgl2588基因下游(824 bp)片段(引物下划线部分分别为限制性内切酶BamHⅠ和XhoⅠ酶切位点),然后混合上、下游片段作为模板,用引物Up F和Down R进行重叠PCR扩增,得到一段1 800 bp左右的重叠片段,经BamHⅠ和XhoⅠ双酶切后将此片段连接至同样经酶切处理的pK18mobsacB质粒,即得到敲除质粒pK18mobsacB-Δ2588。敲除质粒pK18mobsacB-Δ2588的构建流程见图1。
通过电转化方法将此敲除质粒转入谷氨酸棒状杆菌RES167中,在含25 μg/mL卡那霉素的LB平板上筛选第1次同源重组成功的阳性克隆,将此阳性克隆在LB培养基中30 ℃培养过夜后均匀涂布于含有200 g/L蔗糖的LB平板上,筛选发生第2次同源重组的克隆(对卡那霉素敏感),即可获得ncgl2588 基因缺失的突变株。

图1 敲除质粒pK18mobsacB-Δ2588的构建流程
进一步以Up F/Down R为引物,对谷氨酸棒状杆菌野生型菌株和敲除ncgl2588基因的突变菌株、阳性对照pK18mobsacB-Δ2588质粒进行PCR,以验证突变株是否构建成功。
1.4 谷氨酸棒状杆菌野生型和突变株在不同碳源培养基中的生长状况
以苯甲酸钠和苯酚分别为惟一碳源、能源,苯甲酸钠和苯酚的终浓度均为2 mmol/L,将谷氨酸棒状杆菌野生型菌株和突变株用无机盐培养基于30 ℃、180 r/min空气浴中振荡培养,每隔2.5 h取2 mL菌液用721紫外分光光度计测定OD600,根据各个时间点的OD600数值绘制生长曲线。
1.5 ncgl 2588基因的原核表达及纯化
以谷氨酸棒状杆菌基因组DNA为模板PCR扩增ncgl2588基因,正向引物为5′-TCCACATATG-CAGTTTCATTATGAAG-3′(下划线为NdeⅠ位点),反向引物为5′-CAACGCGGCCGCTTAGTTCGCGTTGATTAG-3′(下划线为NotⅠ酶切位点)。PCR反应程序为:95 ℃变性1 min,52 ℃退火1 min,72 ℃延伸2 min,30个循环;72 ℃延伸10 min。纯化后的PCR产物用NdeⅠ和NotⅠ双酶切后与同样酶切处理的表达载体pET28a连接,转化DH5α得到含pET28a-ncgl2588质粒的菌株。将重组的阳性质粒转化到表达宿主BL21(DE3)中,在LB液体培养基中培养至OD600为0.4时,加入0.4 mmol/L IPTG诱导表达蛋白,经SDS-PAGE检测后用6×His亲和柱纯化重组蛋白。SDS-PAGE所用分离胶浓度为12%,浓缩胶浓度为5%,利用考马斯亮蓝R-250染色后观察并拍照[18]。
1.6 ncgl 2588重组蛋白酶活性的测定
酶活反应体系参照Neujahr等[9]的方法并稍作调整。酶促反应体系组成为:0.15 mmol/L NADPH、1 mmol/L FAD、1 mmol/L苯酚、1 mmol/L EDTA、50 mmol/L 磷酸钾缓冲液 (pH 7.5) ,总体积200 μL,最后加入30 μg原核表达的ncgl2588重组蛋白(即苯酚羟化酶)以起始反应。其中FAD为苯酚羟化酶的辅酶,苯酚羟化酶在辅酶FAD的帮助下将苯酚羟基化,羟基化所需要的H+由NADPH提供,因此可以通过测定NADPH在340 nm处吸收值的衰减来计算酶活。
1.7 不同碳源条件下苯酚羟化酶相对活性的测定
以苯酚、葡萄糖、间苯二酚、苯甲酸钠、对甲酚、间羟基苯甲酸和2,4-二羟基苯甲酸分别作为碳源,所有碳源的终浓度均为2 mmol/L,在含不同碳源和能源的无机盐培养基中,于30 ℃、180 r/min条件下培养谷氨酸棒状杆菌RES167菌株16 h;10 000g下收集菌体并用磷酸钾缓冲液(pH 7.5)清洗2次,最终用10 mL磷酸钾缓冲液悬浮细胞;超声破碎菌体(工作3 s,间歇5 s,99次,200 W);12 000g、4 ℃下离心20 min,收集上清可溶部分用Bradford法对总蛋白进行蛋白定量,用牛血清蛋白BSA作为标准品[19]。通过测定全蛋白对苯酚的降解能力(酶活测定方法见1.6节)来计算苯酚羟化酶在全蛋白中所占的比例,即苯酚羟化酶的相对活性。突变株Δ2588 在以葡萄糖为碳源的无机盐培养基中培养后,测定全蛋白中苯酚羟化酶的活性,用于排除其他酶对苯酚的非特异性活性。
1.8 谷氨酸棒状杆菌中苯酚羟化酶的进化树分析
利用MEGA4.0对其他菌株中已经确定为苯酚羟化酶的蛋白氨基酸序列构建生物系统进化树,以分析谷氨酸棒状杆菌中苯酚羟化酶的进化关系及与其他菌中该酶的亲缘关系。
2 结果与分析
2.1 ncgl 2588基因缺失突变株的构建及其对苯酚的利用
2.1.1 突变株的构建 为了敲除ncgl2588基因以鉴定其在谷氨酸棒状杆菌降解苯酚中的作用,首先构建敲除质粒pK18mobsacB-Δ2588,再通过电转化方法将pK18mobsacB-Δ2588质粒转入到谷氨酸棒状杆菌RES167中,并筛选发生2次同源重组的重组子(对卡那霉素敏感),便获得了ncgl2588基因缺失突变株。对谷氨酸棒状杆菌野生型菌株、敲除ncgl2588基因的待测菌株及阳性对照质粒pK18mobsacB-Δ2588进行菌落PCR,结果野生型菌株扩增出一条4 000 bp左右的片段;交换成功菌株与质粒pK18mobsacB-Δ2588均应扩增出一条 1 800 bp左右的片段,筛选ncgl2588基因已被成功敲除的待测菌株(图2),将此菌株命名为CorynebacteriumglutamicumΔ2588,说明ncgl2588基因缺失突变株构建成功。

图2 ncgl 2588基因缺失突变株的PCR鉴定M.Trans2K DNA ladder;1~8.待测缺失突变株;9.野生型菌株;10.pK18mobsacB-Δ 2588质粒
2.1.2 对苯酚的利用 在以苯酚为惟一碳源和能源的无机盐培养基上培养后,发现野生型谷氨酸棒状杆菌能以苯酚为惟一碳源和能源生长,而基因缺失突变株Δ2588完全丧失了利用苯酚的能力(图3-A),证明ncgl2588基因是谷氨酸棒状杆菌降解苯酚所必需的。突变株Δ2588和野生型菌株在以苯甲酸钠为惟一碳源、能源的无机盐培养基中均能正常生长(图3-B),表明ncgl2588基因并不是谷氨酸棒状杆菌降解苯甲酸钠所必需的,这与理论推测一致。
2.2 ncgl 2588基因的克隆表达及其重组蛋白的酶活性
ncgl2588基因原核表达菌株在IPTG诱导后成功表达了1条大小为73 ku的目的蛋白,这与ncgl2588基因翻译产物的理论分子质量相吻合(图4-A)。将此蛋白纯化后测定酶活,可知该蛋白对苯酚的降解活性为0.37 mmol/(min·mg)(图4-B)。进一步证明ncgl2588基因在C.glutamicumRES167中编码了苯酚羟化酶。

图3 谷氨酸棒状杆菌野生型和Δ2588突变株的生长曲线 A.2 mmol/L苯酚为惟一碳源和能源培养基;B.2 mmol/L苯甲酸钠为惟一碳源和能源培养基
Fig.3 Growth of wild-type and deletion mutant ofCorynebacteriumglutamicumRES167 A.Minimal medium with 2 mmol/L phenol as sole carbon and energy source;B.Minimal medium with 2 mmol/L sodium benzoate as sole carbon and energy source
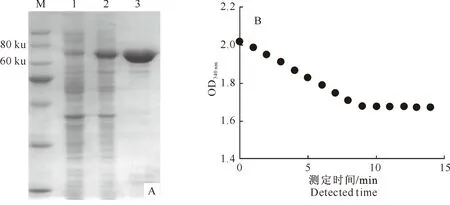
图4 谷氨酸棒状杆菌ncgl 2588基因的表达纯化(A)及重组蛋白的酶活性(B) 1.未诱导BL21上清;2.诱导后BL21上清;3.纯化后的重组蛋白
2.3 碳源对谷氨酸棒状杆菌中苯酚羟化酶相对活性的影响
从表1可以看出,当以葡萄糖为惟一碳源和能源时,谷氨酸棒状杆菌中苯酚羟化酶的相对活性最低;而当以苯酚为惟一碳源和能源时,苯酚羟化酶相对活性较高;以其他不同芳香族化合物作为碳源时苯酚羟化酶相对活性虽然也较高,但除对甲酚外,其余碳源条件下的苯酚羟化酶相对活性均不如苯酚高。以上结果说明,ncgl2588基因所表达的苯酚羟化酶是一个可诱导酶,并且苯酚及其他多种芳香化合物均可诱导其表达;而ncgl2588基因缺失突变株Δ2588仍然能检测到少量苯酚羟化酶活性,很可能是其他酶对苯酚的非特异性酶活。

表1 以不同碳源培养谷氨酸棒状杆菌RES167时ncgl 2588基因所表达的苯酚羟化酶的活性Table 1 Relative activity of phenol hydroxylase expressed by ncgl 2588 inCorynebacterium glutamicum RES167 cultured with various carbon sources
2.4 ncgl 2588基因进化树的构建与分析
通过对谷氨酸棒状杆菌CorynebacteriumglutamicumRES167基因组序列的分析,认为ncgl2588基因与酵母Trichosporoncutaneum中的苯酚羟化酶基因有很高的相似性,可能是该菌中编码苯酚羟化酶的基因。将已知的苯酚羟化酶基因与ncgl2588基因进行序列比对,构建进化树,以确定苯酚羟化酶基因在各种菌株中的进化关系,结果见图5。从图5可以看出,谷氨酸棒状杆菌的苯酚羟化酶属于第3类别,与酵母T.cutaneum中的苯酚羟化酶序列同源性高达43%,明显高于与细菌PseudomonaspickettiiPKO1中苯酚羟化酶的同源性,说明谷氨酸棒状杆菌的苯酚羟化酶与酵母T.cutaneum中苯酚羟化酶的亲缘关系更为接近。
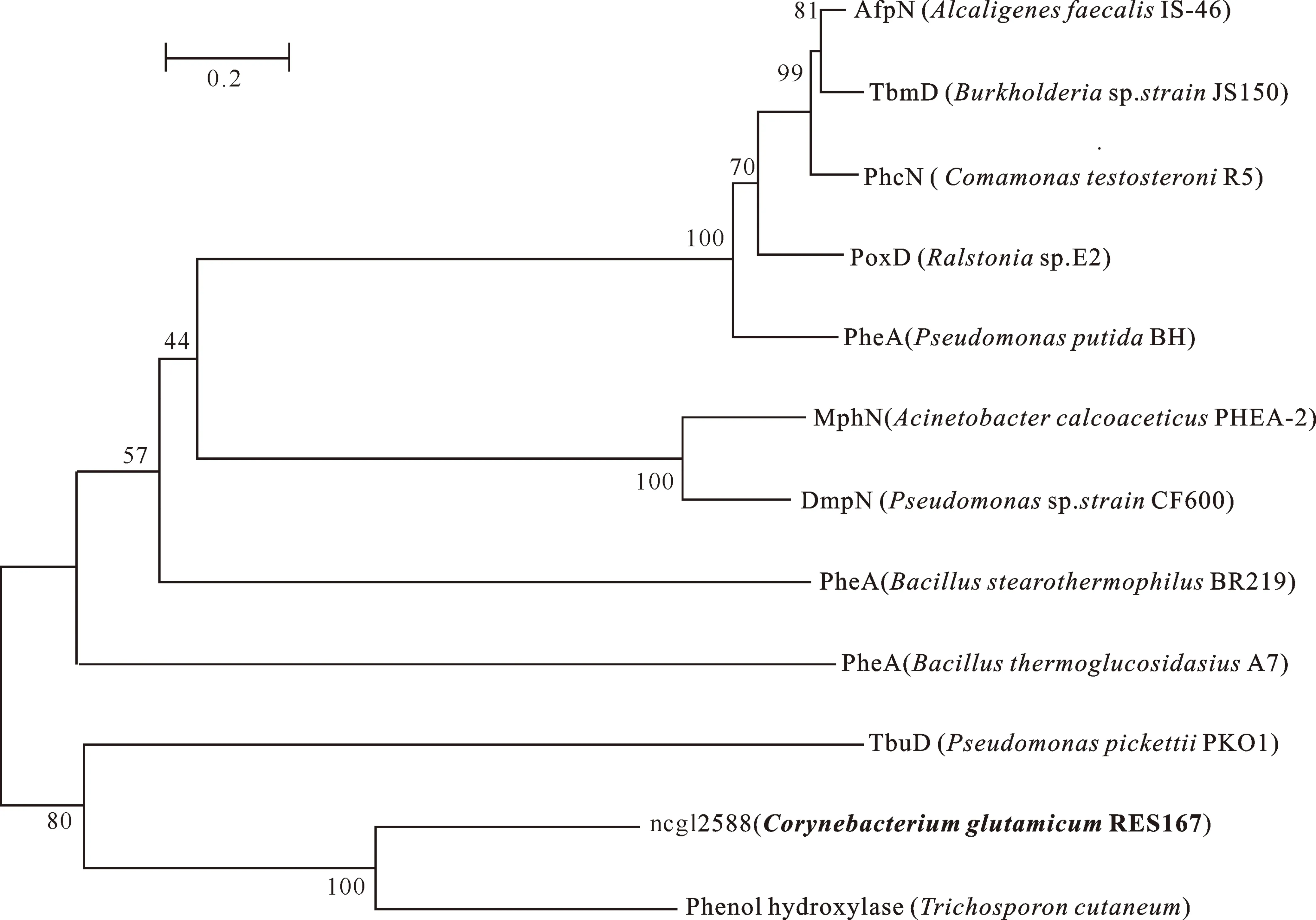
图5 谷氨酸棒状杆菌与其他菌株中苯酚羟化酶的系统发育关系 PhcN (ComamonastestosteroniR5)、PoxD (Ralstoniasp.E2)、AfpN (AlcaligenesfaecalisIS-46)、 MphN (AcinetobactercalcoaceticusPHEA-2)、DmpN (Pseudomonassp.strain CF600)和 PheA (PseudomonasputidaBH) 是这些菌种中苯酚羟化酶最大的组分(此类苯酚羟化酶为多酶复合体); TbmD (Burkholderiasp.strainJS150)是甲苯/苯/氯苯单加氧酶中的一个组分;PheA (BacillusstearothermophilusBR219) 和 PheA(Bacillusthermoglucosidasius)是这2个菌株中最大的亚基;TbuD(PseudomonaspickettiiPKO1)和 Phenol hydroxylase (Trichosporoncutaneum)是这2个菌株中的苯酚羟化酶(此类苯酚羟化酶由单一肽链组成)
Fig.5 Phylogenetic analysis of phenol hydroxylases inCorynebacteriumglutamicumand other strains PhcN (ComamonastestosteroniR5),PoxD (Ralstoniasp.E2),AfpN (AlcaligenesfaecalisIS-46), MphN (AcinetobactercalcoaceticusPHEA-2),DmpN (Pseudomonassp.strain CF600) and PheA (PseudomonasputidaBH) are the largest subunit of phenol hydroxylases.TbmD (Burkholderiasp.strain JS150) is the component of toluene/benzene/chlorobenzene monooxygenase.PheA (BacillusstearothermophilusBR219) and PheA (Bacillusthermoglucosidasius) are the larger subunit of phenol hydroxylase.TbuD (PseudomonaspickettiiPKO1) and Phenol hydroxylase (Trichosporoncutaneum) are the single chain protein of phenol hydroxylase
3 讨 论
本研究通过基因敲除技术,构建了谷氨酸棒状杆菌突变株C.glutamicumΔ2588,并将其对芳香化合物的降解特性与野生型谷氨酸棒状杆菌C.glutamicumRES167进行比较,发现突变株C.glutamicumΔ2588在以苯酚为惟一碳源和能源的培养基中无法生长,说明其已丧失利用苯酚的能力,表明ncgl2588基因与C.glutamicumRES167菌株对苯酚的降解密切相关。另外,本研究构建了表达载体pET28a-ncgl2588,并通过原核表达将其在大肠杆菌BL21(DE3)中成功表达,得到大小为73 ku的目的蛋白,且此蛋白对苯酚具有很高的活性(0.37 mmol/(min·mg)),进一步证明ncgl2588基因在C.glutamicumRES167中编码了苯酚羟化酶。本试验中,以苯酚和对甲酚为惟一碳源培养时,谷氨酸棒状杆菌苯酚羟化酶的活性较高,而在以葡萄糖为惟一碳源时活性最低,说明苯酚羟化酶在谷氨酸棒状杆菌中的表达为不严格的诱导表达型;缺失突变株Δ2588在以葡萄糖为惟一碳源时仍然具有少量的活性,通过Blast蛋白序列同源性分析发现,苯酚羟化酶中并没有与其相同的同源序列,所以突变株Δ2588具有少量活性的原因可能是其他酶对苯酚的非特异性酶活。
通过氨基酸同源性分析与预测,谷氨酸棒状杆菌中的苯酚羟化酶属于硫氧还蛋白超家族,并且很有可能为同源二聚体结构。氨基酸同源比对结果发现,它与酵母T.cutaneum中的苯酚羟化酶相似性最高,为43%;与细菌P.pickettiiPKO1和BacillusthermoglucosidasiusA7中苯酚羟化酶的相似性却很低,这在物种进化上也是一个比较有趣的现象,这种现象可能是基因转移引起的[20]。
虽然微生物广泛分布于环境中, 并经分离得到一些能够降解苯酚等芳香族化合物的微生物[21-22],但是对于微生物在苯酚等芳香族化合物污染生物修复中的作用尚未有足够的重视。谷氨酸棒状杆菌作为1株比较成熟的工业菌株,无论是培养条件还是生物降解作用都非常适合扩大应用。本研究通过克隆、表达及基因敲除分析,确定了ncgl2588基因编码苯酚羟化酶,该酶参与苯酚等芳香族化合物的降解途径,这不仅丰富了具有降解苯酚能力的菌种库,也有望通过后期的基因工程改造,更好地将其应用于苯酚等芳香族化合物的污染治理中。
[1] Gupta S S,Stadler M,Noser C A,et al.Rapid total destruction of chlorophenols by activated hydrogen peroxide [J].Science,2002,296(5566):326-328.
[2] Timmis K N,Pieper D H.Bacteria designed for bioremediation [J].Trends in Biotechnology,1999,17(5):201-204.
[3] Nordlund I,Powlowski J,Shingler V.Complete nucleotide sequence and polypeptide analysis of multicomponent phenol hydroxylase fromPseudomonassp strain CF600 [J].Journal of Bacteriology,1990,172(12):6826-6833.
[4] Takeo M,Maeda Y,Okada H,et al.Molecular cloning and sequencing of the phenol hydroxylase gene fromPseudomonasputidaBH [J].Journal of Fermentation and Bioengineering,1995,79(5):485-488.
[5] Teramoto M,Futamata H,Harayama S,et al.Characterization of a high-affinity phenol hydroxylase fromComamonastestosteroniR5 by gene cloning,and expression inPseudomonasaeruginosaPAO1c [J].Molecular and General Genetics,1999,262(3):552-558.
[6] Kirchner U,Westphal A H,Müller R,et al.Phenol hydroxylase fromBacillusthermoglucosidasiusA7,a two-protein component monooxygenase with a dual role for FAD [J].J Biol Chem,2003,278(48):47545-47553.
[7] Kim I C,Oriel P J.Characterization of theBacillusstearothermophilusBR219 phenol hydroxylase gene [J].Applied and Environmental Microbiology,1995,61(4):1252-1256.
[8] Kukor J J,Olsen R H.Complete nucleotide sequence oftbuD,the gene encoding phenol/cresol hydroxylase fromPseudomonaspickettiiPKO1 and functional analysis of the encoded enzyme [J].Journal of Bacteriology,1992,174(20):6518-6526.
[9] Neujahr H Y,Gaal A.Phenol hydroxylase from yeast purification and properties of the enzyme fromTrichosporoncutaneum[J].Eur J Biochem,1973,35(2):386-400.
[10] Shen X H,Huang Y,Liu S J.Genomic analysis and identification of catabolic pathways for aromatic compounds inCorynebacteriumglutamicum[J].Microbes and Environments,2005,20(3):160-167.
[11] Kalinowski J,Bathe B,Bartels D,et al.The completeCorynebacteriumglutamicumATCC 13032 genome sequence and its impact on the production of l-aspartate-derived amino acids and vitamins [J].Journal of Biotechnology,2003,104(1/2/3):5-25.
[12] Silberbach M,Schäfer M,Hüser A H,et al.Adaptation ofCorynebacteriumglutamicumto ammonium limitation:A global analysis using transcriptome and proteome techniques [J].Applied and Environmental Microbiology,2005,71(5):2391-2402.
[13] Claes W A,Pühler A,Kalinowski J.Identification of twoprpDBCgene clusters inCorynebacteriumglutamicumand their involvement in propionate degradation via the 2-methylcitrate cycle [J].Journal of Bacteriology,2002,184(10):2728-2739.
[14] Schäfer A,Tauch A,Jäger W,et al.Small mobilizable multi-purpose cloning vectors derived from theEscherichiacoliplasmids pK18 and pK19: Selection of defined deletions in the chromosome ofCorynebacteriumglutamicum[J].Gene,1994,145(1):69-73.
[15] Tauch A,Kassing F,Kalinowski J,et al.TheCorynebacteriumxerosiscomposite transposonTn5432 consists of two identical insertion sequences,designatedIS1249,flanking the erythromycin resistance geneermCX[J].Plasmid,1995,34(2):119-131.
[16] Sambrook J,Fritsch E F,Maniatis T.Molecular cloning [M].New York:Cold Spring Harbor Laboratory Press,1989.
[17] Tauch A,Kirchner O,Löffler B,et al.Efficient electrotransformation ofCorynebacteriumdiphtheriaewith a mini-replicon derived from theCorynebacteriumglutamicumplasmid pGA1 [J].Current Microbiology,2002,45(5):362-367.
[18] Laemmli U K.Cleavage of structural proteins during the assembly of the head ofbacteriophageT4 [J].Nature,1970,227(5259):680-685.
[19] Bradford M M.A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding [J].Analytical Biochemistry,1976,72(1):248-254.
[20] Ravi J,Maria C R,James A L.Horizontal gene transfer among genomes:The complexity hypothesis [J].Proc Natl Acad Sci,1999,96:3801-3806.
[21] Sikkema J,de Bont J A.Metabolism of tetralin (1,2,3,4-tetrahydronaphthalene) inCorynebacteriumsp strain C125 [J].Applied and Environmental Microbiology,1993,59(2):567-572.
[22] Itoh N,Yoshida K,Okada K.Isolation and identification of st-yrene-degradingCorynebacteriumstrains,and their styrene metabolism [J].Bioscience,Biotechnology,and Biochemistry,1996,60(11):1826-1830.
Characterization and functional identification of phenol hydroxylase gene inCorynebacteriumglutamicum
YANG Zhi-fang1,XIAO Xiao1,SI Mei-ru1,CHENG Juan-li1,2,SHEN Xi-hui1,WANG Yao1
(1CollegeofLifeSciences,NorthwestA&FUniversity,Yangling,Shaanxi712100,China;2LifeSciencesDepartment,YunchengUniversity,Yuncheng,Shanxi044000,China)
【Objective】 This study aimed to identify the functional and biochemical characterizations of putative phenol hydroxylase encoded byncgl2588 inCorynebacteriumglutamicumRES167. 【Method】 Thencgl2588 deleted mutant ofC.glutamicumRES167 was constructed to determine the difference in phenol degradation compared to wild-type.The recombinant phenol hydroxylase was expressed inE.coliBL21(DE3) by IPTG and purified to determine the activity of catalyzing phenol hydroxylation.【Result】 The obtainedncgl2588 gene deletion mutant ofC.glutamicumRES167 could not grow in the medium that used phenol as sole carbon and energy source,while the wild-type strain grew very well.The pET28a-ncgl2588 plasmid expressed was inE.coliBL21(DE3) as a single band with molecular mass of 73 ku and specific activity to phenol of 0.37 mmol/(min·mg).Phenol hydroxylase reached highest expression when cultured in phenol while it was in low level when cultured in other aromatic compounds.The phenol hydroxylase inC.glutamicumRES167 shared 43% phylogenetic identity with yeastTrichosporoncutaneum,much higher than the identity with bacteria.【Conclusion】 This study confirmed that thencgl2588 gene encoded phenol hydroxylase inC.glutamicumRES167 as an induced enzyme.It could play an important role in phenol degradation.
CorynebacteriumglutamicumRES167;knock out;phenol hydroxylase
时间:2015-11-11 16:16
10.13207/j.cnki.jnwafu.2015.12.027
2014-04-10
国家自然科学基金项目(31170100,31270078);国家“863”高新技术研究与发展计划项目(2013AA102802)
杨志方(1989-),男,山东聊城人,在读硕士,主要从事微生物生理生化研究。E-mail:yangzf1989@126.com
王 瑶(1972-),女,陕西宝鸡人,教授,博士生导师,主要从事微生物生理生化研究。 E-mail:wangyao@nwsuaf.edu.cn
Q936
A
1671-9387(2015)12-0191-08
网络出版地址:http://www.cnki.net/kcms/detail/61.1390.S.20151111.1616.054.html

