基于16S rRNA基因克隆文库技术分析广西富钟水牛瘤胃产甲烷菌组成及多样性
杨承剑 梁 辛 韦升菊 李舒露 梁贤威邹彩霞 杨炳壮 李丽莉
(中国农业科学院广西水牛研究所,农业部(广西)水牛遗传繁育重点实验室,南宁 530001)
广西富钟水牛是我国地方水牛的特色品种之一,2006年被列入《国家级畜禽遗传资源保护名录》[1]。富钟水牛体型较大,具有繁殖力高、成熟早、肉质好、性情温顺、耐粗饲等优良特性[2-3],适合广西壮族自治区东北丘陵地带以低洼水田及黏性土壤旱地的耕作环境条件生长。然而,据2009年统计数据显示,富钟水牛的存栏量仅12.1万头[2]。深入研究富钟水牛的消化生理以及营养特性有助于其优良种质资源的保护。
反刍动物主要依赖瘤胃微生物将结构性碳水化合物降解为挥发性脂肪酸为机体提供能量。不同种类反刍动物其瘤胃微生物结构组成可能存在一定差异,有研究表明不同种类的山羊瘤胃微生物区系不同[4]。产甲烷菌,通常也称为产甲烷古菌,广泛存在于反刍动物胃肠道中[5]。在饲料消化利用过程中,产甲烷菌能够利用各种不同底物,如氢、甲酸、乙酸、甲醇等,还原二氧化碳进而生成甲烷[6]。反刍动物的甲烷生成意味着大量能量损失[7]。目前已有研究显示,多种动物体内均可分离出产甲烷菌[8-9]。部分研究也利用非培养的方式,如16S rRNA基因克隆文库分析等方法研究了绵羊[10]、婆罗门杂交牛[11]、晋南牛[12]瘤胃内产甲烷菌的组成及多样性。更有研究表明,不同肥胖程度的人[13]或者不同品种的猪[14]肠道中的产甲烷菌结构也存在差异。这些研究表明,胃肠道中产甲烷菌多样性可能与物种特异性或与特定功能紧密相关。作为广西壮族自治区特色遗传资源品种之一,富钟水牛瘤胃微生物可能具有其独特的微生物区系,但目前尚未见关于富钟水牛瘤胃微生物方面的研究。因此,本研究利用16S rRNA基因克隆文库技术分析富钟水牛瘤胃产甲烷菌组成及多样性,为进一步研究富钟水牛瘤胃微生物生态系统功能以及品种资源保护提供理论依据。
1 材料与方法
1.1 试验设计
选取3头体况基本一致的5岁左右的健康成年雌性富钟泌乳水牛,饲养于广西壮族自治区水牛研究所水牛场(国家级重点种畜场)。自由采食粗料,粗料为玉米青贮、木薯渣、啤酒渣、新鲜象草;补充适量[5.0 kg/(头·d)]精料。泌乳水牛精料组成及营养水平见表1。每天08:00和16:00定时饲喂。自由饮水。

表1 泌乳水牛精料组成及营养水平(风干基础)Table 1 Composition and nutrient levels of the concentrate for lactating buffalo(air-dry basis)%
试验期30 d,第30天晨饲前通过胃管式瘤胃液采样器采集瘤胃内容物250 mL,装入干净充满二氧化碳厌氧瓶中,置于碎冰块中冷藏迅速带回实验室,保存于-80℃冰箱备用。
1.2 总 DNA 提取
从充分混匀后的250 mL瘤胃样品中用宽口吸头吸取1.5 mL包括瘤胃固、液2相的瘤胃内容物用于总DNA的提取,12 000×g离心5 min,去除上清液,沉淀备用。采用文献[15]所描述的机械破壁及总DNA提取试剂盒相结合的方法提取总DNA。
1.3 PCR扩增及克隆文库构建
产甲烷菌特异性引物Met86F(5'-GCTCAGTAACACGTGG-3')/Met1340R(5'-CGGTGTGTGCAAGGAG-3')[10]扩增 16S rRNA 基因序列。反应体系50μL。反应条件如下:94℃,3 min。94℃,30 s;58℃,30 s;72℃,90 s(40个循环)。72℃,10 min。利用琼脂糖凝胶纯化回收试剂盒(美国Promega)纯化PCR产物。将来自3头动物的等体积PCR产物混合后,按照厂家说明书连接到载体 pMD19-T(日本TaKaRa)上进行克隆,经蓝白斑筛选随机挑取阳性克隆,用引物M13-47(5'-CGCCAGGGTTTTCCCAGTCACGAC-3')和RV-M(5'-GAGCGGATAACAATTTCACACAGG-3')对阳性克隆进行验证,确认PCR片段连接到载体。然后将所有阳性克隆的送检进行测序(上海生工科技有限公司)。
1.4 16S r RNA基因序列分析
用DECIPHER软件去除嵌合体序列[16]。使用Ribosomal Database Project(RDP)Release 11中的分类(classifier)程序对所有序列进行分类分析[17]。用 Mothur 软 件 划 分 分 类 操 作 单 元(OTU),将相似性>97%的序列视为同一个OTU[18]。选取每个 OTU代表序列,利用 Blast程序在GenBank中搜索相似性最高序列。用Clustal X Version 1.83软件进行多重比对,通过Molecular Evolutionary Genetics Analysis(MEGA)4.0 软件计算出序列的系统进化距离,采用Neighbor-Joining方法构建系统发育进化树,1 000次随机抽样,计算自举值(bootstrap)以评估系统发育树的置信度。本研究所获得的序列在DDBJ数据库里的登录号为:AB905820-AB905919。
2 结 果
2.1 16S r RNA基因克隆文库分析
从建立的富钟水牛瘤胃产甲烷菌16S rRNA基因克隆文库中,随机挑选100个阳性克隆进行测序,其中5个可能为嵌合体序列。剩余的95个序列经Mothur软件分析,只有93个独特序列可用于下一步分析。分类分析结果表明,93个序列均属于古细菌域(Archaea)序列,可分为甲烷杆菌目(Methanobacteriales)、热原体目(Thermoplasmatales)、除硫球菌目(Desulfurococcales)3个目,其中Methanobacteriales又可分为甲烷短杆菌属(Methanobrevibacter)、甲烷杆菌属(Methanobacterium)、甲烷球形菌属(Methanosphaera)、未培养的甲烷杆菌(unclassified_Methanobacteriaceae)4个属。以97%序列相似性为阈值,用Mothur软件对剩余的93个序列进行分析,93个序列可分为39个OTU(表2)。Mothur软件计算表明克隆文库库容值为78.49%,满足分析要求。
经Blast与GenBank中序列比对,结果表明,93个序列中60个序列(15个OTU)与已培养细菌16S rRNA序列相似性≥97%,占总序列的64.5%。这些已培养细菌包括:Methanobrevibacter millerae(12 个 OTU,占总序列 55.9%)、Methanobrevibacter smithii(1 个 OTU,占总序列的 1.1%)、Methanobrevibacter ruminantium(1个 OTU,占总序列的5.4%)、Methanobrevibacter boviskoreani(1 个OTU,占总序列的 2.1%)。32 个序列(23个OTU)与已培养菌16S rRNA序列相似性处于90%~(<97%),包括 Methanobrevibacter millerae(5 个 OTU,占总序列的 8.6%)、Methanobrevibacter smithii(9 个 OTU,占总序列的 11.8%)、Methanobrevibacter ruminantium(2个OTU,占总序列的2.1%)、Methanobrevibacter gottschalkii(1 个 OTU,占总序列的 2.1%)、Methanobrevibacter olleyae(2个 OTU,占总序列的 3.2%)、Methanobrevibacter boviskoreani(1 个 OTU,占总序列的 1.1%)、Methanosphaera stadtmanae(2个 OTU,占总序列的4.3%)、Methanobacterium alcaliphilum(1 个 OTU,占总序列的 1.1%)。仅有 1个序列与 Methanomassiliicoccus luminyensis相似性<90%(1个OTU,占总序列的 1.1%)。

表2 富钟水牛瘤胃产甲烷菌16S r RNA基因序列分析Table 2 Sequence analysis of methanogens 16S rRNA gene from the rumen of Fuzhong buffaloes

续表2
2.2 16S r RNA基因系统进化分析
以 Thermococcus aggregans(X99556.1)和 P.fumartus(X99555.1)作为外群(outgroup),将 39个OTU代表序列与11个最相似已知序列构建系统发育树(图1)。进化树分析结果表明,与已知序列Methanomassiliicoccus luminyensis strain B10相似性为89%的代表序列D40在进化树中明显区别于其他分支,属于Thermoplasmatales,其余序列都属于 Methanobacteriales。Methanobacteriales内又分为2个大的分支,其中序列 29、D103、D86、D8、D91、D6、12、15-2、14、21、D35、5、A1、12-3、15、5-2、7共17个代表序列聚集在同一个分支上,在系统发育距离上与Methanobacteriales中任何已知相似序列都相隔较远,因此它们可能代表Methanobacteriales中新的属或种。Methanobacteriales中的其余序列都处于一个分支上,以Methanobrevibacter序列为优势序列,Methanobrevibacter序列之间的遗传距离较远,如代表序列11、D66、12-2、1、D39、D64、D101、D33、D38、9,这意味着它们可能是 Methanobrevibacter中新种;仅有代表序列D53在系统发育树中与Methanosphaera stadtmanae在进化树中距离较近,属于Methanosphaera。
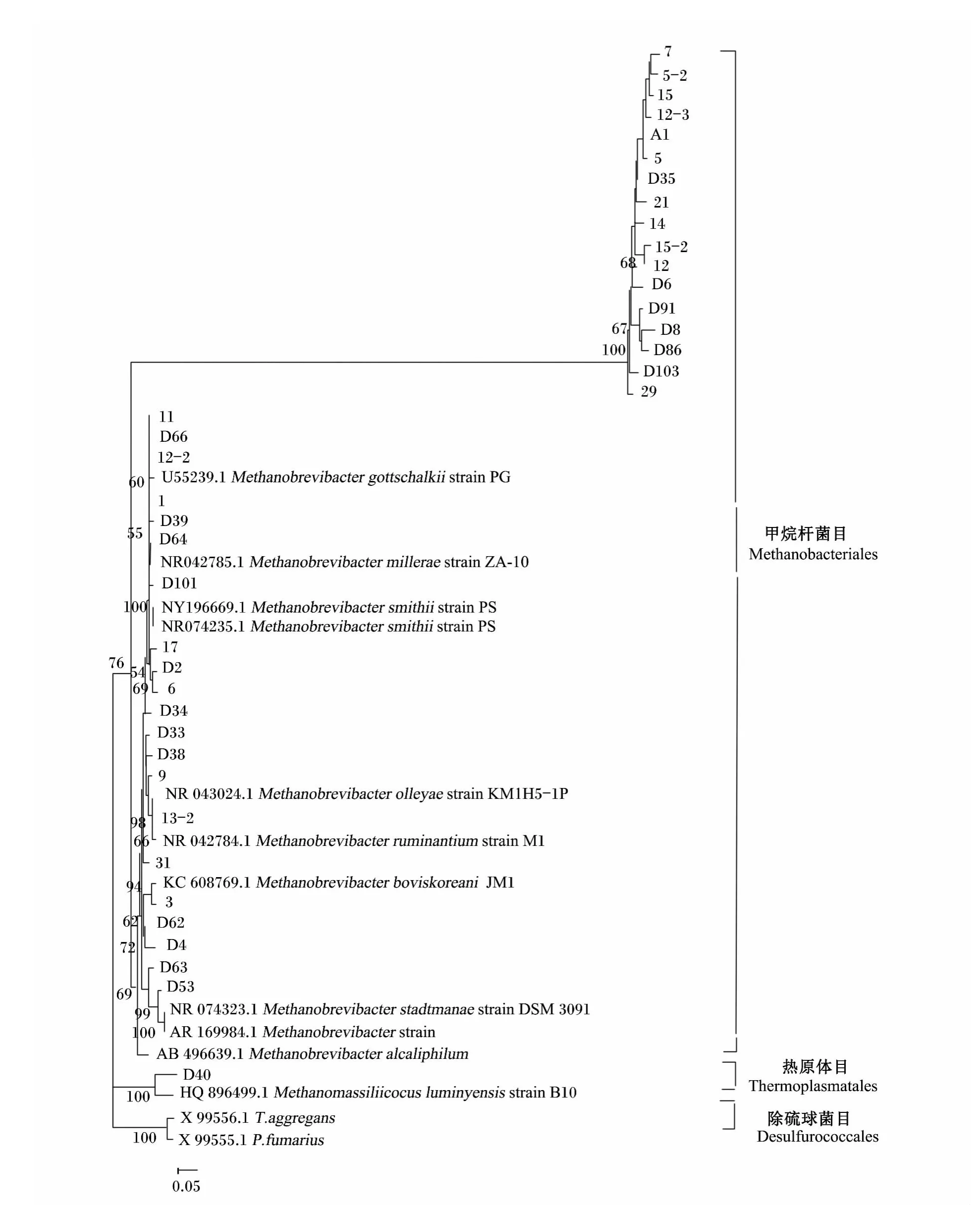
图1 富钟水牛瘤胃产甲烷菌16S r RNA基因序列系统发育分析Fig.1 Phylogenetic analysis of 16S rRNA gene sequences of methanogens from the rumen of Fuzhong buffaloes
3 讨 论
甲烷是反刍动物消化过程中所产生的副产物,它的释放导致能量损失以及温室气体的增加。因而,深入了解水牛瘤胃产甲烷菌的菌群结构及其多样性有助于了解水牛瘤胃甲烷生成的微生物学机理,能够为寻找合适的水牛瘤胃甲烷调控措施提供理论依据。目前已有许多关于绵羊[19-20]、黄牛[11,21]以及水牛[22-24]瘤胃微生物菌群结构组成的报道,其中水牛方面的研究以印度水牛为主。关于我国水牛瘤胃微生物菌群结构组成,尤其是产甲烷菌多样性方面的研究仍尚少。本研究利用16S rRNA基因克隆文库技术分析富钟水牛瘤胃中的产甲烷菌组成及多样性,为进一步研究富钟水牛瘤胃微生物生态系统功能以及品种资源保护提供理论依据。
品种、饲粮以及地理位置之间的复杂互作对于水牛瘤胃微生物区系具有明显作用。如印度的2项研究表明,摩拉水牛瘤胃内的产甲烷菌94.4%~ 97.1% 都 属 于 Methanomicrobium mobile[22,25]。而巴西有研究报道,不考虑饲粮的差异,地中海水牛瘤胃内 91.5%的产甲烷菌属于Methanobrevibacter[26]。本研究通过 16S rRNA 基因克隆文库分析表明,富钟水牛瘤胃内产甲烷菌以Methanobacteriales中 Methanobrevibacter为主,这与国内外许多在其他反刍动物上的研究结果相一致[19,27]。委内瑞拉的绵羊以及澳大利亚袋鼠胃肠中98%以上的序列与Methanobrevibacter gottschalkii 相 似[19,28]。 在 牦 牛 中,与 Methanobrevibacter strain NT7高度相似的瘤胃内的许多产甲烷菌目前仍难以人工分离[29]。本研究的系统进化树分析结果也表明,富钟水牛瘤胃内存在许多未知的Methanobrevibacter。关于不同品种水牛瘤胃内产甲烷菌区系存在显著差异的机理还有待深入研究。Methanobrevibacter能够利用二氧化碳和氢或者甲酸生成甲烷,但是不利用甲醇和甲胺生成甲烷[30]。研究表明,从黄牛瘤胃中分离到的Methanobrevibacter millerae能够利用甲酸进行生长繁殖[31]。Methanobrevibacter smithii PS 可利用二氧化碳和氢或者甲酸生成甲烷[32]。有研究表明,反刍动物体内的Methanobrevibacter smithii可影响饲粮中的多糖成分的利用效率[11]。Facey 等[33]发现Methanosphaera stadtmanae(一种甲醇利用菌)是猩猩胃肠道中的优势产甲烷菌。本研究也发现,在富钟水牛瘤胃内存在序列与Methanosphaera stadtmanae高度相似的产甲烷菌,但是只占总序列数的4.3%。低浓度的Methanosphaera stadtmanae可能是由于Methanobrevibacter占优势或者由于饲料发酵过程中产生的少量甲醇的缘故,这需要进一步研究。
St-Pierre 等[34]提议将与 Methanobrevibacter相似的产甲烷菌序列分为2大类:与Methanobrevibacter smithii(S)、Methanobrevibacter gottschalkii(G)、Methanobrevibacter millerae(M)和 Methanobrevibacter thaurei(T)聚集在一块,统称为SGMT簇;与 Methanobrevibacter ruminantium(R)及Methanobrevibacter olleyae(O)聚集在一起的称为RO簇。已有研究表明,当动物种类不同或饲粮条件不同时,SGMT和RO簇序列之间的分布规律存在差异[34]。研究表明,在饲喂同等饲粮条件下,荷斯坦奶牛瘤胃中的SGMT簇序列的比例要高于娟姗奶牛[21]。在本研究中,SGMT簇序列和RO簇序列所占总序列数比例分别为79.6%、10.8%,即在富钟水牛瘤胃中SGMT簇序列占大多数。关于SGMT簇和RO簇序列在不同动物种类及饲养条件下的分布特点是否可以用于估测瘤胃甲烷生成,进而应用于调控瘤胃甲烷生成,还有待进一步研究。
4 结论
富钟水牛瘤胃产甲烷菌以Methanobacteriales为优势菌群,其中有许多未知的产甲烷菌需进一步分离培养并对其功能进行分析。
[1] 中华人民共和国农业部.国家级畜禽遗传资源保护名录[J].湖北畜牧兽医,2007(1):7.
[2] 苏家联.广西地方畜禽遗传资源现状及保护对策[J].中国畜牧杂志,2009,45(24):13-17.
[3] 陈英姿,何衍琦.富钟水牛[J].广西农学报,2008,23(2):58-61.
[4] SHI PJ,MENG K,ZHOU Z G,et al.The host species affects the microbial community in the goat rumen[J].Letters in Applied Microbiology,2008,46(1):132-135.
[5] HACKSTEIN J H P,VAN ALEN T A.Fecal methanogens and vertebrate evolution[J].Evolution,1996,50(2):559-572.
[6] SAMUEL B S,GORDON JI.A humanized gnotobiotic mouse model of host-archaeal-bacterial mutualism[J].Proceedings of the National Academy of Sciences of the United States of America,2006,103(26):10011-10016.
[7] JOHNSON K A,JOHNSON D E.Methane emissions from cattle[J].Journal of Animal Science,1995,73(8):2483-2492.
[8] JARVIS G N,STRÖMPL C,BURGESS D M,et al.I-solation and identification of ruminal methanogens from grazing cattle[J].Current Microbiology,2000,40(5):327-332.
[9] MILLER T L,WOLIN M J,KUSEL E A.Isolation and characterization of methanogens from animal feces[J].Systematic and Applied Microbiology,1986,8(3):234-238.
[10] WRIGHT A D G,WILLIAMSA J,WINDER B,et al.Molecular diversity of rumen methanogens from sheep in Western Australia[J].Applied and Environmental Microbiology,2004,70(3):1263-1270.
[11] DENMAN S E,TOMKINS N W,MCSWEENEY C S.Quantitation and diversity analysis of ruminal methanogenic populations in response to the antimethanogenic compound bromochloromethane[J].FEMS Microbiology Ecology,2007,62(3):313-322.
[12] PEI C X,MAO SY,CHENG Y F,et al.Diversity,abundance and novel 16SrRNA gene sequences of methanogens in rumen liquid,solid and epithelium fractions of Jinnan cattle[J].Animal,2010,4(1):20-29.
[13] DIBAISE J K,ZHANG H,CROWELL M D,et al.Gut microbiota and its possible relationship with obesity[J].Mayo Clinic Proceedings,2008,83(4):460-469.
[14] LUO Y H,SU Y,WRIGHT A D G,et al.Lean breed Landrace pigs harbor fecal methanogens at higher diversity and density than obese breed Erhualian pigs[J].Archaea,2012,2012:605289.
[15] DENMAN S E,MCSWEENEY C S.Development of a real-time PCRassay for monitoring anaerobic fungal and cellulolytic bacterial populations within the rumen[J].FEMSMicrobiology Ecology,2006,58(3):572-582.
[16] WRIGHT E S,YILMAZ L S,NOGUERA D R.Decipher,a search-based approach to chimera identification for 16S rRNA sequences[J].Applied and Environmental Microbiology,2012,78(3):717-725.
[17] COLE JR,WANG Q,CARDENASE,et al.The ribosomal database project:improved alignments and new tools for rRNA analysis[J].Nucleic Acids Research,2009,37:D141-D145.
[18] SCHLOSS P D,WESTCOTT SL,RYABIN T,et al.Introducing mothur:open-source,platform-independent,community-supported software for describing and comparing microbial communities[J].Applied and Environmental Microbiology,2009,75(23):7537-7541.
[19] WRIGHT A D G,MA X L,OBISPO N E.Methanobrevibacter phylotypes are the dominant methanogens in sheep from Venezuela[J].Microbial Ecology,2008,56(2):390-394.
[20] WRIGHT A D G,TOOVEY A F,PIMM C L.Molecular identification of methanogenic archaea from sheep in Queensland,Australia reveal more uncultured novel archaea[J].Anaerobe,2006,12(3):134-139.
[21] KING E E,SMITH R P,ST-PIERRE B,et al.Differences in the rumen methanogen populations of lactating Jersey and Holstein dairy cows under the same diet regimen[J].Applied and Environmental Microbiology,2011,77(16):5682-5687.
[22] CHAUDHARY P P,SIROHI S K.Dominance of Methanomicrobium phylotype in methanogen population present in Murrah buffaloes(Bubalus bubalis)[J].Letters in Applied Microbiology,2009,49(2):274-277.
[23] TAN H Y,SIEO C C,LEE C M,et al.Diversity of bovine rumen methanogens in vitro in the presence of condensed tannins,as determined by sequence analysis of 16SrRNA gene library[J].The Journal of Microbiology,2011,49(3):492-498.
[24] TATSUOKA N,MOHAMMED N,MITSUMORI M,et al.Phylogenetic analysis of methyl coenzyme-M reductase detected from the bovine rumen[J].Letters in Applied Microbiology,2004,39(3):257-260.
[25] SINGH K M,TRIPATHI A K,PANDYA P R,et al.Methanogen diversity in the rumen of Indian Surti buffalo(Bubalus bubalis),assessed by 16SrDNA analysis[J].Research in Veterinary Science,2012,92(3):451-455.
[26] FRANZOLIN R,ST-PIERRE B,NORTHWOOD K,et al.Analysis of rumen methanogen diversity in water buffaloes(Bubalus bubalis)under three different diets[J/OL].Microbial Ecology,2012,64(1):131-139.doi:10.1007/s00248-012-0007-0.
[27] HOOK SE,WRIGHT A D G,MCBRIDE B W.Methanogens:methane producers of the rumen and mitigation strategies[J/OL].Archaea,2010,2010:945785.doi:10.1155/2010/945785.
[28] EVANS P N,HINDS L A,SLY L I,et al.Community composition and density of methanogens in the foregut of the Tammar wallaby(Macropus eugenii)[J].Applied and Environmental Microbiology,2009,75(8):2598-2602.
[29] JANSSEN P H,KIRS M.Structure of the archaeal community of the rumen[J].Applied and Environmental Microbiology,2008,74(12):3619-3625.
[30] PAUL K,NONOH J O,MIKULSKI L,et al."Methanoplasmatales,"Thermoplasmatales-related archaea in termite guts and other environments,are the seventh order of methanogens[J].Applied and Environmental Microbiology,2012,78(23):8245-8253.
[31] REA S,BOWMAN JP,POPOVSKI S,et al.Methanobrevibacter millerae sp.nov.and Methanobrevibacter olleyae sp.nov.,methanogens from the ovine and bovine rumen that can utilize formate for growth[J].International Journal of Systematic and Evolutionary Microbiology,2007,57(Pt.3):450-456.
[32] SAMUEL B S,HANSEN E E,MANCHESTER J K,et al.Genomic and metabolic adaptations of Methanobrevibacter smithii to the human gut[J].Proceedings of the National Academy of Sciences of the United States of America,2007,104(25):10643-10648.
[33] FACEY H V,NORTHWOOD K S,WRIGHT A D G.Molecular diversity of methanogens in fecal samples from captive Sumatran orangutans(Pongo abelii)[J].American Journal of Primatology,2012,74(5):408-413.
[34] ST-PIERRE B,WRIGHT A D G.Molecular analysis of methanogenic archaea in the forestomach of the alpaca(Vicugna pacos)[J/OL].BMC Microbiology,2012,12:1.doi:10.1186/1471-2180-12-1.

