内源ABA的生物途径及对非生物胁迫的响应机制
韩 璐,王晓萍
(哈尔滨师范大学,黑龙江省分子细胞遗传与遗传育种重点实验室)
0 引言
脱落酸(ABA)于20世纪60年代被首次发现,是一种传统的植物激素,在植物生长和发育的一系列生理进程中起到至关重要的作用,诸如促进种子的成熟,种子的休眠,影响种子萌发以及适应非生物胁迫等等[1],即ABA不仅是细胞信号而且还是系统逆境信号,主要调控这些进程的是内源具有活性的ABA,而植物内源ABA含量的水平是由ABA的合成速率和代谢速率共同决定的[2],因此,为了解ABA水平是如何被调控的,研究参与ABA合成和代谢的相关基因就变的非常重要.
1 内源ABA的生物途径
1.1 高等植物ABA的合成途径
已有的研究证实,高等植物脱落酸是由C40类胡萝卜素前体经过一系列转化所得(如图1所示).目前,高等植物的ABA生物合成通路的生化和分子遗传研究已经很透彻.在水稻C40途径早期阶段,八氢番茄红素脱氢酶(OsPDS)、类胡萝卜素异构酶(OsCRTISO)和番茄红素β-羟化酶(β-OsLCY)和ζ-胡萝卜素去饱和酶(Os-ZDS)、等酶类参与类胡萝卜素合成,编码这些酶的基因若发生了突变,突变体中ABA含量较野生型降低,表型体现均为穗发芽(Pre-harvest sprouting,PHS),PHS是一种ABA缺陷表型,这说明这些酶类是ABA的类胡萝卜素前体合成的重要酶[4].中期阶段产物番茄素转化成玉米黄质,这一步骤需要经过环化及羟基化,质体中的玉米黄质在玉米黄素环氧化酶(ZEP)的催化下变成环氧玉米黄素,再进而催化成堇菜黄素,这一步骤在堇菜黄素脱环氧酶-VDE的作用下可逆.水稻中编码玉米黄素环氧化酶的基因是Os-ABA1,Osaba1突变株是一个由Tos17插入产生的OsABA1缺失突变体,表型为萎蔫,检测发现内源ABA含量很低,干旱胁迫下脱落酸的含量也不会升高.这个突变体被证明是玉米黄素的环氧化过程受阻,即不能合成脱落酸[5].堇菜黄质在酶的催化下转化成一类9'-顺式环氧类胡萝卜素如9'-顺式紫黄质和9'-顺式新黄质,这些产物被9'-顺式-环氧类胡萝卜素双加氧酶(NCED)进一步催化成黄质醛[6],在短链乙醇脱氢酶(SDR)催化下和乙醛氧化酶的氧化作用最终形成ABA.针对拟南芥的分子遗传分析证实,在种子萌发和发育阶段的ABA合成过程中,拟南芥9'-顺式-环氧类胡萝卜素双加氧酶(At-NCED)起到重要作用,9'-顺式-环氧类胡萝卜素双加氧酶家族在拟南芥中有5个家族成员,每个成员在调控过程中的功能都不完全相同[7],AtNCED6和 AtNCED9在种子的发育和萌发阶段ABA的合成起主要作用[8],而AtNCED3是参与高渗条件下种子萌发过程[9].

图1 ABA生物合成途径[5]
1.2 高等植物ABA的代谢途径
与ABA合成途径的研究相比,人们对ABA代谢途径相关酶和他们的调控方式的研究仍不全面[1].目前研究认为ABA的代谢在高等植物中主要有两种方式:一种是糖结合失活途径;另一种是ABA甲基羟基化代谢途径.
糖结合导致ABA失活的产物是脱落酸葡萄糖酯(ABA—Glucosylester,ABA-GE),多以稳定的形式存在于植物的酸性液泡,ABA-GE可在β-葡萄糖苷酶的作用下迅速水解,产物为具有活性、游离的 ABA,以应答环境胁迫[10].糖类代谢的另一作用是参与ABA的合成,已有研究表明可溶性糖类也参与调控植物的发育,生理和代谢过程[11],遗传研究显示葡萄糖和其他糖类作为初级的信号分子影响着种子的萌发和早期种子发育[12-13],糖类伴随着植物激素合成和信号传导共同参与种子萌发的过程,特别是与ABA的联合作用更为显著[14].基因芯片实验结果证实了许多基因的表达受到糖和ABA的共调控作用,包括参与糖类代谢,代谢运输和信号转导的一些基因[15-16].拟南芥中与ABA合成相关的基因如ABA2和NCED3也能被葡萄糖所诱导并参与葡萄糖信号反应[17].ABA缺陷突变体 aba2/gin1和aba3/gin5,ABA不敏感突变体abi4/gin6、abi5和abi8,这些突变体都表现为糖不敏感表型[18],这说明ABA的合成同时也调控葡萄糖信号通路.
高等植物的ABA甲基羟基化途径有三种:9'甲基羟基化、8'甲基羟基化、7'甲基羟基化.产物分别是 9'-OH-ABA、8'-OH-ABA、7'-OH-ABA,这些产物的结构都极不稳定,9'-OH-ABA进一步环化成新红花菜豆酸(neo-PA),8'-OH-ABA发生亲核反应生成红花菜豆酸(PA),PA进一步还原成二氢红花菜豆酸(DPA)或差位异构体 epi-DPA[19-20],其中 7'甲基羟基化途径并不是十分重要,这三种羟化酶都属于细胞色素P450(CYP)家族.高等植物ABA代谢最主要的途径是8'甲基羟基化(如图2所示),产物PA的活性并未完全丧失,但较未8'甲基羟基化的ABA活性要弱很多,而DPA是完全无活性的,若ABA大量降解,植物体内PA和DPA的含量急剧升高.故内源ABA的水平可以通过内源PA和DPA含量的检测来判断.
植物中不同物种编码8'-羟化酶的基因家族成员的数量也不同,如拟南芥中编码此酶的CYP707A成员目前发现四个,玉米中ABA8ox成员有五个,大豆和高粱有三个,小麦钟仅有两个,水稻中有三个可能编码OsABA8ox的基因,命名为OsABA8ox1、2、3.图3所示三种代表性禾本科植物玉米、水稻和高粱的ABA8ox家族基因组结构比对,将同源基因分为一个分支,共三个:ox1、ox2、ox3,结果具有明显的逻辑规律,除ox3中高粱的外显子数较少与另外几个同源基因的外显子数目不符外,另两个分支都有一定的规律性可言:ox2的外显子数都是9;ox1的外显子数是5与ox2相比较少.图4是几种典型的双子叶和单子叶植物ABA8ox家族编码的蛋白序列的系统进化分析.

图2 高等植物ABA的8'羟基化代谢途径[21]
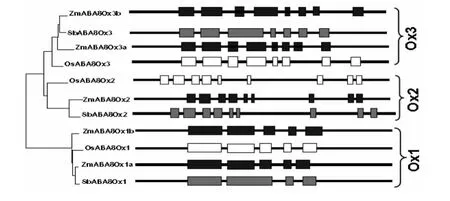
图3 ABA8ox基因家族旁系同源和直系同源基因结构(方块-外显子,直线-内含子)[21]
之前提到可溶性糖类参与种子的萌发和早期种子发育过程,影响ABA的合成,进而影响植物体内ABA含量.植物内源ABA含量不光由合成决定,也取决于代谢的速率.用不同浓度的葡萄糖处理萌发期的水稻种子,荧光定量检测Os-NCED和OsABA8ox家族的mRNA水平,结果发现低浓度的葡萄糖处理对增强OsNCED2表达水平的作用不是很强,仅在高浓度6%葡萄糖处理时显著升高,升高情况与甘露醇处理组一致,推测升高是由于渗透胁迫导致,其他OsNCED的mRNA水平变化不明显;而低浓度外源葡萄糖能明显抑制OsABA8ox基因,尤其是OsABA8ox2和OsABA8ox的表达[22],说明葡萄糖诱导的种子萌发延迟是由于ABA的分解抑制而非ABA的合成增强所介导.去甲二氢愈创木酸(NDGA)是NCED酶的有效抑制剂,可阻碍ABA合成[23];烯唑醇(diniconazole)是ABA8'羟化酶的竞争性抑制物,也是 ABA代谢的有效抑制剂[24].当 ABA的合成被NDGA所抑制,水稻种子萌发无影响,烯唑醇处理抑制ABA的分解后水稻种子萌发受到抑制,表明ABA的分解代谢在种子萌发调节中起主要作用.ABA的合成和代谢有时候会共同作用于植物生理发育和响应胁迫的过程.如水稻在淹水胁迫下,为了伸长茎秆内源ABA的水平会下降,在淹水1 h后OsABA8ox1的mRNA水平升高,而OsZEP和OsNCEDs等合成基因的表达在淹水1~2 h后下降,乙烯及其前体1-氨基环丙烷-1-羧酸(ACC)可诱导水稻OsABA8ox1的表达,而乙烯活性抑制剂可抑制淹水对OsABA8ox1的诱导,OsABA8ox1的表达水平与ABA含量负相关,这说明水稻处于淹水条件下ABA含量降低一部分是由乙烯诱导的OsABA8ox1的所调控,一部分是受不依赖于乙烯的ABA合成基因的表达抑制调控.OsABA8ox1-GFP融合表达后,定位在洋葱表皮细胞的内质网上,证明ABA8ox1 为膜蛋白[22].

图4 代表性双子叶和单子叶植物编码ABA8'羟化酶基因的蛋白系统进化树[21]
2 ABA对非生物胁迫的响应机制
高等植物处于逆境胁迫的条件下,内源ABA的含量会增加,产生诸如溶质外流间接导致保卫细胞气孔关闭等一系列响应胁迫变化,研究表明,ABA对旱胁迫和盐逆境的响应最为显著.胁迫下的植物的变化在细胞水平的体现是:膨压的下降,溶质含量的变化,膜结构的变化[25].例如,在响应干旱时,细胞为了避免失水会增强耐受脱水性,使胞内的膨压维持平衡状态[26];为了抵抗胁迫,形态学性状和发育也会发生改变,如茎根的伸长或生长抑制、叶片的厚度和卷曲度都能使植物适应环境的改变[27];生理生化反应的改变主要体现在有机酸的累积,光合作用缓慢如气孔的关闭和糖代谢途径的改变.干旱胁迫发生后,体内会产生渗透保护剂,溶解和渗透物也会发生聚集,逆境消除后这些物质会随之降解[28].高等植物在应答非生物逆境时在基因调控的分子水平上也会发生变化,近几十年利用基因组学、蛋白质组学和基因表达分析等手段,寻找到很多与非生物胁迫相关的基因,这些基因或者蛋白受到干旱、高盐或者低温的诱导.基因芯片数据表明某些基因会同时受干旱和高盐的诱导,推测这几种非生物胁迫之间存在信号交叉途径[29].响应非生物胁迫的基因同时也受到胁迫激素的调控,如ABA含量会影响这些基因的表达水平.对ABA不敏感突变株或ABA缺陷突变体的研究发现,非生物胁迫能诱导一部分基因的表达,而另一些本应被胁迫诱导表达的基因却无变化,我们就此推测胁迫应答基因的表达有两种调控体系:ABA依赖型和ABA非依赖型(如图5所示),为阐述调控机制,基因相关转录因子及其相应顺式作用原件的研究是必不可少的.

图5 拟南芥应答低温和脱水逆境的基因表达调控网络[30]
2.1 依赖ABA的胁迫应答
基因可以受到ABA的调控是因为在基因的启动子存在相对保守的顺式作用元件—ABRE(ABA-responsive element,PyACGTGGC),这个元件是在小麦调控晚期胚胎发育的EM基因[31]和水稻脱水组织及成熟种子有表达的RAB16[32]中首次发现的,ABA对基因的调控就最主要是通过ABRE.启动子上的G-box元件也可以受ABA的调控,并且通过启动子缺失克隆分析,G-box可以调和不同的启动子区域受到各自相应的刺激.启动子转录激活过程的发生除G-box外至少还需要一个顺式作用元件.对于ABA诱导的转录来说一个拷贝的ABRE元件是远远不够的,ABRE必须和偶联元件-CE组成ABA-敏感复合体,研究证实小麦中,HVA1和HVA22基因就是由 ABRE和 CE1、CE2复合体调控的[33].有些蛋白可以结合 ABRE(ABRE-binding factors)也称作AREB/ABF,这个蛋白的转录激活剂功能是通过原生质体的瞬时表达研究发现的[34],大部分AREB亚蛋白都参与营养组织和根部的 ABA诱导的信号转导途径[35].有些ABA依赖型信号转导过程,基因的启动子区域可以不含ABRE,需要一个或者多个转录因子的结合激活下游基因的调控.拟南芥的RD22基因被MYB和MYC转录因子共同识别,激活基因的表达,MYB和MYC大量上调表达的植株是ABA敏感型,导致渗透胁迫的耐受性增强[36].
2.2 ABA非依赖型传导途径
某些逆境响应基因对ABA并不敏感,表达水平也不完全受ABA的调控.对这些基因的启动子区域利用缺失克隆和碱基突变的技术研究发现,存在着一段保守区域,这段9bp的碱基序列可应答逆境胁迫,命名为DRE(dehydration responsive element).在ABA依赖型的信号途径中ABRE行使功能时必须要存在多个拷贝,而DRE与之不同,单拷贝并且不需要偶联原件的协助作用就能调控下游基因的表达.还有类似DRE的作用元件,他们序列的相似性很高[37],如CRT和LTRE,转录因子同样是结合于这些顺式元件上,AP2/ERF家族就是此类转录因子,家族蛋白成员很多,能激活调控许多逆境应答基因.有趣的是,研究发现同一个转录因子家族的蛋白可能响应各自不同的非生物胁迫,例如在对拟南芥研究中发现DREB1/CBF基因受冷处理诱导,但对干旱和高盐胁迫不敏感;相反的是DREB2基因受干旱和高盐胁迫的诱导而不被低温诱导[38].
3 结束语
ABA与许多逆境反应有关,逆境响应基因的表达和调控.植物在应对环境逆境时,植物体内ABA含量上升,特别是在干旱逆境和盐胁迫时.因此学习ABA水平的调节机制对于理解植物抗逆反应非常重要.正如上所描述,植物中ABA的合成和分解酶类对植物耐逆性和种子休眠非常重要.这说明,我们可以利用一些与ABA合成或者代谢相关的酶来提高植物耐逆性,这些相关基因可以用来调节内源ABA水平.如,过量表达ABA的合成途径的关键基因或者抑制ABA代谢途径的关键酶的合成,这些方法都可能导致内源ABA的含量升高,体现在较强的耐逆性.
[1] Nambara E,Marion-Poll A.Annual Review of Plant Biology,2005,56:165-185.
[2] Sawada Y,Aoki M,Nakaminami K,et al.Phytochromeand gibberellin-mediated regulation of abscisic acid metabolism during germination of photoblastic lettuce seeds.Plant Physiol,2008;146:1386-1396.
[3] Marion-Poll A,et al.Abcisic acid synthesis,metabolism and signal transduction.In Plant Hormone Signaling,Annual Plant Reviews,2006:1-35.
[4] Fang J,Chu C.Abscisic acid and the pre-harvest sprouting in cereals.Plant Signal Behav,2008:1046-1048.
[5] Agrawal G K,Yamazaki M,Kobayashi M,et al.Screening of the rice viviparous mutants generated by endogenous retrotransposon Tos17 insertion.Tagging of a zeaxanthin epoxidase gene and a novel OsTATC gene.Plant Physiol,2001,125:1248–1257.
[6] Aleksandra Wasilewska,Florina Vlad,Caroline Sirichandra,et al,An Update on Abscisic Acid Signaling in Plants.Mol Plant,2008,1(2):198-217.
[7] Seo M,Aoki H,Koiwai H,et al.Comparative studies on the Arabidopsis aldehyde oxidase(AAO)gene family revealed a major role of AAO3 in ABA biosynthesis in seeds.Plant Cell Physiol,2004,45:1694-1703.
[8] Lefebvre V,North H,Frey A,et al.Marion-Poll A Functional analysis of Arabidopsis NCED6 and NCED9 genes indicates that ABA synthesized in the endosperm is involved in the induction of seed dormancy.Plant J,2006,45:309-319.
[9] Ruggiero B,Koiwa H,Manabe Y,et al.Uncoupling the effects of abscisic acid on plant growth and water relations:analysis of sto1/nced3,an abscisic acid-deficient but salttolerant mutant in Arabidopsis.Plant Physiol,2004,136:3134-3147.
[10] Lee K H,Piao H L,Kim H Y.Activation of glucosidase via stress-induced polymerization rapidly increases active pools of abscisic acid,2006.
[11] Koch K E.Carbohydrate-modulated gene expression in plants.Annu Rev Plant Physiol Plant Mol Biol,1996,47:509-540.
[12] Gibson S .Sugar and phytohormone response pathways:navigating a signalling network.J Exp Bot,2004,55:253-264.
[13] Dekkers B,Schuurmans J,Smeekens S.Glucose delays seed germination in Arabidopsis thaliana Planta,2004,218:579-588.
[14] Finkelstein R,Reeves W,Ariizumi T,et al.Molecular aspects of seed dormancy.Annu Rev Plant Biol,2008,59:387-415.
[15] Price J,Laxmi A,St Martin S K,et al.Global transcription profiling reveals multiple sugar signal transduction mechanisms in Arabidopsis.Plant Cell,2004,16:2128 – 2150.
[16] Li Y,Lee K K,Walsh S,et al.Establishing glucose-and ABA-regulated transcription networks in Arabidopsis by microarray analysis and promoter classifi cation using a relevance vector machine.Genome Res,2006,16:414-427 .
[17] Chen Y,Ji F,Xie H,et al.The regulator of G protein signaling proteins involved in sugar and abscisic acid signaling in A rabidopsis seed germination.Plant Physiol,2006,140:302-310.
[18] Dekkers B,Schuurmans J W,J A M J,et al.Interaction between sugar and abscisic acid signalling during early seedling development in Arabidopsis.Plant Mol Biol,2008,67:151-167.
[19] Kushiro T,Okamoto M,Nakabayashi K,et al.The Arabidopsis cytochrome P450 CYP707A encodes ABA 8'-hydroxylases:key enzymes in ABA catabolism.EMBO J,2004,23:1647-1656.
[20] SaitoS, HiraiN, MatsumotoC, etal. Arabidopsis CYP707As encode(+)-abscisic acid 8'-hydroxylase,a key enzyme in the oxidative catabolism of abscisic acid.Plant Physiol,2004,134:1439-1449.
[21] Ratnakar Vallabhaneni,Eleanore T.Wurtzel.From epoxycarotenoids to ABA:The role of ABA 80-hydroxylases in drought-stressed maize roots.Archives of Biochemistry and Biophysics,2010,504:112-117.
[22] Zhu Guohui,Ye Nenghui,Zhang Jianhua.Glucose-Induced Delay of Seed Germination in Rice is Mediated by the Suppression of ABA Catabolism Rather Than an Enhancement of ABA Biosynthesis.China Academy of Agricultural Sciences,2013.
[23] Ren H,Gao Z,Chen L,et al.Dynamic analysis of ABA accumulation in relation to the rate of ABA catabolism in maize tissues under water defi cit.J Exp Bot,2007,58:211-219.
[24] Kitahata N,Saito S,Miyazawa,et al.Chemical regulation of abscisic acid catabolism in plants by cytochrome P450 inhibitors.Bioorg Med Chem,2005,13:4491-4498.
[25] Chaves M M,et al.Understanding plant responses to drought-from gene to the whole plant.Funct Plant Biol,2003,30:239-264.
[26] Kramer PJ,et al.Water Relations of Plants.Academic press,1995.
[27] Pathan M S,et al,Moleculer dissection of abiotic stress tolerance in sorghum and rice.In Physiology and biotechnology Integration for Plant Breeding.Edited by Nguyen HT,Blum A Marcel Dekker,Inc,2004.525-569.
[28] Tabaeizadeh Z.Drought-induced responses in plant cells.Int Rev Cytol,1998,182:193-247.
[29] Seki M,et al.Molecular responses to drought,salinity and frost:common and different paths for plant protection.Curr Opin Biotechnol,2003,14:194-199.
[30] Kazuko Yamaguchi-Shinozaki et al.Transcriptional Regulatory Networks in Cellular Responses and Tolerance to Dehydration and Cold Stresses.Plant Biol,2006,57:781-803.
[31] Guiltinan M J,Marcotte W R,Quatrano R S.A plant leucine zipper protein that recognizes an abscisic acid response element.Science,1990,250:267-71.
[32] Mundy J,Yamaguchi-Shinozaki K,Chua N H.Nuclear proteins bind conserved elements in the abscisic acid-responsive promoter of a rice rab gene.Proc Natl Acad Sci USA,1990,87:1406-1410.
[33] Shen Q,Zhang P,Ho T H.Modular nature of abscisic acid(ABA)response complexes:composite promoter units that are necessary and sufficient for ABA induction of gene expression in barley.Plant Cell,1996(8):1107-19.
[34] Uno Y,Furihata T,Abe H,et al.Arabidopsis basic leucinezipper transcription factors involved in an abscisic acid-dependent signal transduction pathway under drought and highsalinity conditions.Proc Natl Acad Sci USA,2000,97:11632-11637.
[35] Choi H,Hong J,Ha J,et al.ABFs,a family of ABA-responsive elementbinding factors.J Biol Chem,2000,275:1723-30.
[36] Abebe T,et al.Tolerance of mannitol-accumulating transgenic wheat to water stress and salinity.Plant Physiol,2003,131:1748-1755.
[37] Jiang C ,et al.Requirement of a CCGAC cis-acting element for cold induction of the BNI15gene from winter Brassica napus.Plant Mol Biol,1996,30:678-684.
[38] Nakashima K,Shinwari Z K,Sakuma Y,et al.Organization and expression of two Arabidopsis DREB2 genes encoding DRE-binding proteins involved in dehydration and high-salinity-responsive gene expression.Plant Mol Biol,2000,42:657-65.

