7-脱氢胆甾醇合成功能模块与底盘细胞的适配性
张莹,张璐,刘夺,丁明珠,周晓,元英进
天津大学化工学院 系统生物工程教育部重点实验室,天津 300072
胆甾相液晶材料在显示、电磁场检测、生物医学等方面均有着广泛的应用。作为胆甾相液晶材料的重要前体物,7-脱氢胆甾醇 (7-DHC)需求量巨大,但其来源和产量问题无法满足当前日益增长的需求[1]。现有的7-DHC生产方法主要是化学全合成,但其工艺流程长、收率低、能耗高、副产物去除过程复杂,且易对环境造成严重的污染[2-3]。已有研究者实现了利用酶促转化合成7-DHC,虽然反应温和,但底物与酶的成本问题成为限制该技术工业化应用的瓶颈[4]。
合成生物技术在合成重要产品、生产清洁能源、维护人类健康等方面均取得了令人瞩目的成果,引起极大关注[5]。利用合成生物技术构建7-DHC人工细胞可以弥补化学法和酶促法的不足,且生产过程绿色清洁,具有很大的优势和广阔的市场前景。目前国内并无利用合成生物技术生产 7-DHC的报道,国际上也多以7-DHC为中间产物,进一步合成雄烯二酮等甾体激素类药物,鲜有以7-DHC为目标产物的相关文献。Christine和 Markus于2006年公开了酵母中生产7-DHC的方法。通过表达来自小鼠和人的C-8甾醇异构酶、C-5甾醇去饱和酶和甾醇C24-还原酶的基因,并将ERG5和ERG6失活且 tHMG1基因被过表达,获得了能合成7-DHC的酵母菌[6]。荷兰的Hohmann Hans-Peter等也通过上调 HMG1的截短基因,失活 ERG5和 ERG6,并引入脊椎动物来源的甾醇 C24-还原酶,实现了在酵母中生产7-DHC[7]。
酿酒酵母中存在固醇类物质的合成途径。作为单细胞真核生物,酿酒酵母不仅拥有转录后修饰功能,基因工程操作简单,便于培养和大规模发酵;且遗传背景较清晰,次生代谢产物较单一,能简化代谢工程产物的纯化[8]。因此酿酒酵母是理想的真核蛋白表达系统之一,适合作为固醇类物质人工合成的底盘细胞。通过对功能模块内启动子、质粒等指标的改变,调节模块与底盘细胞的适配性,从而增加目的产物的产量,是合成生物技术发展中的关键问题[9-10]。以酿酒酵母为底盘细胞生产7-DHC,国外以基因工程手段为主,并没有对合成功能模块与底盘细胞的适配关系进行研究。因此,以合成生物学为基础,注重挖掘功能模块与底盘细胞间的适配关系,降低酵母底盘细胞的代谢压力,提高与功能模块的适配性,从而增强人工合成细胞的鲁棒性,具有很大的研究价值。
7-DHC 在酵母中的生物合成过程见图 1[7,11-12],是以酵母固醇为底物,引入 C-24还原酶将侧链C-24位双键还原,并与内源基因ERG2编码的C-8异构酶、ERG3编码的C-5脱氢酶共同实现。本研究利用合成生物技术,敲除非必需基因固醇 C-24甲基转移酶基因(ERG6)、固醇 C-22脱氢酶基因 (ERG5)以抑制主要竞争路径中麦角固醇的合成[13-15],减少酵母固醇向麦角固醇的转化,增加底物的积累;过表达截短的HMG-CoA还原酶基因 (tHMGR)[16-17]、角鲨烯环氧酶基因 (ERG1)[18-19]等基因元件,提高酵母固醇合成途径中限速步骤的酶表达,以调节内源功能模块与底盘细胞的适配性,优化前体酵母固醇的供给代谢;同时引入人源 C-24还原酶基因 (DHCR24)[20-22]设计并构建7-DHC合成外源功能模块。通过对模块内不同强度启动子、不同底盘细胞等参数的考察,从异源基因表达的密码子优化、合成途径的标准化组装、合成途径的精细调控等方面研究外源功能模块与酿酒酵母底盘细胞的适配关系,得到稳定高产7-DHC的人工细胞。整体研究思路见图2。

图1 7-脱氢胆甾醇在酵母中的生物合成路径[7,11-12]Fig. 1 7-DHC biosynthetic pathway in Saccharomyces cerevisiae[7,11-12].

图2 本研究的整体思路Fig.2 Research purpose in this study.
1 材料与方法
1.1 菌株、质粒、培养基
大肠杆菌Escherichia coli BMDH5α购于博迈德公司。酿酒酵母 Saccharomyces cerevisiae YSG50由美国伊利诺伊大学香槟分校赵惠民教授馈赠[23],W303a、BY4742、YML 和SyBE_001344、SyBE_001358均由本实验室保存。本研究所用酵母细胞见表1。
本研究所用质粒包括大肠杆菌质粒pSB1A2、pUC18,酵母整合型质粒pRS403,酵母复制型质粒pRS425和pYES2均为本实验室保存。
大肠杆菌BMDH5α的保存和培养用LB培养基 (10 g/L胰蛋白胨,5 g/L酵母提取物,10 g/L NaCl;固体培养基添加1.5%琼脂粉),筛选大肠杆菌转化子用含有 100 μg/mL氨苄青霉素的LB培养基。酵母菌保存和培养用YPD培养基 (10 g/L酵母提取物,20 g/L细菌蛋白胨,20 g/L葡萄糖;固体培养基添加2%的琼脂粉),酵母重组菌株筛选用 SC drop-out培养基(6.7 g/L酵母氮源,2 g/L drop-out氨基酸混合物,20 g/L葡萄糖;固体培养基添加2%的琼脂粉)。

表1 本研究所用酵母细胞Table 1 Yeast cells used in this study
1.2 工具酶和试剂
本研究所用限制性内切酶、T4 DNA连接酶、高保真Fast Pfu DNA聚合酶、Taq DNA聚合酶均购自 Fermentas公司;DNA凝胶回收试剂盒、普通质粒提取试剂盒、酵母质粒提取试剂盒均购自天根公司;醋酸锂购自天津苏庄化学试剂厂;酵母氮源、PEG4000购自北京鼎国生物技术有限责任公司;蛋白胨、酵母提取物购自北京奥博星生物技术有限责任公司;葡萄糖、NaCl、KOH购自北方天医化学试剂厂;琼脂粉购自天津英博生化试剂有限公司;甲醇购自康科德公司;正己烷购自天津大茂化学试剂厂;7-脱氢胆甾醇标准品、衍生化试剂MSTFA、吡啶购自Sigma公司。
1.3 基因元件的引物设计、PCR扩增与合成
为实现标准化组装,采用biobrick同尾酶的方法构建质粒[24]。对内源基因tHMGR、启动子TDH3p、终止子 PGK1t进行引物设计,5′端分别引入 EcoRⅠ、XbaⅠ两个限制性酶切位点,3'端分别引入PstⅠ、SpeⅠ两个酶切位点。内源基因 ERG1、启动子 PGK1p、终止子 PGK1t的引物设计,5′端分别引入 SacⅠ、XhoⅠ两个限制性酶切位点,3´端分别引入BamHⅠ、SalⅠ两个酶切位点。使用酵母选择标记LEU实现内源基因ERG5的敲除,根据GenBank中酿酒酵母ERG5上下游的基因序列设计引物,选择标记LEU 的 5′端、3′端分别引入 34 bp、38 bp的同源臂。内源基因、启动子、终止子均以 GenBank中酿酒酵母 S288c的基因组为模板,进行高保真的PCR扩增。选择标记以含有该标记的酵母质粒pRS425为模板,进行高保真的PCR扩增。本研究所用引物见表2。
选择人体来源的C-24还原酶 (DHCR24),在GenBank中查找相应氨基酸序列,根据底盘酵母偏好性进行密码子优化,优化后的核苷酸序列与终止子PGK1t的核苷酸序列相连,并在5′端分别引入 EcoRⅠ、XbaⅠ两个限制性酶切位点,3′端分别引入 PstⅠ、SpeⅠ两个酶切位点,全合成该基因。合成的基因片段通过HindⅢ、BsrGⅠ两个酶切位点,连接于穿梭载体pYES2上。
1.4 功能模块的构建
EcoRⅠ、PstⅠ酶切PCR产物PGK1t,并与相同酶切的质粒 pSB1A2连接,构建质粒pSB1A2-PGK1t后,使用EcoRⅠ、XbaⅠ酶切该质粒。EcoRⅠ、SpeⅠ酶切PCR产物tHMGR,与酶切后的质粒 pSB1A2-PGK1t连接,构建质粒pSB1A2-tHMGR-PGK1t。再次EcoRⅠ、SpeⅠ酶切 PCR产物 TDH3p,与使用 EcoRⅠ、XbaⅠ酶切的质粒pSB1A2-tHMGR-PGK1t连接,构建质粒pSB1A2-TDH3p-tHMGR-PGK1t。EcoRⅠ、SpeⅠ酶切该质粒,得到TDH3p-tHMGR- PGK1t片段,与相同酶切的质粒pRS403连接,构建功能模块pRS403-TDH3p-tHMGR-PGK1t,命名为SyBE_000916。

表2 本研究所用引物Table 2 Primers used in this study
SacⅠ、SalⅠ酶切 PCR产物 PGK1t,并与相同酶切的质粒 pUC18连接,构建质粒pUC18-PGK1t后,使用SacⅠ和XhoⅠ酶切该质粒。SacⅠ和 SalⅠ酶切 PCR产物 ERG1,并与酶切后的质粒 pUC18-PGK1t连接,构建质粒pUC18-ERG1-PGK1t。再次 SacⅠ、SalⅠ酶切PCR产物PGK1p,与使用SacⅠ、XhoⅠ酶切的质粒 pUC18-ERG1-PGK1t连接,构建质粒pUC18-PGK1p-ERG1-PGK1t。XhoⅠ、Hind III酶切该质粒,得到PGK1p-ERG1-PGK1t片段,并与相同酶切的质粒 pRS425连接,构建质粒pRS425-PGK1p-ERG1-PGK1t。ApaⅠ、SalⅠ酶切该质粒,得到PGK1p-ERG1-PGK1t片段,并与ApaⅠ、XhoⅠ酶切后的质粒pRS403-TDH3ptHMGR-PGK1t连接,得到内源功能模块pRS403-PGK1p-ERG1-PGK1t-TDH3p-tHMGRPGK1t,命名为SyBE_000923。
EcoRⅠ、SpeⅠ酶切 PCR产物 TDH3p,与使用 EcoRⅠ、XbaⅠ酶切的质粒 pYES2-DHCR24-PGK1t连接,构建外源功能模块pYES2-TDH3p-DHCR24-PGK1t,命名为SyBE_000937。同理,启动子可替换为PGK1p、TDH1p,得到包含不同启动子的外源功能模块分别命名为SyBE_000938、SyBE_000939。本研究构建的功能模块见表3。
1.5 DNA操作
大肠杆菌的转化参照文献[25]进行。构建的功能模块通过 LiAc法[26]导入不同的底盘酵母细胞内,利用营养缺陷型培养基筛选转化子,提取酵母基因组,通过PCR检测目的片段的存在,从而确定阳性克隆。
1.6 产物提取方法
将筛选出的阳性克隆接种到种子培养基中,30 ℃传代培养,待种子培养至第3代时转接到发酵培养基中,使发酵初始OD600为0.2。30 ℃培养72 h后取出发酵液,1 000 r/min离心2 min,弃上清液留下层沉淀,反复水洗2次后收集细胞,用液氮冷冻并研磨;称取200 mg研磨后细胞加入甲醇配制的1.5 mol/L KOH,在85 ℃恒温水槽中进行皂化反应90 min;然后加入1 mL正己烷,漩涡振荡混合均匀,1 000 r/min离心5 min,取上清液真空冷冻干燥,–40 ℃下保存在 1 mL离心管中备用。向离心管中加入50 μL 吡啶,80 μL N-甲基-N-三甲基硅基三氟乙酰胺 (MSTFA),在30 ℃恒温水槽中进行衍生化反应30 min[27]。

表3 本研究所构建的功能模块Table 3 Functional modules constructed in this study
1.7 GC-TOF/MS检测
对提取的产物进行气相色谱-飞行时间质谱(GC-TOF/MS)分析检测。分析条件如下:仪器为 Waters公司的 GC-TOF/MS (Waters Corp.,USA);硅胶毛细管柱为 30 m×0.25 mm×0.25 μm DB-5MS,J&W Scientific,Folsom。电离方式为电子轰击电离EI+,电子束能量70 eV,离子化电流40 μA。质谱扫描范围在50~800 m/z,离子源温度为250 ℃,进样口温度为260 ℃,氦气(99.999 5%)用作载气,91 kPa恒压模式下操作。柱温在70 ℃保持 2 min,以30 ℃/min的速度升至 250 ℃,然后以 10 ℃/min的速度升到280 ℃,在280 ℃维持15 min;再以5 ℃/min的速度升到290 ℃,在290 ℃维持5 min。
2 结果与分析
本研究对表 1所列酵母细胞进行改造,得到改造后底盘细胞 SyBE_000954 (YML,erg5::LEU2);引入内源功能模块SyBE_000916、SyBE_000923以调节模块与底盘的适配性,获得酵母细胞SyBE_000955 ( SyBE_000954,HIS::SyBE_000916)和 SyBE_000956 (SyBE_000954,HIS:: SyBE_000923)。在此基础上引入人源C-24还原酶构建外源功能模块,得到9株7-DHC人工合成细胞,如表4所示。
2.1 主要竞争途径麦角固醇合成的抑制
在酿酒酵母的代谢中,麦角固醇的生物合成是主要途径。因此为了提高底物酵母固醇浓度,需抑制酵母固醇向麦角固醇的转化。以酿酒酵母ERG6单敲菌YML为酵母细胞,采用同源重组实现内源基因 ERG5的敲除,获得麦角固醇合成途径中的底盘细胞。通过PCR扩增将ERG5基因序列前后的同源臂引入酵母选择标记LEU,PCR产物转化底盘细胞YML,利用营养缺陷互补在 SC drop-out培养基上筛选转化子,提取酵母基因组。以LEU的特异引物进行PCR反应,检验选择标记LEU的存在,从而确定阳性克隆。改造后的底盘细胞命名为SyBE_000954。
对原始菌株 BY4742和改造后底盘细胞SyBE_000954的发酵产物进行 GC-TOF/MS检测。各发酵产物对比见图 3。结果发现,SyBE_000954的麦角固醇 (Egosterol)含量低于 BY4742,而酵母固醇 (Zymosterol)的含量在 SyBE_000954中明显高于 BY4742。这说明内源基因ERG6编码的固醇C-24甲基转移酶、ERG5编码的固醇C-22脱氢酶催化的反应是麦角固醇生物合成途径中的重要限速步骤[14],对它们的敲除可以有效抑制麦角固醇的合成,增加7-DHC的合成底物酵母固醇的积累。

表4 本研究所构建的人工合成细胞Table 4 Artificial synthetic cell obtained in this study
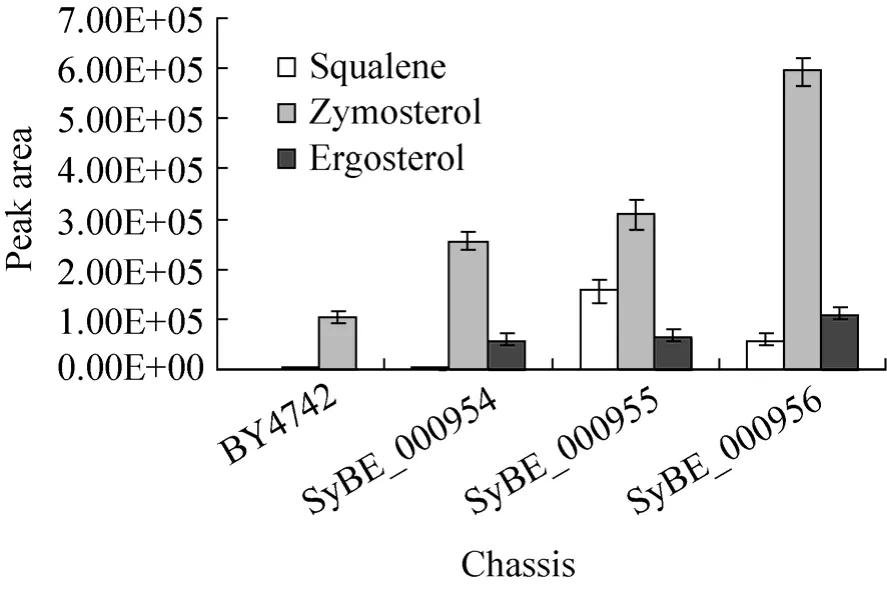
图3 不同酵母细胞发酵产物的对比Fig.3 Comparison of product in different yeast cells.
2.2 内源功能模块与底盘细胞的适配关系
研究表明,HMGR是酵母甲羟戊酸途径中的第一个限速酶,是固醇类物质代谢中的重要调控靶点[28]。HMGR在麦角固醇含量高时会受到反馈抑制,过表达截短的HMGR (tHMGR)可以解除途径中的反馈抑制[29],虽能大量积累前体物鲨烯,却对后续固醇类的产量无较大影响。因此,本工作以鲨烯为节点,将底物酵母固醇合成路径分为上游和下游两部分,研究内源功能模块与底盘细胞的适配性。
2.2.1 上游路径的优化
以功能模块 SyBE_000916转化底盘细胞SyBE_000954,利用营养缺陷互补在SC drop-out培养基上筛选转化子,提取酵母基因组。分别以TDH3p的正向引物和tHMGR的反向引物、tHMGR的正向引物和 PGK1t的反向引物进行PCR反应,检测目的基因tHMGR的存在。改造后的酵母细胞命名为SyBE_000955。
将对照菌株SyBE_000954和改造后酵母细胞 SyBE_000955的发酵产物进行 GC-TOF/MS检测,结果见图3。由图可知,与SyBE_000954相比,SyBE_000955的发酵产物中含有较多鲨烯 (Squalene),而酵母固醇、麦角固醇等固醇类物质的产量并无明显增加。这表明内源基因tHMGR的上调虽然会促进固醇前体物鲨烯的生成,但鲨烯的大量积累导致原有代谢路径的平衡被破坏,造成代谢流不通顺,鲨烯无法继续转化,因此对固醇类物质产量的增加影响较小[17]。
2.2.2 下游路径的优化
为促进鲨烯的代谢以进一步增加底物积累,过表达酵母固醇合成途径中的另一关键基因 ERG1[19]。用功能模块 SyBE_000923转化底盘细胞SyBE_000954,利用营养缺陷互补在SC drop-out培养基上筛选转化子,提取酵母基因组。分别以PGK1p 的正向引物和ERG1的反向引物、ERG1的正向引物和PGK1t的反向引物、TDH3p的正向引物和 tHMGR的反向引物、tHMGR的正向引物和 PGK1t的反向引物进行PCR反应,检测目的基因ERG1和tHMGR的存在。改造后的酵母细胞命名为SyBE_000956。
将对照菌株SyBE_000955和改造后酵母细胞 SyBE_000956的发酵产物进行 GC-TOF/MS检测,结果见图3。由图可知,与SyBE_000955相比,SyBE_000956中鲨烯含量降低,而酵母固醇的含量明显提高。这说明过表达内源基因ERG1编码的角鲨烯环氧酶,可以促进鲨烯向酵母固醇的转化[15];功能模块SyBE_000923的引入,将内源基因tHMGR和ERG1同时上调,会使整个代谢路径达到新的平衡,从而优化底物酵母固醇的供给代谢,加强与底盘细胞的适配关系。
2.3 外源功能模块与酵母细胞的适配
以优化后的SyBE_000956和本实验室已有的 SyBE_001344、 SyBE_001358为酵母细胞,研究外源功能模块与底盘的适配性。将构建的外源功能模块 SyBE_000937、SyBE_000938、SyBE_000939分别转化酵母细胞SyBE_000956、SyBE_001344、SyBE_001358,利用营养缺陷互补在SC drop-out培养基上筛选转化子,提取酵母质粒。以DHCR24的特异引物进行PCR反应,验证 DHCR24的存在,从而确定阳性克隆。得到的 9种 7-脱氢胆甾醇人工合成细胞分别命名为 SyBE_000969、SyBE_000970、SyBE_000971、SyBE_000978、SyBE_000979、SyBE_000980、SyBE_000987、SyBE_000988、SyBE_000989。
对上述 9种人工合成细胞的发酵产物进行GC-TOF/MS检测,其中SyBE_000969发酵产物的气质图见图 4B。从图 4B可以看出,SyBE_000969的发酵产物在保留时间22.62 min检测到的物质与标准品 7-DHC的保留时间(图4A)相同。对该组分进行质谱分析,与标准品7-DHC的碎片峰 (图4A)一致,证明该物质即为7-DHC。经定性分析,另外8种人工合成细胞的发酵产物均为7-DHC。
2.3.1 不同强度启动子的影响

图4 7-DHC标准品、SyBE_000969发酵产物的气质检测图Fig.4 GC-MS identification of 7-DHC and product in SyBE_000969.
研究表明,功能模块中启动子的强度对于目标产物产量的优化具有重要影响。Siavash Partow等发现,在发酵培养基中葡萄糖尚未耗尽时,启动子TDH3p、PGK1p的调控强度大体相同,而当葡萄糖耗尽时PGK1p的调控强度略大于TDH3p[30]。然而Sun的研究表明,TDH3p为强启动子,PGK1p仅为中强型启动子[31]。由此可见,相同启动子在不同培养条件和表达体系中的调控强度不完全一致,这可能跟外源功能模块中启动子与底盘细胞的适配程度、目标产物的差异等因素有关。
本研究根据7-DHC标准曲线,对上述人工合成细胞中的7-DHC进行定量分析,分析结果见图5。可以看出,在相同底盘细胞的情况下,外源功能模块中启动子调控强度的不同会影响外源基因的表达量,其中 TDH3p调控的基因DHCR24表达量最高,PGK1p居中,受TDH1p控制的基因表达量最低。结果说明受TDH3p调控的 7-脱氢胆甾醇合成功能模块与底盘细胞的适配性最佳,PGK1p的调控强度居中,TDH1p调控的功能模块与酵母底盘的适配性最差。
2.3.2 不同酵母细胞的影响
由图 5可以看出,不同酵母细胞与外源功能模块的适配性对7-DHC的合成产生重要的作用。以SyBE_000956为底盘的人工合成细胞,7-DHC含量较高;以 SyBE_001344、SyBE_001358为底盘的人工合成细胞,7-DHC产量相对较低,且两种底盘对产量的影响稍有差异,以 SyBE_001358为底盘的人工细胞,7-DHC产量最低。分析原因,可能是由于SyBE_001344和SyBE_001358两种底盘中麦角固醇合成途径未受抑制,酵母固醇向麦角固醇转化导致。Souza等和Denis Pompon等在分别研究能够生产胆固醇的酵母工程菌时,也都发现敲除了内源基因ERG6和ERG5的酵母菌株能够更稳定的合成胆固醇[15,22]。此外,由于SyBE_001344和SyBE_001358两种底盘过表达了内源基因ERG20,使FPP产量提高并向萜类物质GGPP方向转化,不利于底物酵母固醇的积累。Zhou等在萜类合成的研究中便发现,内源基因ERG20与BTS1的融合表达,可以有效提高FPP由GGPP向次丹参二烯的转化[32]。这也是造成以 SyBE_001344、SyBE_001358为底盘的人工合成细胞中7-DHC产量较低的原因。但与 3种底盘细胞适配性最强的功能模块均为启动子TDH3p所调控,体现出功能模块与底盘细胞适配关系的规律性。
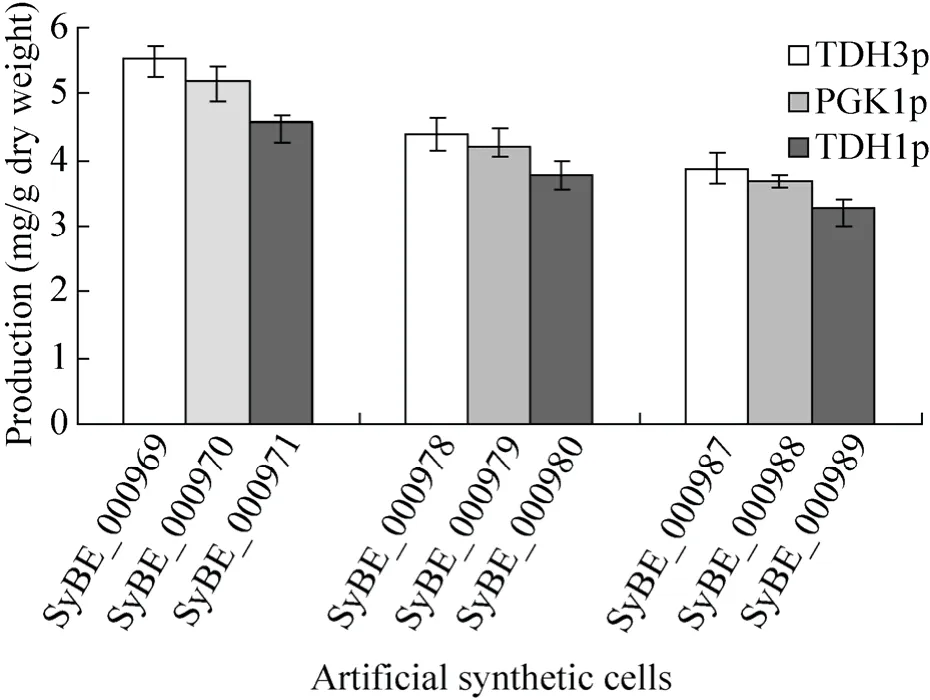
图5 9种人工合成细胞的7-DHC产量Fig. 5 7-DHC production of different artificial synthetic cells.
3 结论
在底盘细胞相同的情况下,外源功能模块中启动子的调控强度会影响外源基因的表达。与启动子PGK1p、TDH1p相比,TDH3p调控的7-脱氢胆甾醇合成功能模块与底盘细胞的适配性最佳。
受同一启动子调控的功能模块与不同底盘细胞的适配性不同。以SyBE_000956为底盘的人工细胞具有更强的适配性,其 7-DHC产量最高。
[1]Li PJ, Zhao CS, Li J. Synthesize of cholesteric liquid crystal. Chem Eng, 2005(1): 57–58 (in Chinese).李佩瑾, 赵春山, 李佳. 胆甾相液晶的合成. 化学工程师, 2005(1): 57–58.
[2]Wei CJ, Tan TW. Prospect for production of vitamin D3. Chem Ind Eng Prog, 2003, 22(10):1098–1101 (in Chinese).韦昌金, 谭天伟. 维生素D3合成新工艺的初步设想. 化工进展, 2003, 22(10): 1098–1101.
[3]Huang SH, He XP, Yu PP, et al. Study on the transformation of cholesterol to 7-dehydrocholesterol by 7,8-dehydrogenase from shrimps’ Y-organ. J Anhui Agri Sci, 2011, 39(25): 15397–15399 (in Chinese).黄时海, 何鑫平, 于配配, 等. 虾Y-器官7、8-脱氢酶转化胆固醇为7-脱氢胆固醇研究. 安徽农业科学, 2011, 39(25): 15397–15399.
[4]Xia XB, Wu KY, Li XP, et al. Research progress on biological synthesis of 7-dehydrocholesterol. Sci Tech Food Ind, 2009, 30(11): 339–342.夏小斌, 吴孔阳, 李湘萍, 等. 生物法合成 7-脱氢胆固醇研究进展. 食品工业科技, 2009, 30(11):339–342.
[5]Liu D, Du J, Zhao GR, et al. Applications of synthetic biology in medicine and energy. CIESC J,2011, 62(9): 2391–2397.刘夺, 杜瑾, 赵广荣, 等. 合成生物学在医药及能源领域的应用. 化工学报, 2011, 62(9):2391–2397.
[6]Christine L, Markus V. Preparation of 7-dehydrocholesterol and/or the biosynthetic intermediates and/or secondary products thereof in transgenic organisms: US, 2006/0240508.2006-10-26.
[7]Hohmann HP, Lehmann M, Merkamm M, et al.Production of non-yeast sterols by yeast: US,20120231495. 2012-09-13.
[8]Kong JQ, Shen JH, Huang Y, et al. Production of amorpha-4,11-diene in engineered yeasts. Acta Pharm Sin, 2009, 44(11): 1297–1303 (in Chinese).孔建强, 沈君豪, 黄勇, 等. 酵母工程菌制备紫穗槐-4,11-二烯的研究. 药学学报, 2009, 44(11):1297–1303.
[9]Keasling JD. Synthetic biology and the development of tools for metabolic engineering.Metab Eng, 2012, 14(3): 189–195.
[10]Yadav VG, De Mey M, Lim CG, et al. The future of metabolic engineering and synthetic biology:towards a systematic practice. Metab Eng, 2012,14(3): 233–241.
[11]Lees ND, Skaggs B, Kitsch DR, et al. Cloning of the late genes in the ergosterol biosynthetic pathway of Saccharomyces cerevisia. Lipids, 1995,30(3): 221–226.
[12]Dai ZB, Liu Y, Huang LQ, et al. Production of miltiradiene by metabolically engineered Saccharomyces cerevisiae. Biotechnol Bioeng,2012, 109(11): 2845–2853.
[13]Zhang ZY, He XP, Li WW, et al. Regulation role of sterol C-24 methyltransferase and sterol C-8 isomerase in the ergosterol biosynthesis of Saccharomyces cerevisiae. Acta Microbiol Sin,2009, 49(8): 1063–1068 (in Chinese).张振颖, 何秀萍, 李巍巍, 等. 甾醇 C-24甲基转移酶和甾醇C-8异构酶在酿酒酵母麦角甾醇生物合成中的调控作用. 微生物学报, 2009, 49(8):1063–1068.
[14]Catherine D, Roberto S, Eric D, et al.Self-sufficient biosynthesis of pregnenolone and progesterone in engineered yeast. Nat Biotechnol,1998, 16: 186–189.
[15]Cleiton MS, Tatjana ME, Harald P, et al. A stable yeast strain efficiently producing cholesterol instead of ergosterol is functional for tryptophan uptake, but not weak organic acid resistance. Metab Eng, 2011, 13: 555–569.
[16]John ED, Gabriel CW, Malmirchegini GR, et al.Synthetic protein scaffolds provide modular control over metabolic flux. Nat Biotechnol, 2009, 27(8):753–761.
[17]Polakowski T, Ulf S, Christine L. Overexpression of a cytosolic hydroxymethylglutaryl-CoA reductase leads to squalene accumulation in yeast.Appl Microbiol Biotechnol, 1998, 49: 66–71.
[18]Lin XS, Jiang HW, Zhang Y. Study on biosynthesis of ergosterol in yeast and its gene regulation. J Biol, 2010, 27(6): 83–86 (in Chinese).林晓珊, 江宏文, 张毅. 酵母麦角固醇生物合成及其基因调控的研究. 生物学杂志, 2010, 27(6):83–86.
[19]Markus V, Ulf S, Christine L. Combined overexpression of genes of the ergosterol biosynthetic pathway leads to accumulation of sterols in Saccharomyces cerevisiae. FEMS Yeast Res, 2003, 4: 87–95.
[20]Hou FF, Liu ZQ, Liu JL, et al. Progress in study on 3β-hydroxysteroid-Δ24 reductase (DHCR24). Chin J Cell Biol, 2010, 32(6): 942–950 (in Chinese).侯芳芳, 刘振青, 刘剑利, 等. 24-脱氢胆固醇还原酶 DHCR24的研究进展. 中国细胞生物学学报, 2010, 32(6): 942–950.
[21]Florence MS, Cathy C, Coralie V, et al. Total biosynthesis of hydrocortisone from a simple carbon source in yeast. Nat Biotechnol, 2003, 21:143–149.
[22]Denis P, Bruno D, Roberto S. Cholesterolproduction yeast strains and uses thereof: US,2009/0239837. 2009-09-24.
[23]Shao ZY, Zhao H, Zhao HM. DNA assembler, an in vivo genetic method for rapid construction of biochemical pathways. Nucleic Acids Res, 2009,37(2): 1–10.
[24]Reshma PS, Drew E, Thomas FK. Engineering BioBrick vectors from BioBrick parts. J Biol Eng,2008, 2(5): 1–12.
[25]Sambrook J. Molecular Cloning: A Laboratory Manual. 3rd ed. New York: Cold Spring Harbor Laboratory Press, 2001: 29.
[26]Gietz RD, Woods RA. Yeast transformation by the LiAc/SS carrier DNA/PEG method. Methods Mol Biol, 2006, 313: 107–120.
[27]Zhou X, Zhou J, Tian HC, et al. Dynamic lipidomic insights into the adaptive responses of Saccharomyces cerevisiae to the repeated vacuum fermentation. Omics J Integr Biol, 2010, 14(5):563–574.
[28]Yang H, Gao YG, Li FY, et al. Research progress on biosynthetic pathway of terpenoids containing ginsenoside and the HMGR. China Biotechnol,2008, 28(10): 130–135 (in Chinese).杨鹤, 郜玉钢, 李璠瑛, 等. 人参皂苷等萜类化合物生物合成途径及 HMGR的研究进展. 中国生物工程杂志, 2008, 28(10): 130–135.
[29]Donald KA, Hampton RY, Fritz IB. Effects of overproduction of the catalytic domain of 3-hydroxy-3-methylglutaryl coenzyme A reductase on squalene synthesis in Saccharomyces cerevisiae.Appl Environ Microbiol, 1997, 63(9): 3341–3344.
[30]Siavash P, Verena S, Sara B, et al. Characterization of different promoters for designing a new expression vector in Saccharomyces cerevisiae.Yeast, 2010, 27: 955–964.
[31]Sun J, Shao ZY, Zhao H, et al. Cloning and characterization of a panel of constitutive promoters for applications in pathway engineering in Saccharomyces cerevisiae. Biotechnol Bioeng,2012, 109(8): 2082–2092.
[32]Zhou YJ, Gao W, Rong QX, et al. Modular pathway engineering of diterpenoid synthases and the mevalonic acid pathway for miltiradiene production. J Am Chem Soc, 2012, 134:3234–3241.

