肾癌、前列腺癌、膀胱癌及睾丸癌全基因组关联研究计量和热点的分析
田玉旺 许春伟 方美玉
肾癌、前列腺癌、膀胱癌及睾丸癌全基因组关联研究计量和热点的分析
田玉旺 许春伟 方美玉
目的 分析肾癌、前列腺癌、膀胱癌和睾丸癌全基因组关联研究(GWAS)英文文献数据的构成特征。方法对国际最新GWAS研究数据库之一——美国国立人类基因组研究所(NHGRI)数据库进行数据挖掘。结果 应用文献计量学方法,客观分析了NHGRI的文献基本特征、第一第二阶段样本量、染色体区域相关(核苷酸多态性)SNP、风险等位基因频率、P值文献中最强SNP、优势比(odds ratio,OR)或β相关系数、实验平台及检测SNP 数量8项数据,找出了泌尿生殖系统肿瘤GWAS的一些共性特征。结论 8号染色体区域SNP多态性,可能是泌尿生殖系统肿瘤遗传易感区域,深入此易感区域研究,可能为阐述肿瘤发生、治疗及预后评估提供一定线索。
全基因组关联研究;文献研究;数据挖掘
(ThePracticalJournalofCancer,2014,29:1548~1552)
全基因组关联研究(genome-wide association study,GWAS)是应用高通量基因芯片技术,对人类全基因组范围内常见遗传变异——单核苷酸多态性(single nucleotide polymorphism,SNP)和拷贝数变异(copy number variation,CNV)进行总体关联分析的研究方法[1]。它基于DNA是可遗传的且和临近的等位基因从上一代传递给下一代这一理论。它可以在病例和对照中比较全基因组范围内所有变异的等位基因频率,从中发现与疾病相关联的序列变异[2-3]。GWAS统计分析的基本原则是减少系统偏倚和加大研究强度,譬如进行强有力的SNP设定分析[4]。2007年4月Duggan等[5]和Murabito等[6]利用Illumina及Affymetrix的基因芯片对前列腺癌进行了GWAS,其结果同时首次在Science杂志上发表,自此以后越来越多肿瘤方面的GWAS研究结果陆续发表。现通过对最新GWAS数据库之一——NHGRI,进行肾癌、前列腺癌、膀胱癌和睾丸癌文献特征数据分析挖掘,所获得的GWAS数据信息或许能对国内泌尿生殖系统肿瘤GWAS产生参考作用。
1 资料与方法
研究资料来源于NHGRI,该数据库由Hindorff等始建于2008年11月25日。登入页面http://www.genome.gov/page.cfm?pageid=26525384#searchForm。在页面下Disease/Trait一栏中输入“Renal cell carcinoma”,点击Search后,再点击“Download Spreadsheet of Search Result”保存“MyGWASSearch.xls”,并改名为“Renal cell carcinoma.xls”,并用同样的步骤获得“Prostate cancer.xls”、“Bladder cancer.xls”和“Testicular cancer.xls”。
2 结果
2.1 文献基本特征
数据库原记录包含30篇论文[5-34](肾癌3篇,前列腺癌21篇,膀胱癌4篇、睾丸癌2篇)、涉及135个SNP(肾癌8个SNP,前列腺癌112个SNP,膀胱癌12个SNP,睾丸癌3个SNP)。论文分别发表在Nat Genet(IF:35.209/2012)16篇,BMC Med Genet(IF:2.536/2012)1篇,Cancer Biol Ther(IF:3.287/2012)1篇,Cancer Res(IF:8.65/2012)1篇,Hum Genet(IF:4.633/2012)1篇,Hum Mol Genet(IF:7.692/2012)7篇,J Natl Cancer Inst (IF:14.336/2012)1篇,Cancer Epidemiol Biomarkers Prev(IF:4.559/2012)2篇。
2.2 第一(筛选)阶段样本量
30篇泌尿生殖系统肿瘤GWAS研究文献,第一阶段病例组样本量从202~6 621例;对照组从100~41 603例。按病例数/对照数的研究文献的分布见表1。3.33%的文献没有明确报告对照组样本量。

表1 泌尿生殖系统肿瘤GWAS文献第一阶段样本量分布
注:NR为未报道。
2.3 第二(验证) 阶段样本量
第二阶段病例组样本量从439~22 957例,没有报告第二阶段病例数据的文献占3.33%。对照组从860~24 726例。按病例数/对照数的研究文献的分布见表2。
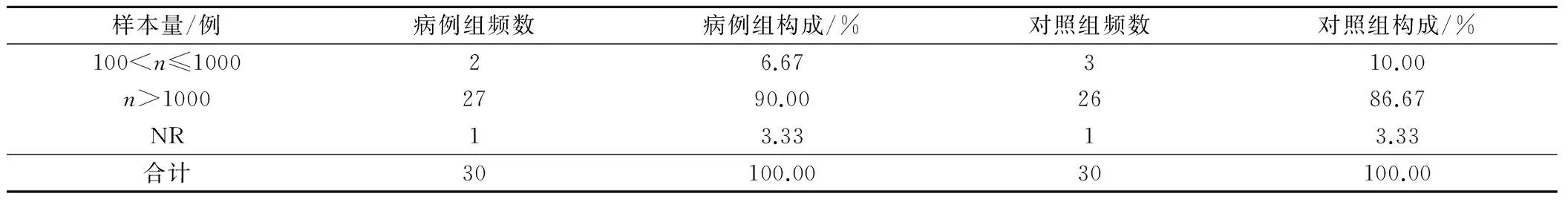
表2 泌尿生殖系统肿瘤GWAS文献第二阶段样本量分布
2.4 染色体区域相关SNP(NCBI rs登记号)
所定位的染色体区域分布大致与染色体长度近似。其中8号染色体明显多于邻近的染色体,见表3。
2.5 对照组相关风险等位基因频率
泌尿生殖系统肿瘤GWAS文献中对照组相关风险等位基因P频率的分布见表4,其中未发现稀有变异(即在对照人群中的发生频率<0.01)。
2.6 优势比(odds ratio,OR)或β相关系数
文献中最强SNP相关风险等位基因优势比(或β相关系数),其中β相关系数最小值为1.04,最大值为3.07,见表5。
2.7P值文献中最强SNP
相关风险等位基因P值选择<1×10-6为录入标准,最小P值3×10-33。P值分布见表6。
2.8 实验平台及检测核苷酸多态性(SNP)数量
文献中SNP试验平台见表7。超过一半(73.33%)的泌尿生殖系统肿瘤GWAS独立使用了Illumina研究平台,六分之一(16.67%)使用Affimetrix芯片系统。SNP数量从60 275~5 340 737个。16.67%研究试验平台的SNP数量在100万~1 000万间,见表7,8。
3 讨论
3.1 概述

表3 泌尿生殖系统肿瘤GWAS文献染色体区域相关SNP (NCBI rs 登记号)

表4 泌尿生殖系统肿瘤GWAS文献中对照组相关风险等位基因频率分布
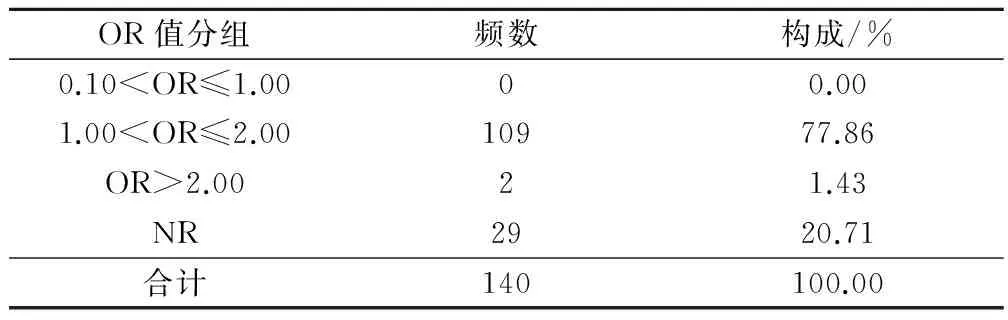
表5 最强SNP相关风险等位基因优势比(或β相关系数)分布

表6 P值分布

表7 泌尿生殖系统肿瘤GWAS文献实验的试验平台分布
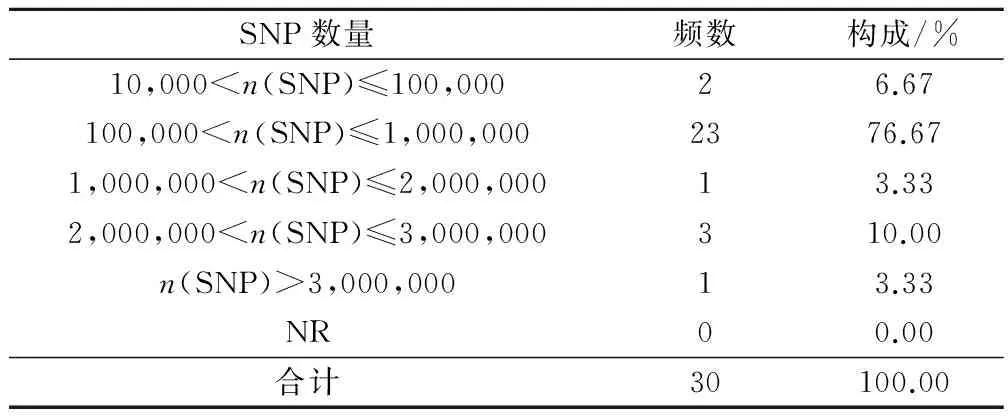
表8 泌尿生殖系统肿瘤GWAS文献使用的试验平台检测核苷酸多态性(SNP)数量
目前泌尿生殖系统肿瘤GWAS方面的报道主要在肾癌、前列腺癌、膀胱癌和睾丸癌领域,其中肾癌GWAS方面Purdue等[9]研究发现2p21区域(EPAS1基因)rs7579899、12q24.31区域(SCARB1基因)rs4765623和11q13.3区域(基因间隔区)rs7105934位点多态性与肾细胞癌遗传易感性相关联。Wu等[8]研究发现12p11.23区域(ITPR2基因)rs718314位点多态性与肾细胞癌遗传易感性相关联。前列腺癌GWAS方面2007年Murabito等[6]研究发现3p22区域(CTDSPL基因)rs9311171、6p12区域(PKHD1基因)rs10498792、5q14区域(HAPLN1基因)rs4466137、3q24区域(基因间隔区)rs345013和13q33区域(基因间隔区)rs1529276与前列腺癌遗传易感性相关联。并在在同一年Duggan等[5]也报道前列腺癌GWAS结果但未报道位点具体未知,以上这两篇是GWAS首次在肿瘤中的报道,从此也拉开了肿瘤GWAS的序幕,此后在前列腺癌GWAS领域陆续发现上百个基因多态性位点。膀胱癌GWAS方面Wu等[32]研究发现8q24.3区域(PSCA基因)rs2294008位点多态性与膀胱癌遗传易感性相关联。睾丸癌GWAS方面Kanetsky等[34]研究发现9p24区域(DMRT1基因)rs7040024和rs755383与睾丸癌遗传易感性相关。一年后Kanetsky等[33]又发现12q21.3区域(KITLG基因)rs4474514也与睾丸癌遗传易感性相关。
3.2 NHGRI数据库分析
通过对NHGRI数据库中泌尿生殖系统肿瘤中八项数据进行不完全统计后,我们发现泌尿生殖系统肿瘤有如下特点:①在文献基本特征方面:泌尿生殖系统肿瘤在Nat Genet发表的文献最多,占53.33%,其次Hum Mol Genet占23.33%和Cancer Epidemiol Biomarkers Prev占6.67%,BMC Med Genet、Cancer Biol Ther、Cancer Res、Hum Genet和J Natl Cancer Inst各一篇。笔者推测Nat Genet以其影响力将是优质泌尿生殖系统肿瘤GWAS的主要阵地。②泌尿生殖系统肿瘤GWAS第一、二阶段的病例组样本量方面:一般从几百例到几万例不等,且以千例为主,最大样本量均不超过25,000例;对照组样本量也从几百例到几万例不等,但以千例以上为主,最大样本量均为超过50,000例。③染色体区域相关SNP方面:以8号染色体区域SNP居多,其中8号染色体区域SNP在胃肠消化系统肿瘤中也居多。④对照组相关风险等位基因频率方面:泌尿生殖系统肿瘤GWAS中风险等位基因以Risk Allele Frequency=0.40左右最多,其次是Risk Allele Frequency=0.50,但未发现稀有变异。⑤P值文献中最强SNP方面:泌尿生殖系统肿瘤P值以10-10为主,其中最强P值2×10-33。⑥优势比(odds ratio,OR)或β相关系数方面:泌尿生殖系统肿瘤OR以1.00左右的弱相关占多数,2.00以上的强相关也占相当一部分比例。⑦实验平台及检测SNP 数量方面:泌尿生殖系统肿瘤实验平台方面Illumina研究平台占大多数,其次是Affymetrix芯片系统,未见Perlegen或Invader研究平台的报道。检测SNP数量从60 275个到5 340 737个不等,1/6试验平台的SNP数量在100万~1 000万之间。
肿瘤方面GWAS的最终目标是应用于临床,主要体现在对正常人群进行肿瘤的危险度预测评估和指导肿瘤的个体化治疗。个体化治疗推崇在肿瘤遗传谱指导下的个体化医学(personalized medicine),为肿瘤发生和治疗疗效提供早期预测,促进肿瘤预防和治疗。泌尿生殖系统肿瘤GWAS经过7年的发展,已在肾癌、前列腺癌、膀胱癌和睾丸癌遗传易感性研究方面取得了很大成功,为个体化治疗打下了基础。本文通过运用文献计量学方法客观分析了NHGRI的文献基本特征、第一、二阶段样本量、染色体区域相关SNP、对照组相关风险等位基因频率、P值文献中最强SNP、优势比(odds ratio,OR)或β相关系数和实验平台及检测SNP 数量8项数据,找到了泌尿生殖系统肿瘤GWAS的一些共性特征,为把握泌尿生殖系统肿瘤GWAS的研究现状和发展趋势奠定基础,并对国内泌尿生殖系统肿瘤GWAS产生参考作用。
[1] 张学军.全基因组关联分析对银屑病遗传学研究的启示〔J〕.浙江大学学报(医学版),2009,38(4):333-337.
[2] 严卫丽.复杂疾病全基因组关联研究进展——研究设计和遗传标记〔J〕.遗传,2008,30(4):400-406.
[3] 严卫丽.复杂疾病全基因组关联研究进展——遗传统计分析〔J〕.遗传,2008,30(5):543-549.
[4] Kraft P,Cox DG.Study designs for genome-wide association studies〔J〕.Adv Genet,2008,60:465-504.
[5] Duggan D,Zheng SL,Knowlton M,et al.Two genome-wide association studies of aggressive prostate cancer implicate putative prostate tumor suppressor gene DAB2IP〔J〕.J Natl Cancer Inst,2007,99(24):1836-1844.
[6] Murabito JM,Rosenberg CL,Finger D,et al.A genome-wide association study of breast and prostate cancer in the NHLBI's Framingham Heart Study〔J〕.BMC Med Genet,2007,8 (Suppl 1):S6.
[7] Henrion M,Frampton M,Scelo G,et al.Common variation at 2q22.3 (ZEB2) influences the risk of renal cancer〔J〕.Hum Mol Genet,2013,22(4):825-831.
[8] Wu X,Scelo G,Purdue MP,et al.A genome-wide association study identifies a novel susceptibility locus for renal cell carcinoma on 12p11.23〔J〕.Hum Mol Genet,2012,21(2):456-462.
[9] Purdue MP,Johansson M,Zelenika D,et al.Genome-wide association study of renal cell carcinoma identifies two susceptibility loci on 2p21 and 11q13.3〔J〕.Nat Genet,2011,43(1):60-65.
[10] Nam RK,Zhang W,Siminovitch K,et al.New variants at 10q26 and 15q21 are associated with aggressive prostate cancer in a genome-wide association study from a prostate biopsy screening cohort〔J〕.Cancer Biol Ther,2011,12(11):997-1004.
[11] Cheng I,Chen GK,Nakagawa H,et al.Evaluating genetic risk for prostate cancer among Japanese and Latinos〔J〕.Cancer Epidemiol Biomarkers Prev,2012,21(11):2048-2058.
[12] FitzGerald LM,Kwon EM,Conomos MP,et al.Genome-wide association study identifies a genetic variant associated with risk for more aggressive prostate cancer〔J〕.Cancer Epidemiol Biomarkers Prev,2011,20(6):1196-1203.
[13] Sun J,Zheng SL,Wiklund F,et al.Sequence variants at 2- 2q13 are associated with prostate cancer risk〔J〕.Cancer Res,2009,69(1):10-15.
[14] Tao S,Feng J,Webster T,et al.Genome-wide two-locus epistasis scans in prostate cancer using two European populations〔J〕.Hum Genet,2012,131(7):1225-1234.
[15] Amin Al Olama A,Kote-Jarai Z,Schumacher FR,et al.A meta-analysis of genome-wide association studies to identify prostate cancer susceptibility loci associated with aggressive and non-aggressive disease〔J〕.Hum Mol Genet,2013,22(2):408-415.
[16] Schumacher FR,Berndt SI,Siddiq A,et al.Genome-wide - association study identifies new prostate cancer susceptibility loci〔J〕.Hum Mol Genet,2011,20(19):3867-3875.
[17] Xu J,Mo Z,Ye D,et al.Genome-wide association study in Chinese men identifies two new prostate cancer risk loci at 9q31.2 and 19q13.4〔J〕.Nat Genet,2012,44(11):1231-1235.
[18] Kote-Jarai Z,Olama AA,Giles GG,et al.Seven prostate c- ancer susceptibility loci identified by a multi-stage genome-wide association study〔J〕.Nat Genet,2011,43(8):785-791.
[19] Haiman CA,Chen GK,Blot WJ,et al.Genome-wide association study of prostate cancer in men of African ancestry identifies a susceptibility locus at 17q21〔J〕.Nat Genet,2011,43(6):570-573.
[20] Takata R,Akamatsu S,Kubo M,et al.Genome-wide association study identifies five new susceptibility loci for prostate cancer in the Japanese population〔J〕.Nat Genet,2010,42(9):751-754.
[21] Eeles RA,Kote-Jarai Z,Al Olama AA,et al.Identification of seven new prostate cancer susceptibility loci through a genome-wide association study〔J〕.Nat Genet,2009,41(10):1116-1121.
[22] Gudmundsson J,Sulem P,Gudbjartsson DF,et al.Genome-wide association and replication studies identify four variants associated with prostate cancer susceptibility〔J〕.Nat Genet,2009,41(10):1122-1126.
[23] Eeles RA,Kote-Jarai Z,Giles GG,et al.Multiple newly identified loci associated with prostate cancer susceptibility〔J〕.Nat Genet,2008,40(3):316-321.
[24] Gudmundsson J,Sulem P,Rafnar T,et al.Common sequen- ce variants on 2p15 and Xp11.22 confer susceptibility to prostate cancer〔J〕.Nat Genet,2008,40(3):281-283.
[25] Thomas G,Jacobs KB,Yeager M,et al.Multiple loci identified in a genome-wide association study of prostate cancer〔J〕.Nat Genet,2008,40(3):310-315.
[26] Gudmundsson J,Sulem P,Steinthorsdottir V,et al.Two variants on chromosome 17 confer prostate cancer risk,and the one in TCF2 protects against type 2 diabetes〔J〕.Nat Genet,2007,39(8):977-983.
[27] Gudmundsson J,Sulem P,Manolescu A,et al.Genome-wide association study identifies a second prostate cancer susceptibility variant at 8q24〔J〕.Nat Genet,2007,39(5):631-637.
[28] Yeager M,Orr N,Hayes RB,et al.Genome-wide association study of prostate cancer identifies a second risk locus at 8q24〔J〕.Nat Genet,2007,39(5):645-649.
[29] Garcia-Closas M,Ye Y,Rothman N,et al.A genome-wide association study of bladder cancer identifies a new susceptibility locus within SLC14A1,a urea transporter gene on chromosome 18q12.3〔J〕.Hum Mol Genet,2011,20(21):4282-4289.
[30] Rafnar T,Vermeulen SH,Sulem P,et al.European genome-wide association study identifies SLC14A1 as a new urinary bladder cancer susceptibility gene〔J〕.Hum Mol Genet,2011,20(21):4268-4281.
[31] Rothman N,Garcia-Closas M,Chatterjee N,et al.A multi-stage genome-wide association study of bladder cancer identifies multiple susceptibility loci〔J〕.Nat Genet,2010,42(11):978-984.
[32] Wu X,Ye Y,Kiemeney L A,et al.Genetic variation in the prostate stem cell antigen gene PSCA confers susceptibility to urinary bladder cancer〔J〕.Nat Genet,2009,41(9):991-995.
[33] Kanetsky PA,Mitra N,Vardhanabhuti S,et al.A second independent locus within DMRT1 is associated with testicular germ cell tumor susceptibility〔J〕.Hum Mol Genet,2011,20(15):3109-3117.
[34] Kanetsky PA,Mitra N,Vardhanabhuti S,et al.Common variation in KITLG and at 5q31.3 predisposes to testicular germ cell cancer〔J〕.Nat Genet,2009,41(7):811-815.
(编辑:吴小红)
Measurement and Hot Research of Genome-wide Association Study in Renal Cell Carcinoma、Prostate Cancer、Bladder Cancer and Testicular Cancer
TIANYuwang,XUChunwei,FANGMeiyu.
DepartmentofPathology,theMilitaryGeneralHospitalofBeijingPLA,Beijing,100700
Objective To analyze the data constitution features of genome-wide association study (GWAS) in Renal cell carcinoma、Prostate cancer、Bladder cancer and Testicular cancer in English literature.Methods National human genome institute GWAS database,one of the latest international research database of GWAS,was meticulously data mined.Results Using literature metrology method objectively analyzed 8 data of the NHGRI's basic characteristics of the literature,the first and second stage of sample size,chromosomal regions associated SNPS,related risk allele frequency in the control group,P value with the strongest SNP in the literature,odds ratio or beta coefficient of correlation and experimental platform including testing the SNP number,and found out the common characteristics of the urogenital system tumor GWAS.Conclusion Chromosome 8 regional SNP polymorphism may be the urogenital system tumor genetic susceptibility region,and through into the susceptible area can provide some clues in the treatment of cancer and prognosis assessment.
Genome-wide Association Study( GWAS);Literature research;Data Mining
100700 中国人民解放军北京军区总医院(田玉旺,许春伟);310021 浙江省肿瘤医院中西医结合科(方美玉)
许春伟
10.3969/j.issn.1001-5930.2014.12.012
R73-36
A
1001-5930(2014)12-1548-05
2014-02-24
2014-03-18)

