聚苹果酸的微生物合成及应用研究进展
王媛媛,全玉芬,宋存江
南开大学生命科学学院 分子微生物学与技术教育部重点实验室,天津 300071
综 述
聚苹果酸的微生物合成及应用研究进展
王媛媛,全玉芬,宋存江
南开大学生命科学学院 分子微生物学与技术教育部重点实验室,天津 300071
王媛媛, 全玉芬, 宋存江. 聚苹果酸的微生物合成及应用研究进展. 生物工程学报, 2014, 30(9): 1331−1340.
Wang YY, Quan YF, Song CJ. Progress in microbial synthesis and application of polymalic acid. Chin J Biotech, 2014, 30(9): 1331−1340.
聚苹果酸是一种优良的完全可生物降解的生物活性材料,在医学和药学领域有着极大的应用潜力。文中结合我们的工作总结了近年来聚苹果酸代谢、聚苹果酸的微生物发酵合成以及聚苹果酸在医药领域的应用等方面的研究进展,并对聚苹果酸的深入研究作了展望。
聚苹果酸,微生物合成,生物活性材料
Keywords: polymalic acid, microbial synthesis, bioactive material
1 聚苹果酸概述
聚苹果酸 (Polymalic acid,PMLA) 是一种完全可生物降解的聚酯类高分子材料。它分解后的终产物安全无毒,不会对环境造成二次污染。作为一种可溶性脂肪族聚酯,聚苹果酸具有高度的水溶性、化学衍生性和生物相容性。这些优良特性使聚苹果酸成为一种极具应用前景的生物活性材料。
聚苹果酸以L-苹果酸为唯一单体,由一分子苹果酸的–OH和另一分子苹果酸的α-或β-COOH脱水聚酯而成,聚苹果酸有3种结构,即α型、β型、γ型 (图1)。目前研究发现生物体内存在的聚苹果酸只有β型。
与其他许多天然多聚物不同,聚苹果酸还具有两个显著的优点:1) 易代谢性,由于L-苹果酸是生物体内三羧酸循环 (TCA) 的中间体,β-聚苹果酸容易在生物体内通过正常的三羧酸循环代谢途径分解;2) 易修饰性,聚苹果酸具有许多自由羧基,容易与其他官能团反应而制得聚苹果酸衍生物,或引入功能基团或小分子药物,从而改变聚苹果酸的性质得到具有特殊功能的产物。聚苹果酸可以通过化学合成法和微生物发酵法制备。化学合成法可以得到3种构型的聚苹果酸,且技术成熟,成本低。与化学合成法不同,微生物发酵制备的聚苹果酸具有很高的化学纯度、光学纯度、很高的分子量及突出的生物学特性[1]。因此微生物发酵生产聚苹果酸成为目前研究的热点,本文总结了近年来相关的研究,并对聚苹果酸的进一步研究作出了展望。

图1 苹果酸及聚苹果酸的结构[1]Fig. 1 Structure of malate and polymalic acid[1].
2 聚苹果酸的微生物合成
早在1969年,Shimada等[2]从圆弧青霉Penicillium cyclopium中分离得到一种酸性的大分子化合物,这种物质能抑制蛋白酶的活性,最终证实该化合物为聚苹果酸。1989年,Fischer等[3]从多头绒泡菌Physarum polycephalum细胞中分离出聚苹果酸,并发现它是DNA聚合酶α的抑制剂。β-PMLA可以由多种黏菌和丝状真菌合成,主要的生产细胞类型是多头绒泡菌的变形体和短梗霉属的出芽短梗霉。
2.1 多头绒泡菌发酵合成聚苹果酸
多头绒泡菌属于粘菌门,它的生活史包括微观的变形虫阶段和巨大多核原质团阶段。其中,只有多核原质团才能以L-苹果酸为前体合成PMLA,这种水溶性的大分子集中分布在细胞核中起作用,多余部分则以纯聚苹果酸的形式分泌到胞外,分子量在10–300 kDa之间。PMLA在细胞中结合、运输核酸相关蛋白,并暂时性地抑制一些酶和结构蛋白的活性,以起到维持原质团形态的作用。能够和PMLA结合的蛋白包括:组蛋白 (H1、H2B、H3和H4)[4]、DNA聚合酶α引物酶[3,5]、DNA聚合酶δ-复合体[6]以及DNA聚合酶ε-复合体[7]。Göttler提出,利用亲和层析的方法加上蛋白序列测序可以获得PMLA结合蛋白的更多信息,对解析PMLA在原质体中的作用提供更有效的方法[8]。
目前研究证实的聚苹果酸合成途径是,以L-苹果酸为前体,在聚合酶的催化作用下合成聚苹果酸。L-苹果酸前体的获得可以分为两种情况:培养基有CaCO3条件下,通过丙酮酸羧化和草酰乙酸还原反应两步反应获得;培养基缺乏CaCO3的条件下,通过TCA循环和乙醛酸支路获得 (图2)。
在发酵培养基中,菌体通过丙酮酸羧化酶进行葡萄糖代谢和二氧化碳固定,生成草酰乙酸,进一步还原生成L-苹果酸,此过程并不消耗TCA循环的中间产物。当培养基中加入CaCO3后,在丙酮酸羧化酶的作用下,草酰乙酸的合成量会增加,其中一部分进一步还原生成L-苹果酸,一部通过苹果酸-天冬氨酸穿梭进入TCA循环,防止草酰乙酸的过量积累造成代谢上的不平衡[9]。该种假说否定了之前认为的L-苹果酸前体的积累是丙酮酸羧化酶和TCA循环造成的,强调了CaCO3的开关作用,即CaCO3通过调节体内的草酰乙酸的水平来调节L-苹果酸前体的积累途径。
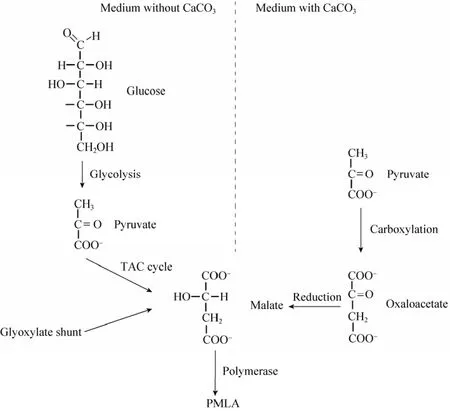
图2 聚苹果酸合成途径Fig. 2 Synthetic pathway of polymalic acid.
Willibald等[10]通过向多头绒泡菌中注射14C标记的L-苹果酸,来跟踪聚苹果酸的合成,并检测了KCN、砷酸盐、Desulfo CoA、ATP类似物等物质对PMLA合成的影响。作者在文章中提出了一个PMLA合成酶系的模型,即包括1个malyl-AMP连接酶和1个malyl-转移酶 (聚合酶) 活性。通过向原质团细胞注射ATP类似物发现,5′-(α,β-亚甲基)-三磷酸-腺苷 (Apcpp)的加入能够100%抑制PMLA的合成,而5′-(β, γ-亚甲基)-三磷酸-腺苷 (Appcp) 的加入对PMLA合成的抑制率只有40%,并且Desulfo CoA的加入并不会抑制PMLA的合成,该结果可以得出在PMLA的合成过程中,L-苹果酸被活化成malyl-AMP。作者试图体外合成PMLA,但是原质团裂解物只具备将苹果酸单体转化成malyl-AMP的能力,并不能将苹果酸单体聚合成PMLA。注射磷酸酪氨酸酶的抑制剂和非水解GTP类似物均能抑制PMLA的合成。作者推测,原质团裂解物不能合成PMLA是细胞对损伤所作出的应激反应使malyl-转移酶失活造成的。到目前为止,研究者未能从细胞提取物中分离出有活性的PMLA聚合酶蛋白。
Pinchai等[11]鉴定出一种调节PMLA水平的蛋白质NKA48,但是没有证据证明该多肽是PMLA合成酶的转录产物。他们提取多头绒泡菌的阿米巴虫和原质团两个生长阶段细胞的cDNA,利用抑制消减杂交技术 (SSH) 分析了PMLA代谢相关基因,鉴定出一个原质团细胞特有的spherulin 3a样多肽序列——NKA48 (Spherulin 3b),利用RNAi技术敲除NKA48-cDNA后,PMLA的水平严重降低,说明spherulin 3b有调节PMLA水平的功能。
研究人员在探索PMLA的生理作用时发现PMLA在原质团中不是游离的,推测和PMLA结合的分子伴侣是聚苹果酸降解酶(Polymalatase, PMase)。PMase在体外可以和PMLA的游离羟基末端结合,并且在细胞质环境中 (pH 6.5) 是没有降解活性的,分泌到胞外后会把聚苹果酸降解成苹果酸单体。Miachael等[12]通过向多头绒泡菌原质团中注射荧光标记的PMLA,发现细胞质中的PMLA与PMase及一些蛋白结合成复合体被运送到细胞核表面,然后复合体解离,PMLA进入细胞核中,PMase及一些蛋白则留在细胞核表面。PMase在细胞核表面的定位可能涉及到结合蛋白、原位离子效应、脂肪酸乙酰化和细胞膜结合等问题,还有待深入研究。
PMase可特异性地降解PMLA为苹果酸单体,可以作为修饰PMLA的分子工具。由于PMase蛋白在体外很容易被剪切,研究者起初推测它的激活机制可能是多肽片段的切割。但是Mueller等[13]发现蛋白酶抑制剂并不能抑制PMase的激活,因此肯定存在其他途径。细胞膜损伤启动的酪氨酸磷酸化信号能够使PMLA合成酶失活,他们推测可能是相同的机制启动PMase的活性,并通过一个酶反应体系验证该论断。在提取的多头绒泡菌细胞膜溶液中加入2 mmol/L MgATP温育,然后测定PMase的活性,发现酶活比只有细胞膜存在时提高的很多。在体系中加入Tyrphostin-47 (一种酪氨酸激酶抑制剂) 或者酪氨酸特异性蛋白磷酸酶时,激活失败。他们的实验结果证明,PMase的激活是细胞膜上酪氨酸蛋白激酶启动的,并成功激活了酿酒酵母重组表达的PMase。
对多头绒泡菌合成PMLA的研究多集中在合成机制及调控方面,发酵方面的研究较少。目前利用P. polycephalum发酵合成PMLA多是采用Lee等[14]在1999年建立的原质团细胞发酵的方法。该方法中菌株M3CVII (ATCC204388)培养6 d后PMLA的产量可以达到2.7 g/L。2000年,该研究组用非生长型的P. polycephalum原质团细胞发酵合成了PMLA,使PMLA的合成和P. polycephalum的生长解离开来,使小的生物量合成大量的PMLA成为可能。
2.2 出芽短梗霉发酵合成聚苹果酸
出芽短梗霉Aurobasidium pullulans分类属于半知菌纲,是一种具有酵母型和菌丝型的多形态真菌,合成的聚苹果酸一部分以β-聚苹果酸-葡聚糖结合体的形式分泌出来,另一部分被分解为分子量为5–11 kDa的自由β-聚苹果酸分泌到细胞外。利用β-聚苹果酸在有机溶剂丙酮中的高溶解性可以去除与之相连的葡聚糖,得到纯β-聚苹果酸。目前聚苹果酸产量最高的菌株是陆生半知菌的短梗霉属,随菌株的不同,产量在12–60 g/L聚苹果酸钙盐或聚苹果酸钠盐之间变化。
出芽短梗霉属菌株可以利用D-葡萄糖、蔗糖、果糖、葡萄糖醇和琥珀酸为碳源,发酵合成聚苹果酸。刘双江等研究人员以出芽短梗霉为研究对象,研究发现三氟乙酸可以抑制聚苹果酸的产生,它会使柠檬酸合成酶失活,是柠檬酸循环和乙醛酸支路的强抑制剂,苹果酸、丁二酸、富马酸对聚苹果酸的积累有促进作用,马来酸和丙二酸会产生抑制作用,马来酸不参与代谢且对机体有毒害,丙二酸是琥珀酸脱氢酶的竞争性抑制剂,因此刘双江等指出出芽短梗霉聚苹果酸的合成途径与多头绒泡菌相似[15]。
到目前为止,对于出芽短梗霉发酵合成聚苹果酸的研究集中在菌种的筛选和发酵过程优化方面。
Naoki等[16]从日本各地采集的木质标本中分离到Aureobasidium sp. A-91,以葡萄糖为碳源,培养基中加入30 g/L的CaCO3,25 ℃摇床培养7 d,聚苹果酸的浓度达到47 g/L,质量的转化率为0.4 g聚苹果酸/g葡萄糖,分子量为6–11 kDa。Toshiaki等[17]利用Aureobasidium sp. A-91的固定化细胞生产聚苹果酸,第一步获得大量的细胞,第二步利用静止的细胞进行转化,实验进行了5次重复,聚苹果酸浓度为80 g/L左右。刘双江[18]从荷兰和德国菌种保藏中心筛选出两株具有较高产聚苹果酸能力的出芽短梗霉菌株CBS591.75和DSM3497,并对CBS591.75进行2 L和20 L发酵条件的摸索,培养基参照Nagata等的方法配制。菌株CBS591.75在20 L规模发酵144 h后发酵液中聚苹果酸浓度为8.0 g/L。王丽燕等[19]对分离的菌株A. pullulans BS02摇瓶发酵聚苹果酸的条件进行了研究,以葡萄糖为碳源,培养基中加入50 g/L的CaCO3,pH 4.0–4.5、24 ℃发酵10 d,聚苹果酸产量可达到30.0 g/L。Zhang等[20]从新鲜的植物样品中分离得到一株A. pullulans。通过优化发酵条件,摇瓶试验中,以葡萄糖为碳源,培养基中加入30 g/L的CaCO3,该菌合成聚苹果酸的浓度达到62.3 g/L;10 L发酵罐试验中,合成聚苹果酸的浓度为57.2 g/L,生产强度为0.35 g/(L·h)。徐玲芬等[21]从自然界植物样本中分离得到一株产PMLA的菌株,命名为A. pullulans ZUCC-41,以葡萄糖为碳源,培养基中加入30 g/L的 CaCO3,该菌株发酵合成聚苹果酸的浓度为26.2 g/L。
以上分离得到的菌株均以葡萄糖为碳源发酵合成聚苹果酸,Pennapa等[22]则利用生物质能合成聚苹果酸。他们首先对从冰岛和泰国等地采样分离得到的56株A. pullulans菌株进行了系统进化上的分类,并研究了它们产PMLA的能力。不同类群的A. pullulans合成PMLA的能力是有差别的,可能某类遗传背景的A. pullulans是比较适合PMLA的合成的。然后以其中代表性的菌株,利用过氧化氢处理的小麦秸秆和玉米芯为唯一碳源,发酵合成PMLA。在优化条件下:限氮培养基中加入5% (W/V)预处理的小麦秸秆、辅助酶和3% (W/V)的CaCO3发酵7 d,A. pullulans NRRL 50383合成PMLA的能力达到20 g/L。农作物秸秆是农业生产过程中的副产品,如果能够加以利用合成PMLA,则会很大程度地降低PMLA的生产成本。
在发酵过程优化方面,中国科学院过程工程研究所的万印华等[23]公开了利用A. pullulans ipe-1发酵生产聚苹果酸的新工艺,通过控制溶氧、pH、有机氮和无机氮交配使用,在7 L发酵罐中发酵4 d,发酵液中β-聚苹果酸的浓度为35.2 g/L。该研究组还研究了A. pullulans ipe-1合成PMLA和菌体生长的关系[24]。他们认为对数生长期晚期菌体的生长和PMLA的合成无关是由于高的PMLA浓度和培养基中低的还原力抑制了菌体的生长。重复分批发酵(Repeated-batch fermetation) 和细胞再循环发酵(Cell-recycle fermetation) 或在两种发酵过程中维持培养基氧化还原电位 (ORP) 低于70 mV,均能提高PMLA的产率。控制ORP的重复分批发酵中,PMLA的产量和细胞的生长分别提高了21.2%和11.7%;控制ORP的细胞再循环发酵中,每单位葡萄糖合成PMLA的产量提高了33.2%。该研究成果为PMLA合成的过程优化提供了新的思路。
对已分离出芽短梗霉菌株合成聚苹果酸的能力总结见表1。出芽短梗霉作为一种重要的工业微生物,除聚苹果酸外,可以合成多种胞外产物,包括普鲁兰多糖、淀粉酶、木糖醇脱氢酶、抗菌素等,而且在培养过程中会产生黑色素,使产品在下游加工过程中必须采用活性炭多次脱色。因此,副产物和黑色素的去除在出芽短梗霉合成聚苹果酸及产物纯化的过程中也是很有必要的。

表1 已分离出芽短梗霉发酵合成聚苹果酸的能力Table 1 Polymalic acid producing strains of A. pullulans and their productivity

3 聚苹果酸的应用
聚苹果酸作为一种脂肪族聚酯,具有良好的水溶性、可生物降解性、生物相容性,并且易代谢、易修饰,诸多优点使其在生物医药和生物材料等领域极具应用前景,有望成为手术缝合线、药物缓释系统、组织工程支架材料、透析袋等。
3.1 聚苹果酸作为药物载体的应用
1975年,Ringsdorf[25]首次提出用具有药理活性的基团的大分子作为药物的概念,这种高分子药物体系的性能很大程度上取决于所选用的大分子。可用作药物的大分子必须具有无毒性、可生物降解性、免疫惰性、可控释放性及窄分布的高分子。聚苹果酸具有上述优良特性,近几年其作为药物载体的研究成为热点。
目前为止,最新的应用研究是以PMLA为载体,共价键合反义核苷酸的纳米共聚物“Polycefin”。该模型采用的是多重靶向给药系统,在PMLA的主链上还共价结合了肿瘤特异抗体和很多功能单元,使给药系统能够定位并进入肿瘤细胞,并释放寡核苷酸或是药物。该系统能够同时携带多个反义寡核苷酸药物和抗体,载体通过抗体对肿瘤表面的高度特异性,大大降低了药物对循环系统及其他部位的毒性,这对于抗癌药物来说极其重要[26-30]。
为了解决PMLA自身在水环境中降解过快的问题,Portilla-Arias等[31]将PMLA部分甲基化,研究了其特性,发现25%和50%甲基化的PMLA (coPMLA-Me25H75和coPMLA-Me50H50)在一定条件下可以破坏脂质体的膜,随着甲基化程度的提高,PMLA衍生物的可溶性降低,并且对脑癌细胞和乳腺癌细胞系的毒性越大,该研究表明,一定程度甲基化的PMLA可以用作药物传递中的纳米共聚物载体。Inoue等[32-33]通过在载体上连接2C5 mAb、TfR mAb 和AON来抑制HER2/neu Receptor和蛋白EGFR的合成,研究将Polycefin用于乳腺癌的治疗。
3.2 聚苹果酸作为生物材料的应用
Cammas等[34]研究发现改性后的聚苹果酸透析袋有极好的胶体渗透压,是一种理想的透析材料。此外由于具有可生物降解性和免疫惰性,聚苹果酸及其衍生物还可以作为生物医学材料如烧伤治疗的绷带、手术缝合线等。Viviane等[35]在其研究中发现聚苹果酸的衍生物本身不能治愈伤口,但是它能与交联生长因子的乙酰肝素结合,刺激骨骼的修复,提高伤口的愈合速度。李世普等还利用添加有聚苹果酸的高分子聚合物制成一种可降解的导电生物医学材料,该材料同时具有可降解性和导电性的功能[36]。
4 展望
笔者所在研究室建立了出芽短梗霉双产物发酵合成PMLA和普鲁兰,及其分离纯化的方法。在研究出芽短梗霉的生长周期和产物合成的关系时,我们发现PMLA在对数生长期中后期增长速度最快,对菌株Aureobasidium pullulans IFO6353来说,膨胀型细胞是合成PMLA的主要细胞形态[37]。目前,笔者正在利用分子生物学技术对普鲁兰合成酶基因及黑色素合成相关基因进行敲除。
聚苹果酸 (PMLA) 是一种具有巨大潜在应用价值的聚酯类物质,但相关应用研究的文献不多。究其原因:首先,聚苹果酸的生产强度较低。目前分离得到的合成聚苹果酸能力较高的菌株为出芽短梗霉,聚苹果酸作为一种次级代谢产物,自然状态下菌体不会大量合成。又由于该菌生活史复杂,生长繁殖速度慢,致使发酵周期延长,单位时间的产量较低。另外,聚苹果酸在水环境中过快的降解速度也限制了它的应用。若要改变聚苹果酸微生物合成与应用的现状,应从以下几方面入手:1) 继续选育高产菌株,从自然界中筛选稳定高产的聚苹果酸合成菌;2) 发酵工艺优化与原位分离发酵的应用,继续优化聚苹果酸合成菌的发酵条件,采用原位分离发酵技术提高聚苹果酸的产率;3) 合成途径优化,利用合成生物学在分子水平改造合成途径——提高前体物质的合成、聚苹果酸合成酶纯化及改性等,进一步提高菌株合成聚苹果酸的能力,减少副产物的合成,提高聚苹果酸的分子量;4) 聚苹果酸分子的化学修饰,通过键合保护基团或环境应答基团等对聚苹果酸进行化学修饰,使其具有更优良的性质。随着研究的不断深入,聚苹果酸微生物合成的工业化肯定会成为现实。
REFERENCES
[1] Ding R, Li GJ. Synthesis and application prospects of biopolymers poly (malic acid) and its derivatives. Polym Bull, 2005(2): 48–56 (in Chinese).丁锐, 李光吉. 生物高分子聚苹果酸及其衍生物的合成与应用前景. 高分子通报, 2005(2): 48–56.
[2] Shimada K, Matsushima K, Fukumoto J, et al. Poly-(L)-malic acid, a new protease inhibitor from Penicillium cyclopium. Biochem Bioph Res Commun, 1969, 35(5): 619–624.
[3] Fischer H, Erdmann S, Holler E. An unusual polyanion from Physarum polycephalum that inhibits homologous DNA polymerase α in vitro. Biochemistry, 1989, 28(12): 5219–5226.
[4] Angerer B, Holler E. Large complexes of β-poly (L-malate) with DNA polymerase α, histones, and other proteins in nuclei of growing plasmodia of Physarum polycephalum. Biochemistry, 1995, 34(45): 14741–14751.
[5] Holler E, Achhammer G, Angerer B, et al. Specific inhibition of Physarum polycephalum DNA-polymerase-α-primase by poly (L-malate) and related polyanions. Eur J Biochem, 1992, 206(1): 1–6.
[6] Achhammer G, Winkler A, Angerer B, et al. DNA polymerase-δ of Physarum polycephalum. Curr Genet, 1995, 28(6): 534–545.
[7] Doerhoefer S, Khodyreva S, Safronov IV, et al. Molecular consitituents of the replication apparatus in the plasmodium of Physarum polycephalum: identification by photoaffinity labeling. Microbiology, 1998, 144(11): 3181–3193.
[8] Göttler T, Holler E. Screening for β-poly (L-malate) binding proteins by affinity chromatography. Biochem Bioph Res Commun, 2006, 341(4): 1119–1127.
[9] Lee BS, Maurer T, Kalbitzer HR, et al. β-poly-(L-malate) production by Physarum polycephalum13C: nuclear magnetic resonance studies. Appl Microbiol Biot, 1999, 52(3): 415–420.
[10] Willibald B, Bild W, Lee BS, et al. Is β-poly (L-malate) synthesis catalysed by a combination of β-L-malyl-AMP-ligase and β-poly (L-malate)polymerase? Eur J Biochem, 1999, 265(3): 1085–1090.
[11] Pinchai N, Lee BS, Holler E. Stage specific expression of poly (malic acid)-affiliated genes in the life cycle of Physarum polycephalum. Spherulin 3b and polymalatase. FEBS J, 2006, 273(5): 1046–1055.
[12] Miachael K, Bernd G, René CK, et al. Localization of fluorescence-labeled poly (malic acid) to the nuclei of the plasmodium of Physarum polycephalum. Eur J of Biochem, 2003, 270(7): 1536–1542.
[13] Mueller W, Haind M, Holler E. Physarum polymalic acid hydrolase: recombinant expression and enzyme activation. Biochem Bioph Res Commun, 2008, 377(3): 735–740.
[14] Lee BS, Holler E. β-Poly (L-malate) production by non-growing microplasmodia of Physarum polycephalum. Effects of metabolic intermediates and inhibitors. FEMS Microbiol Lett, 2000, 193(1): 69–74.
[15] Liu SJ, Steinbuechel A. Production of poly (malic acid) from different carbon sources and its regulation in Aureobasidium pullulans. Biotech Lett, 1997, 19(1): 11–14.
[16] Naoki N, Tadaatsu N, Takeshi T. Fermentative production of poly (β-L-malic acid), a polyelectrolytic biopolyester, by Aureobasidium sp.. Biosci Biotech Biochem, 1993, 57(4): 638.
[17] Toshiaki NK, Naoko H, Tadaatsu N. Poly (β-malic acid) production by the non-growing cells of Aureobasidium sp. strain A-91. J Ferm Bioen, 1996, 82(4): 411–413.
[18] Liu SJ. Production of polymalic acid by Aureobasidium pullulans CBS591.75 and DSM2404 in 2 L and 20 L fermenter. Chin J Biotech, 1997, 13(3): 279–283 (in Chinese).刘双江. 出芽短梗霉菌株CBS591.75 和DSM2404发酵生产聚苹果酸的研究. 生物工程学报, 1997, 13(3): 279–283.
[19] Wang LY, Zheng YF, Liu TT. Optimization of fermentation conditions for poly (β-malic acid) production. Chin J Biopro Eng, 2010, 8(2): 41–45 (in Chinese).王丽燕, 郑谊丰, 刘婷婷. 聚苹果酸的发酵培养条件优化. 生物加工过程, 2010, 8(2): 41–45.
[20] Zhang HL, Cai J, Dong JQ, et al. High-level production of poly (β-L-malic acid) with a new isolated Aureobasidium pullulans strain. Appl Microbiol Biotech, 2011, 92(2): 295–303.
[21] Xu, LF, Chen ZH, Zhao J, et al. Screening and identification of high-yield poly (β-malic acid) bacterial strain. J Zhejiang Univer: Sci Med, 2012, 41(4): 434–440 (in Chinese).徐玲芬, 陈忠华, 赵婧, 等. β-聚苹果酸高产菌株的筛选及鉴定. 浙江大学学报: 医学版, 2012, 41(4): 434–440.
[22] Manitchotpisit P, Skory CD, Peterson SW, et al. Poly (β-L-malic acid) production by diverse phylogenetic clades of Aureobasidium pullulans. J Ind Microbiol Biotech, 2012, 39(1): 125–132.
[23] Wan YH, Cao WF. β-poly malic acid fermentation technology: CN 101696434A. 2010-04-21.万印华, 曹伟峰. β-聚苹果酸发酵新工艺: CN 101696434A. 2010-04-21.
[24] Cao W, Luo J, Zhao J, et al. Intensification of β-poly (L-malic acid) production by Aureobasidium pullulans ipe-1 in the late exponential growth phase. J Ind Microbiol Biotech, 2012, 39(7): 1073–1080.
[25] Ringsdorf H. Structure and properties of pharmacologically active polymers. J Polym Sci Pol Symp, 1975, 51(1): 135–153.
[26] Lee BS, Fujita M, Khazenzon NM, et al. Polycefin, a new prototype of a multifunctional nanoconjugate based on poly (β-L-malic acid) for drug delivery. Bioconjugate Chem, 2006, 17(2): 317–326.
[27] Fujita M, Khazenzon NM, Ljubimov AV, et al. Inhibition of laminin-8 in vivo using a novel poly (malic acid)-based carrier reduces glioma angiogenesis. Angiogenesis, 2006, 9(4): 183–191.
[28] Fujita M, Lee BS, Khazenzon NM, et al. Brain tumor tandem targeting using a combination ofmonoclonal antibodies attached to biopoly (β-L-malic acid). J Control Release, 2007, 122(3): 356–363.
[29] Ljubimova JY, Fujita M, Khazenzon NM, et al. Nanoconjugate based on polymalic acid for tumor targeting. Chem Biol Interact, 2008, 171(2): 195–203.
[30] Ljubimova JY, Fujita M, Ljubimov AV, et al. Poly (malic acid) nanoconjugates containing various antibodies and oligonucleotides for multi-targeting drug delivery. Nanomedicine, 2008, 3(2): 247–265.
[31] Portilla-Arias J, Pati R, Hu J, et al. Nanoconjugate platforms development based in poly (β-L-malic acid) methyl esters for tumor drug delivery. J Nanotechnol, 2010, doi:10.1155/2010/825363.
[32] Inoue S, Ding H, Portilla-Arias J, et al. Polymalic acid-based nanobiopolymer provides efficient systemic breast cancer treatment by inhibiting both HER2/neu receptor synthesis and activity. Cancer Res, 2011, 71(4): 1454–1464.
[33] Inoue S, Pati R, Portilla-Arias J, et al. Nanobiopolymer for direct targeting and inhibition of EGFR expression in triple negative breast cancer. PLoS ONE, 2012, 7(2): e31070.
[34] Cammas S, Bear MM, Moine L. Polymers of malic acid and 3-alkylmalic acid as synthetic PHAs in the design of biocompatible hydrolyzable devices. Int J Biol Macromol, 1999, 25(1-3): 273–282.
[35] Jeanbat-Mimaud V, Barbaud C, Caruelle JP, et al. Bioactive funtionalized polymer of malic acid for bone repair and muscle regeneration. J Biomat Sci Polym Ed, 2000, 11(9): 979–991.
[36] Li SP, Yan YH, Zhang QS, et al. Biodegradable conductive biomedical polymer materials: ZL200810197694.7. 2009-04-08.李世普, 闫玉华, 张青松, 等. 可降解导电生物医用高分子材料: ZL200810197694.7. 2009-04-08.
[37] Sun XM. β-Poly (L-malate) production by Aureobasidium pullulans IFO6353[D]. Tianjin: Nankai University, 2011 (in Chinese).孙秀梅. 利用出芽短梗霉发酵生产β-聚苹果酸的研究[D]. 天津: 南开大学, 2011.
(本文责编 郝丽芳)
Progress in microbial synthesis and application of polymalic acid
Yuanyuan Wang, Yufen Quan, and Cunjiang Song
Key Laboratory of Molecular Microbiology and Technology for Ministry of Education, College of Life Sciences, Nankai University, Tianjin 300071, China
Polymalic acid, known as a bioactive material, is completely biodegradable, and has far reaching application potential in medical field. Combined with our own findings, we summarized advances in polymalic acid metabolism, microbial fermentation synthesis, and application research in the medical field. Finally, prospect for further research was addressed.
December 12, 2013; Accepted: March 28, 2014
Cunjiang Song. Tel/Fax: +86-22-23503866; E-mail: songcj@nankai.edu.cn
Supported by: National Basic Research Program of China (973 Program) (No. 2012CB725204), National High Technology Research and Development Program of China (863 Program) (No. 2012AA021505), National Natural Science Foundation of China (Nos. 31070039, 31170030, 51073081), Tianjin Municipal Key Program (Nos. 13JCYBJC24900, 13JCZDJC27800).
国家重点基础研究发展计划 (973计划) (No. 2012CB725204),国家高技术研究发展计划 (863计划) (No. 2012AA021505),国家自然科学基金 (Nos. 31070039, 31170030, 51073081),天津市重点项目 (Nos. 13JCYBJC24900, 13JCZDJC27800) 资助。
时间:2014-04-01 网络出版地址:http://www.cnki.net/kcms/doi/10.13345/j.cjb.130632.html
Received: December 12, 2013; Accepted: March 28, 2014
Supported by: National Basic Research Program of China (973 Program) (No. 2012CB725204), National High Technology Research and Development Program of Chnia (863 Program) (No. 2012AA021505), National Natural Science Foundation of China (Nos. 31070039, 31170030, 51073081), Tianjin Municipal Key Program (Nos. 13JCYBJC24900, 13JCZDJC27800).
Corresponding author: Cunjiang Song. Tel/Fax: +86-22-23503866; E-mail: songcj@nankai.edu.cn
国家重点基础研究发展计划(973计划)(No. 2012CB725204),国家高技术研究发展计划(863计划)(No. 2012AA021505),国家自然科学基金(Nos. 31070039,31170030,51073081),天津市重点项目(Nos. 13JCYBJC24900,13JCZDJC27800)资助。
网络出版时间:2013-11-27 网络出版地址:http://www.cnki.net/kcms/detail/11.1998.Q.20131127.1120.003.html

