金霉素对畜禽养殖废水厌氧生物处理及细菌多样性的影响
陈峻峰,颜智勇,邵继海,舒 鹏,王 杰,文树龙
(湖南农业大学资源环境学院,湖南长沙 410128)
畜禽养殖废水是一种具有高COD、高氨氮、高SS且难处理的废水[1,2]。四环素类抗生素是规模化养猪场常用的抗生素之一,具有较强的抗菌效应。金霉素是一种典型的四环素类生物抑制药物,对废水生物处理过程中的大多数微生物有明显的抑制作用,含金霉素的畜禽养殖废水的生物处理效果较差[3]。厌氧处理工艺是利用厌氧微生物在无氧情况下通过自身代谢过程将废水中的有机物转化为无机物和少量细胞物质的过程[4]。厌氧生物处理技术可用来处理难降解有机废水也可以提高废水可生化性[5]。
目前研究者对畜禽养殖废水金霉素对厌氧微生物的影响机理尚不清楚,不过现代分子生物学技术为深入研究环境系统中的微生物提供了先进的技术手段[6]。已有一些文献报道了利用分子生物学技术对环境微生物的研究[7,8],但是鲜有利用分子生物学技术针对畜禽养殖废水中金霉素对厌氧微生物群落的影响报道。
通过从受金霉素影响的厌氧颗粒活性污泥中提取总DNA,应用聚合酶链式反应(PCR)-梯度变形凝胶电泳(DGGE)、割胶回收再做聚合酶链式反应(PCR)、克隆后测序以及构建系统发育树等新的分子生物学技术,对受金霉素影响的厌氧活性污泥中的细菌群落进行研究,获得厌氧颗粒污泥中细菌组成和功能以及动态变化规律,从而为进一步更好处理畜禽养殖废水提供科学依据。
1 材料与方法
1.1 厌氧处理工艺及取样
试验用金霉素购自合肥博美生物科技有限责任公司。试验用颗粒污泥取自长沙市某畜禽养殖废水处理厂的UASB反应器,在实验室(35±2)℃稳定培养 48 h,污泥浓度为 40.75 g/L。
试验装置如图1所示。反应瓶容积为500 mL,瓶口用橡胶塞密封,橡胶塞上设置导管,将产生的气体引入3%NaOH溶液,以吸收气体中的CO2,排出的液体即为甲烷产量。向反应瓶中加入100 mL活性颗粒污泥、400 mL配水,一定量的金霉素并且摇匀,调节pH至7.0~7.5,以此探明金霉素对养殖废水厌氧消化的抑制作用。根据金霉素对养猪场废水厌氧消化抑制作用的研究[9],设置金霉素的浓度分别为 0.4、0.8、1.0、2.0、4.0 mg/L,编号分别 1、2、3、4、5进行试验。对照瓶中不添加金霉素,其他条件与受试瓶相同。各反应瓶均做平行试验。将反应瓶置于恒温振荡水浴锅中,定时摇动以保证污泥处于悬浮状态[10]。每天定时测定产甲烷量,直至基本停止产气。每天测定COD浓度,反应结束后取活性污泥于-20℃保存。
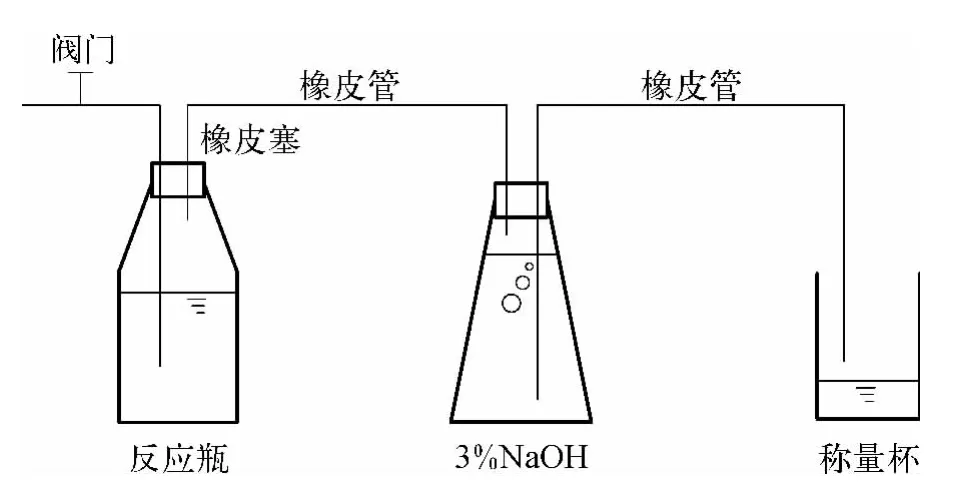
图1 试验装置Fig.1 Experimental Apparatus
1.2 活性污泥总DNA提取
百泰克试剂盒法:采用土壤基因组DNA快速提取试剂盒(离心柱型),购自北京百泰克生物技术有限公司,具体试验步骤参照说明书,提取完成后以1%琼脂糖凝胶电泳检测。
1.3 PCR 扩增
采用上海生工合成的细菌16S rDNA通用引物341F(5’-CCT ACG GGA GGC AGC AG-3’)[11-15]和517R(5’-ATT ACC GCG GCT GCT GG-3’)[15-17]直接对总DNA进行PCR扩增,其中在341F的5’末端加40 bp的 GC夹(5’-CGC CCG CCG GGC CCC GGG CCC GGC CCG CCC CCG CCC G-3’)[12-15],用于后续的DGGE。PCR扩增体系为Easy Taq DNA Polymeerase(TransGen,北京)1 μL(5 U),10 × Easy Taq Buffer 5 μL(200 mmol/L Tris-HCl、200 mmol/L KCl、100 mmol/L(NH4)2SO4、20mmol/L MgSO4),2.5 mmol/L dNTPs 4 μL(0.2 mmol/L),引物为1 μL(20 μmol/L),模板 DNA 为1 μL,其余用双蒸水补足至50 μL体系。PCR反应在上扩增,反应条件:95℃预变性3 min;95℃预变性30 s;65℃退火1 min;72℃延伸1 min,循环33次;72℃延伸7 min;4℃保温。PCR反应后以1%琼脂糖凝胶电泳检测。
1.4 DGGE 分析
将上面步骤得到的PCR产物在DCode System(Bio-Rad,USA)上进行电泳,变性浓度范围为45% ~65%,聚丙烯酰胺凝胶浓度是8%,电压设置为70 V,电泳时间为16 h,温度设定为60℃。电泳结束后,凝胶电泳以0.5 μg/mL的溴化乙锭溶液染色,洗涤数次后于Gel Doc2000凝胶成像系统(Bio-Rad,USA)上检测。
1.5 统计分析
为了了解菌种在受金霉素影响的反应器中的动态变化,对DGGE条带图谱进行了统计分析,重点对细菌种群的丰富度和相关性进行分析。丰富度分析以图谱中所有的条带数为1,不同样品的细菌种群丰富度为该样品的条带数除以图谱中的总条带数(同一位置的条带只能算一条),具体公式如下。

式中,Rsi为第i泳道的细菌种群丰富度值;
Li为第i条泳道上的条带数;
Lt为图谱中的总条带数。
相关性分析主要分析不同样品之间细菌种群的相似性,两条带之间的相似系数可以用Sorenson配对比较相似性系数(Cs)公式[18,19]计算。
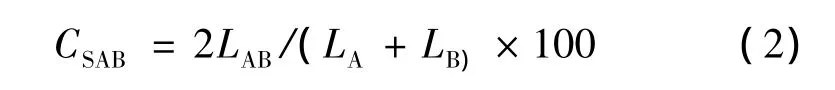
式中,CSAB为泳道A和泳道B之间的相似性系数;
LAB为泳道A上的与泳道B上位置相同的条带数;
LA为泳道A上的条带数;
LB为泳道B上的条带数。
1.6 切胶PCR、克隆和系统发育分析
在紫外灯下切下DGGE条带,切下后放入干净离心管,然后用胶回收试剂盒做胶回收,再对回收的DNA用不含GC夹的341F和517R进行扩增,反应体系同上,得到的PCR产物以1%琼脂糖电泳检测。将得到的PCR产物用pEASY-T1 Cloning Kit(TransGen,北京)进行克隆,最后对挑选的克隆子送交测序公司(Sangon,上海)测序,测序结果提交NCBI获得接受号。使用 NCBI的 GenBank的BLAST对测序结果进行同源性分析,运用分子进化遗传分析软件MEGA5.0中的Kimura2-parameter模型计算各序列间的距离和序列相似性等,采用邻接法(neighbor-joining method,NJ)构建NJ树。
2 结果与讨论
2.1 反应器运行情况
厌氧反应器驯化结束后,投加不同量的金霉素,连续运行7 d,每天监测运行参数(包括pH、COD和产甲烷量),第8 d后产甲烷体积忽略不计且COD的变化也甚微。具体结果如图2、图3所示,图2纵坐标为产甲烷体积以10为底的对数,图3纵坐标为COD的浓度以10为底的对数。
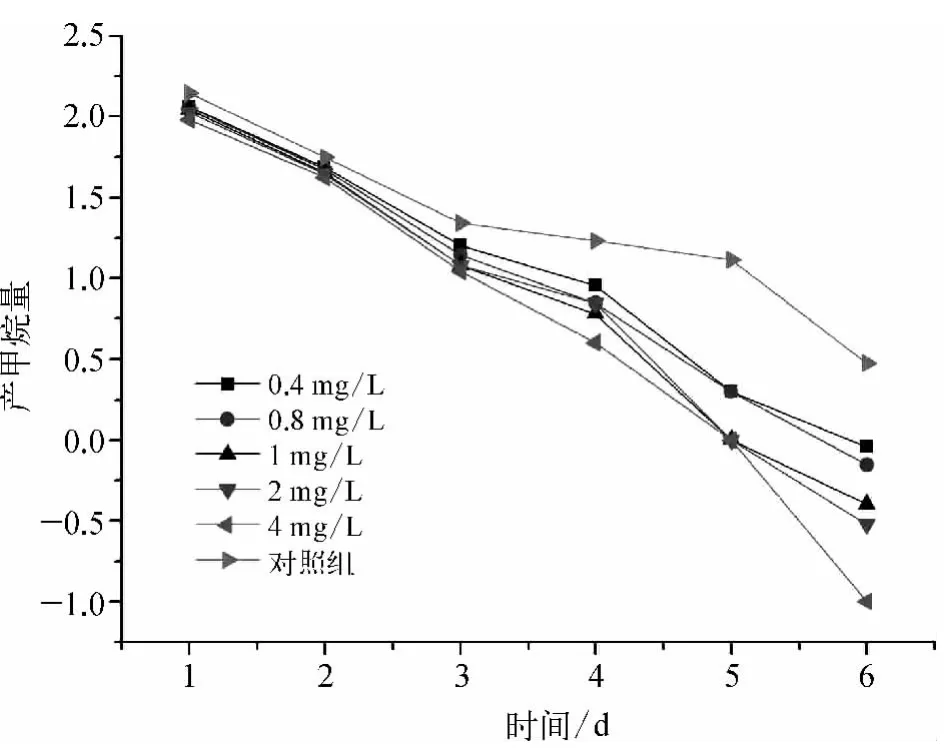
图2 金霉素对禽养殖废水厌氧生物处理系统甲烷产量的影响Fig.2 Effect of Chlortetracycline on Methane Yield in Livestock Wastewater in Anaerobic Biological Treatment System
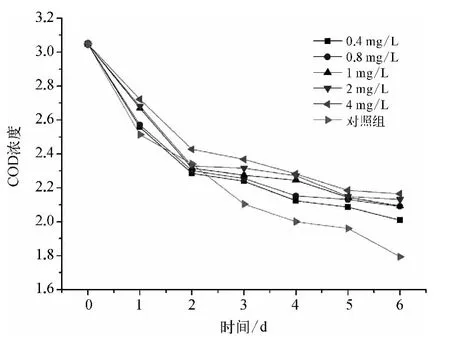
图3 禽养殖废水厌氧生物处理系统中COD浓度的变化情况Fig.3 Changes of COD Concentration in Anaerobic Biological Treatment System
由图2可知在系统运转的前5 d对照组的产甲烷量明显高于试验组,计算得 1、2、3、4、5、6 组的CH4的 总 产 量 分 别 是 242、232、220、196、184、260 mL,试验组与对照组的差值率分别是6.9%、10.8%、15.4%、24.6%、29.2%,说明金霉素对厌氧微生物产甲烷的能力起到抑制作用;随着金霉素浓度的增加,其抑制作用依次增大,两者成正比例关系。到第6 d时所有的试验瓶基本停止产气。由图3可知COD在第1、2 d时降解速度最快,第3 d后平稳降解,对照组COD的浓度明显低于金霉素的试验组,试验组的COD去除率分别是90.89%、89.02%、88.93%、87.95%、86.96%,对照组的 COD 去除率达到94.46%,这说明金霉素对厌氧微生物处理COD的能力起到抑制作用。随着金霉素浓度的增加,其抑制作用依次增大,两者是正比例关系,与其对产甲烷能力的趋势是一致的。
2.2 DNA提取以及PCR扩增
提取的污泥微生物DNA经1%琼脂糖凝胶电泳检测,结果表明初提的微生物总DNA片段长度正常,且获得了较高的量(如图4a)。PCR扩增产物以1%琼脂糖电泳后以溴化乙锭(EB)溶液染色,经凝胶成像系统检测拍照,由图4b可知PCR产物长度为250 bp左右。
图4 DNA提取和PCR产物扩增的琼脂糖电泳图Fig.4 Map of DNA and PCR Products after Agarose Gel Electrophoresis
2.3 DGGE图谱的细菌多样性统计分析
在Gel Doc2000凝胶成像系统下,使用Quantity One V4.52(Bio-Rad,USA)软件对经溴化乙锭(0.5 μg/mL)染色的DGGE凝胶进行成像,在6条泳道上一共观察到23条不同的条带如图5a所示;图5b是根据凝胶成像系统的成像照片,经过系统软件分析后的DGGE图谱示意图,各条带的丰富度值、分布及相关性分析的统计分析结果如表1、表2所示。
由图5、表1可知在1~5泳道中出现的条带都可以在第6泳道中找到,金霉素处理组的条带数均小于对照;而且从第1泳道到第5泳道的Rs值开始变小,这说明随着金霉素浓度的增加,对细菌群落的影响在变大,细菌群落数量在减少。由表2可知从第1泳道到第5泳道依次与第6泳道的对照组相比较发现随着金霉素浓度的增加,其与对照组的相似性系数在减小,同样可以说明金霉素对微生物群落产生了抑制作用。

表1 细菌群落丰富度值Tab.1 Richness Value of Bacterial Community
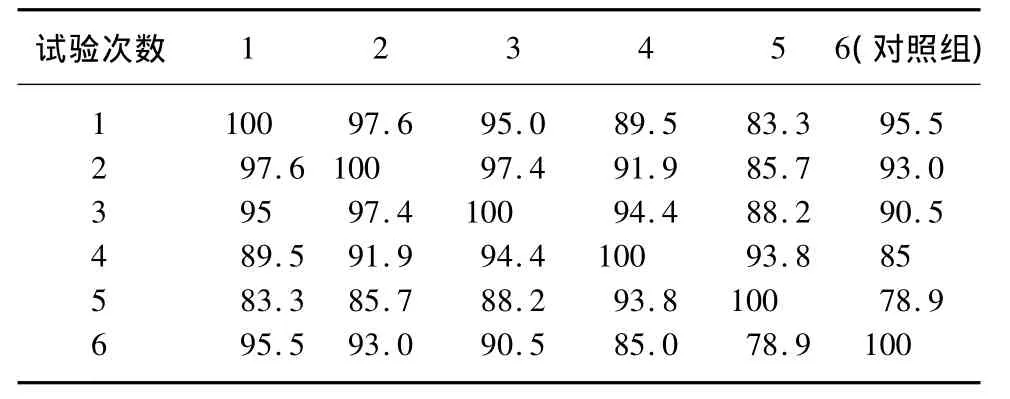
表2 各组细菌种群相似性系数Tab.2 Comparability Index of Bacterial Population in Different Groups

图5 DGGE凝胶成像图及DGGE图谱示意图Fig.5 Gel Imaging and Analysis Map of DGGE
2.4 细菌系统发育分析
由于23条带亮度不同,选取典型且亮度很高的差异性条带进行切胶回收、PCR重扩增、克隆后测序,获得图5a中 A、B、C、D、E 中不同的 16S rDNA序列,提交给GenBank注册,获得临时登录号,条带与临时登录号之间对应关系如表3所示。使用GenBank的BLAST程序,将获得的序列与数据库中的序列进行比对,获得各序列的同源性信息(表3),然后运用分子进化遗传分析软件MEGA5.0中的Kimura2-parameter模型计算各序列间的距离和序列相似性等,采用邻接法(neighbor-joining method,NJ)构建NJ树,如图6所示。

表3 序列的BLAST结果Tab.3 Results of Sequences using BLAST
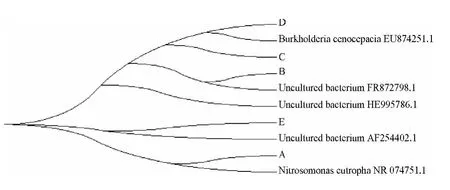
图6 根据序列和BLAST建立的系统发育树Fig.6 Phylogenetic Trees of Bacterial Based on Results of BLAST of Sequences
由表3、图6可知 C与Burkholderia cenocepacia的亲缘关系比较接近,而C在1、2、3、4、对照组中的条带比较弱,这与Chopra等[20]研究发现的四环素类抗生素促使细菌产生耐药性是一致的;A与Nitrosomonas eutropha的亲缘关系比较接近,是硝化过程中一类常见的自养菌,它可以从氧化氨氮的过程中获得能量而生长繁殖,A在6组中均有此类,这与Purkhold等[21]在研究金霉素影响氨氧化细菌多样性的调查中也发现了此菌种;B、C、E在BLAST比对与Uncultured bacterium的亲缘性比较接近,是一类不能纯培养的细菌,此类的细菌与Burkholderia cenocepacia和Nitrosomonas eutropha也存在一定的亲缘关系,说明此类细菌在金霉素的影响下产生耐药性,组成了整个厌氧活性污泥细菌多样性,且对于废水的除碳氮有着一定的作用。
3 结论
(1)经过一段时间的连续监测,受到金霉素影响的试验组会比对照组的COD浓度要高,产甲烷量要小,而且是随着金霉素浓度的增加,产甲烷量差值从6.9%扩大到29.2%,COD去除率从90.89%降至86.96%,而对照组COD去除率达到94.46%。
(2)通过PCR-DGGE技术对厌氧活性污泥中细菌多样性统计的分析,发现随着金霉素浓度的增加,厌氧活性污泥中的细菌种群丰富度Rs从0.913到0.652;对各组的系数比较也可以看出随着金霉素浓度的增加,其与对照组的差距也在增大,相似性系数 CS从97.6 到 78.9。
(3)PCER-DGGE技术对于监测微生物群落结构和动态变化进行监测,结合DNA测序、序列同源性分析和系统发育性分析对厌氧活性污泥中微生物做出种属鉴定[6],可为研究畜禽养殖废水中金霉素处理机理提供一定的理论依据。
[1]段妮娜,董滨,何群彪,等.规模化养猪废水处理模式现状和发展趋势[J].净水技术,2008,27(4):9-15,37.
[2]颜智勇,吴根义,刘宇赜,等.UASB/SBR/化学混凝工艺处理养猪废水[J].中国给水排水,2007,23(14):66-68.
[3]孙建平,郑平,胡宝兰.金霉素对猪场废水厌氧消化抑制作用[J].哈尔滨工业大学学报,2010,42(4):652-655
[4]王宝贞.水污染治理新技术、新工艺、新概念、新理论[M].北京:科学出版社,2004.
[5]Speece R E.李亚新译.工业废水的厌氧生物技术[M].北京:中国建筑工业出版社,2001.
[6]肖勇,杨朝晖,曾光明,等.PCR-DGGE研究处理垃圾渗滤液序批式生物膜反应器(SBBR)中的细菌多样性[J].环境科学,2007,28(5):1095-1101.
[7]Kawai M,Matsutera E,Kanda H,et al.16S ribosomal DNA-based analysis of bacterial diversity in purified water used in pharmaceutical manufacturing processes by PCR and denaturing gradient gel electrophoresis [J].Appl.Environ.Microbiol.,2002,68(2):699-704.
[8]苏俊峰,马放,王弘宇,等.利用 PCR-DGGE技术分析生物陶粒硝化反应器中微生物群落动态[J].环境科学学报,2007,27(3):386-390.
[9]孙建平.抗生素与重金属对猪场废水厌氧消化的抑制效应及其调控对策[D].杭州:浙江大学,2009.
[10]朱晓磊,王路光,王靖飞,等.土霉素对厌氧生物处理的抑制作用研究[J].中国给水排水,2010,26(1):93-95.
[11]Yoshida N,Yagi K,Sato D,et al.Bacterial communities inpetroleum oil in stockpiles[J].Journal of Bioscience and Bioengineering,2005,99(2):143-149.
[12]Ishii K,Fukui M.Opt imization of annealing temperature to reducebias caused by a primer mismatch in multitemplate PCR[J].Applied and Environmental Microbiology,2001,67(8):3753-3755.
[13]Veeh R H,Sumithraratne P.Monitoring of microbial souring in chemically treated,produced-water biofilm systems using molecular techniques[J]. Journal of Industrial Microbiology and Biotechnology,2005,32(4):163-170.
[14]Ferris M J,Muyzer G,Ward D M.Denaturant gradient gel electrophoresis profiles of 16S rRNA-defined populations inhabiting a hot spring microbial community[J].Applied and Environmental Microbiology,1996,62(1):340-346.
[15]Muyzer G,Brinkhoff T,Ulrich N,et al.Denaturing gradient gel electrophoresis(DGGE)in microbialecology[J].Molecular Microbial Ecology Manual,1998,34(4):1-27.
[16]Suzuki K,Koyanagi M,Yamashita H.Genetic characterization and specific detect ion of beer-spoilage Lactobacillus sp.LA2 and related strains[J].Journal of Applied Microbiology,2004,96(4):677-683.
[17]Hashizume T,Takai C,Naito M,et al.Characteristics of the mucus layer on the surface of the bluegill(Lepomis macrochirus)and the bacterial flora in the mucus[J].Microbes and Environments,2005,20(1):69-80.
[18]Gillian D C,Speksnijder A G C L,Zwart G,et al.Genetic diversity of the biofilm covering Montacuta ferruginosa(Mollusca,Bivalvia)as evaluated by denaturing gradient gel electrophoresis analysis and cloning of PCR amplified gene fragment coding for 16SrDNA[J].Applied and Environmental Microbiology,1998,64(9):3464-3472.
[19]Murray A E,Hollibaugh J T,Orrego C.Phylogenetic composition of bacterioplankton from two California estuaries compared by denaturing gradient gel electrophoresis of 16S rDNA fragments[J].Applied and Environmental Microbiology,1996,62(7):2676-2680.
[20]Ian Chopra,Marilyn Roberts.Tetracycline Antibiotics:Mode of Action,Applications,Molecular Biology,and Epidemiology of Bacterial Resistance[J].Microbiology and Molecular Biology Reviews,2001,65(2):232-260.
[21]Purkhold U,Pommerening-Röser A, Juretschko S, et al.Phylogeny of all recognized species of ammonia oxidizers based on comparative 16S rRNA and amoA sequence analysis:implications for molecular diversity surveys[J].Applied and Environmental Microbiology,2000,66(12):5368-5382.

