无患子SRAP-PCR反应体系的优化
彭珠清,范辉华,刘 宝,刘 燕,李 静
(1.福建农林大学 林学院,福建 福州 350002;2.福建林业科学研究院,福建 福州 350012)
无患子Sapindus mukurossi属于无患子科Sapindaceae无患子属Sapindus,是一种非常有潜力的经济树种,其果可以用来制作洗涤剂,种仁能提取生物柴油,树形美观,可用于园林观赏等。日本、法国、中国台湾等对其蕴含的经济价值有一定的开发利用,而中国大陆虽早在几百年前就已对其有初步利用,但深度开发这方面却尚在起步阶段[1-3]。目前,对无患子的研究主要集中在生物学特性、繁殖技术[1,4]、化学成分和分离提取技术及其利用价值的开发研究[5-7]等方面,而在分子生物学方面的研究报道不多[8-9]。分子标记技术是研究种质资源遗传背景的重要手段,2001年美国加州大学蔬菜作物系Li与Quiros博士提出了一种新型的基于聚合酶链式反应(PCR)的标记方法的相关序列扩增多态性(sequence-related ampli-fied polymorphism,SRAP)标记方法[8,10]。该标记通过独特的双引物设计对基因 的 ORFs(open reading frames)的特定区域进行扩增。与其他分子标记相比,该方法具有简单高效、多态性高、重复性较好等特点,并已成功应用于多种蔬菜、经济树种的种质资源分析中[5-7,11-13]。但在无患子的遗传多样性以及种质鉴定等方面还鲜见报道。为此,本研究以无患子叶片为材料,采用单因素优化的方法建立无患子SRAPPCR的最佳反应体系,旨在为不同地理种源无患子的遗传多样性分析、亲缘关系鉴定、遗传图谱构建、遗传改良等奠定基础。
1 材料与方法
1.1 材料来源
试验材料采于福建顺昌无患子种源收集区,共12个种源地36份种质材料。本优化试验以福建南平种源地的基因组DNA为试验材料。
1.2 主要试剂
试验中所用的三磷酸碱基脱氧核苷酸(dNTP)s,TaqDNA酶,琼脂糖和DNA标记物均购自上海生工生物工程有限公司,其中100 bp标记物是加DNA梯度。所用的SRAP引物序列由上海生物工程(上海)股份有限公司合成,其序列参照 Li等[10]和 Ren 等[14]的方法。
优化试验引物选用me2-em7组合,体系验证引物选用me2-em7和me6-em1,序列具体如表1。
1.3 无患子总DNA的提取
无患子总DNA提取采用改良的十六烷基三甲基溴化铵(CTAB)法[15-16]。DNA 的浓度和纯度,分别用质量浓度为1%的琼脂糖凝胶电泳和紫外分光光度计分析检测。样品稀释至 10 μg·L-1后,置-20 ℃冰箱保存备用。
1.4 反应体系的优化
SRAP-PCR基本反应体系:在20.0 μL总体积中,含 2.0 μL 10×PCR 缓冲液(free Mg2+),2.0 mmol·L-1镁离子(Mg2+),0.2 mmol·L-1dNTPs,2.0×16.67 nkat(2.0 U) TaqDNA 聚合酶,正反引物各 0.5 μmol·L-1,用双蒸水补足至20 μL。
SRAP-PCR扩增程序为:94℃预变性3 min;94℃变性l min,35℃复性l min,72℃延伸l min;共5个循环。随后94℃变性l min,50℃复性1min,72℃延伸l min,共35个循环;最后72℃延伸10 min。PCR仪为ABI梯度PCR仪。
本试验采用单因素法对反应体系进行优化,分别调整模板DNA,镁离子,dNTPs,引物,TaqDNA聚合酶各因子的用量(表2)。
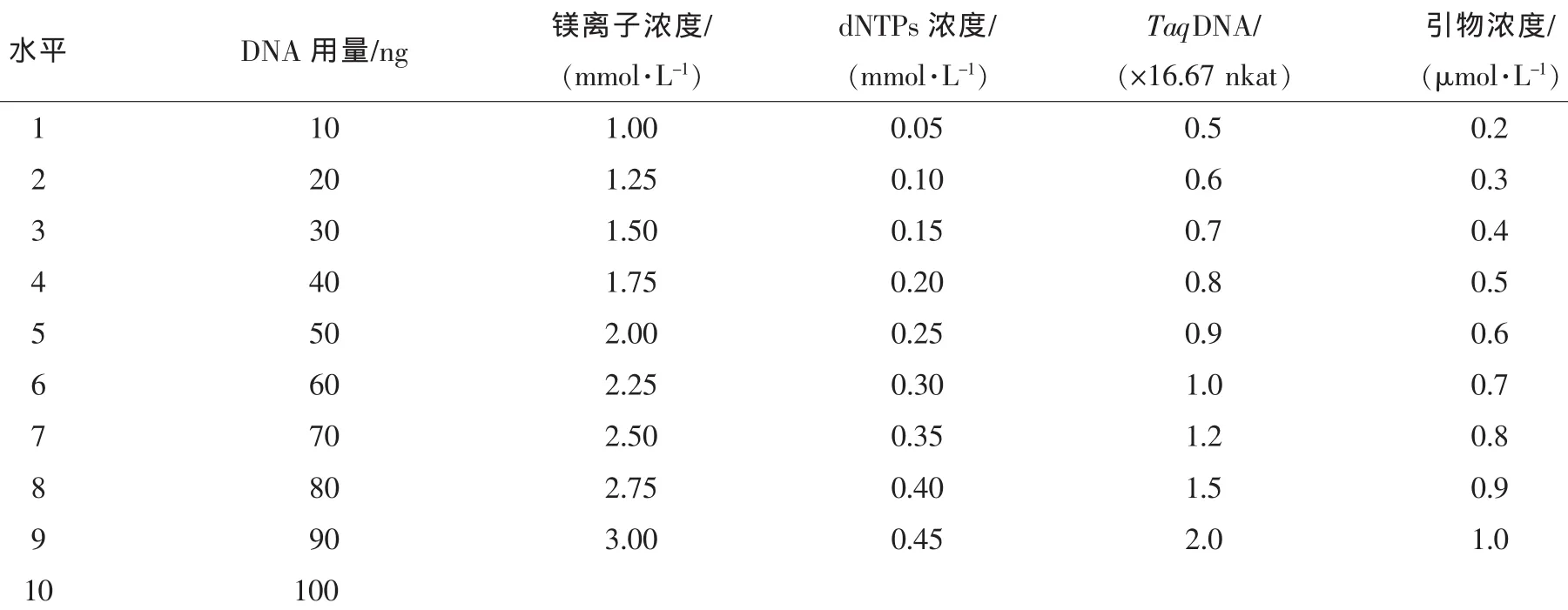
表2 反应体系各因子水平Table2 Reaction system in each factor level
1.5 PCR产物的检测
将PCR产物与上样缓冲液(5:1)混合后于质量分数为1.5%琼脂糖凝胶上电泳,用溴化乙锭(EB)染色后,在凝胶成像系统(AlphaImager HP)上观察。
2 结果与分析
2.1 无患子基因组DNA检测
无患子叶片内富含多酚、多糖、蛋白质及其他次生代谢物质[14],操作时易发生褐变,干扰DNA的提取,这些物质若残留在DNA样品中将会严重影响PCR的效果,导致DNA的酶切、扩增失败,对后续试验研究的开展有很大的影响。本研究采用改良的CTAB法,成功提取出质量较高的基因组DNA,条带较为清晰、完整、无拖尾,符合SRAP分子标记对DNA质量的要求(图1)。
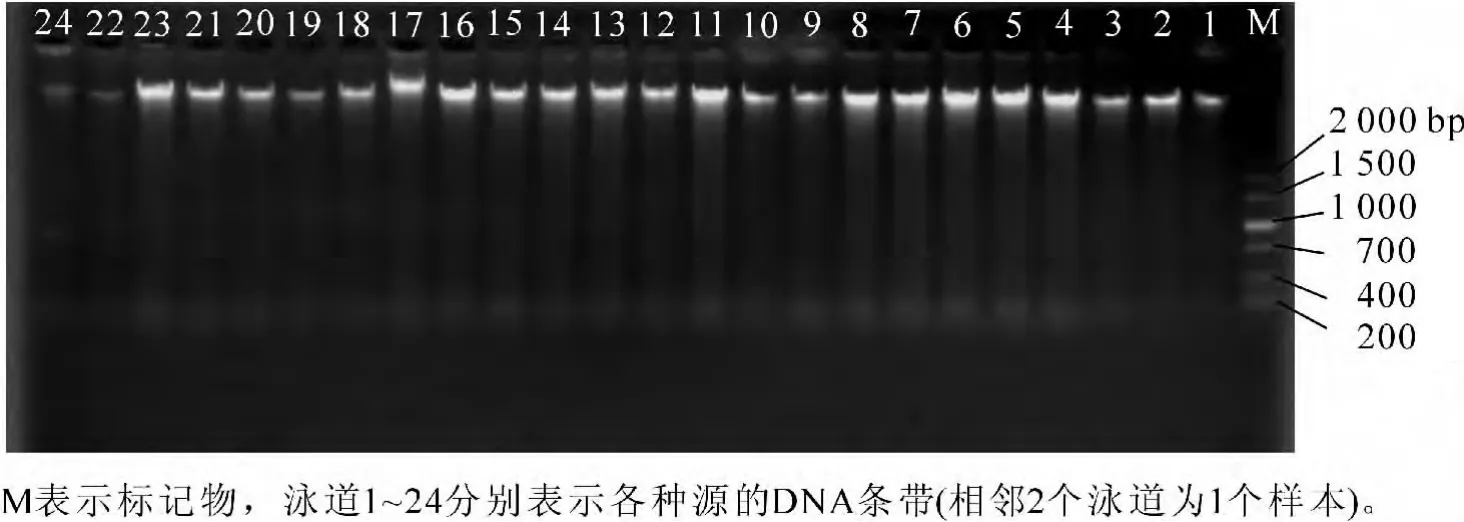
图1 不同无患子总DNA电泳检测图Figure1 Electrophoresis of total genomic DNA from 12 different Sapindus mukurossi provenances
经紫外分光光度计测定,其D260/D280为1.2~2.2(表3),经纯化均能达到1.8左右的比值。所有样品质量浓度均大于430 μg·L-1,在SRAP分析中可获得较为清晰、稳定的扩增结果产物。
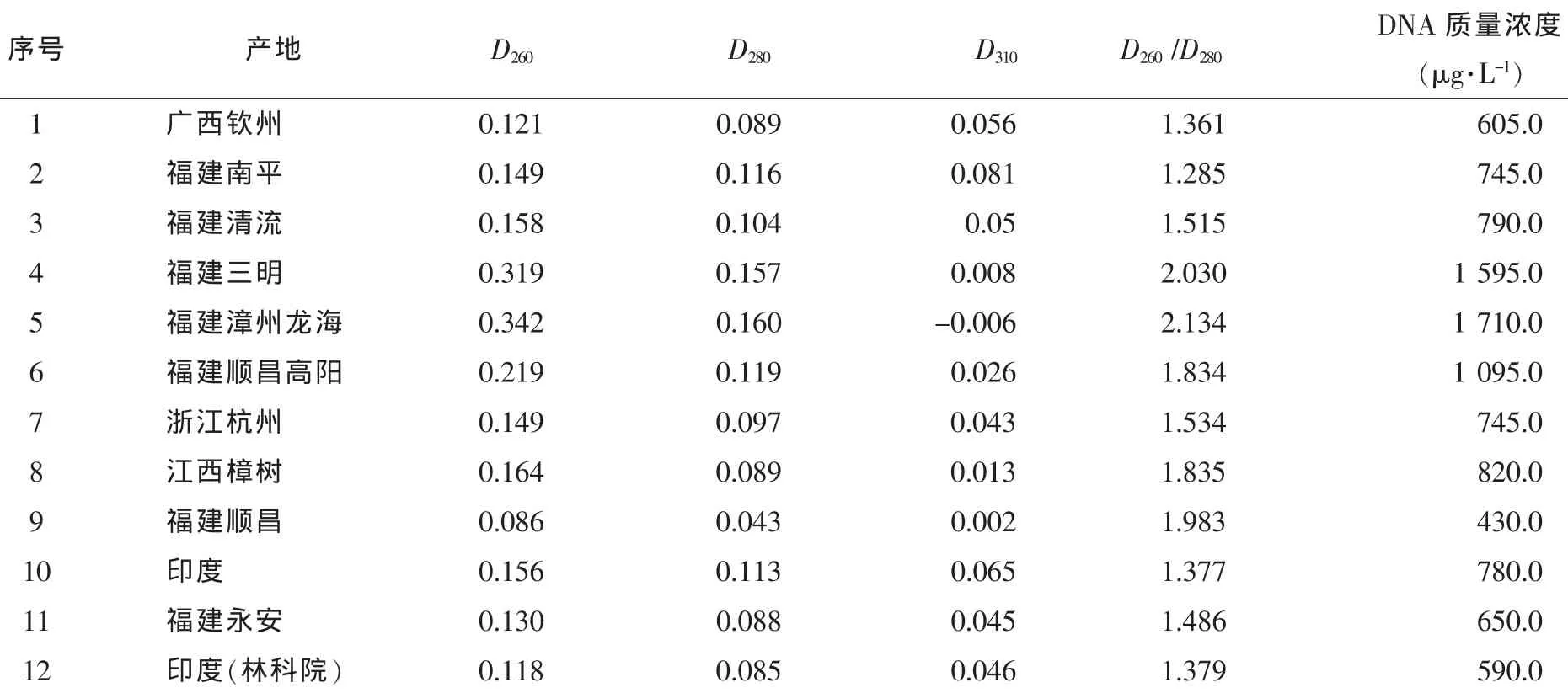
表3 无患子总DNA浓度检测Table3 Concentration detection of total genomic DNA from 12 different Sapindus mukurossi provenances
2.2 模板DNA对PCR扩增的影响
本试验设定了10个用量梯度(10~100 ng),研究不同模板DNA用量对PCR扩增的影响。模板DNA使用量在设定范围内,都能扩增出几乎相同的清晰条带(图2)。10 ng时条带比较模糊;70~100 ng时,条带较淡、呈波浪状,20~60 ng时主带比较清晰。DNA较长时间保存会部分降解,致使有效含量降低。考虑到试验时间较长,为避免DNA降解对试验结果的影响,最终选择50 ng作为模板优化用量。
2.3 镁离子浓度对PCR扩增的影响
镁离子浓度对PCR扩增影响较大。镁离子浓度过高时,TaqDNA聚合酶催化非特异性扩增,降低反应的忠实性;反之,镁离子浓度过低时,则又会使TaqDNA聚合酶的活性下降,反应产物减少。当镁离子浓度为 1.00 mmol·L-1时,未扩增出条带;当 Mg2+浓度为 1.25~1.75 mmol·L-1时,条带模糊,出现缺失现象;当镁离子浓度为2.00~2.50 mmol·L-1时,扩增带型基本一致;当镁离子浓度增至2.75,3.00 mmol·L-1时,条带呈弥散趋势(图3)。高浓度镁离子易产生非特异性产物,为避免高浓度镁离子对后续试验的影响。本研究最终选择2.00 mmol·L-1作为镁离子的优化浓度。
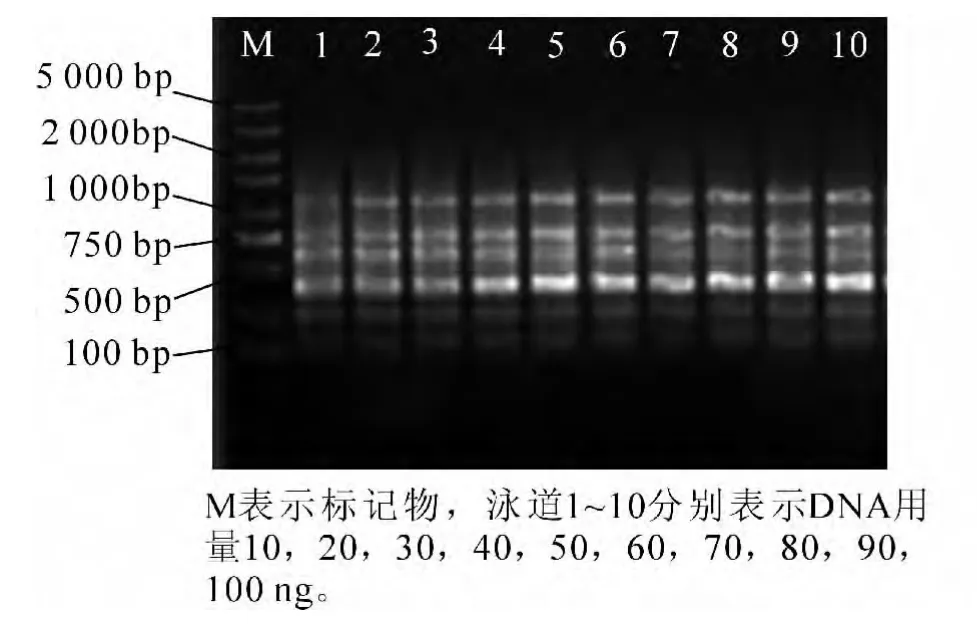
图2 模板DNA用量对PCR扩增的影Figure2 Effect of DNA template concentration on PCR amplification
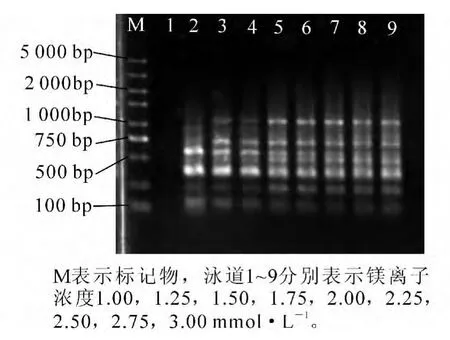
图3 镁离子浓度对PCR扩增的影响Figure3 Effect of Mg2+concentration on PCR amplification
2.4 dNTPs浓度对PCR扩增的影响
dNTPs对SRAP-PCR反应有极其重要的作用,dNTPs浓度过高会增加DNA聚合酶的错配率,而浓度过低又会降低产量。dNTPs浓度为 0.05~0.15 mmol·L-1时,条带较少且模糊,0.20~0.25 mmol·L-1时条带较为清晰,在0.30~0.45 mmol·L-1时条带又开始模糊,最终确定0.20 mmol·L-1为dNTPs的优化浓度(图 4)。
2.5 TaqDNA聚合酶用量对PCR扩增的影响
TaqDNA聚合酶的用量与反应体积、酶活性等因素有关。一般酶用量过多会使非特异性增加、弥散背景增强,用量过少则会降低产量,使条带不够明亮。当TaqDNA聚合酶用量为0.5×16.67 nkat(0.5 U)和1.0×16.67 nkat(1.0 U)时,条带均较多,但0.5×16.67 nkat(0.5 U)时,条带亮度更佳;当TaqDNA聚合酶用量(0.6~0.9)×16.67 nkat(0.6~0.9 U)时,条带较少;当 TaqDNA 聚合酶用量为(1.2~2.0)×16.67 nkat(1.2~2.0 U)时,条带少(图 5)。综上,当 TaqDNA 聚合酶用量为 0.5×16.67 nkat(0.5 U)时,扩增效果最理想,以此作为TaqDNA聚合酶的优化用量。
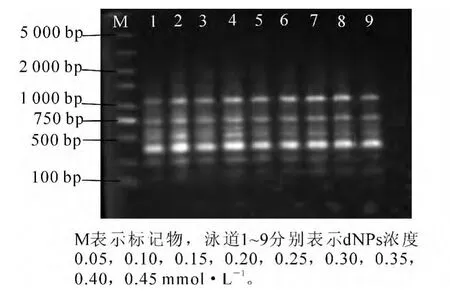
图4 dNTPs浓度对PC R扩增的影响Figure4 Effect of dNTPs concentration on PCR amplification
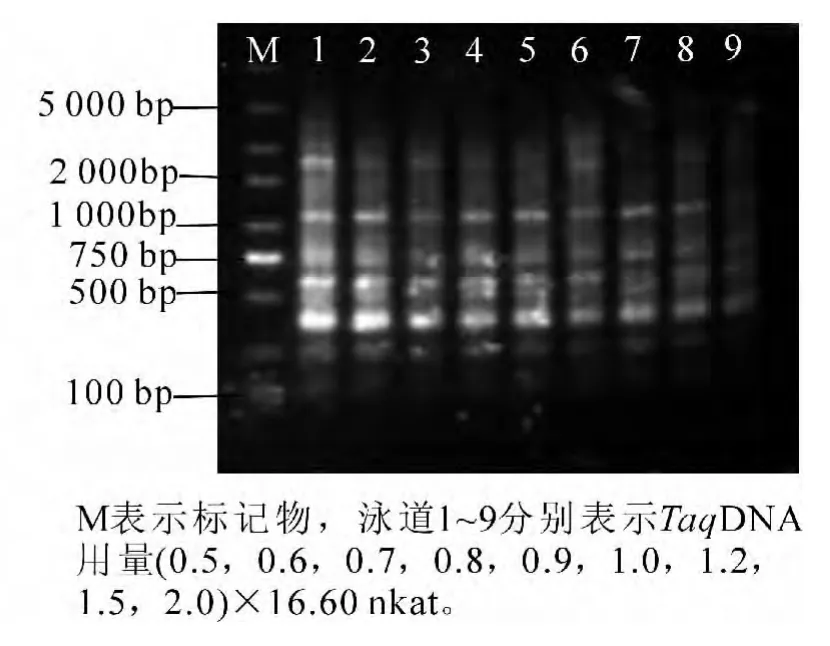
图5 TaqDNA用量对PCR扩增的影响Figure5 Effect of TaqDNA concentration on PCR amplification
2.6 引物浓度对PCR扩增的影响
引物过多会产生引物二聚体,引物过低则降低产量。在设定的引物浓度范围内,PCR产物量基本相同(图 6)。当引物浓度为 0.2~0.4 μmol·L-1有较清晰、 一致的条带,且 0.4 μmol·L-1时,条带较亮;当引物浓度为 0.5~1.0 μmol·L-1开始条带渐变弱,扩增效果不佳,均较模糊。综上,选择 0.4 μmol·L-1作为引物的优化浓度。
2.7 SRAP-PCR反应体系的确立
本研究用引物组合m2e7和m6e1在无患子材料中进行SRAP-PCR优化反应体系验证,扩增结果比较稳定、条带清晰(图7),且引物组合m6e1扩增出的多态性条带较引物组合m2e7多,表现出更高的多态性。由此说明,该体系适宜无患子SRAP-PCR反应。
通过对反应体系的验证,从结果的稳定性和经济性出发,确立了适合无患子的SRAP-PCR反应体系为:在20 μL反应体系中,含模板DNA为50 ng,镁离子 2.0 mmol·L-1,dNTPs 0.2 mmol·L-1,TaqDNA聚合酶 0.5×16.67 nkat(0.5 U),引物 0.4 μmol·L-1。
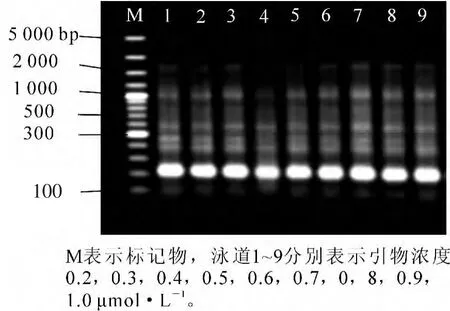
图6 引物浓度对PCR扩增的影响Figure6 Effect of primers concentration on PCR amplification
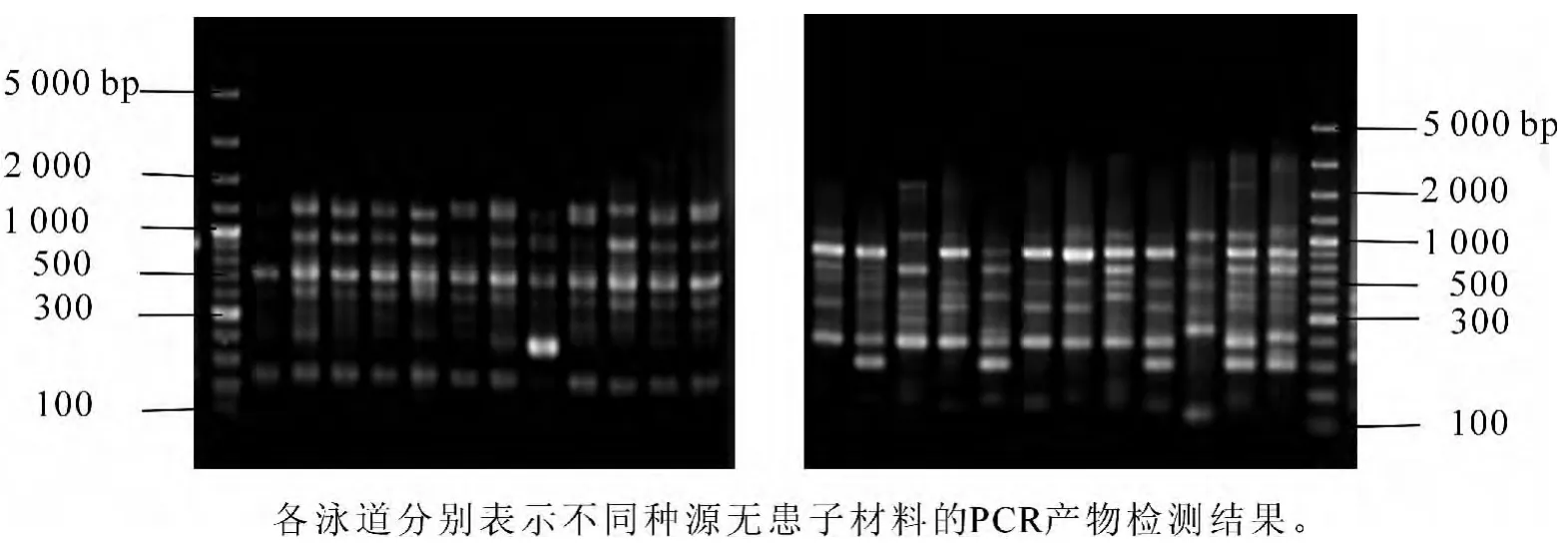
图7 优化体系验证图(左m2e7,右m6e1)Figure7 Optimized system verification(left m2e7,right m6e1)
3 讨论
SRAP标记是一种基于PCR的新型分子标记技术,具有以下优点:①有一套通用引物,可任意搭配,通过筛选获取适合引物对,极大降低了引物合成费用。②反应条件相对固定,试验重复性较好,操作简易。因此,在林木种质资源遗传多样性、品种鉴定等方面应用前景广阔[17-19]。
SRAP-PCR反应体系主要包含5个影响因素,即DNA模板、镁离子、dNTPs、引物和TaqDNA聚合酶。各个因素用量不同,将明显影响扩增效果,进而影响SRAP分子标记的可靠性。高质量DNA的获取是PCR成功的根本性因素,DNA中残留的蛋白、酚类等物质,会抑制DNA聚合酶。本研究中,采用改良的CTAB法提取了较高质量的DNA,符合SRAP-PCR反应要求。
PCR混合物中,DNA模板、引物和dNTPs的磷酸碱基均可与镁离子结合,降低镁离子的实际浓度;而对TaqDNA聚合酶起作用的是游离的镁离子,因此,镁离子加入量应高于dNTPs[13]。本试验中,镁离子浓度比dNTPs高出1.8 mmol·L-1。PCR扩增产物的大小是由引物限定的,引物浓度过高,不仅会促进非特异性产物的合成,而且还会增加引物二聚体的形成,本研究最终选择0.4 μmol·L-1作为引物的优化浓度。
本试验采用传统的单因素法,确立了适合无患子的SRAP-PCR反应体系。利用该体系对12个种源地的无患子进行扩增,得到了稳定清晰的条带,效果良好,说明优化后的体系适宜无患子种质资源的分子标记分析,但需对SRAP引物进行筛选。SRAP-PCR优化体系的建立,为SRAP分子标记在无患子地理种源多态性分析、遗传图谱构建、品种鉴定等方面打下良好的基础。
[1]黄素梅,王敬文,杜孟浩,等.无患子的研究现状及其开发利用[J].林业科技开发.2009,23(6)∶1-5.HUANG Sumei,WANG Jingwen,DU Menghao,et al.Research Situation and Utilization of Sapindus mukorossi Gaerth.[J].China For Sci Technol,2009,23(6)∶1 - 5.
[2]贾黎明,孙操稳.生物柴油树种无患子研究进展[J].中国农业大学学报,2012,17(6)∶191-196.JIA Liming,SUN Caowen.Research progress of biodiesel tree Sapindus mukorossi[J].J China Agric Univ,2012,17(6)∶191 - 196.
[3]姜翠翠,叶新福,卢新坤,等.无患子研究进展概述[J].福建农业学报,2013,28(4)∶405-411.JIANG Cuicui,YE Xinfu,LU Xinkun,et al.Research progress on Sapindus mukorossi[J].Fujian J Agric Sci,2013,28(4)∶405 - 411.
[4]林文荣.无患子扦插繁殖试验研究[J].现代园艺,2007(7)∶9-10.LIN Wenrong.Research cutting propagation of Sapindus mukorossi Gaerth.[J].Mod Hortic,2007(7)∶9 - 10.
[5]乔燕春,林顺权,刘成明,等.SRAP分析体系的优化及在枇杷种质资源研究上的应用[J].果树学报,2008,25(3)∶348 - 352.QIAO Yanchun,LIN Shunquan,LIU Chengming,et al.Optimization of SRAP analysis and its application in germplasm research of loquat(Eriobotrya japonica) [J].J Fruit Sci,2008,25(3)∶348 - 352.
[6]刘丽娟,刘灶长,陈海荣,等.SRAP标记技术及其在蔬菜作物遗传多样性分析中的应用[J].中国农学通报,2009,25(21)∶43 - 48.LIU Lijuan,LIU Zaochang,CHEN Hairong,et al.SRAP marker technique and its application in genetic diversity analyses of vegetable crops [J].Chin Agric Sci Bull,2009,25(21)∶43 - 48.
[7]昝逢刚,吴转娣,曾淇,等.荔枝SRAP-PCR反应体系的优化[J].基因组学与应用生物学,2009,28(1)∶132-136.JIU Fenggang,WU Zhuandi,ZENG Qi,et al.Optimization of SRAP-PCR reaction system in litchi[J].Genomics Appl Biol,2009,28(1)∶132 - 136.
[8]MAHAR K S,RANA T S,RANADE S A.Molecular analyses of genetic variability in soap nut (Sapindus mukurossi Gaertn.) [J].Ind Crops Prod,2011,34(1)∶1111 - 1118.
[9]洪莉,柏明娥,张加正,等.无患子种质资源多样性与亲缘关系的ISSR和SRAP分析[J].浙江农业科学,2013(5)∶556 - 568,570.HONG Li,BAI Ming’e,ZHANG Jiazheng,et al.Genetic diversity and relationship analysis of Sapindus mukorossi germplasm by ISSR and SRAP molecular markers [J].J Zhejiang Agric Sci,2013(5)∶556 - 568,570.
[10]LI G,QUIROS C F.Sequence-related amplified polymorphism (SRAP),a new marker system based on a simple PCR reaction∶its application to mapping and gene tagging in Brassica [J].TAG Theor Appl Gen,2001,103(2)∶455 -461.
[11]柳李旺,龚义勤,黄浩,等.新型分子标记:SRAP与TRAP及其应用[J].遗传,2004,26(5)∶777-781.LIU Liwang,GONG Yiqin,HUANG Hao,et al.Novel molecular marker systems:SRAP and TRAP and their application[J].Hereditas,2004,26(5)∶777 - 781.
[12]赵玉辉,郭印山,傅嘉欣,等.龙眼SRAP反应体系的建立和优化[J].中国农学通报,2009,25(18)∶409-412.ZHAO Yuhui,GUO Yinshan,FU Jiaxin,et al.Establishment and optimization of SRAP reaction system in longan[J].Chin Agric Sci Bull,2009,25(18) ∶409 - 412.
[13]唐琴,曾秀丽,廖明安,等.大花黄牡丹遗传多样性的SRAP分析[J].林业科学,2012,48(1)∶70-76.TANG Qin,ZENG Xiuli,LIAO Ming’an,et al.SRAP analysis of genetic diversity of Paeonia ludlowii in Tibet[J].Sci Silv Sin,2012,48(1)∶70 - 76.
[14]REN Na,LIU Jiajia,YANG Dongliang,et al.Sequence-related amplified polymorphism (SRAP) marker as a new method for identification of endophytic fungi from Taxus[J].World J Microbiol Biotechnol,2012,28(1)∶215 - 221.
[15]刘成明,梅曼彤.无患子科果树植物DNA的高效提取及RAPD分析体系的优化[C]∥中国园艺学会.中国园艺学会第5届青年学术讨论会论文集.广州∶[出版者不详],2002∶297-302.
[16]周延清.DNA分子标记技术在植物研究中的应用[M].北京:化学工业出版社,2005:5-45.
[17]徐操,赵宝华.SRAP分子标记的研究进展及其应用[J].生命科学仪器,2009,7(4)∶24-27.XU Cao,ZHAO Baohua.The development and application of SRAP molecular markers [J].Life Sci Instrum,2009,7(4)∶24 - 27.
[18]林萍,姚小华,王开良,等.油茶长林系列优良无性系的SRAP分子鉴别及遗传分析[J].农业生物技术学报,2010,18(2)∶272 - 279.LIN Ping,YAO Xiaohua,WANG Kailiang,et al.Identification and genetic analysis of Camellia oleifera Changlin series superior clones by SRAP molecular marker [J].J Agric Biotechnol,2010,18(2)∶272 - 279.
[19]AMAR M H,BISWAS M K,ZHANG Z,et al.Exploitation of SSR,SRAP and CAPS-SNP markers for genetic diversity of Citrus germplasm collection [J].Sci Hort,2011,128(3)∶220 - 227.

