反硝化酶及其环境影响因子的研究进展
李卫芬 郑佳佳 张小平 邓 斌
(浙江大学动物科学学院, 杭州 310058)
反硝化酶及其环境影响因子的研究进展
李卫芬 郑佳佳 张小平 邓 斌
(浙江大学动物科学学院, 杭州 310058)
随着全球经济的迅速发展, 环境问题特别是水环境污染问题日益严重。现代农业长期大量使用化肥, 养殖水体积累了大量的鱼类粪便和饵料残渣, 导致水体氮素含量严重超标, 形成水体氮污染, 其中亚硝酸盐能氧化鱼虾体内的亚铁血红蛋白, 使其成为高铁血红蛋白, 丧失运输氧气的能力, 导致水生生物大批患病甚至死亡, 严重影响水质和水产品安全[1]。近年来国内外对氮污染的治理日益重视, 目前微生态技术是当前水环境污染治理的研究热点[2]。微生物的反硝化作用是地球氮素循环的重要分支, 在水产养殖污染水体脱氮修复中起重要作用。其关键酶与细菌反硝化性能的强弱密切相关, 许多学者对反硝化细菌的各种反硝化酶进行研究[3]。本文从反硝化酶系和环境因子两方面, 对反硝化作用的影响进行了系统论述, 旨在为反硝化细菌在水体氮污染控制的深入研究提供理论基础。
1 反硝化关键酶
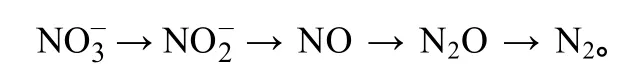
1.1 硝酸还原酶
细菌好氧反硝化的第一步是硝酸盐还原为亚硝酸盐,由硝酸还原酶催化。反硝化菌通常包含一种或两种硝酸还原酶, 一种是膜结合硝酸还原酶(NAR), 另一种为周质硝酸还原酶(NAP)。NAR在厌氧条件下优先表达, 且仅在厌氧状态下发挥作用; NAP在有氧条件下优先表达, 也存在于厌氧生长的细胞中, 当 NAR被氧抑制时, 它仍具有硝酸还原能力[4]。
NAR NAR是一个由 3个亚基组成的复合物NarGHI, 属于钼喋呤氧化还原酶家族[5]。亚基 N arI将酶固定到膜上, 接受来自醌的电子, 通过两个亚铁血红素 b将电子传递到 NarH; NarH通过铁硫键将电子传递到NarG胞质一侧的活性位点—钼协作因子或核苷钼辅因子;同时硝酸盐在还原过程中形成 N和 H2O, 释放到周质,电子传递到NarI[6]=。氧气以限制硝酸根通过细胞质膜来间接抑制此酶的活性, 使之在有氧条件下失活。NAR存在于Escherichia coli、Pseudomons denitrificans、Bacillus licheniformis、Bacillus stearothermophilus、Paracoccus denitrification等微生物中[7]。
NAP NAP是由2 个亚单位(NapA和NapB)组成的二聚体。大亚基NapA由基因napA编码, 为还原硝酸盐的作用位点, 包含辅因子钼蝶呤鸟苷二核苷酸(MGD)和一个铁硫中心。NapB由基因napB编码, 为二血红素细胞色素C552[8]。铁硫中心协助血红素c和MGD之间的电子传递。NapC编码一种四血红素c型细胞色素, 有两个结构域, 每个结构域包含一个双血红素, 每个血红素末梢都有一个组氨酸残基。NapC在周质中表达, 负责将电子从泛醌传递到NapAB[7]。与NAR相比, NAP对硝酸根有更高的底物专一性, 对叠氮化物的抑制不敏感; 硫氰酸盐可以竞争性地与NAP结合, 是 N AP的一种抑制剂[7]。目前, 一些细菌, 如Haemophilus influenzae、Rhodobacter sphaeroides、Pseudomonas sp.和Sulfurimonas denitrifcans等编码NAP的基因先后被鉴定[9]。
1.2 亚硝酸还原酶
在反硝化过程中, 由亚硝酸还原酶(NIR)催化亚硝酸盐转变成一氧化氮, 实现了水体中的氮向大气中氮的转变, 在反硝化过程中扮演了重要角色。NIR分布于细胞壁与细胞膜之间, 根据其辅基的不同一般分为由 nirS编码的血红素cd1型亚硝酸还原酶(cd1-NiRs)和由nirK编码的铜型亚硝酸还原酶(Cu-NiRs), 它们分别以血红素 cd1和Cu作为辅基[5]。通常由于反硝化NIR类型有排外性, NirK和 NirS虽然等价, 生态位分化导致每个菌体只存在其中的一种[10]。
Cu-NiR Cu-NiR是可溶性三角形酶, 每个单体有两个铜中心, Ⅰ型和Ⅱ型[11]。Ⅰ型Cu为电子进入位点, 主要的电子供体为天青蛋白和假天青蛋白[12], Ⅱ型Cu为底物结合位点, 受His100, His135和 His306调控。电子从Ⅰ型Cu中心, 通过Asp和His的化学途径传递到Ⅱ型Cu中心, 还原 N为 N O。有研究证实 T hiosphoera pantotropha、Bdellovibrio bacteriovorus、Chromobacterium violaceum、Pseudoalteromonas haloplanktis和 Bacillus azotoformans中都存在Cu-NiR[12]。此外, 一些Neisseria菌中存在另一种位于外部膜的亚硝酸盐还原酶 AniA, 为Cu-NiR的同源体[13]。
cd1-NiR cd1-NiR是一种双功能酶, 催化 N得到一个电子转变成 N O, 使O2得到 4 个电子生成水[14]。细胞色素cd1-NiR为同二聚体周质蛋白质, 每个单体上含有两个亚铁血红素基团, c型和d1型[7]。其电子供体为天青蛋白、细胞色素c551或假天青蛋白, 电子通过c型血红素传递到d1型血红素, NO2–结合在d1血红素上还原为NO[14](图 3)。研究表明, 许多菌中都可分离到细胞色素cd1-NiR, 包括Stenotrophomonas maltophilia、Pseudomonas aeruginosa、Thiosphaera pantotropha、proteobacteria, Pseudomonas denitrificans和Paracoccus pantotrophus[7]。
1.3 一氧化氮还原酶
一氧化氮还原酶(NOR)催化一氧化氮还原为一氧化二氮形成 N -N键, 该酶属于亚铁血红素-铜氧化酶, 根据其一级结构和空间结构, 可分为三种: cNor、qNor和qCuNor[12]。
cNor cNor为膜结合细胞色素bc型酶, 也称短链Nor(scNor), 由cnorB基因编码, 包含一个小亚基NorC和一个大亚基NorB。NorC固定于质膜, 包含一个亚铁血红素 c, 配合一个保守组氨酸和一个蛋氨酸, 将电子传递到催化单元NorB[15]。NorB为一膜锚定蛋白, 包含两个亚铁血红素 b和一个非亚铁血红素铁。其中一个亚铁血红素b, 配合两个保守组氨酸, 将电子转移到二金属双核催化反应中心, 释放 N2O到周质[12]。在亚铁细胞色素 c 作为电子供体的条件下, 此酶显示出很高的一氧化氮还原酶活性[7]。研究发现 cnorB基因类型的菌有 Pseudomonas aeruginosa、Pseudomonas srutzeri、Paracoccus denitrfican、Halomonas halodenitrficans、Rhodobacter sphaerroides、Bradyrhizobium japonicum和Alcaligenes faecalis[16]。
qNor 在Ralstonia eutropha中发现另一种一氧化氮还原酶qNor, 也称NorZ或长链Nor(lcNor)。该酶是单体酶, 由qnorB基因编码。qNor多肽链与cNor有很高的同源性, 由一个N端延伸和一个C端域组成[17]。其N端为对苯二酚结合位点[17], C端类似于cNor酶的催化亚基NorB[16]。该酶在反硝化和非反硝化细菌中都有, 而后者经常有致病性[12]。Hendriks, et al.[18]研究表明含有 q Nor酶的菌主要有Staphylococcus aureus、Bacillus stearothermophilus、Mycobacterium avium、Corynebacterium diphtheriae、Synechocystis sp.和Ralstonia eutropha。
qCuANor 革兰氏阳性菌中存在第三种类型的一氧化氮还原酶qCuANor。该酶含有两个亚基, 最大的催化亚基类似NorB, 含有两个亚铁血红素b, 小亚基利用CuA位点获得电子传递到催化亚基[6]。该酶利用延胡索酸和细胞色素c551为电子供体, 前者作用于一氧化氮脱毒作用,后者作用于反硝化[19]。qCuANor仅在 Bacillus azotoformans中, 而其编码基因尚未发现[5]。
1.4 一氧化二氮还原酶
一氧化二氮还原生成氮气, 为完全反硝化的最终步骤, 催化该反应的一氧化二氮还原酶(N2OR), 为多铜二聚体酶, 由 nosZ 基因编码其催化中心。在革兰氏阴性菌中为周质酶, 革兰氏阳性菌中为膜结合酶[6]。
好氧与厌氧反硝化菌中分离出的 N2OR在分子性质上非常相似[7]。根据氧化还原特性和光谱学分析, 可分为Ⅰ型(purple型)和Ⅱ型(pink型), 其中Ⅱ型是从有氧状态下的培养物中分离而来[5]。该同型二聚体酶每个亚基都包含6个Cu原子, 它们排列成2个不同类型的多聚铜活性中心, 一个是双核电子传递位点CuA; 另一个是四核催化位点 CuZ。CuA从细胞色素 c、延胡索酸或假天青素[6,7]得到电子, 传递到亚基CuZ, 在此N2O还原成N2。
N2OR从Pseudomonas stutzeri首次分离得到, 其后陆续在其他菌中发现: Pseudomonas stutzeri、Rhodobacter capsulatus、Wolinella succinogenes、Achromobacter cycloclastes、Pseudomonas aeruginosa、Paracoccus pantotrophus、Alcaligenes xylosoxidans、Pseudomonas nautica等。通常携带 nirS的菌也带有 nosZ, 极少例外, 而携带 nirK的菌更多使用不完全反硝化途径, 推测与其进化机制有关[20]。
2 影响反硝化的因素
对微生物生长发育具有直接或间接影响的环境要素,被称为环境因子。环境因子如电子受体、碳源、碳氮比、溶解氧、pH和温度等, 为反硝化的重要影响因素。
2.1 电子受体
作为电子受体, 硝酸盐氮和亚硝酸盐氮是控制反硝化效率的最直接因素。在Verbaenderta, et al.[21]的研究中, 70.5%反硝化菌以硝酸盐为电子受体, 45.5%以亚硝酸盐为电子受体, 18.2%该两种电子受体都能利用, 95.5%能以两者的复合物为电子受体。Paracoccus pantotrophus以氧和硝酸盐共同作为电子受体时其生长速率高于以单一底物作为电子受体时的速率, 最后获得的蛋白质产率也最高,缺少或O2都会降低细菌的生长率和反硝化率[22]。有研究表明,浓度越高, Pseudomonas mandelii反硝化基因表达更持久[23]。
2.2 碳源
反硝化菌以有机碳为主要电子供体, 碳源含量为影响好氧反硝化的主要因素[4]。Bernat, et al.[24]利用活性污泥处理无碳源的合成废水时脱氮率为1.54 mg[N]/L, 而处理投加醋酸钠的配制废水时脱氮率达到 22.50 mg[N]/L,说明好氧反硝化菌外源呼吸比内源呼吸效率高。在对群落和碳源关系的研究发现, 醋酸对污水处理厂发现的反硝化菌Thauera和Acidovorax具有选择作用; 乙醇或甲醇可以诱导出独特的反硝化群落, 而且对cd1-NiR反硝化菌影响更大。在乙酸钠和丁二酸钠为碳源时, 反硝化菌对亚硝酸盐氮的去除率最高, 而对乳酸、蔗糖的碳源利用率较低,亚硝酸盐氮去除率相对较小, 可见菌对小分子碳源的利用率较高, 而对大分子碳源的利用率较低[25]。
2.3 碳氮比
C/N 在反硝化过程中同样也起着很重要的作用, 脱氮率随C/N的提高而增加。Citrohucter diversus以醋酸为碳源且C/N为4—5时可得到最佳的反硝化活性, 该C/N较缺氧反硝化时所需要的 C/N高; 生物反应器中反硝化菌群以蔗糖为碳源C/N为2.5亚硝酸盐氮去除率最大达到5 mg/L[26]。有研究发现, 还原性的碳源越多, 周质硝酸盐还原酶的活性越高[27]; 低C/N、NOR和N2OR等竞争力较弱的还原酶(其中N2OR最弱)活性就会受抑制[4]。随C/N降低, 反硝化相关基因表达减弱, 且无法获得足够的碳源合成足够的反硝化酶系, 影响了菌体的生长以及对硝酸盐氮或亚硝酸盐氮的去除[4]; 碳源受到限制时, 好氧反硝化现象消失[27]。
2.4 溶解氧
多数研究者发现, 在一定范围内, 反硝化率不受 DO值的影响; 当DO降低到某个限值时, 随DO降低反应效率迅速升高, 说明 DO存在一个阈值[4]。Pseudomonas stuteri的硝酸盐还原酶、亚硝酸还原酶和一氧化二氮还原酶的 DO 阈值分别是 5.10、2.50、3.80 mg/L。而Pseudomonas nautica三种还原酶的DO 阈值分别是4.05、2.15、0.25 mg/L。研究表明, 阈值随着底物、微生物的种类以及环境条件(温度、气压、离子强度等)不同而不同, DO阈值的范围一般在0. 08—7. 70 mg/L[8]。此外, Huang, et al.[26]在利用Citrobacter diversus进行好氧反硝化的研究时指出, ρ(DO)为2—6 mg/L对细菌生长和反硝化最合适。
2.5 pH
环境中的pH主要作用于细胞膜的电荷, 影响微生物对营养物质的吸收和代谢过程中酶的活性。Šimek, et al.[28]表示, 细菌生长及反硝化酶活性的最适pH为中性或微碱性, 而cnorB和nirS的表达水平在pH 6—8可保持相对稳定[29]。Pseudomonas putida的亚硝酸盐氮去除率于 pH 6—9时均较高[30]; 而Thioalkalivibrio nitratireducen的亚硝酸盐还原酶在pH 6.7—7.5范围内活性最高, pH 9—10会抑制其80%的活性, 导致中间产物亚硝酸盐氮、一氧化氮和一氧化二氮的积累[31]。当pH小于5.0时, 生长及反硝化效能都急剧下降; pH为4.0时, 细菌表面的负电荷环境被改变, 营养物质难于进入细胞, 菌株的生长受阻, 基质中 H+浓度过高, 酶结构不稳, 硝酸还原酶活性遭到抑制, 菌株生长和反硝化效能效果均最差[1]。
2.6 其他因素
细菌生长及反硝化活性最适温度范围是 25—35 ℃ ,当温度超过这一范围时, 均会抑制细菌生长和反硝化性能[25]。好氧反硝化过程对温度的耐受性比较好, 在17.5—33.1℃时, 硝酸盐氮平均去除率大于90%[23]。有研究表明, Pseudomonas putida亚硝酸盐去除率 20—30℃时的显著高于15℃[30]。在低温10—15℃时, 由于菌株胞膜不饱和脂肪酸含量较高, 保证了膜的通透性, 仍具有一定的脱氮能力[1]。
高盐度对反硝化还原酶活性有一定抑制作用。当盐度范围在5—15时, 细菌对亚硝酸盐氮的去除率接近100%,当盐度超过15 g/L, 细菌亚硝酸盐氮去除率逐渐降低[25]。Dincer和Kargi[32]研究表明当NaCl浓度超过20 g/L, 硝酸盐消耗和含氮化合物的脱除效率显著减少。
反硝化过程包含一些金属离子为协作因子的蛋白质,因此, 金属对反硝化有显著影响。添加 Fe3+和 Mo6+能增加 NAR 和 NAP活性[33]; Catarina, et al.[34]研究发现, 60 μg/g Cu可抑制杜罗河沉淀物中85%反硝化过程; Cu的添加还可致使一氧化二氮和亚硝酸盐氮的积累, 显著降低了nirK、nirS和nosZ的丰度和酶的多样性。
在适宜磁场条件下, 磁作用使细胞内线粒体数目和线粒体内的嵴明显增加, 为细胞的呼吸、氧化还原提供足够的场所, 使能量代谢明显提高, 水解酶和氧化还原酶活性增强, 提高生物细胞膜的通透性, 好氧反硝化菌数量和活性产生明显变化[3]。当磁粉投加量在 0.5—2 g/L,硝酸盐氮去除率在 93.96%以上, 并随着磁粉投加量的增加而逐渐上升; 当投加量为2 g/L时, 硝酸盐氮去除率达到 100%; 磁场过强, 会抑制菌株的生长活性, 硝酸盐氮去除率下降[3]。
3 结语
养殖水体的富营养化已经成为农业发展最为突出的问题之一, 利用反硝化菌实现养殖水体生物脱氮已有成功应用的报道。陈瑞芳等[35]将一株海水养殖池底泥中分离得到的假单胞菌SF1, 接种到鱼池底泥36h后, 底泥的亚硝酸盐氮去除率达到 67%。张小玲等[36]从土壤中分离出一株反硝化芽孢杆菌 DNF 409, 其硝酸盐氮和亚硝酸盐氮的降解率可分别达到94.79%和99.94%, 并且在国内多个水塘中进行了中试试验, 均取得了良好的脱氮效果。
利用反硝化细菌代谢转化去除含氮有害物质已成为世界公认的最经济有效的绿色途径之一, 具有重要的研究意义。反硝化酶是反硝化过程中的催化因子, 前人对其结构及其作用机制初步开展了的研究。同时, 综合影响反硝化速率的限制因素包括: 电子受体、碳源、碳氮比、pH、溶解氧和温度等, 研究这些限制因素并建立数学模型,可以为反硝化脱氮系统的设计, 提供生物学参数。目前虽已取得了不少成果, 但对微生物的反硝化作用还有待进一步深入研究, 主要包括以下几方面: (1) 反硝化酶功能基因表达的机制研究; (2) 反硝化酶活性的调控机理; (3)碳源、温度等环境因子影响反硝化过程的深入机制。应当加快反硝化菌作用机理的研究, 以便进一步开发利用反硝化细菌, 为生物脱氮提供充分的理论依据。
[1] Lü Y S, Qi S T, Wang X Y, et al. Screening of efficient denitrobacteria induced by ultraviolet irradiation and lithium chloride [J]. Marine Sciences, 2011, 35(11): 35—40 [吕玉珊,齐树亭, 王晓宇, 等. 用于海水养殖水脱氮的反硝化细菌的诱变及筛选. 海洋科学, 2011, 35(11): 35—40]
[2] Zhang Q H, Feng Y H, Wang J, et al. Study on the characteristics of the ammonia-nitrogen and residual feeds degradation in aquatic water by Bacillus licheniformis [J]. Acta Hydrobiologica Sinica, 2011, 35(3): 498—503 [张庆华, 封永辉, 王娟, 等. 地衣芽孢杆菌对养殖水体氨氮、残饵降解特性研究. 水生生物学报, 2011, 35(3): 498—503]
[3] Wang Q. Study on biological nitrogen removal mechanisms and performance of magnetic strengthening aerobic denitrifiers [D]. Thesis for Doctor of Science. Harbin Institute of Technology, Harbin. 2010 [王强. 磁强化好氧反硝化菌的生物脱氮机制与效能. 硕士学位论文, 哈尔滨工业大学,哈尔滨. 2010]
[4] Fan L R, Huang S B. Research progress of aerobic denitrification technology [J]. Industrial Water & Wastewater, 2008, 39(2): 5—9 [范利荣, 黄少斌. 好氧反硝化脱氮技术研究进展. 工业用水与废水, 2008, 39(2): 5—9]
[5] Zumft W G. Cell biology and molecular basis of denitrifcation [J]. Microbiology and Molecular Biology Reviews, 1997, 61(4): 533—616
[6] Kraft B, Strous M, Tegetmeyer H E. Microbial nitrate respiration-Genes, enzymes and environmental distribution [J]. Journal of Biotechnology, 2011, 155(1): 104—117
[7] Yang H, Huang J, Liu B. Advances in research of heterotrophic nitrification-aerobic denitrification strain Paracoccus pantotrophus ATCC 35512 [J]. Chinese Journal of Applied and Environmental Biology, 2008, 14(4): 585—592 [杨航,黄钧, 刘博. 异养硝化–好氧反硝化菌 Paracoccus pantotrophus ATCC 35512的研究进展. 应用与环境生物学报, 2008, 14(4): 585—592]
[8] Li P, Zhang S, Liu D L. Study progress of bacterial aerobic denitrification [J]. Journal of Microbiology, 2005, 25(1): 60—64 [李平, 张山, 刘德立. 细菌好氧反硝化研究进展.微生物学杂志, 2005, 25(1): 60—64]
[9] Sievert S M, Scott K M, Klotz M G, et al. Genome of the epsilonproteobacterial chemolithoautotroph Sulfurimonas denitrificans [J]. Applied and Environment Microbiology, 2008, 74(4): 1145—1156
[10] Smith J M, Ogram A. Genetic and functional variation in denitrifier populations along a short-term restoration chronosequence [J]. Applied and Environmental Microbiology, 2008, 74(18): 5615—5620
[11] Nojiri M, Koteishi H, Nakagami T, et al. Structural basis of inter-protein electron transfer for nitrite reduction in denitrification [J]. Nature, 2009, 462: 117—120
[12] Heylen K. Study of the genetic basis of denitrification in pure culture denitrifiers isolated from activated sludge and soil [D]. Thesis for Ph.D. of Science. Ghent: Ghent University. 2007
[13] Boulange M J, Murphy M E P. Crystal structure of the soluble domain of the major anaerobically induced outer membrane protein (AniA) from pathogenic Neisseria: a new class of copper-containing nitrite reductases [J]. Journal of Molecular Biology, 2002, 315(5): 1111—1127
[14] Fülöp V, Moir J W B, Ferguson S J, et al. The anatomy of a bifunctional enzyme: structural basis for reduction of oxygen to water and synthesis of nitric oxide by cytochrome cd1 [J]. Cell, 1995, 81(3): 369—377
[15] Tavares P, Pereira A S, Moura J J G, et al. Metalloenzymes ofthe denitrification pathway [J]. Journal of Inorganic Biochemistry, 2006, 100(12): 2087—2100
[16] Li M Z. Application study of cnorB gene, encoding the catalyzing subunit, in population analysis for denitrifying bacteria [D]. Thesis for Master of Science. Zhejiang University, Hangzhou. 2004 [李明振. 一氧化氮还原酶催化亚基编码基因(cnorB)在反硝化细菌种群分析中的应用研究. 硕士学位论文, 浙江大学, 杭州. 2004]
[17] de Vries S, Pouvreau S, Pouvreau L A M. Nitric oxide reductase: structural variations and catalytic mechanism [A]. In: Bothe H, Ferguson S J, Newton W E (Eds.), Biology of the nitrogen cycle [C]. Amsterdam: Elsevier. 2007, 57—66
[18] Hendriks J, Oubrie A, Castresana J, et al. Nitric oxide reductases in bacteria [J]. Biochimica et Biophysica Acta, 2000, 1459(2-3): 266—273
[19] Suharti, Heering H A, de Vries S. NO reductase from Bacillus azotoformans is a bifunctional enzyme accepting electrons from menaquinol and a specific endogenous membrane-bound cytochrome c551 [J]. Biochemistry, 2004, 43(42): 13487—13495
[20] Jones C M, Stres B, Rosenquist M, et al. Phylogenetic analysis of nitrite, nitric oxide, and nitrous oxide respiratory enzymes reveal a complex evolutionary history for denitrifcation [J]. Molecular Biology and Evolution, 2008, 25(9): 1955—1966
[21] Verbaenderta I, Boon N, De Vos P, et al. Denitrifcation is a common feature among members of the genus Bacillus [J]. Systematic and Applied Microbiology, 2011, 34(5): 385—391
[22] Robertson L A, Kuenen J G. Combined heterotrophic nitrification and aerobic denitrification in Thiosphaera pantotropha and other bacteria [J]. Antonie Van Leeuwenhoek, 1990, 57(3): 139—152
[23] Saleh-Lakha S, Shannon K E, Henderson S L, et al. Effect of Nitrate and Acetylene on nirS, cnorB, and nosZ expression and denitrifcation activity in Pseudomonas mandelii [J]. Applied and Environmental Microbiology, 2009, 75(15): 5082—5087
[24] Bernat K, Wojnowska-Baryła I. Carbon source in aerobic denitrification [J]. Biochemical Engineering Journal, 2007, 36(2): 116—122
[25] An J, Song Z F, Yang X L, et al. Characteristics of aerobic denitrifying strain YX-6 and identification [J]. Journal of Fishery Sciences of China, 2010, 17(3): 561—569 [安健, 宋增福, 杨先乐, 等. 好氧反硝化细菌 YX-6 特性及鉴定分析. 中国水产科学, 2010, 17(3): 561—569]
[26] Huang H K, Tseng S K. Nitrate reduction by Citrobacter diversus under aerobic environment [J]. Applied Microbiology and Biotechnology, 2001, 55(1): 90—94
[27] Pang D H, Liu Z F. The mechanism of aerobic denitrification and the outlook of dealing with NOxwaste gas [J]. Pollution Control Technology, 2010, 23(3): 74—77, 84 [庞德红, 柳知非. 好氧反硝化脱氮机理及处理NOX废气的展望. 污染防治技术, 2010, 23(3): 74—77, 84]
[29] Wang Y, Wang S Y, Sun H W, et al. Effects of pH and MLSS on nitrite reduction rate in shortcut denitrification [J]. China Water & Wastewater, 2010, 26(13): 25—28 [王燕, 王淑莹,孙洪伟, 等. pH和MLSS对短程反硝化过程N还原速率的影响. 中国给水排水, 2010, 26(13): 25—28]
[30] Zheng J J, Shen T, Fu L Q, et al. Identification and denitrification characteristics of a denitrifer Pseudomonas putida F6 [J]. Acta Hydrobiologica Sinica, 2012, 36(1): 161—167 [郑佳佳, 沈涛, 傅罗琴, 等. 一株硝化反硝化菌的筛选鉴定及反硝化特性研究. 水生生物学报, 2012, 36(1): 161—167]
[31] Gao Y, Cornwell J C, Stoecker D K, et al. Effects of cyanobacterial-driven pH increases on sediment nutrient fuxes and coupled nitrifcation-denitrifcation in a shallow fresh water estuary [J]. Biogeosciences Discuss, 2012, 9: 1161—1198
[32] Dincer A R, Kargi F. Salt inhibition of nitrification and denitrification in saline wastewater [J]. Environmental Technology, 1999, 20(11): 1147—1153
[33] Pintathong P, Richardson D J, Spiro S, et al. Influence of metal ions and organic carbons on denitrification activity of the halotolerant bacterium, Paracoccus pantotrophus P16 a strain from shrimp pond [J]. Electronic Journal of Biotechnology, 2009, 12(2): 1—11
[34] Magalhães C M, Machado A, Matos P, et al. Impact of copper on the diversity, abundance and transcription of nitrite and nitrous oxide reductase genes in an urban European estuary [J]. FEMS Microbiology Ecology, 2011, 77(2): 274—284
[35] Chen R F, Xu C A, Liu L M, et al. Nitrite degradation of a Pseudomonas sp. strain SF1 and its application in fish pond sediment [J]. Marine Sciences, 2011, 35(6): 30—34 [陈瑞芳,徐长安, 刘丽梅, 等. 一株假单胞菌的亚硝酸盐降解特性及其对鱼池底泥的净化作用. 海洋科学, 2011, 35(6): 30—34]
[36] Zhang X L, Liang Y X. Screening of a strain denitrobacteria and its denitrofication characteristic [J]. Freshwater Fisheries, 2006, 36(5): 28—32 [张小玲, 梁运祥. 一株反硝化细菌的筛选及其反硝化特性的研究. 淡水渔业, 2006, 36(5): 28—32]
PROGRESS IN STUDIES ON DENITRIFICATION ENZYMES AND ENVIRONMENTAL IMPACT FACTORS
LI Wei-Fen, ZHENG Jia-Jia, ZHANG Xiao-Ping and DENG Bin
(College of Animal Sciences, Zhejiang University, Hangzhou 310058, China)
反硝化; 酶; 环境因子
Denitrification; Enzymes; Environmental factors
Q938.8
A
1000-3207(2014)01-0166-05
10.7541/2014.22
2012-09-17;
2013-06-02
国家重点基础研究发展规划(973)项目(2009CB118705); 公益性行业(农业)科研专项(201203083)资助
李卫芬(1965—), 女, 江苏张家港人; 研究员, 博士; 研究方向为微生物与基因工程。E-mail: wfli@zju.edu.cn

