福建省禽流感病毒非结构蛋白基因特征分析
朱春华,林 甦,陈 珍,刘斌琼,万春和,傅光华,黄 瑜
禽流感(Avian Influenza)是由A型流感病毒引起的一种禽类(家禽和野禽)的感染和/或疾病综合征[1],该病毒由8个基因片段构成,其中最短的一个基因为非结构基因(non-structural gene,NS)。病毒复制过程中,NS基因编码一个mRNA转录本,该转录本可被选择性的剪切并表达两个重要的功能蛋白,分别是 26 kDa的NS1蛋白和14 kDa的NEP(nuclear export protein,NEP)蛋白(通常被称为NS2蛋白)。NS1蛋白以二聚体形式存在,在病毒的复制过程中发挥着重要的作用,mRNA的合成、剪切、蛋白磷酸化甚至诱导细胞凋亡都与这个蛋白相关[2-3]。同时,有研究表明该蛋白在非特异性免疫反应中会抑制Ⅰ型干扰素的合成[3]。NS1蛋白含有两个核定位信号区,其中34~38位氨基酸残基的Asp-Arg-Leu-Arg-Arg信号区非常保守,在所有的甲型流感病毒中都相同。第二个信号区位于203~230位氨基酸残基处,大多数甲型流感病毒中都有这一序列[4]。NS1蛋白是决定AIV对感染细胞的破坏力的关键因素,影响AIV的致病性和毒力[5-6];NS2具有调节非结构蛋白合成的作用,在病毒感染细胞过程发挥重要的作用[3]。在细胞感染流感病毒的早期就可发现细胞核内有大量的NS1聚集,细胞浆内也有NS1聚集;而NS2合成较晚,主要存在于细胞浆,也可在细胞核内发现[4]。
目前福建省分离的禽源流感毒株包括H5N1、H6N6、H9N2等亚型,本研究拟对福建省鸡源、鸭源、鹅源等不同源的流感毒株的NS基因进行遗传进化分析,进一步探讨福建省不同亚型之间和同一亚型内不同毒株的NS基因与国内代表株禽流感病毒NS基因的特性差异,为进一步了解福建省不同亚型禽流感病毒的NS基因遗传进化关系及毒力变异奠定基础,为深入了解流感病毒的致病机制与NS基因之间交互作用提供理论依据。
1 材料与方法
1.1材料 9~10日龄SPF鸡胚购自北京梅利亚实验动物有限公司。2株鸡源禽流感病毒FZ-04、FZ-11株,5株鸭源禽流感病毒A/duck/Fujian/FQ2/2007(H9N2)、A/Duck/Fujian/FQ107/2007(H9N2)、A/duck/Fujian/MH/2003(H9N2)、A/Duck/Fujian/FZ01/08(H9N2)、A/Muscovy duck/Fujian/CL/1997(H9N2)[7-8]毒株均由本禽病室于1997-2011年从福建地区分离保存,毒株分离与增殖见文献[8]。大肠杆菌DH5α由本室保存。
1.2仪器和试剂 凝胶成像分析系统(BIO-RAD)、PCR仪(Eppendorf 公司),Trizol购自Invitrogen。胶回收试剂盒和质粒提取试剂盒均购自美国Omega Bio-Tek公司。大肠杆菌(Escherichiacoli)DH5α感受态细胞自制。AMV反转录酶、Ex Taq DNA聚合酶和pMD18-T载体均购自宝生物工程(大连)有限公司。
1.3引物设计 FZ-04株禽流感病毒NS基因特异引物参考文献[9]。反转录引物序列为5′-AGCAAAAGCAGG-3′。引物由宝生物工程(大连)有限公司合成。
1.4毒株NS基因扩增 毒株分离、增殖以及病毒RNA提取详见文献[8]。用流感的反转录引物Uni-12,按照AMV反转录酶说明书进行反转录合成cDNA。PCR扩增以cDNA为模板,反应体系为50 μL:10×Buffer 5 μL,dNTP mixture (2.5 nmol/L)4 μL,Ex-Taq DNA polymerase 0.5 μL,引物(20 μmol/L)各1 μL,模板cDNA 1 μL,ddH2O 37.5 μL。反应条件:94 ℃预变性5 min,按94 ℃30 s,55 ℃ 35 s,72 ℃ 2 min,进行35个循环,最后72 ℃延伸10 min。取5 μL产物经1%琼脂糖凝胶电泳检测扩增结果。
1.5NS基因的遗传进化分析 将毒株NS基因的PCR产物经切胶回收试剂盒纯化后与pMD18-T载体连接,转化DH5α感受态细胞,PCR方法鉴定重组质粒,阳性质粒送大连宝生物公司测序。分别应用DNAstar 5.0和MEGA 4.0分析软件(By Clustal W Method)对NS基因序列与GenBank中登录的国内流感病毒典型代表株及福建省不同时期分离的H5N1、H9N2和H6N6亚型流感毒株进行核苷酸序列同源性比较和遗传进化分析。遗传进化分析所涉及的毒株见表1。
2 结 果
2.1NS基因核苷酸测序结果及核苷酸同源性分析 FZ-04、FZ-11毒株NS基因扩增的长度均为887 bp,GenBank基因登录号分别为KF550969、KF550968。由DNAstar软件分析可知,两个毒株NS1基因完整的阅读框为654 bp(从27~680 bp),编码217个氨基酸;NS2 基因编码区则由27~56 bp及529~864 bp构成,共编码121个氨基酸,不存在氨基酸缺失或插入现象。MegAlign软件分析可知,FZ-04株与福建株CK/FJ/G9/09(H9N2)同源性最高(99.0%),FZ-11株与广西省马源分离株EQ/GX/3/2011(H9N2)同源性最高(99.4%)。FZ-04,FZ-11毒株与福建H5N1毒株同源性分别为88.2%~89.3%,88.3%~89.2%;与福建H9N2毒株的同源性分别为92.7%~99.0%,93.2%~99.4%;与疫苗株CK/SD/6/96同源性分别为92.6%、93.2%,与国内H5N1代表株GS/GD/1/96同源性最低,分别为70.8%和70.6%,与福建省H6N6代表株同源性分别为89.6%和90.1%。
2.2NS基因遗传进化分析 从遗传进化树分析可知(图1),福建省H5、H9亚型禽流感毒株分别聚合在两个不同的大分支上。GenBank中与本研究的两株病毒NS基因亲缘关系最近的毒株为 CK/FJ/G9/09株,与国内高致病性禽流感代表株GS/GD/1/96遗传距离最远,CK/BJ/1/94毒株位于进化分析的根部,表明其余的H9N2亚型毒株都是该毒株与其他亚系毒株之间基因交流的产物。NS基因与广西省马源流感株EQ/GX/3/2011遗传距离很近,可能是禽源流感病毒基因重组后感染了马类造成的[10]。
福建省禽流感NS1氨基酸序列比对结果(图2和表2),FZ-04和FZ-11毒株NS1蛋白羧基末端存在13个氨基酸缺失,与福建省分离的H9N2亚型毒株序列长度相同,而福建省H5N1毒株NS1蛋白80-84位有5个氨基酸缺失,总长度为225 bp。本研究中福建省H9N2毒株的NS1蛋白羧基端不存在PL基序(ESEV/EPEV),而国内首次分离的高致病性GS/GD/1/96代表株和福建9株H5N1病毒SW/FJ/F1/01、GS/FJ/bb/03、CK/FJ/9821/05等存在ESEV基序,有一株病毒CK/FJ/1/07(H5N1)则呈现GSEV遗传多态性。
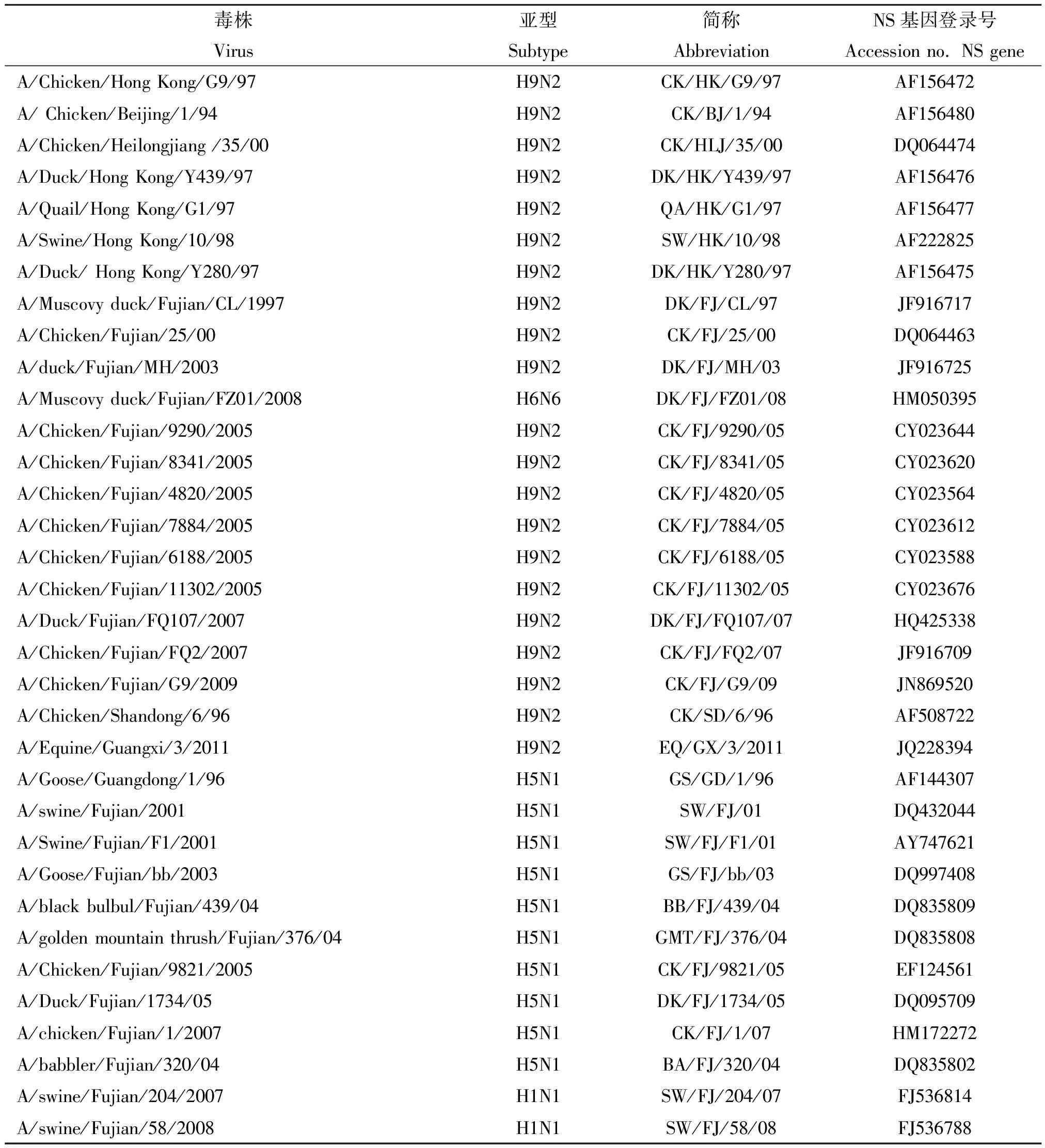
表1 本研究所涉及的毒株

图1 NS基因的遗传进化树
3 讨 论
根据NS基因核苷酸同源性不同分为A和B两个等位基因群,群内同源性相对比较高。A群包含禽源和哺乳动物源流感病毒,而B群主要包含禽源流感病毒和一种马流感[11-12],有研究报道A等位基因群中欧亚H9N2亚型流感毒株又被分为3个不同的亚群,分别为DK/HK/Y280/97株为代表的Y280亚群、DK/HK/Y439/97株为代表的Y439亚群及QA/HK/G1/97株为代表的G1亚群。本研究中所涉及的毒株GS/GD/1/96属于B等位基因群,其他的都属于A等位基因群。根据GenBank提交的序列信息可知,福建省分离的禽源流感毒株目前已见报道的有3种亚型,分别是H5N1、H9N2和H6N6亚型,福建H9N2亚型毒株都属于Y280亚群,而福建H5N1流感毒株则属于Y439亚群,H6N6亚型鸭源DK/FJ/FZ01/08株则是独立的一分支。
本研究中FZ-04和FZ-11的NS1基因编码217个氨基酸,其羧基末端存在13个氨基酸缺失,早期报道野外分离的禽流感病毒的某些毒株,NS1蛋白的羧基端有氨基酸缺失现象,而近几年研究发现多数低致病性毒株羧基端存在氨基酸缺失现象,本研究中福建省H9N2毒株都存在羧基末端氨基酸缺失现象,与其亚群代表株Y280不同,Y280株不存在基因缺失现象。令人感兴趣的是,本研究的21株病毒中仅福建H5N1高致病性流感毒株NS1蛋白80-84位有5个氨基酸缺失(图2),而H9N2亚型毒株和H6N6毒株在这几个位点不存在缺失现象,这是否可作为福建省区分高致病性和低致病性禽流感病毒的标志之一,有待于进一步研究。近年来关于NS1蛋白这几个位点氨基酸缺失的报道比较多,欧新华等[13]报道从湖南长沙市禽类农贸市场分离到8株H5N1病毒的NS1蛋白80-84位点均存在氨基酸缺失。
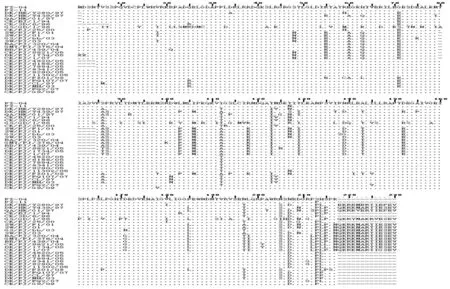
图2 禽流感病毒NS1氨基酸序列比对
Fig.2ComparisononaminoacidsequenceofNS1ofavianinfluenzaviruses
In the figure, dots indicate residues identical to those of the FZ-04 strain; dashes indicate the deletion positions.
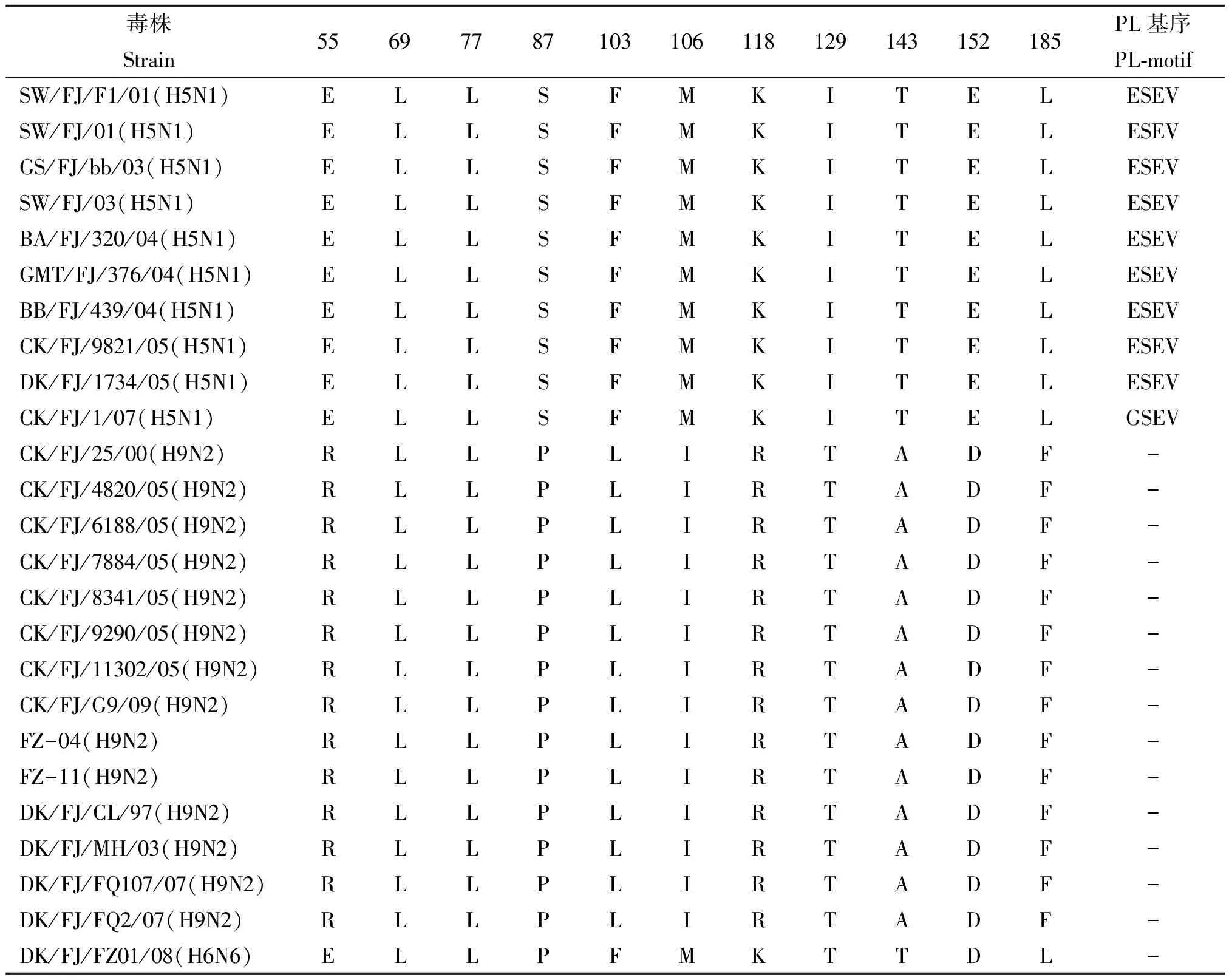
表2 福建省禽流感NS1氨基酸位点分析
Note: Dashes indicate the deletion positions in the table.
福建省AIV的NS1氨基酸序列中有11个氨基酸位点高度保守(分别是第55、69、77、87、103、106、118、129、143、152和185位),已有研究证实第69位和第77位氨基酸位点突变会影响NS蛋白正确的亚细胞定位,从而减弱病毒的复制能力[14];同时,有研究报道称103、106位氨基酸位点分别从L(亮氨酸)突变成F(苯丙氨酸)和I(异亮氨酸)突变成M(蛋氨酸),将会增强禽源和人源流感毒株的毒力[15],其他7个氨基酸位点在不同亚型上(H5N1或H9N2)氨基酸不同,而同一亚型内(H5N1或H9N2)的毒株的7个氨基酸位点相同(见表2),这是否能够成为福建省高致病性H5N1株和低致病性H9N2株流感病毒的区分标志之一,有待于后续深入研究。本研究中仅QA/HK/G1/97株NS蛋白92位氨基酸为E(谷氨酸), 其他毒株该位点氨基酸均为D(天冬氨酸),有研究分析该位点由D突变为E,会降低NS1蛋白磷酸化程度,并增强NS1对dsRNA的结合能力,从而提高病毒对干扰素的抗性[5,16]。因此,研究禽流感病毒NS基因遗传进化关系,有助于进一步了解福建省流感病毒变异情况,为深入了解流感病毒的致病机制与NS基因间交互作用提供理论依据。
参考文献:
[1]Gan MH. Avian influenza[M]. Bejing: Beijing Agricultural University Press, 1995: 74. (in Chinese)
甘孟侯. 禽流感[M]. 北京:北京农业大学出版社,1995:74.
[2]Hale BG, Randall RE, Ortin J, et al. The multifunctional NS1 protein of influenza A viruses[J]. J Gen Virol, 2008, 89(10): 2359-2376. DOI: 10.1099/vir.0.2008/004606-0
[3]Chervyakova OV, Strochkov VM, Sultankulova KT, et al. Molecular and genetic analysis of NS gene from high pathogenic strains of the avian influenza (H5N1) virus isolated in Kazakhstan[J]. Gene, 2011, 476(1-2): 15-19. DOI: 10.1016/j.gene.2011.02.003
[4]Yin Z, Liu JH. Animal virology[M]. 2nded. Beijing: Science Press, 1997: 581. (in Chinese)
殷震,刘景华. 动物病毒学[M]. 北京:科学出版社,1997:581.
[5]Li JL, Chen EL, Li HP, et al. Genetic analysis of the nonstructural gene (NS1) of H9N2 avian influenza viruses isolated in China[J]. Chin J Virol, 2008,24(3): 220-226. (in Chinese)
李建丽,陈恩林,李和平,等. H9N2亚型禽流感病毒NS1基因的进化分析[J]. 病毒学报, 2008,24(3): 220-226.
[6]Salvatore M, Basler CF, Parisien JP, et al. Effects of influenza A virus NS1 protein on protein expression: the NS1 protein enhances translation and is not required for shutoff of host protein synthesis[J]. J Virol, 2002, 76(3): 1206-1212. DOI: 10.1128/JVI.76.3.1206-1212.2002
[7]Wan CH, Fu GH, Cheng LF, et al. Sequence comparison of the hemagglutinin gene of the duck-origin H9N2 subtype avian influenza viruses[J]. Chin J Virol, 2012, 28(2): 158-164. (in Chinese)
万春和,傅光华,程龙飞,等. 鸭源H9N2 AIV血凝素基因序列比较[J]. 病毒学报, 2012,28(2): 158-164.
[8]Zhu CH, Jiang B, Liu BQ, et al. Isolation and preliminary identification of H9 subtype influenza A viruses[J]. Fujian J Agr Sci, 2011, 26(4): 537-540. DOI: 10.3969/j.issn.1008-0384.2011.04.007 (in Chinese)
朱春华,江斌,刘斌琼,等. H9亚型禽流感病毒的分离及初步鉴定[J]. 福建农业学报,2011,26(4):537-540.
[9]Wan CH, Fu GH, Cheng LF, et al. Sequencing of the eight genes of A/Chicken/Zhejiang/HJ/2007 (H9N2) Subtype avian influenza virus and their phylogenetic analysis[J]. J Agr Biotech, 2009, 17(5): 750-757. DOI: 10.3969/j.issn.1674-7968.2009.05.002 (in Chinese)
万春和,傅光华,程龙飞,等. A/Chicken/Zhejiang/HJ/2007(H9N2)禽流感病毒8基因测序及遗传进化分析[J]. 农业生物技术学报, 2009,17(5):750-757.
[10]Kawaoka Y, Gorman OT, Ito T, et al. Influence of host species on the evolution of the nonstructural (NS) gene of influenza A viruses[J]. Virus Res, 1998, 55(2): 143-156. DOI: 10.1016/S0168-1702(98)00038-0
[11]Wang S, Shi WM, Mweene A, et al. Genetic analysis of the nonstructural (NS) genes of H9N2 chicken influenza viruses isolated in China during 1998-2002[J]. Virus Genes, 2005, 31(3): 329-335. DOI: 10.1007/s11262-005-3251-2
[12]Ludwig S, Schultz U, Mandler J, et al. Phylogenetic relationship of the nonstructural (NS) genes of influenza A viruses[J]. Virology, 1991, 183(2): 566-577. DOI: 10.1016/0042-6822(91)90985-k
[13]Ou XH, Zhang RS, Song KY, et al. Genetic analysis of the NS genes of H5N1 avian influenza viruses isolated from sewage in poultry markets[J]. Chin J Virol, 2012, 28(3): 265-271. (in Chinese)
欧新华,张如胜,宋克云,等. 家禽市场污水来源的H5N1亚型禽流感病毒NS基因分析[J]. 病毒学报,2012,28(3):265-271.
[14]Li W, Noah JW, Noah DL. Alanine substitutions within a linker region of the influenza A virus non-structural protein 1 alter its subcellular localization and attenuate virus replication[J]. J Gen Virol, 2011, 92(8): 1832-1842. DOI: 10.1099/vir.0.031336-0
[15]Dankar SK, Wang S, Ping J, et al. Influenza A virus NS1 gene mutations F103L and M106I increase replication and virulence[J]. Virol J, 2011, 8: 13. DOI:10.1186/1743-422X-8-13
[16]Lipatov AS, Andreansky S, Webby RJ, et al. Pathogenesis of Hong Kong H5N1 influenza virus NS gene reassortants in mice: the role of cytokines and B- and T-cell responses[J]. J Gen Virol, 2005, 86(4): 1121-1130. DOI:10.1099/vir.0.80663-0

