中国南方沿海13种石斑鱼类的分子系统进化关系分析*
陈兴汉,郭 梁,李明明,蒙子宁,林浩然
(1. 阳江职业技术学院生命科学与技术系,广东 阳江 529566;2. 中山大学水生经济动物研究所暨广东省水生经济动物良种繁育重点实验室,广东 广州 510275)
石斑鱼类在分类学上隶属于鲈形目(Perciformes)、鲈亚目(Percoidei)、鮨科(Serranidae)、石斑鱼亚科(Epinephelinae)。俗称的石斑鱼是石斑鱼亚科鱼类的统称。石斑鱼除少数种类生活在河口,多数栖息于热带或亚热带的珊瑚礁水域,主要以小型鱼类、底栖甲壳类和头足类等为食,是珊瑚礁生态系统的主要掠食者[1]。石斑鱼为世界性名贵海产经济鱼类,也是我国南方沿海省份(广东、海南、福建、广西)重要的海水增养殖对象。然而近年来受过度捕捞、养殖群体近亲繁殖、珊瑚礁破坏、海洋环境污染等因素影响,加之石斑鱼本身有性逆转的特性,致使资源日渐衰退,部分种类已被国际自然保护联盟(IUCN, International Union for Conservation of Nature)列为濒危或近危[2]。
石斑鱼除资源状况堪忧外,还存在着种类鉴定上的难题[3]。石斑鱼的种类较多,全世界范围内已记录的有100多种,我国已记录的有50多种。石斑鱼的种类鉴定通常是根据体色、身体和头部形态、鳍的结构和数量[1],而进一步的系统进化研究则主要是依据一些骨学特征以及幼体发育过程中仔鱼的形态特征[4-7]。然而,由于石斑鱼种类繁多、形态特征类似且体色和斑纹多变,因此,仅依据这些形态特征往往难以准确的鉴别,并导致了石斑鱼分类学上的混淆以及重建进化关系研究上的困难,至今石斑鱼的分类依然是鱼类分类学的难题。目前依据Nelson[8]的鱼类分类系统,将鮨科分为鮨亚科、花鲈亚科和石斑鱼亚科3个亚科,共63属449种。其中石斑鱼亚科又可细分为5个族:石斑鱼族、东洋鲈族、鲈族、长鲈族和黄鱼族。
近年来分子生物学的快速发展,使得在分子水平上进行鱼类的分类鉴定和系统进化研究成为现实。分子系统学(Molecular Phylogeny)通过检测生物大分子的遗传信息,因其含有信息量大,且趋同效应弱,因而其结论相对于经典的形态系统分类更具客观性和可比性。其中线粒体DNA由于其结构简单、无重组、多数母性遗传等特点,已成为群体遗传分析和系统进化研究的有力工具[9-11]。线粒体特定基因序列分析,已经成功的应用于许多种鱼类系统进化的研究,如圆罩鱼(Cyclothone)、南乳鱼(Galaxias)、突吻鳕(Coryphaenoides)等[12-14]。Craig等[15]最先研究报道了南北美洲42种石斑鱼类的分子系统进化关系。国内学者仅见丁少雄等[16]针对中国近海30种石斑鱼类,进行了基于16S rDNA的分子系统进化研究。
文章中应用线粒体细胞色素b基因(Cyt b)以及两个核糖体RNA基因(16S和12S)序列对中国近海石斑鱼亚科的3个属:石斑鱼属(Epinephelus)、九棘鲈属(Cephalopholis)和鳃棘鲈属(Plectropomus)共13个种的种类鉴定及亲缘关系进行分析,采用了线粒体基因组不同区域的多组序列数据(Cyt b、16S rRNA、12S rRNA)的联合分析,联合构建系统进化树,以探讨石斑鱼类的分子系统进化关系。
1 材料和方法
1.1 材料
文章中的石斑鱼亚科3个属13个种共61个个体为采自中国南方沿海省份广东(广州、深圳、珠海、惠州、湛江)和海南(海口)沿海的科研基地、养殖场和鲜鱼市场(见表1)。
1.2 方法
1.2.1 DNA提取 剪取鱼体尾部组织约50 mg,参照Sambrook等[17]方法进行基因组DNA提取。
1.2.2 线粒体Cyt b、16S rRNA以及12S rRNA基因扩增及测序 所用引物序列见表2(上海生工合成)。PCR反应总体积为25 μL,包括2.0 mmol/L MgCl2,0.2 mmol/L dNTP,0.2 μmol/L每种引物,1 U Taq plus DNA聚合酶(MBI),1×Taq 聚合酶缓冲液以及30 ng基因组DNA。反应条件为94 ℃预变性2 min后经过30个循环,每个循环包括94 ℃ 45 s,50 ℃ 1 min,72 ℃ 1 min,最后72 ℃延伸10 min。PCR产物用UNIQ-5(上海生工)柱式纯化试剂盒纯化后,在ABI377型自动测序仪(PE)上进行双向测序。
1.2.3 序列分析 采用DNAStar软件中的SeqMan程序对测序所得到的正向序列和反向序列进行拼接,使用Clustalx1.8进行对位排序,应用Genedoc软件进行人工校正。用DNASP3.51软件计算变异位点、简约信息位点数、核苷酸多样性、平均核苷酸差异数、种间平均每位点核苷酸替代数。
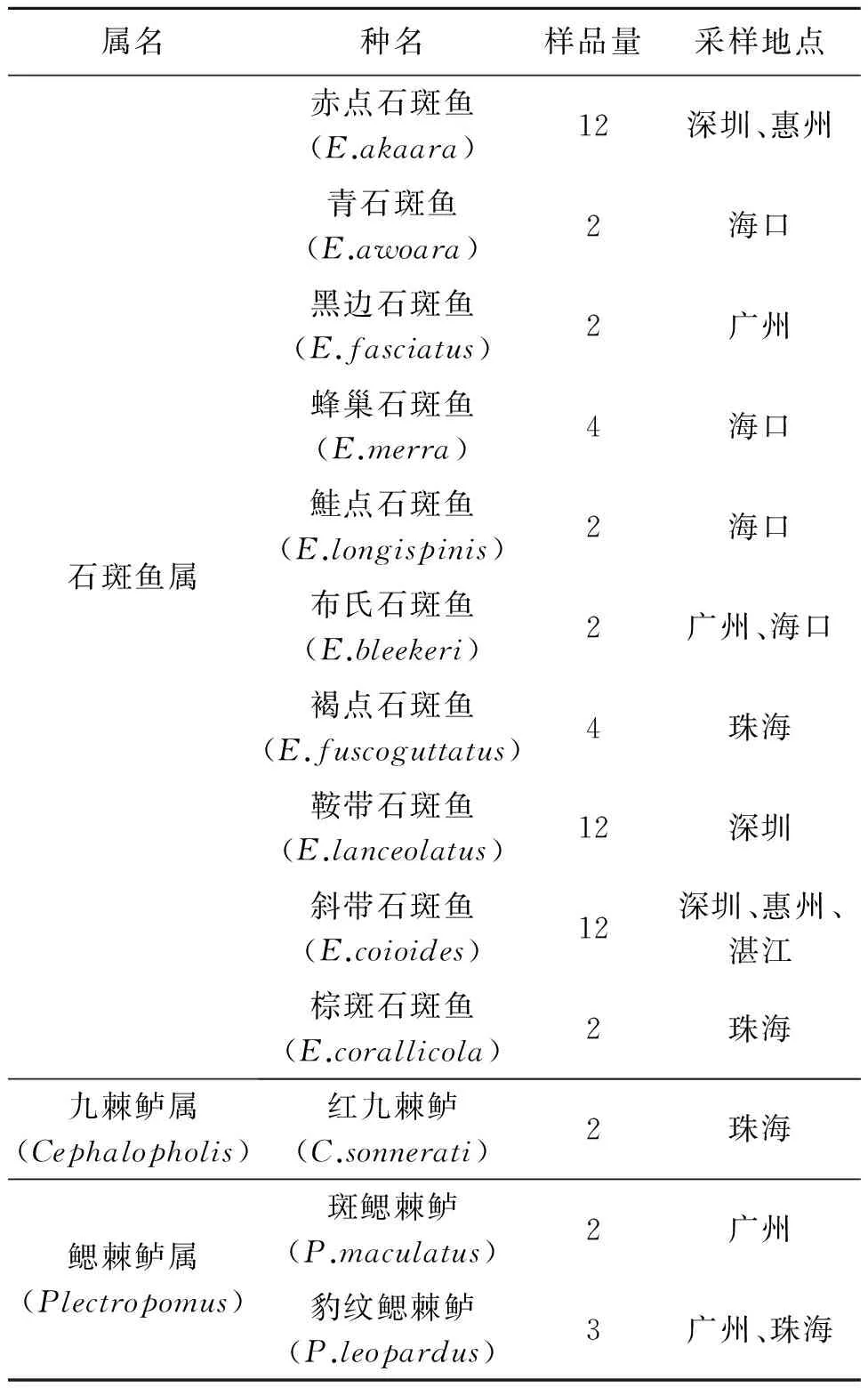
表1 样品来源Table1 Origin of the samples

表2 引物及其序列Table 2 Primers and their sequences
1.2.4 聚类分析 利用MEGA3.1软件并根据Kimura双参数模型计算遗传距离,对3段基因序列分别构建邻接(Neighbor-joining,NJ)和最大简约(Maximum-parsimony,MP)系统树,之后用3段基因序列的联合基因序列(Combined genes)构建 NJ 和 MP 系统树,采用Bootstrap1000检验分枝的置信值。
2 结 果
2.1 序列及变异
测序所得13种石斑鱼类的Cyt b基因片段长423 bp,无插入和缺失,转换/颠换之比为 2.5。共存在25种单倍型序列,序列间共有153个变异位点,其中简约信息位点139个。核苷酸多样性指数为0.14,平均核苷酸差异数达59.84(表3)。从种内变异来看,序列差异较小。青石斑鱼、黑边石斑鱼、鮭点石斑鱼、布氏石斑鱼、棕斑石斑鱼和斑鳃棘鲈种内序列相同,其余石斑鱼种内存在变异位点1~8个(表4)。
去除7个插入/缺失,测序所得13种石斑鱼类的16S rRNA基因片段长592 bp,转换/颠换之比为 2.2。共存在21种单倍型序列,序列间共有139个变异位点,其中简约信息位点119个。核苷酸多样性指数为0.08,平均核苷酸差异数达46.62(表3)。种内序列差异较小,青石斑鱼、鮭点石斑鱼、布氏石斑鱼、棕斑石斑鱼、红九棘鲈和斑鳃棘鲈种内序列相同,其余石斑鱼种内存在变异位点1~3个(表4)。
13种石斑鱼类的12S rRNA基因片段存在5个插入/缺失,去除后为434 bp,转换/颠换之比为 2.2。共存在14种单倍型序列,序列间共有87个变异位点,其中简约信息位点70个。核苷酸多样性指数为0.08,平均核苷酸差异数达34.74(表3)。种内变异极小(变异位点1个、单倍型序列14种),除豹纹鳃棘鲈种内存在1个位点差异外,其余石斑鱼种内序列相同(表4)。
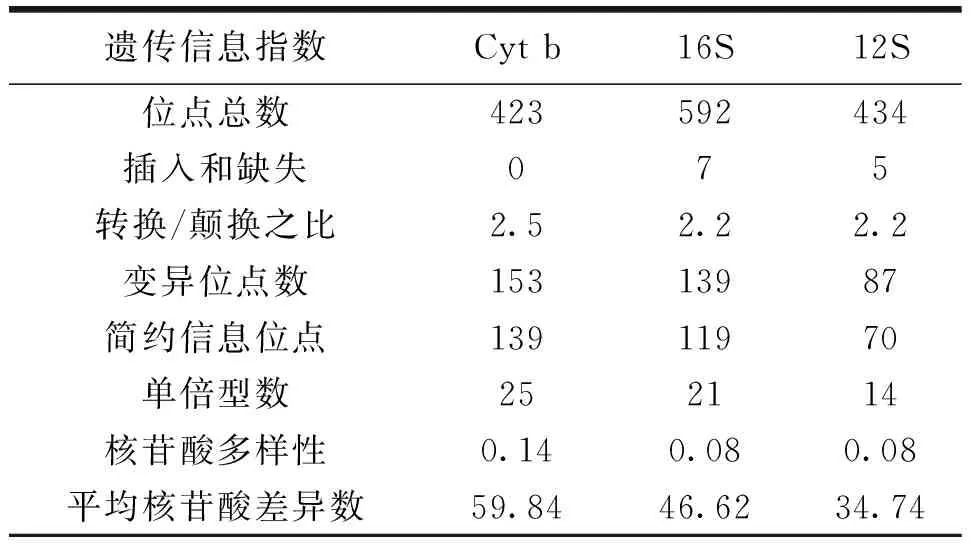
表3 13种石斑鱼类线粒体基因序列的遗传信息指数Table 3 Genetic informative indices of the mitochondrial genes of 13 Epinepheline species
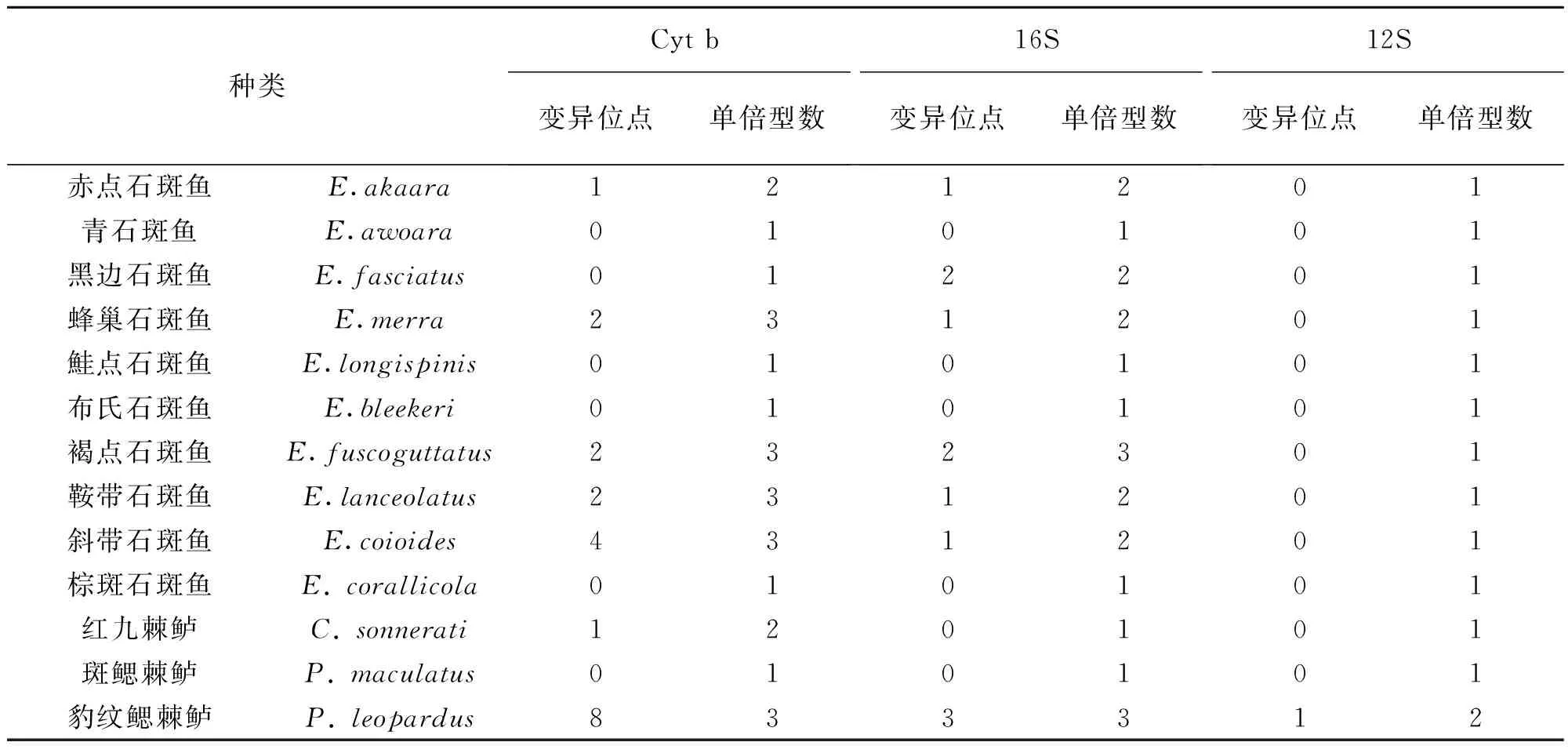
表4 13种石斑鱼类的种内遗传差异Table 4 Intraspecies genetic variations of 13 Epinepheline species
2.2 聚类分析
从属水平来看,线粒体3个基因以及联合基因序列计算的遗传距离均为石斑鱼属与九棘鲈属的最小,与鳃棘鲈属的最大,表明石斑鱼属与九棘鲈属有较近的亲缘关系(表5)。
从种水平来看,不同基因序列得出的种间遗传距离不同:13种石斑鱼类种间遗传距离最大的分别为黑边石斑鱼与斑鳃棘鲈(Cyt b、联合基因)、棕斑石斑鱼与斑鳃棘鲈(16S)、鮭点石斑鱼与斑鳃棘鲈(12S);最小的分别为豹纹鳃棘鲈与斑鳃棘鲈(Cyt b、12S、联合基因)、赤点石斑鱼与青石斑鱼(16S)(表6)。
另一方面,就石斑鱼属9个种而言,遗传距离最大的为鞍带石斑鱼与青石斑鱼(Cyt b)、鞍带石斑鱼与黑边石斑鱼(16S、联合基因)、褐点石斑鱼与黑边石斑鱼(12S);遗传距离最小的均为赤点石斑鱼与青石斑鱼。
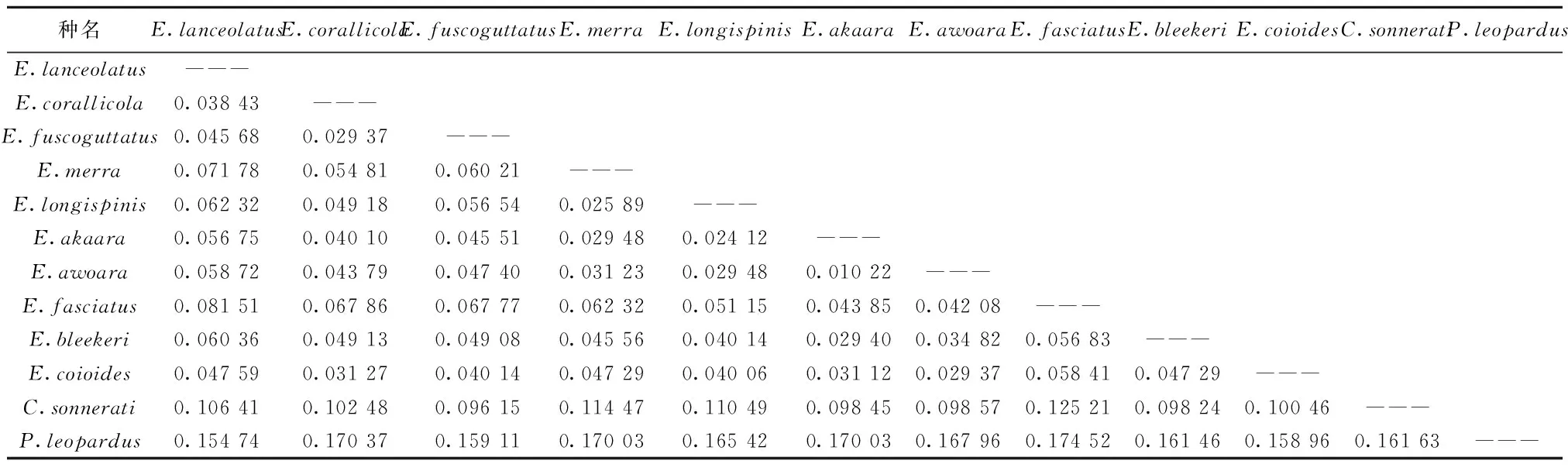
根据线粒体Cyt b基因序列构建NJ和MP系统树(图1),均表明13种石斑鱼类分别按所属的石斑鱼属、九棘鲈属和鳃棘鲈属各自聚类,形成3个单系的聚类群。石斑鱼属与九棘鲈属亲缘关系较近,首先聚类;鳃棘鲈处于系统树的最基部。在NJ系统树,石斑鱼属形成两个聚类群(为方便描述起见,简称为A群和B群),A群由赤点石斑鱼、青石斑鱼、黑边石斑鱼、蜂巢石斑鱼、鮭点石斑鱼以及布氏石斑鱼组成,B群则包含褐点石斑鱼、鞍带石斑鱼、斜带石斑鱼以及棕斑石斑鱼。MP系统树支持了B群的聚类,而A群中的种类则表现为阶梯式的姐妹枝。在两种系统树中,两两相聚且有相对较高的置信值(>50)有:赤点石斑鱼和青石斑鱼、蜂巢石斑鱼与鮭点石斑鱼、斜带石斑鱼与棕斑石斑鱼,表明它们具有较近的亲缘关系。
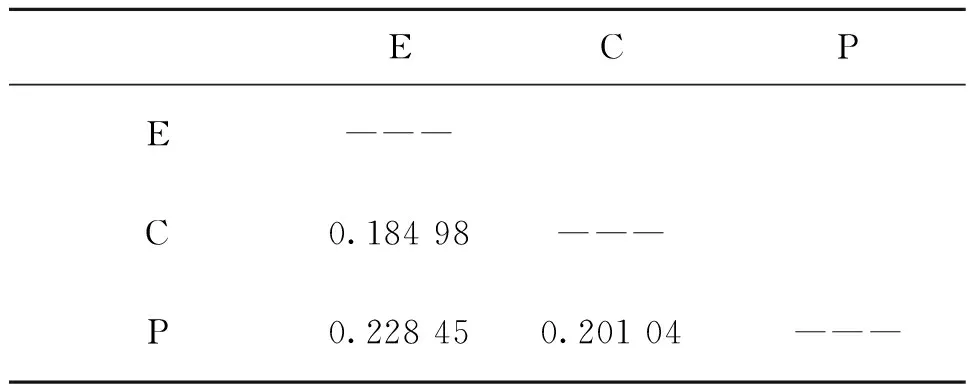
表5 13种石斑鱼类属间的遗传距离Table 5 Intergeneric genetic distance of 13 Epinepheline species
a.Intergeneric genetic distance——Cyt b

ECPE―――C0.10577―――P0.164280.16270―――
b.Intergeneric genetic distance——16S

ECPE―――C0.06768―――P0.141620.11340―――
c.Intergeneric genetic distance——12S

ECPE―――C0.11570―――P0.176400.15820―――
d.Intergeneric genetic distance——combined genes
(注: E=Epinephelus,C=Cephalopholis,P=Plectropomus)

表6 13种石斑鱼类种间的遗传距离Table 6 Interspecies genetic distance of 13 Epinepheline species
a.Interspecies genetic distance——Cyt b
种名E.lanceolatusE.corallicolaE.fuscoguttatusE.merraE.longispinisE.akaaraE.awoaraE.fasciatusE.bleekeriE.coioidesC.sonneratiP.leopardusE.lanceolatus―――E.corallicola0.03843―――E.fuscoguttatus0.045680.02937―――E.merra0.071780.054810.06021―――E.longispinis0.062320.049180.056540.02589―――E.akaara0.056750.040100.045510.029480.02412―――E.awoara0.058720.043790.047400.031230.029480.01022―――E.fasciatus0.081510.067860.067770.062320.051150.043850.04208―――E.bleekeri0.060360.049130.049080.045560.040140.029400.034820.05683―――E.coioides0.047590.031270.040140.047290.040060.031120.029370.058410.04729―――C.sonnerati0.106410.102480.096150.114470.110490.098450.098570.125210.098240.10046―――P.leopardus0.154740.170370.159110.170030.165420.170030.167960.174520.161460.158960.16163―――
b.Interspecies genetic distance——16S

种名E.awoaraE.akaaraE.bleekeriE.fasciatusE.merraE.longispinisE.fuscoguttatusE.coioidesE.lanceolatusE.corallicolaC.sonneratiP.leopardusE.awoara―――E.akaara0.01871―――E.bleekeri0.050470.03813―――E.fasciatus0.050310.040470.05031―――E.merra0.037940.028280.043020.04525―――E.longispinis0.057900.047950.058100.055210.02354―――E.fuscoguttatus0.073730.063250.076080.086470.076230.07874―――E.coioides0.070820.065610.078470.078230.068100.067990.03320―――E.lanceolatus0.078600.068100.080990.078130.075830.075710.040530.03560―――E.corallicola0.073320.065510.078340.070490.073200.073090.038060.028280.02833―――C.sonnerati0.080990.075710.083650.083200.078230.086060.052910.047870.042820.04531―――P.leopardus0.149610.140370.158130.151350.157470.163260.117900.129200.117770.123370.11248―――
c.Interspecies genetic distance——12S

种名E.lanceolatusE.fuscoguttatusE.coioidesE.corallicolaE.akaaraE.awoaraE.fasciatusE.bleekeriE.merraE.longispinisC.sonneratiP.leopardusE.lanceolatus―――E.fuscoguttatus0.06490―――E.coioides0.070370.06183―――E.corallicola0.065090.058080.05050―――E.akaara0.088420.072570.074070.08216―――E.awoara0.100740.078910.070930.087000.03783―――E.fasciatus0.109940.099410.100230.097810.079660.07561―――E.bleekeri0.090610.077910.085010.083530.058850.065750.08362―――E.merra0.100450.081260.080210.093360.059550.062500.089090.06720―――E.longispinis0.090610.078070.083400.079540.062570.075020.085140.068760.04832―――C.sonnerati0.111000.102780.105420.111290.115280.114440.141490.111780.122630.12093―――P.leopardus0.162540.158470.165420.167420.179860.178300.183020.172700.182300.183090.15330―――
d.Intergeneric genetic distance——combined genes
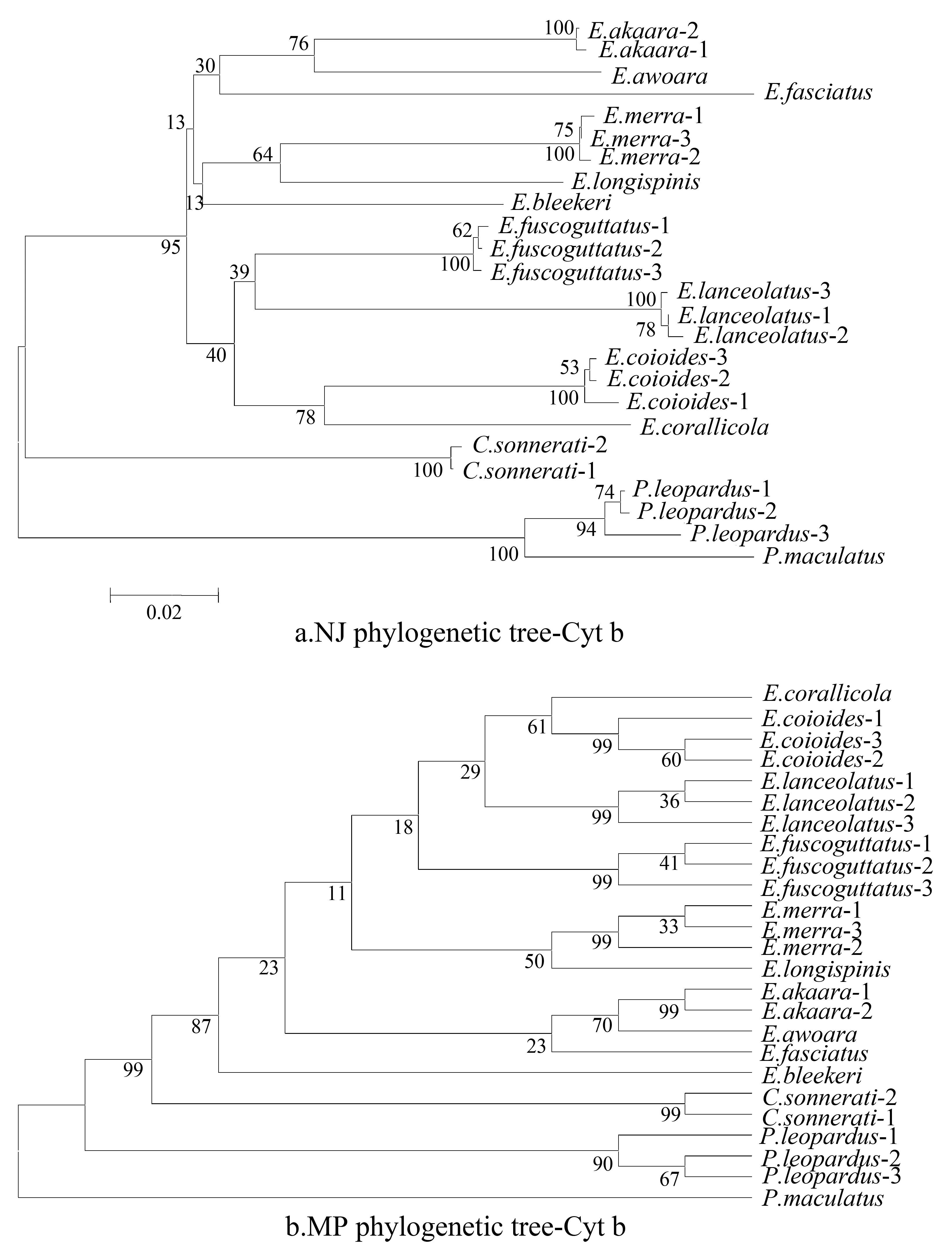
图1 基于13种石斑鱼类Cyt b基因序列的NJ和MP系统树Fig.1 NJ and MP phylogenetic trees based on Cyt b gene sequences of 13 Epinepheline species
基于16S和12S rRNA基因序列构建的NJ和MP系统树(图2,3)的拓扑结构与线粒体Cyt b基因的相似,如13个种各自按所属聚类形成3个单系群,三个属在系统树所处的位置以及石斑鱼属形成的两个聚类群等。不同之处在于石斑鱼属中一些种所处的位置,如在16S-NJ系统树中,斜带石斑鱼与A群相聚(而在Cyt b/12S-NJ系统树中斜带石斑鱼归类于B群);此外16S和12S-MP系统树支持的是A群的聚类,而B群中的种类则表现为阶梯式的姐妹枝;16S和12S系统树均不支持斜带石斑鱼与棕斑石斑鱼具有较近的亲缘关系。
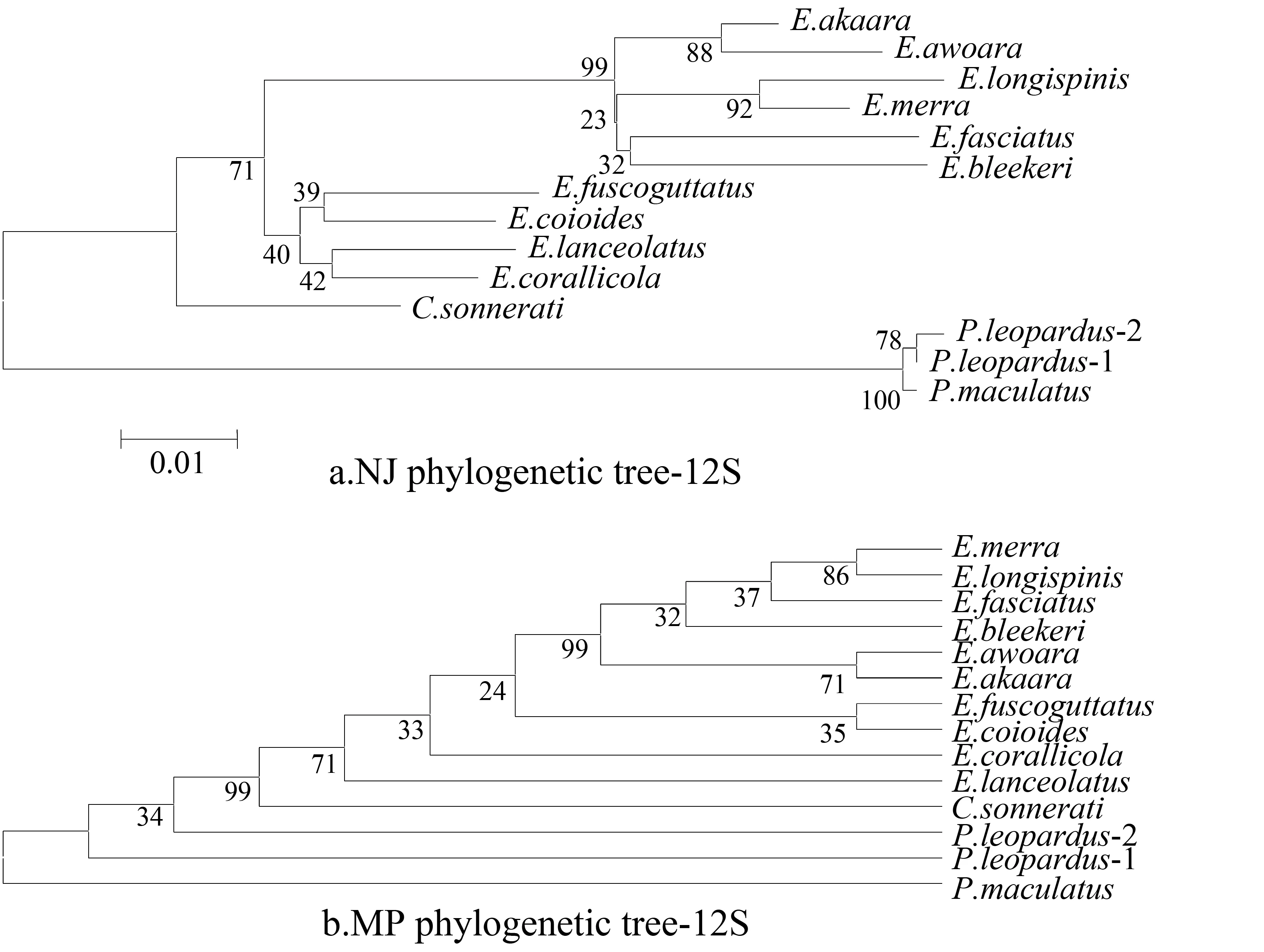
图3 基于13种石斑鱼类12S rRNA基因序列的NJ和MP系统树Fig.3 NJ and MP phylogenetic trees based on 12S rRNA gene sequences of 13 Epinepheline species
相比较与上述单个基因所构建的系统树,联合基因序列的系统树明显具有更好的一致性(NJ和MP的拓扑结构基本一致)和更高的置信度(系统树分枝的置信值高)(图4)。联合基因系统树不仅支持13个种各自按所属聚类形成3个单系群以及3个属在系统树所处的位置,而且以极高的置信值(100)支持石斑鱼属的两个聚类群。此外它还支持了Cyt b系统树分析得到的赤点石斑鱼和青石斑鱼、蜂巢石斑鱼与鮭点石斑鱼、斜带石斑鱼与棕斑石斑鱼具有较近的亲缘关系的结论。

图4 基于13种石斑鱼类联合基因序列的NJ和MP系统树Fig.4 NJ and MP phylogenetic trees based on combined gene sequences of 13 Epinepheline species
3 讨 论
3.1 线粒体基因序列变异与石斑鱼类的种类鉴定
准确鉴定石斑鱼类的种类是进一步研究其分类关系和系统演化过程的基础;在生产实践中,对石斑鱼类的产品交易、鱼类自然资源的保护和利用也有着重要的意义。鱼类分类学家Heemstra和Randall[1]在编著的《Groupers of the world》引言的第一段即指出:“准确种类鉴定的重要性对于每一个生物学家是显而易见的,将得到养殖者和分类学家的重视,同时对正确管理该物种的渔业资源也极为重要”。然而,由于石斑鱼类的种类繁多、形态类似、体色易变等,使得基于形态特征的传统种类鉴定较为复杂困难,并出现了许多鉴定错误的事例。分子标记(如线粒体基因序列)反映的是物种内在的遗传差异,不受外在形态的影响,故已经成为鱼类种类鉴定的有力工具[18-19]。
文章中对13种石斑鱼类的线粒体Cyt b、16S rRNA以及12S rRNA基因的部分片段进行了扩增和测序,结果表明:石斑鱼类种间基因序列存在较大的变异,如Cyt b基因片段的变异位点高达153个,更为保守的rRNA基因也存在139(16S)和88(12S)个变异位点;另一方面,13种石斑鱼类种内基因序列的变异较小,在Cyt b和16S rRNA基因中均有近半数(6~7种)石斑鱼类种内序列完全相同,而其余种类通常只有1~3个变异位点。12S rRNA基因种内变异极小,除一种石斑鱼(豹纹鳃棘鲈)种内存在1个位点差异外,其余石斑鱼种内序列相同。显然,种间较大的遗传差异以及极小的种内变异使得这些线粒体基因十分适合用于石斑鱼类的鉴定。通过线粒体基因的变异位点,就可以非常准确的鉴别这13种石斑鱼类。
通过计算变异位点数、简约信息位点数、核苷酸多样性、平均核苷酸差异数等遗传信息指数,可知12S基因的保守性最大,16S次之,而Cyt b基因保守性最小。Cyt b基因相对较小的保守性使其能提供更多用于种类鉴定的变异位点,因而作为鉴定标记较rRNA基因更为准确。但这并不表明保守性越小的基因就更适合用于石斑鱼类的种类鉴定。
3.2 13种石斑鱼类的系统进化及亲缘关系分析
近年来,许多鱼类学者已经从单纯的分类区系研究转向鱼类系统进化方面。他们综合利用形态学、生物化学、分子生物学等方法来辨别和分析祖征和离征,藉以探求鱼类系谱,探索共同起源,真正反映出彼此间的亲缘关系[20]。
文章中根据所测定的13种石斑鱼类的线粒体Cyt b、16S rRNA以及12S rRNA基因的部分片段,计算遗传距离并构建NJ和MP系统树进行亲缘关系分析。结果表明,3个基因构建的系统树拓扑结构大体一致,如石斑鱼属、九棘鲈属和鳃棘鲈属在系统树所处的位置以及石斑鱼属形成的两个聚类群等。但在石斑鱼属中,一些种类在系统树中所处的位置随基因的不同或建树方法的不同而改变,且分枝节点的置信值较低。
单个基因(特别是保守性较高的基因)能提供的遗传信息往往较为有限[21]。在无其它背景资料的情况下,为了获得较为客观的系统树,不少学者主张线粒体基因组不同区域的多组序列数据的联合分析[22]。Miller 等[23]研究发现,多基因联合比单独进行分析得到的系统树分辨率及置信值更高,且被形态性状所支持,即使基因间的进化速率差异较大。随后更多研究发现同样现象,支持了将同一样本所有具有系统发育信息的基因合并在一起进行分析,可以获得更真实系统进化关系[24-25]。
文章中将所测序的线粒体3个基因进行联合分析。相比较单个基因,联合基因序列构建的系统树明显具有更好的一致性(NJ和MP的拓扑结构基本一致)和更高的置信度(系统树分枝的置信值高)。结合本文的结果与上述其他学者的研究结果,作者认为基因联合分析所得到的石斑鱼类系统进化关系可信度更高,现描述如下:13种石斑鱼类分别按所属的石斑鱼属、九棘鲈属和鳃棘鲈属各自聚类,形成3个单系的聚类群。石斑鱼属与九棘鲈属亲缘关系较近,首先聚类。石斑鱼属处于系统树的顶端,表明其最近分化的进化地位;鳃棘鲈处于系统树的最基部,表明其为较为原始的种类。石斑鱼属形成两个聚类群,一枝由赤点石斑鱼、青石斑鱼、黑边石斑鱼、蜂巢石斑鱼、鮭点石斑鱼以及布氏石斑鱼组成,另一枝则包含褐点石斑鱼、鞍带石斑鱼、斜带石斑鱼以及棕斑石斑鱼。其中,赤点石斑鱼和青石斑鱼、蜂巢石斑鱼与鮭点石斑鱼、斜带石斑鱼与棕斑石斑鱼两两相聚且有相对较高的置信值,表明它们具有较近的亲缘关系。
[1] HEEMSTRA P C,RANDALL J E.Groupers of the world (Family Serranidae,Subfamily Epinephelinae):An annotated and illustrated catalogue of the grouper,rock cod,hind,coral grouper and lyretail species known to date[M].FAO Fisheries Synopsis,No 125,Vol 16.Rome: Food and Agriculture Organization of the United Nations,1993.
[2] IUCN.IUCN Red List Categories and Criteria[M].Ver 3.1.Gland and Cambridge:IUCN Species Survival Commission,2001.
[3] 蒙子宁,杨丽萍,吴丰,等.斜带石斑鱼、赤点石斑鱼RAPD和线粒体Cyt b基因序列变异分析[J].中山大学学报:自然科学版,2007,1(46):75-80.
[4] SMITH C L.A revision of the American Groupers:Epinephelusand allied genera[J].Bull Am Mus Nat Hist,1971,146(2):67-242.
[5] RANDALL J E,BEN-TUVIA A.A review of the groupers (Pisces:Serranidae: Epinephelinae) of the Red Sea,with description of a new species ofCephalopholis[J].Bulletin of marine Science,1983,33(2):373-426.
[6] JOHNSON G D.Niphonspinosus,a primitive epinepheline serranid:corroborative evidence from the larvae[J].Japanese Journal of Ichthyology,1988,35(1):7-18.
[7] BALDWIN C C,JOHNSON D G.Phylogeny of the Epinephelinae (Teleostei: Serranidae)[J].Bulletin of Marine Science,1993,52(1):240-283.
[8] NELSON J S. Fishes of the World[M].4th ed. New York: John Wiley & Sons Inc, 2006.
[9] LIU Z J,CORDES J F.DNA marker technologies and their applications in aquaculture genetics[J].Aquaculture,2004,238(4):1-37.
[10] 杨慧荣,赵会宏,蒙子宁,等.赤眼鳟线粒体D-loop和Cyt b基因序列的对比分析[J].中山大学学报:自然科学版,2012,(51)5:100-107.
[11] 高天翔,毕潇潇,赵林林,等.基于线粒体 Cyt b 基因全序列的松江鲈群体遗传结构分析[J].水生生物学报,2013,37(2):199-207.
[12] MIYA M,NISHIDA M.Molecular phylogenetic perspective on the evolution of the deep-sea fish genusCyclothone(Stomiiformes:Gonostomatidae)[J].Ichthyological Research,1996,43(4):375-398.
[13] MORITA T.Molecular Phylogenetic Relationships of the Deep-Sea Fish GenusCoryphaenoides(Gadiformes:Macrouridae) Based on Mitochondrial DNA[J].Molecular Phylogenetics and Evolution,1999,13(3):447-454.
[14] WATERS J M,WALLIS G P.Mitochondrial DNA phylogenetics of theGalaxiasvulgariscomplex from South Island,New Zealand:rapid radiation of a species flock[J].Journal of Fish Biology,2001,58(4):1166-1180.
[15] CRAIG M T,PONDALLA D J,FRANCK J P C,et a1.On the status of the serranid fish genusEpinephelus:evidence for paraphyly based upon 16S rDNA sequence[J].Mol Phylogenet,2001,19:121-130.
[16] 丁少雄,王颖汇,王军,等.基于16S rDNA 部分序列探讨中国近海 30 种石斑鱼类的分子系统进化关系[J].动物学报,2006,52(3):504-513.
[17] SAMBROOK J,FRITSCH E F,MANIATIS T.Molecular cloning[M].New York:Cold spring harbor laboratory press,1989.
[18] SUNEETHA K B,DAHLE G,N☞VDAL G.Analysis of mitochondrial DNA sequences from two Maurolicus taxa:evidence for separate species?[J].Journal of fish biology,2000,57(6):1605-1609.
[19] HAN Z Q,GAO T X,YANAGIMOTO L,et a1.Genetic population structure ofNibeaalbiflorain the yellow and east China seas[J].Fisheries Science,2008,74(3):544-552.
[20] 孟庆文,苏锦祥,缪学祖.鱼类分类学[M].北京:中国农业出版社,1995.
[21] CAO Y,SORENSON M D,KUMAZAWA Y,et al.Phylogenetic position of turtles among amniotes:evidence from mitochondrial and nuclear genes[J].Gene,2000,259(1):139-148.
[22] WIENS J J,REEDER T W.Combining data sets with different numbers of taxa for phylogenetic analysis[J].Systematic Biology,1995,44(4):548-558.
[23] MILLER J S,BROWER A V,DESALLE R.Phylogeny of the neotropical moth tribe Josiini (Notodontidae:Dioptinae):comparing and combining evidence from DNA sequences and morphology[J].Biological Journal of the Linnean Society,1997,60(2):297-316.
[24] WIENS J J.Combining data sets with different phylogenetic histories[J].Systematic Biology,1998,47(4):568-581.
[25] OBERMILLER L E,PFEILER E.Phylogenetic relationships of elopomorph fishes inferred from mitochondrial ribosomal DNA sequences[J].Molecular Phylogenetics and Evolution,2003,26(2):202-214.

