东北地区耐盐芽胞杆菌筛选统计及初步鉴定
杜传英 李海涛 刘荣梅 谢滨姣 张金波 高继国
(东北农业大学生命科学学院,哈尔滨 150030)
东北地区耐盐芽胞杆菌筛选统计及初步鉴定
杜传英 李海涛 刘荣梅 谢滨姣 张金波 高继国
(东北农业大学生命科学学院,哈尔滨 150030)
采用温度筛选与表面定向培养相结合的方法对东北地区土壤中可培养耐盐芽胞杆菌进行分离和筛选,得到137株芽胞杆菌,其中耐盐芽胞杆菌74株,占总芽胞杆菌数量的54%,最适盐浓度均为1%,耐盐能力在4%-14%之间。通过扩增耐盐菌株的16S rRNA基因序列,对其进行分子鉴定和分类,获得东北地区土壤中可培养的耐盐芽胞杆菌的多样性信息。通过同源性比对和耐盐性差异,确定36株差异耐盐芽胞杆菌,分属于芽胞杆菌属中的7个种。其中多数菌株为苏云金芽胞杆菌(Bacillus thuringiensis)(14株,占总数38.9%,最高耐盐性在4%-9%NaCl之间)。其次依次为蜡样芽胞杆菌(Bacillus cereus)(7株,19.4%,4%-8%NaCl),枯草芽胞杆菌(Bacillus subtilis)(7株,19.4%,8%-11%NaCl),炭疽芽胞杆菌(Bacillus anthracis)(4株,11.1%,5%-7%NaCl),弯曲芽胞杆菌(Bacillus flexus)(2株,5.6%,9%-14%NaCl),球形芽胞杆菌(Bacillus sphaericus)(1株,2.8%,5%NaCl)和阿氏芽胞杆菌(Bacillus aryabhattai)(1株,2.8%,6%NaCl)。弯曲芽胞杆菌和枯草芽胞杆菌的耐盐能力较好。从中选取3株代表菌株,明确了其培养特性、形态特征及生理生化特性,并对其分别进行了系统发育分析。
耐盐 芽胞杆菌 分离 初步鉴定 16S rRNA
盐碱地多数情况下较为平坦便于灌溉,同时又是世界性的低产土壤,因此长期以来都受到人们广泛的关注[1]。东北地区素以肥沃的黑土地著称,是我国重要的农、林业和畜牧业生产基地。但随着气候的变化和土壤的不合理利用,东北地区盐碱土面积达到了384万hm2,占该地区总面积的3.1%,其
中耕种盐碱地面积占该地区总耕地面积的6.8%,已成为我国盐碱土四大分布区之一[2],严重制约着该地区的农林畜牧业和经济发展。
目前我国对于盐碱地的改良措施主要以物理、水利、生物和化学四大体系为核心[3]。 其中生物改良主要应用高效耐盐微生物与植物的协同作用[4],在恢复盐碱地的同时还可以保护生态平衡,在改善盐渍土质方面发挥着重要的作用。由于多数植物和微生物肥料不具有抵抗高盐的能力,而细菌与植物在耐盐方面又有较大的相似性,也有些植物可以与耐盐菌共生[5]。因此,构建高效耐盐细菌资源库以及发掘耐盐功能基因应运而生。
芽胞杆菌为革兰氏阳性菌,菌体杆状,直或近直,(0.3-2.2)μm×(1.2-7.0)μm,多数运动,鞭毛典型侧生,形成抗热内生孢子[6]。2004年,El-Komy等[7]发现多粘芽胞杆菌(Bacillus polymexa)E.M1231菌株能增加盐胁迫下玉米鲜重和干物质产量。2006年,尹汉文[8]发现在一定浓度氯化钠胁迫下,枯草芽胞杆菌可以在一定程度内提高黄瓜植株的耐盐性。对于模式菌株枯草芽胞杆菌耐盐性的研究也一直备受关注[9-11]。芽胞杆菌由于产生内生孢子,可以有效的抵抗外界环境的压力,维持自身生长不受影响,是抗逆性最强的生命体。近年来在工、农、医等各个科学领域都有广泛的应用,研究总体呈加速上升的趋势[12],因此统计发掘耐盐芽胞杆菌,旨在为构建高效耐盐转基因植物提供重要数据支持以及基因资源储备。20 mim后进行试验。定向耐盐筛选培养基:LB培养基中加入梯度浓度的NaCl。
1.2 方法
1.2.1 耐盐芽胞杆菌的筛选与统计
1.2.1.1 土样采集与芽胞杆菌的分离 广泛采集东北各地区土壤样品,先将表土层铲去,取距表层5-10 cm的土壤作为样本,装入无菌封口袋中,标明采集日期、地点、周围植被等,每个采样地点至少取10处(表1)。
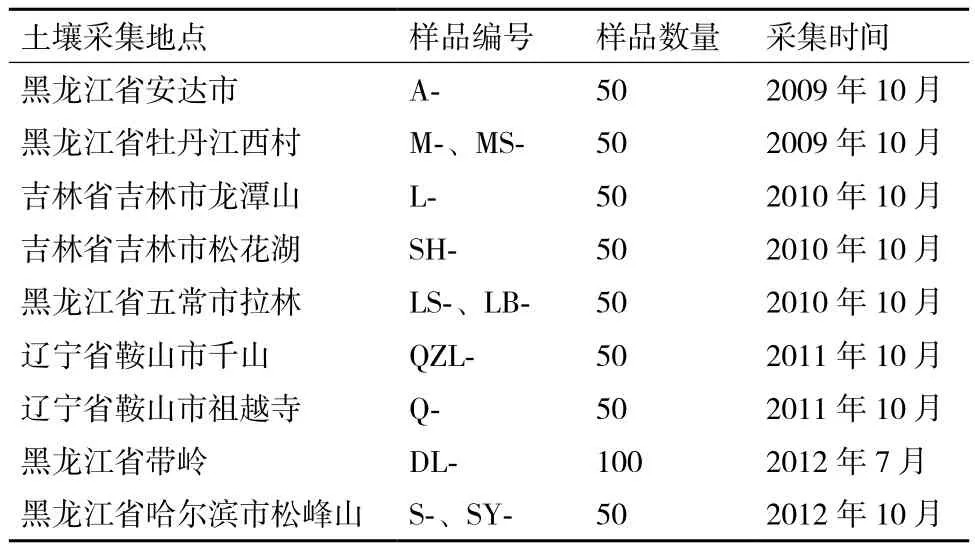
表1 供试土壤样品
1.2.1.2 土壤样品中芽胞杆菌的分离及纯化 取1-2 g土样放入装有15 mL灭菌水和适量玻璃珠的50 mL灭菌的大离心管中,在旋涡振荡器上振荡约1 min,将土壤颗粒打碎,然后80℃水浴20 min,充分将非芽胞菌杀死。待稍静置后,稀释500倍后吸取100 μL菌悬液于1/2 LB板上,涂布均匀,30℃培养3 d。挑取菌落涂片,用石炭酸复红染色,100倍油镜下观察。检出菌株划线于LB培养基上培养,4℃保存。
1.2.1.3 菌株生长温度测试 将分离得到的纯培养芽胞杆菌接种于LB液体培养基中,分别置于16℃、22℃、30℃、37℃条件下,220 r/min震荡培养48 h,取样稀释10倍,测量600 nm下OD值,绘制曲线。
1.2.1.4 菌株耐盐性测试 将分离得到的纯培养芽胞杆菌分别接种于含有0%-15%NaCl的LB培养基平板上,30℃恒温培养48 h,观察生长情况,统计不同菌株的最高耐盐度。
1.2.1.5 基因组DNA提取 菌株在固体LB培养基上划线培养24 h后收集到1.5 mL EP管中,约100 mg,加入200 μL TE,200 μL 2% SDS,充分混匀,然后加入200 μL Tris饱和酚,200 μL氯仿,混匀后,
1 材料与方法
1.1 材料
1.1.1 主要仪器与试剂 恒温培养箱购自上海一恒科技有限公司;PCR仪购自百瑞德进出口贸易有限公司;凝胶成像分析仪购自赛智创业科技有限公司;PCR相关试剂购自北京康为世纪生物科技有限公司。T4 DNA ligase、DNA Fragment Purification Kit和DNA Marker均购自TaKaRa公司。生理生化鉴定试剂盒购于海博生物有限公司。
1.1.2 培养基 LB液体培养基:trytone 1%,yeast extract 0.5%,NaCl 1%,pH调为7.0。LB固体培养基:LB液体培养基中加入1.3% 琼脂,121℃灭菌
12 000 r/min离心10 min。取上清,加入1.5倍体积的异丙醇,50 μL的5 mol/L NaCl,轻轻振荡混匀,12 000 r/min离心10 min,弃上清,沉淀用无水乙醇洗涤3次,室温晾干。用适量的ddH2O溶解沉淀,短期-20℃保存,长期-70℃保存备用。
1.2.1.6 基于16S rRNA基因序列的系统发育分析 对菌株基因组的16S rRNA序列进行PCR扩增,采用的通用引物[13]为16S rRNA-F:5'-AGAGTTTGATCCTGGCTCAG-3';16S rRNA-R1:5'-ACGGCTACCTTGTTACGACT-3';16S rRNA-R2:5'- AAGGAGGTGATCCAGCCGCA-3'。反应条件为:94℃ 8 min;94℃ 1 min,52℃ 1 min,72℃ 2 min,共30个循环;72℃ 10 min。扩增产物经DNA纯化试剂盒纯化后连接pMD19-T载体,转化大肠杆菌JM109,蓝白斑筛选阳性克隆,PCR验证后,提质粒送交测序。引物合成和序列测定均由金唯智生物科技有限公司完成。所得序列应用NCBI Blast在线比对,找出各序列在NCBI数据库中最亲缘种的模式菌株序列,采用Clustal X和MEGA5.0软件进行多序列比对,然后用Neighbor-Joining方法选择 Bootstrap为1 000个重复构建系统发育学进化树。新菌株16S rRNA序列提交GeneBank数据库,获取序列号。
1.2.2 优势菌株的初步鉴定
1.2.2.1 最高盐浓度下菌株的生长情况 将优势菌株接种于LB液体培养基中,30℃,200 r/min培养12 h,离心收集菌体,用无菌水洗涤菌体后制成菌悬液,取1 mL菌悬液以1∶100的比例接种于该菌株最高耐盐浓度的液体培养基中,30℃,200 r/min摇床培养,每隔1 h测其菌液600 nm下的OD值,并绘制生长曲线。
1.2.2.2 菌株形态特征观察 菌株接种于LB固体培养基中,30℃培养2 d,挑取菌体,石炭酸复红染色,在10×100倍的光学显微镜下观察芽胞形成后,刮取培养菌用无菌水洗2-3遍,12 000 r/min离心1 min收集菌体,将沉淀悬浮于1 mL无菌水中,取适量胞晶混合液滴于玻璃片上,待干燥后,经锇酸固定,而后经酒精梯度脱水,临界点干燥,离子溅射喷金(2 nm),进一步的扫描电镜观察。电镜(HITACHI S-3400N)分析在东北农业大学大型仪器开放平台电镜室完成。
1.2.2.3 生理生化特性鉴定 参照生理生化鉴定试剂盒操作说明书。
2 结果
2.1 耐盐芽胞杆菌筛选与统计
采用温度筛选法从东北地区采集的500份土样中筛选得到137株芽胞杆菌,本研究中东北地区芽胞杆菌出土率约为27.4%。进一步采用表面定向培养的方法,调整基本LB培养基中NaCl浓度,分别配成0%-20%NaCl的培养基,进行耐盐芽胞杆菌筛选,得到可以在含有4%-14%NaCl的培养基中生长的耐盐芽胞杆菌74株,占总芽胞杆菌数量的54%,说明半数以上的芽胞杆菌对于外界高盐环境具有一定的耐受能力。所有芽胞杆菌在NaCl含量为1%时,菌落形态饱满,生长旺盛,在高盐环境下菌落形态明显变小、生长缓慢或不能生长,在不含NaCl环境下生长速度明显下降,菌落形态小,光学显微镜下观察营养体少,有大量芽胞产生,故本试验中分离出的所有芽胞杆菌最适生长的NaCl浓度均为1%,为耐盐菌没有嗜盐菌。图1为137株芽胞杆菌在基本LB培养基和高盐培养基中可生长的菌株数量,本试验中大部分芽胞杆菌的耐盐性在8%以下,得到两株耐盐性10%以上的芽胞杆菌。
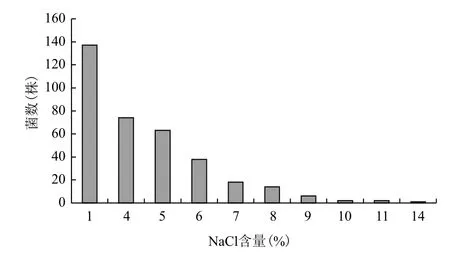
图1 东北地区耐盐芽胞杆菌的统计
2.2 基于16S rRNA基因序列的系统发育分析
提取74株耐盐芽胞杆菌的基因组,用通用引物扩增16S rRNA基因序列,测序结果应用DNAMAN软件进行序列分析比对,结合菌株的耐盐性差异,得到36株差异耐盐芽胞杆菌用于后续分析菌株多样性。将得到的16S rRNA基因序列与在线NCBI数据库中的序列比对,选择相似度最高的模式菌株应用
上述方法构建Neighbor Joining Tree(图2)。由于苏云金芽胞杆菌与蜡样芽胞杆菌的16S rRNA序列的同源性非常高,鉴于苏云金芽胞杆菌在芽胞期分泌伴孢晶体,结合光学显微镜下观察的结果,区分苏云金芽胞杆菌与蜡样芽胞杆菌。结果显示,该36株耐盐芽胞杆菌分属于芽胞杆菌属中的7个种:苏云金芽胞杆菌14株(占总数38.9%)、蜡样芽胞杆菌7株(19.4%)、枯草芽胞杆菌7株(19.4%)、炭疽芽胞杆菌4株(11.1%)、弯曲芽胞杆菌2株(5.6%)、球形芽胞杆菌1株(2.8%),阿氏芽胞杆菌1株(2.8%)。该36株代表菌株与最近缘种的模式菌株的16S rRNA基因序列相似性在99.7%-100%之间,其中有22株菌株相似性在99.7%-99.9%之间,17株菌株的16S rRNA基因序列已提交GenBank获得登录号分别为:KJ022700、KJ496370- KJ496385。
2.3 各种芽胞杆菌耐盐性统计
耐盐性最好的菌株为LBR-4(Bacillus flexus)最高耐盐度为14% NaCl和S5-1(Bacillus subtilis)最高耐盐度为11%。但在高盐胁迫下这两株菌株的菌落形态都较正常培养基中小,再转接到正常培养基中会恢复到正常生长状态,说明高盐胁迫对菌株的正常生长有一定的影响。总体菌株耐盐性统计结果显示,耐盐性较好的菌株为弯曲芽胞杆菌(最高耐盐度9%-14%NaCl)和枯草芽胞杆菌(8%-11%)。其它种芽胞杆菌耐盐性差别不大,多数菌株的耐盐性在7%以下(苏云金芽胞杆菌,4%-9%;蜡样芽胞杆菌,4%-8%;枯草芽胞杆菌,8%-11%;炭疽芽胞杆菌,5%-7%;球形芽胞杆菌,5%;阿氏芽胞杆菌,6%)。图3显示了各芽胞杆菌在不同盐含量的培养基中可生长菌的数量。
2.4 代表菌株形态特征
选取3株代表菌株分别为耐盐性超过10%的弯曲芽胞杆菌LBR-4、枯草芽胞杆菌S5-1以及最高耐盐9%的苏云金芽胞杆菌DL1-5进行初步鉴定分析。代表菌株的菌体形态如图4,总体上菌体均为杆状,石炭酸复红染色后显微镜下观察呈红色,芽胞为椭球形,染色后呈空心粉色。苏云金芽胞杆菌DL5-1菌株可以在芽胞期分泌出球形伴孢晶体,染色后呈均一的红色。弯曲芽胞杆菌LBR-4的营养体相对较长,而枯草芽胞杆菌S5-1的芽胞相对较圆。
2.5 代表菌株的培养特性及生理生化指标
将3株菌株分别扩增繁殖后,按一定比例分别接种于LB培养基和相应最高耐盐度的培养基中进行摇床培养,每隔1 h取样测量600 nm处OD值,绘制如图5的菌株生长曲线。菌株在LB培养基中2.5 h左右开始进入对数生长期,而在相应最高盐胁迫下有将近10-12 h的调整期。说明芽胞杆菌有很强的适应能力,菌株最开始并不能适应高盐环境,但是经过一段时间的调整后仍能生长。菌株在LB培养基中从对数期到稳定期经过近14 h,与高盐胁迫下经历的时间基本相同,但在高盐胁迫下进入稳定期后的OD值有2-2.5倍的差异。
代表菌株LBR-4、S5-1及DL1-5最适生长盐浓度均为1%,最高耐盐度分别为14%、11%和9%,最适生长温度为30℃(图6)。V-P反应均为阳性。菌株LBR-4可以利用葡萄糖、半乳糖和蔗糖作为唯一碳源生长,不能利用纤维二糖、甘露糖、七叶苷、水杨苷。可以利用L-精氨酸,不能利用尿素酶。菌株S5-1可以利用葡萄糖、蔗糖、纤维二糖、七叶苷和水杨苷作为唯一碳源生长,不能利用半乳糖和甘露糖。不能利用L-精氨酸,可以利用尿素酶。菌株DL1-5可以利用葡萄糖、蔗糖、纤维二糖和七叶苷作为唯一碳源生长,不能利用半乳糖、甘露糖、水杨苷。不能利用L-精氨酸和尿素酶(表2)。
2.6 代表菌株的系统发育分析
将同为弯曲芽胞杆菌的LBR-4和MS14-1菌株16S rRNA基因序列与已鉴定的模式菌株16S rRNA基因序列比较后,结果(图7)显示这两株菌株均与模式菌株Bacillus flexus IFO 15715聚在一起,形成一个单独的分支。通过序列两两比较,LBR-4菌株与模式菌株Bacillus flexus IFO 15715的相似性为99.87%,分别在第167和173个碱基处有2个碱基的差异。MS14-1菌株与模式菌株Bacillus flexus IFO 15715的相似性为99.93%,在第173个碱基处有1个碱基差异,LBR-4菌株与MS14-1菌株相似性为99.93%。
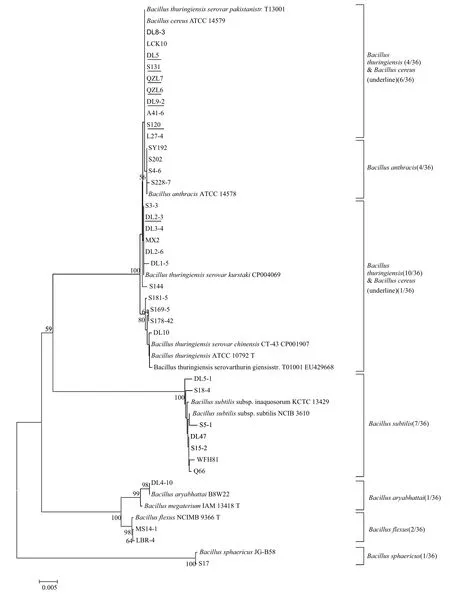
图2 基于16S rRNA基因序列构建的东北地区耐盐芽胞杆菌系统发育树
将同为枯草芽胞杆菌的7株菌株16S rRNA基因序列与已鉴定的模式菌株比较后,结果(图8)显
示这7株菌株均与模式菌株Bacillus subtilis的3个不同亚种聚在一起,形成一个单独分支。其中S5-1菌株和S18-4与模式菌株Bacillus subtilis subsp. subtilis NCIB 3610的亲缘关系最近,其他5株菌与模式菌株Bacillus subtilis subsp. inaquosorum KCTC 13429的亲缘关系较近。通过序列两两比较,S5-1菌株与模式菌株Bacillus subtilis subsp. subtilis NCIB 3610的相似性为99.80%,分别在第222、456和1019个碱基处有3个碱基差异。S18-4菌株与模式菌株Bacillus subtilis subsp. subtilis NCIB 3610的相似性也为99.80%,分别在第276、476和1061个碱基处有3个碱基差异。S5-1菌株与S18-4菌株的相似性为99.60%,有6个碱基的差异。

图3 各种芽胞杆菌耐盐性统计
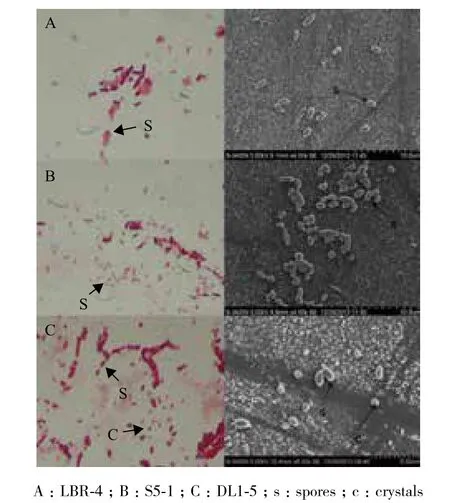
图4 3株代表菌株的革兰氏染色和扫描电镜图片
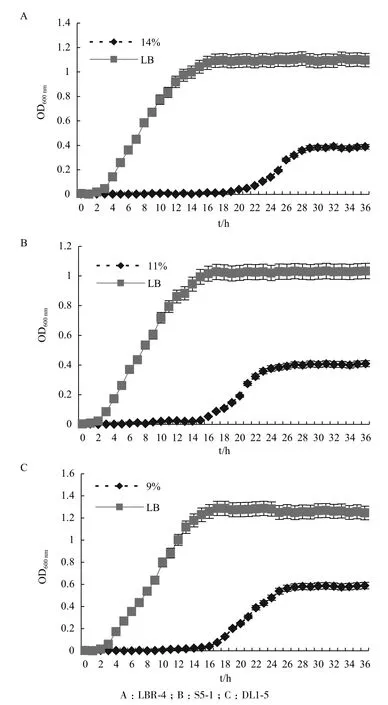
图5 三株代表菌株在正常LB培养基和最高耐盐度培养基中的生长曲线

图6 三株代表菌株不同温度下的生长曲线
通过16S rRNA基因序列的两两比对,将与
DL1-5菌株同源性较高的4株Bt菌株与Bt各亚种模式菌株的16S rRNA基因序列比较后,结果(图9)显示,这5株Bt菌株与kurstaki、chinensis、berliner和huazhongensis亚种的Bt模式菌株聚在一起,形成一个单独的分支。序列两两比对结果显示DL1-5菌株与模式菌株Bacillus thuringiensis serovar kurstaki CP004069的相似性为99.81%,分别在第554、630和720处有3个碱基的差异。菌株S3-3和菌株DL2-6与该模式菌株相似性为100%,与DL1-5相似性为99.81%。菌株MX2和菌株DL3-4与该模式菌株相似性为99.94%,在第53个碱基处有1个碱基的差异,与DL1-5菌株的相似性为99.74%。

表2 三株代表菌株生化指标
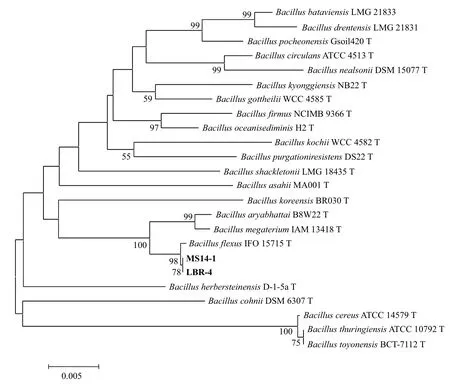
图7 邻接法构建菌株LBR-4基于16S rRNA基因序列的系统发育树
3 讨论
本研究在东北各地区采集分离的137株芽胞杆菌筛选出74株耐盐性高于4%的芽胞杆菌,说明大部分芽胞杆菌具有较好的适应高盐环境的能力。另外在国内,大量从不同地区分离得到的耐盐、嗜盐菌的报道中都有芽胞杆菌属的细菌分布。例如,新疆达坂盐湖[14]、云南省一平浪盐矿古老岩盐[15]和昆明盐矿古老岩盐[16]地区均以芽胞杆菌属为最优势属。因此研究统计芽胞杆菌耐盐性,可以为今后发掘高效耐盐的细菌提供有力的数据依据。

图8 邻接法构建菌株S5-1基于16S rRNA基因序列的系统发育树
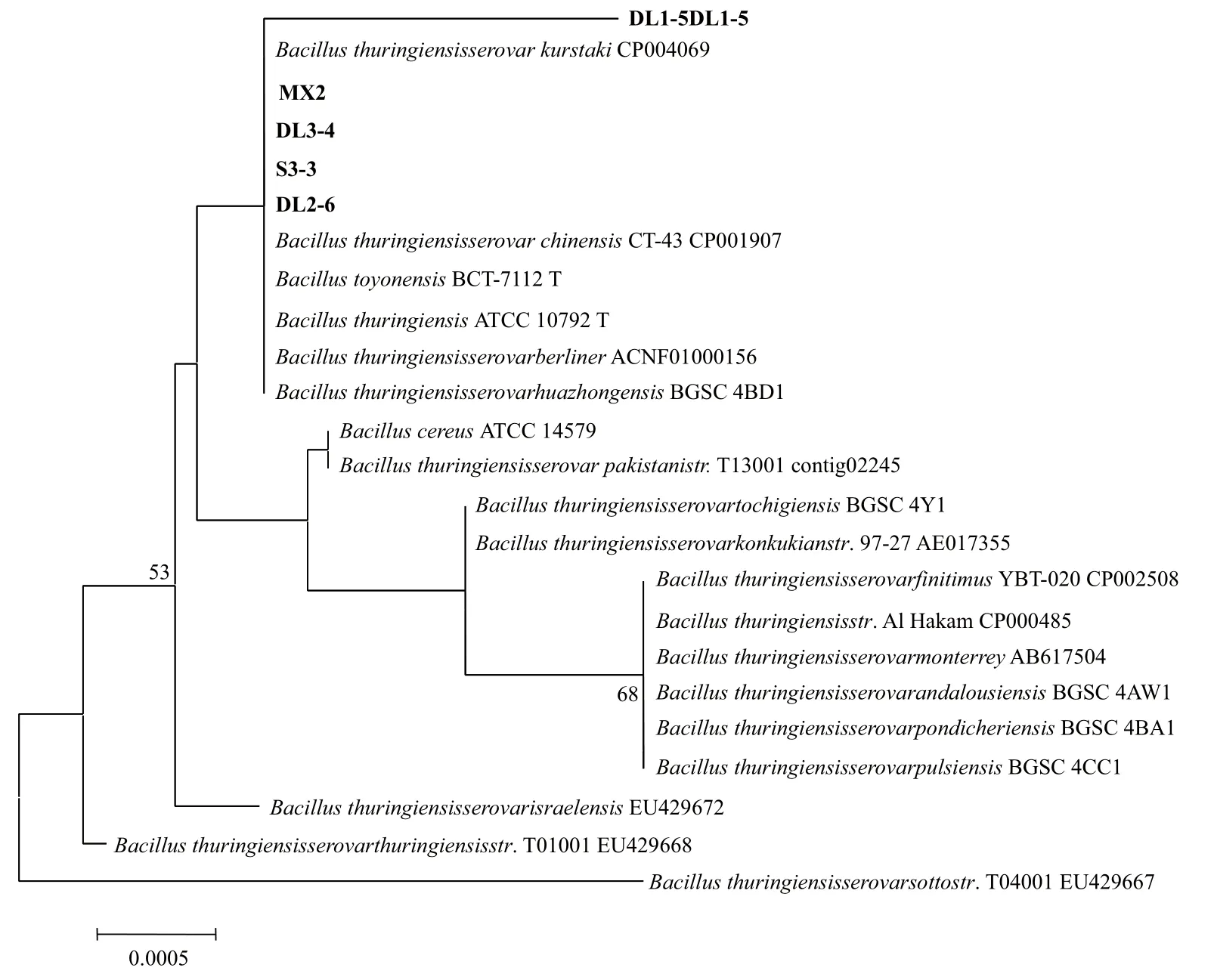
图9 邻接法构建菌株DL1-5基于16S rRNA基因序列的系统发育树
本研究基于16S rRNA基因序列构建了分离得到的36株不同耐盐芽胞杆菌和3株代表菌株的系统发育树。但是在分析过程中,苏云金芽胞杆菌和蜡样芽胞杆菌的16S rRNA基因序列具有高度的保守性,并不能单凭16S rRNA基因序列来区分这两个种的菌株,本研究应用了苏云金芽胞杆菌可以在芽胞期分泌伴孢晶体来区分,下一步工作可以采用de la Haba等[17]提出的多为点序列分析方法(MLSA)继续进
行深入的分类研究。
通过对3株代表菌株在正常情况下和最高耐受盐浓度下生长曲线的测定中,看出这3株芽胞杆菌在刚进入高盐环境中需要一段很长的适应时间,3株菌株调整期时间基本一致,在进入对数期到达稳定期的时间和正常环境下的菌株也基本相同。但是高盐胁迫下的对数生长期的生长曲线斜率明显小于正常情况下的曲线斜率,同时在达到稳定期后3株菌株的OD600nm值均比正常情况下的OD600nm值要小很多。推测有可能高盐胁迫影响了菌株正常繁殖分裂的速率。但是本研究并未涉及耐盐机理,为了寻找耐盐芽胞杆菌适应高盐环境的原因,我们正在继续研究在高盐胁迫下的耐盐芽胞杆菌的蛋白表达差异,以确定其是否通过蛋白调节来改变自身适应性。
通过比较3株代表菌株的16S rRNA基因序列发现耐盐性较好的这3株菌株在16S rRNA基因序列上均有不同程度的碱基替换,可能这3株菌在进化过程中有了一些改变,使之能够更好地适应逆境。同时Bt菌株MX2与DL3-4和DL2-6与S3-3在16S rRNA基因序列上完全一致,但是却表现出不同的耐盐性,甚至相差近1倍,说明同一亚种的芽胞杆菌之间也有着不同的适应逆境能力。
4 结论
本研究对东北各地区土壤样品中的芽胞杆菌进行了耐盐性的统计与分析,得到该地区芽胞杆菌总体耐盐性的数据。初步测定了分属于芽胞杆菌属不同种的3株耐盐性较好的代表菌株的形态学特征、生理生化特征、盐胁迫下生长情况及16S rRNA基因序列系统发育进化关系。
[1]王春裕. 中国东北盐渍土[M]. 北京:科学出版社, 2004, 333-337.
[2]姚荣江, 杨劲松, 刘广明. 东北地区盐碱土特征及其农业生物治理[J]. 土壤, 2006, 38(3):256-262.
[3]朱伟, 刘晓静. 中国盐碱地改良专利技术发展概述[N]. 中国知识产权报, 2013-01-16007.
[4]阿吉艾克拜尔, 邵孝侯, 常婷婷, 等. 我国盐碱地改良技术和方法综述[J]. 安徽农业科学, 2013, 16:7269-7271.
[5]马芸, 谢占玲, 李德英, 马志杰. 青藏高原药用植物内生细菌多样性研究[J]. 生物技术通报, 2010(6):234-239.
[6]Buchanan RE, Gibbens NE. 伯杰氏细菌鉴定手册[M]. 北京:科学出版社, 1984.
[7]El-Komy HMA, Abdel-Samad HM, Hetta AMA, Barakat NA. Possible roles of nitrogen fixation and mineral uptake induced by rhizobacterial inoculation on salt tolerance of maize[J]. Polish Journal of Microbiology, 2004, 53(1):53-60.
[8]尹汉文, 郭世荣, 刘伟, 陈海丽. 枯草芽孢杆菌对黄瓜耐盐性的影响[J]. 南京农业大学学报, 2006, 29(3):18-22.
[9]Horsburgh MJ, Moir A. σM, an ECF RNA polymerase sigma factor of Bacillus subtilis 168, is essential for growth and survival in high concentrations of salt[J]. Mol Microbiol, 1999, 32(1):41-50.
[10]Hahne H, Mäder U, Otto A, et al. A comprehensive proteomics and transcriptomics analysis of Bacillus subtilis salt stress adaptation[J]. Journal of Bacteriology, 2010, 192(3):870-882.
[11]Höper D, Bernhardt J, Hecker M. Salt stress adaptation of Bacillus subtilis:a physiological proteomics approach[J]. Proteomics, 2006, 6(5):1550-1562.
[12]刘国红, 林乃铨, 林营志, 刘波. 芽孢杆菌分类与应用研究进展[J]. 福建农业学报, 2008, 23(1):92-99.
[13]李玉红. 苏云金芽胞杆菌的抑菌活性及新型cry基因研究[D]. 哈尔滨:东北农业大学, 2012:41-42.
[14]吴海平, 王真辉, 等. 新疆达坂盐湖沉积土壤嗜盐细菌的定向富集与多样性分析[J]. 微生物学通报, 2010, 37(7):956-961.
[15]陈义光, 李汇明, 李沁元, 等. 一平浪盐矿古老岩盐沉积中可培养细菌的系统发育多样性研究[J]. 微生物学报, 2007, 47(4):571-577.
[16]肖炜, 杨亚玲, 刘宏伟, 等. 昆明盐矿古老岩盐沉积中可培养细菌多样性研究[J]. 微生物学报, 2006, 46(6):967-972.
[17]De la Haba R R, Sánchez-Porro C, Márquez M C, et al. Taxonomy of halophiles[M]. Extremophiles Handbook, 2011.
(责任编辑 李楠)
Isolation and Primary Identification of Salt-tolerant Bacillus in Northeast of China
Du Chuanying Li Haitao Liu Rongmei Xie Binjiao Zhang Jinbo Gao Jiguo
>(College of Life Science,Northeast Agricultural University,HarBin 150030)
To analyze the biodiversity of salt-tolerant Bacillus in the northeast of China, the temperature screening and gradient plate methods were used. 137 Bacillus strains were obtained, in which has 74 salt-tolerant Bacillus strains, 54% of total Bacillus strains. The optimum salt concentrations of these salt-tolerant Bacillus strains were all 1% and the range were 4%-14%. 16S rRNA sequences were amplified and analyzed for the determination of phylogenetic relationships. The 36 salt-tolerant Bacillus strains were different from each other in their 16S rRNA sequences or the salt tolerance, classified into 7 species of Bacillus genera. Among them, Bacillus thuringiensis was majority(14 strains, 38.9% of all, highest salt tolerance were 4%-9%NaCl), second were Bacillus cereus(7, 19.4%, 4%-8%)and Bacillus subtilis(7, 19.4%, 8%-11%), then Bacillus anthracis(4, 11.1%, 5%-7%), Bacillus flexus(2, 5.6%, 9%-14%), Bacillus sphaericus(1, 2.8%, 5%)and Bacillus aryabhattai(1, 2.8%, 6%). Bacillus flexus and Bacillus subtilis were shown better salt tolerance. Cultural and morphology characteristics, biochemical indexes and phylogenetic analysis of 3 typical strains were given. Provide an effective data support for the discovery of salt-tolerant Bacillus.
Salt-tolerant Bacillus Isolation Primary identification 16S rRNA
2014-02-23
国家高技术研究发展计划资助项目(2011aa10a203),转基因生物新品种培育(国家科技重大专项)(2014ZX0800913B-002),国家基础科学人才培养基金资助项目(J1210069),东北农业大学博士启动基金项目(2010RCB54)
杜传英,女,硕士,研究方向:生物化学与分子生物学;E-mail:duchuanying@126.com
高继国,教授,博士生导师,研究方向:生物化学与分子生物学;E-mail:gaojiguo1961@hotmail.com

