利用基因芯片技术鉴别山羊绒和绵羊绒
吕雪峰 阿布力孜·阿布力米提 帕娜尔 陶卫东 邢巍婷 采复拉 郑文新
(农业部种羊与羊毛羊绒质量监督检验测试中心,乌鲁木齐 830000)
利用基因芯片技术鉴别山羊绒和绵羊绒
吕雪峰 阿布力孜·阿布力米提 帕娜尔 陶卫东 邢巍婷 采复拉 郑文新
(农业部种羊与羊毛羊绒质量监督检验测试中心,乌鲁木齐 830000)
为了建立山羊绒与绵羊绒的基因芯片鉴别方法,达到快速、高通量鉴别山羊绒与绵羊绒的目的,文章根据山羊和绵羊遗传物质的特异性,选择线粒体基因组细胞色素b基因(cyt b)为目的基因,在cyt b基因通用引物区间设计出一对能鉴别绵羊绒与山羊绒的探针,经过与含有不同比例绵羊源性的PCR产物杂交,结果显示当山羊源性成分中绵羊源性含量为3% 时,仍然可见杂交信号,说明基因芯片鉴别毛绒的方法具有非常高的特异性,能够定性的鉴别山羊绒和绵羊绒。
山羊绒 绵羊绒 线粒体DNA Cyt b基因 基因芯片
山羊绒是一种珍稀而昂贵的动物纤维,其制品的品质极高,而且非常舒适,深受消费者青睐。为了追求利润,有些生产者将与山羊绒外观相似的绵羊毛或其它纤维混入织物中,严重损害了消费者的利益,因而准确鉴别山羊绒与绵羊毛及其它动物纤维显得尤为重要。以前常用的鉴别方法主要有显微镜法[1]、溶液法[2,3]和光谱分析法[4,5]等,但由于山羊绒与绵羊毛同属蛋白质纤维,化学性质差异不大,所以存在误判率较高、操作复杂、主观因素强等缺点[6]。特别是粗毛羊毛被中的底绒,又称绵羊绒[7],因其细度、光泽、鳞片都与山羊绒及其相似,故使用传统的方法很难区别。绵羊和山羊为不同种的动物,遗传物质的区别才是物种间根本差别,因此检测DNA的种间差异性不失为鉴定毛绒的理想途径[8]。
基因芯片具有高通量、自动化、检测速度快、特异性强的特点,目前已经广泛用于动物源性成分检测[9]、病原微生物、细菌检测[10-12]、基因多态性[13-15]、转基因食品检测[16]等领域。我国是毛绒生产大国,但毛绒掺杂检测技术相对比较滞后,检测手段单一,专业人员少,国内也没有成熟的毛绒鉴别技术来支撑毛绒市场以及进出口检验检疫,因此急需一项新型检测技术来弥补这方面的缺陷。基因芯片技术在高通量检测方面有绝对的优势。文章根据毛绒DNA的特点和基因芯片核酸分子杂交原理,在通用引物扩增区间设计出一对能鉴别山羊和绵羊源性的探针,旨在建立能同时检验山羊和绵羊源性的基因芯片方法,并确定检验的最低限量。
1 材料与方法
1.1 材料
从绵羊和山羊体上剪下的毛绒,剪毛时尽量贴近皮肤。
PCR扩增仪(德国Biometra公司)、凝胶成像系统(美国Bio-Rad公司)、基因芯片点样仪、芯片扫描仪、杂交仪、芯片清洗仪(北京博奥生物芯片公司),其余为实验室常用仪器。
Chelex-100(SIGMA公司)、蛋白酶K(Genviw公司)、TaqDNA 聚合酶、dNTPs、SSC、Denhardt’s液(上海生工生物公司)、Cy5-dCTP(Health Care公司)、醛基化基片及盖片(北京博奥生物公司)。
1.2 方法
1.2.1 引物和探针设计与合成 根据文献选用了一对可扩增绵羊和山羊线粒体基因cyt b的通用引物[3],在引物的扩增区间设计出数对探针,将这些探针序列用NCBI的 Blast进行比对分析,结合试验验证,最终选择特异性较好的序列作为鉴别绵羊绒与山羊绒的基因探针,阳性定位探针和阴性质控探针见石丰运报道[9]。引物和探针的合成、修饰由上海生工生物技术有限公司完成,具体序列见表 1。
1.2.2 毛干DNA提取 改进的chelex-100法:称取毛发样本10 mg左右,液氮研磨粉碎后置于2 mL离心管中。先后用无水乙醇,去离子水各浸泡30 min,每管中依次加入1720 μL 20% Chelex-100(SIGMA)、80 μL 1 mol/L DTT、200 μL 20 mg/mL 蛋 白 酶K,56℃温浴5-6 h,剧烈振荡30 s,100℃,8 min,使蛋白质变性。振荡30 s。12 000 r/min 离心15 min,取上清。
1.2.3 PCR产物制备 利用上述引物,分别对绵羊毛和山羊绒DNA进行扩增,PCR 反应体系为:10×PCR buffer 5 μL、Mg2+2.5 μL、dNTPs(2.5 mmol/L)4 μL、Cy5-dCTP 0.5 μL、正向引物和反向引物各1 μL(10 μmol/L)、DNA 模板(10 ng/μL)5 μL、Taq酶 1 μL(5 U/μL)、ddH2O 补足总体积到50 μL。PCR扩增条件为:94℃预变性 4 min;94℃变性45 s,55℃退火 45 s,72℃ 延伸 1 min,共35个循环;72℃延伸7 min。
1.2.4 芯片的制备 将合成的探针用 1×TE 溶解至终浓度为 40 μmol/L,与 50% DMSO 等体积混匀后作为探针点样混合液,加入384 孔板中,空白对照为 50% DMSO,按预先设计的探针点阵点至基片上。将芯片点有探针的一面在65℃水合10 s两次,自然晾干,紫外交联5 min。将芯片在42℃预热,0.5% SDS清洗10 min,然后用42℃预热的去离子水清洗2 min,离心甩干,4℃避光保存备用。
1.2.5 芯片杂交与清洗 将Cy5荧光标记的PCR产物和杂交液(25%甲酰胺,3×SSC,5×Dehartdt液,0.2% SDS)等量混合,总体积为80 μL;将其转移到PCR管中,在PCR仪上95℃热变性5 min,冰浴骤冷5 min;将杂交混合液点到芯片后盖上盖玻片,放入杂交仪中,42℃杂交2 h。杂交完成后分别预热洗液A(2×SSC,0.2% SDS)和洗液B(0.2% SDS)至42℃,洗液A洗两次,各2 min;洗液B洗一次,2 min。
2 结果
2.1 毛绒DNA提取
分别剪下山羊绒和绵羊毛毛根部分毛样,通过改进的chelex-100法提取毛绒mtDNA作为模板,进行 PCR 扩增,电泳检测结果见图1。
从图1可以看出,通用引物能有效扩增出山羊绒和绵羊毛cytb部分基因,大小在500 bp左右,与预期扩增产物大小相同。
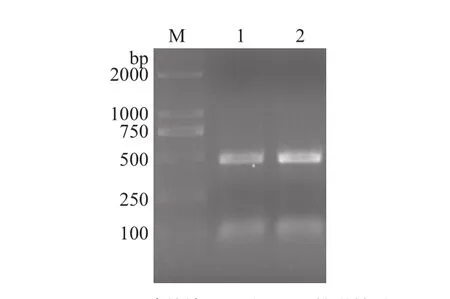
图 1 毛绒cytb基因扩增结果
2.2 探针杂交特异性试验
基因芯片杂交及扫描结果显示,两条探针与目的产物有较好的杂交信号,绵羊毛和山羊绒PCR产物均可与芯片内相应的探针进行杂交,而与其它的探针无特异性交叉反应,阳性质控探针及阴性质控探针杂交信号正常,表明这两条探针有很好的特异性,探针点样和杂交结果分别见图2和图3。

图2 探针点样矩阵图
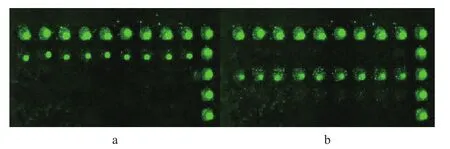
图 3 基因探针特异性杂交结果图
2.3 芯片最低检测限量试验
分别以在山羊源性PCR产物中掺入50%、10%、3%、1% 比例的绵羊源性产物,然后与绵羊源性探针杂交。根据芯片杂交扫描分析后的图像,得出能检测出绵羊源性最小掺入量,这也间接的反应出混合毛绒样品中的山羊绒和绵羊绒的检出限量。试验结果(图4,图5)表明,该芯片能够检出到3%的绵羊绒掺杂量。
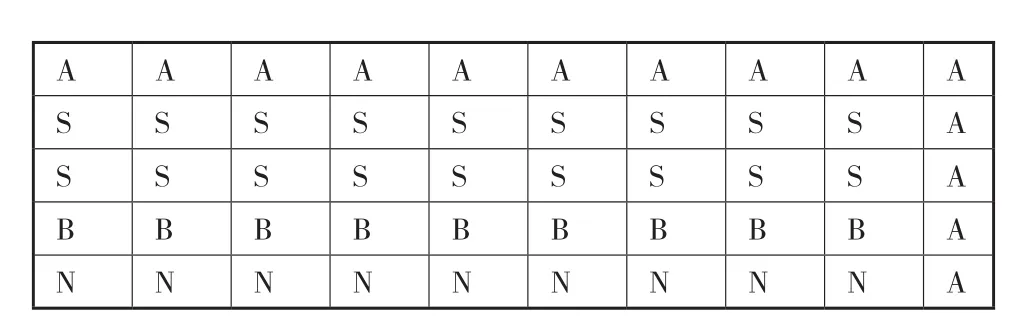
图4 基因芯片点样矩阵图

图5 不同比例绵羊源性成分与芯片杂交信号图
3 讨论
动物毛纤维中 DNA 含量极其微量,核 DNA 主要存在于毛囊部位,毛干中几乎没有核 DNA,仅含有微量 mtDNA,且取材部位随着与毛囊距离的增加而呈减少趋势[17],另外,毛干外层的角质蛋白对酸、碱和很多化学物质都具有较好的抗性,裂解非常困难,所以毛绒DNA提取一直非常困难。有关动物毛绒DNA提取方法有一些报道,如PCR缓冲液法、SDS-蛋白酶K法,试剂盒法等都容易从毛囊部位提取DNA[6],提取毛干DNA比较成功的方法有chelex-100法、碱裂解法和 QIAamp@DNA Investigator试剂盒法,从安全性和价格上考虑,chelex-100法仍是毛绒DNA提取的首选。无论何种方法,关键是消化毛干外层的角质层,液氮研磨毛干,chelex -100结合蛋白酶K有助于增加蛋白酶K与毛绒的接触面积,破坏毛干保护层,从而提高DNA的抽提效率。
线粒体DNA是高等动物唯一的核外遗传物质,所有组织细胞中均含有大量的线粒体,mtDNA 主要以编码序列构成,种内的异质基因很少[18],而在不同的物种间具有高度的变异性[19]。山羊、绵羊都属于牛科山羊亚科,说明它们之间存在着很近的亲缘关系[20]。 而Cyt b基因是线粒体自身编码的为数不多的蛋白质之一,其进化速度适中,进化模式清楚,加上能用一些通用引物扩增,是鉴别亲缘关系较近品种的理想工具之一[21-23]。
基因芯片检测的关键之一在于设计出特异性探针,探针的特异性和引物的通用性是成功建立基因芯片检测技术的关键[24-26]。由于山羊和绵羊mtDNA基因组的相似度达到了90%,在通用引物的扩增区间设计特异性探针,具有一定困难。本研究所用的通用引物扩增长度为500 bp,山羊和绵羊的相似度达到了96%,探针的设计除了使用软件外,还需手动增减碱基,然后用 NCBI的blast验证特异性,最后经试验杂交验证才能最终确定出特异性强的探针。芯片检测过程中的各项条件也是影响检测结果的因素,合适的反应条件可以降低杂交背景、提高杂交特异性,严格控制温度和试剂的离子强度是避免假阳性和假阴性结果的先决条件,在杂交过程中,温度太低或离子强度太高都会降低杂交的严格性,还会影响检测信号的特异性。相反,提高温度或降低离子强度会增加杂交的严格性。研究发现杂交温度应在42℃为宜,杂交时间2 h 即可,与杂交12 h 相比无显著差异。
目前,国内用于羊绒羊毛鉴别依然采用传统常规的国标方法[27],而分子鉴别手段有 SNP 分型检测和 DNA 分析。SNP 操作复杂、所需时间长、步骤分散、易出错、价格昂贵、专业要求高、难以实现高通量快速检测等。而 DNA 分析常常是通过PCR法来实现,对通过 PCR 产物进行凝胶电泳完成检测,必要时需进行酶切鉴定[28,29]或测序鉴定。基因芯片检测最大的优点在于高通量,对于从事毛绒检测的机构来说是非常必要的,因为毛绒检测机构和进出口检验机构常常面临成百上千的样品,利用基因芯片进行检测,可同时并行检测大批量的样品,大大地缩短了检测的时间,节约了人力,提高了检测的准确性。石丰运[9]等利用 mtDNA 16S rRNA 基因为目标基因,在通用引物扩增区间设计了16条特异性基因芯片检测探针对16种动物源性成分进行检测,体现出了基因芯片客观、准确、高通量的优点。
4 结论
通过对山羊绒和绵羊绒DNA提取和基因芯片鉴别方法的研究,初步建立了一种快捷、特异、灵敏且稳定地鉴别绵羊绒和山羊绒的基因芯片方法,可以应用于山羊绒与绵羊绒的定性判断。
[1] 杨桂芬, 付妍, 红霞, 等. 扫描电子显微镜与光学显微镜鉴别山羊绒纤维的技术探讨[J].中国纤检, 2006, 6:15-20.
[2] 倪广菊, 张毅. 细羊毛与羊绒纤维的鉴别[J]. 现代纺织技术, 2004, 12(4):40-42.
[3] 胡志宇. 多参数毛绒类纤维图像识别系统研究[M]. 北京:北京服装学院, 2005.
[4] 赵国, 徐静. 利用近红外光谱技术进行羊毛、羊绒鉴别[J].毛纺科技, 2006(1):42-45.
[5] 吴桂芳, 朱登胜, 何勇, 等. 可见-近红外光谱用于鉴别山羊绒与细支绵羊毛的研究[J]. 光谱学与光谱分析, 2008, 28(6):1260-1263.
[6] 金美菊, 阮勇, 石东亮, 等. 羊绒与羊毛纤维的鉴别检测综述[J]. 山东纺织科技, 2007, 48(4):28-30.
[7] 郑文新, 王乐, 赵晓丽. 关于“绵羊绒”纤维分类以及名称的考证研究[J]. 草食家畜, 2009, 2:4-9.
[8] 王晓梅, 杨秀荣.DNA分子标记研究进展[J]. 天津农学院学报, 2000, 7(1):21-24.
[9] 石丰运. 应用基因芯片技术鉴别检测动物源性成分[D].兰州:甘肃农业大学, 2010.
[10] 丁耀忠, 刘永生, 张杰.基因芯片技术在口蹄疫检测中的应用[J]. 浙江农业科学, 2009, 2:423-426.
[11] Wang LC, Pan CH, Severinghaus LL, et al. Simultaneous detection and differentiation of Newcastle disease and avian influenza viruses using oligo nucleotide microarrays[J]. Vet Microbiol, 2008, 127(34):217-226.
[12] 杨忠苹, 王秀荣, 石霖, 等.区分禽流感病毒亚型诊断芯片的构建[J]. 中国动物检疫, 2008, 25(10):29.
[13] Cowell JK, Hawthorn L. The application of microarray technology to the analysis of the cancer genome[J]. Curr Mol Med, 2007, 7(1):
103-120.
[14] Ito T, Tanaka E, Kadowaki T, et al. An ultrasensitive new DNA microarray chip provides gene expression profiles for preoperative esophageal cancer biopsies without RNA amplification[J]. Oncology, 2007, 73(5):366-375.
[15] 赖铭裕. 基因芯片及其在肿瘤研究中的应用[J]. 医学综述, 2009, 13:1960-1962.
[16] 于凤池. 基因芯片技术及其在植物研究中的应用[J]. 中国农学通报, 2009, 6:64-65.
[17] 华育平, 张琼, 徐艳, 等. 细胞色素b基因序列虎物种特异性鉴定的PCR方法研究[J]. 兽类学报, 2004, 24(2):103-108.
[18] Momcilovic D, Rasooly A. Detection and analysis of animal materials in food and feed[J]. Journal of Food Protection, 2000, 63:1602-1609.
[19] Tartaglia M, Saulle E, Pestalozza S, et al. Detection of bovine mitochondrial DNA in ruminant feeds:a molecular approach to test for the presence of bovine-derived materials[J]. Journal of Food Protection, 1998, 61:513-518.
[20] 李均祥. 岩羊染色体核型研究[J]. 甘肃畜牧兽医, 1999, 29, 4:13-15.
[21] Kouch TD, Thomas WKA, Meyer SV, et al. Dynamics of mitochondrial DNA evolution in animal:amplification and sequencing with conserved primers[J]. Proc Acad Sci USA, 1989, 86:6190-6200.
[22] Irwin DM, Kocher TD, Wilson AC, et al. Evolution of cytochrome b gene of mammals[J]. J Mol Evol, 1991, 32:128-144.
[23] Vladimir OA, Poltoraus LA, Zhivotovsky V. Mitochondrial DNA sequence diversity in Russians[J]. Federation of European Biochemical Societies Letter, 1999, 197-201.
[24] Dorris DR, Nguyen A, Gieser L, et al. Oligo deoxyribose nucleotide probe accessibility on a three-dimensional DNA microarray surface and the effect of hybridization time on the accuracy of expression ratios[J]. BMC Biotechnol, 2003, 3:6.
[25] Chou CC, Chen CH, Lee TT, et al. Optimization of probe length and the number of probes per gene for optimal microarray analysis of gene expression[J]. Nucleic Acids Res, 2004, 32(12):99.
[26] Hans B, Stephan P. Specific and nonspecific hybridization of oligonucleotide probes on microarrays[J]. Biophys J, 2005, 89:337-352.
[27] 曹渭芳, 杨桂芬, 邱桂芬, 等. GB/T 14593-2008 山羊绒、绵羊毛及其混合纤维定量分析方法扫描电镜法[S]. 北京:中国标准出版社, 2008:11.
[28] Subramanian S, Karthik T, Vijayaraaghavan NN. Single nucleotide polymorphism for animal fiber identification[J]. Journal of Biotechnology, 2005, 116(2):153-158.
[29] 金美菊, 阮勇, 石东亮, 等. 单核苷酸多态性技术在羊绒、羊毛纤维鉴别中的应用[J]. 毛纺科技, 2007, 11:10-14.
(责任编辑 李楠)
Identification of Cashmere and Sheep down by Microarray Technology
Lü Xuefeng Abliz Ablimit Pa Naer Tao Weidong Xing Weiting Cai Fula Zheng Wenxin
(Department of Agriculture-Breeding Sheep and Wool and Cashmere Quality Supervision Test Center,Urumqi 830000)
This study is to explore the method for identification of cashmere and sheep down by gene chip, thereby enabling rapid, highthroughput identification of cashmere and sheep down. According to the specificity of the genetic material of cashmere and sheep down, cyt b was selected as the target gene, a pair of probes that can identify cashmere and sheep down was designed in the universal primers interval of cyt b gene. Then PCR products of different proportions derived from sheep were hybridized with probe. Results showed that in goat derived PCR products, when content of PCR products derived from sheep was 3%, hybridization signal was still visible. It indicates that the gene chip method can be used to qualitatively identify the cashmere and sheep down, and has a very high specificity.
Cashmere Sheep down Mitochondria DNA Cyt b gene Gene chip
2013-10-07
新疆维吾尔自治区公益性科研院所基本科研业务经费
吕雪峰,男,中级,研究方向:动物遗传育种;E-mail:lxf00700@163.com
郑文新,男,研究员,研究方向:绒山羊育种与毛绒标准化质量控制技术;E-mail:zwx2020@126.com

