泌乳分子机制及基因调控网络
泌乳分子机制及基因调控网络
任洪辉1,2王永1江明锋1,2
(1.动物遗传育种学国家民委-教育部重点实验室,成都 610041;2.西南民族大学生命科学与技术学院,成都 610041)
为了提高泌乳家畜的产奶性能,使得进一步理解乳腺的功能及其调控机制变得十分必要。早期的研究倾向于从组织学和激素层面研究分析乳腺的生理功能,随着近年来分子生物学技术的发展,芯片等高通量技术被应用到乳腺泌乳机制的研究中,泌乳生物学进入了一个全新的发展时期。综述了近年来国内外关于乳腺泌乳机制方面的研究进展,如乳腺各个泌乳时期的基因调控网络、乳汁主要成分的合成调控网络、参与乳腺泌乳周期循环的信号传导通路等。
乳腺 泌乳 分子机制 调控网络 信号通路
提高乳用家畜的乳产量和质量是乳业相关领域科学家的主要目标。研究者已经从乳用家畜的生理学、营养学等角度进行了充分的研究。显然,泌乳生物学研究的重点应从乳腺自身着手,研究乳腺的泌乳机制、乳汁合成和分泌、影响乳腺生长发育的各种因素。虽然泌乳生物学研究历史很长,但由于大型动物的价格相对高昂且实际操作困难,很多研究只能从大鼠等试验动物着手。例如,与泌乳持续性[1]相关的乳腺细胞增殖-凋亡机理、通过控制营养条件调节乳汁的生物合成[2]和青春期乳腺的生长速率[3]等研究结果均通过对大鼠和小鼠等模式动物的研究得到。近年来,运用转录组学技术对荷斯坦奶牛等大家畜泌乳的分子机制进行研究成为热点,美国伊利诺伊大学的 Loor[4]和Bionaz[5]等在乳腺泌乳周期基因调控网络、乳汁各成分合成机制及相关参与泌乳调控分子机制等方面的研究处于国际领先地位。近几年我国在该方向的研究也取得了实质性的进展,东北农业大学的李庆章[6]、侯晓明[7]等率先开展了奶牛乳腺相关基因表达谱的研究,为我国在该领域的研究起到了带头作用。
目前,信号传导网络是乳腺发育和泌乳分子机制中研究最多的领域,这些结果大多通过对培养的乳腺细胞和转基因大鼠乳腺组织的研究获得。随着现代分子生物学技术的发展,泌乳生物学进入了一
个全新的发展时期。生物信息学、转录组学、蛋白质组学等技术逐渐应用到泌乳生物学的研究领域,科学家试图从组学层面阐释泌乳分子机制。本文综述了国内外泌乳生物学研究进展,试图从基因调控网络和信号传导途径等角度对乳腺泌乳的分子机制进行阐释,为相关领域的研究提供参考。
1 转录组学在泌乳生物学中的研究概述
随着芯片等高通量分析技术的发展,转录组分析技术和相关生物信息学工具可以很好的应用于复杂的基因网络[8]。一些生物软件广泛的用于生命科学(如GeneGo,Ingenuity Pathway Analysis),研究者可以手工操作软件中的一些功能(如分子间的相互作用、基因与表型之间的相互关系等),使得对乳腺整个泌乳周期分子调控网络的研究成为了可能。目前,对哺乳动物乳腺泌乳分子机制的研究已经从模式动物过渡到大型哺乳动物。
1.1 大鼠乳腺转录组学研究
Rudolph 等[9]对大鼠整个泌乳期进行芯片分析,大约4 832个差异表达基因(Differentially expressed gene,DEG)参与泌乳循环,主要是参与乳汁主要成分合成及调控、泌乳周期调节、乳腺生理变化、信号传导等有关泌乳的基因,各个基因参与不同的调节机制。根据基因变化的差异性,将芯片数据中变化较明显的基因分成3个部分以解析相关基因的调控机制[10](图1)。第一部分描述了分析结果中大约50%的基因,这些基因在妊娠后期开始持续表达直至泌乳期保持稳定,最后在退化初期开始快速下调。第二个部分和第三个部分代表一些次重要的基因,分别包含13.6%和11.6%。第二部分的基因在妊娠期和泌乳期未发生明显变化,但在退化期开始表达。在第三部分中,基因在妊娠后期开始表达,到泌乳期时开始下降,直至退化期这些基因又开始持续性高表达。大鼠妊娠期乳腺组织的表皮细胞发生了很大的生理和生化变化,乳房的体积逐渐变大,尤其是在妊娠期第12天体积增长最迅速。结合已知的生理学知识进行分析表明,表皮细胞在妊娠期的增殖可能是第三部分妊娠期基因差异性表达的主要驱动力。通过对第一部分的分析发现,只有一小部分基因(n=592)在整个泌乳周期中发生较大的差异性变化,这些基因可能参与整个泌乳周期中乳汁的合成,同时这也说明在妊娠后期或分娩前,乳腺组织在分子水平上已做好了产奶准备。断奶后,乳头失去了必要的刺激,乳房的生理和生化功能在断奶后第2天开始退化,乳腺组织恢复到妊娠期的状态。第二部分的DEG在L9(Lactation L)和I2(Involution I)时段开始表达,但在妊娠期和泌乳期未发生变化,因此这部分基因的表达可能与乳腺退化有关。
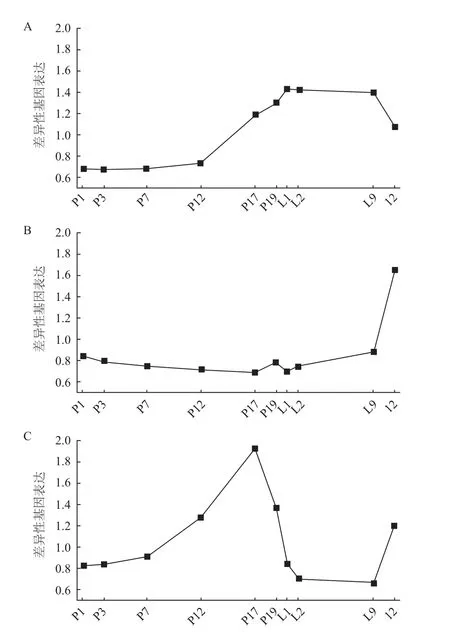
图1 DEG在不同泌乳时期的变化趋势
1.2 奶牛乳腺转录组学研究
Bionaz等[5]利用芯片技术对荷斯坦奶牛乳腺泌乳基因调控网络进行了研究,提出了DIA(Dynamic impact approach)分析技术,并成功应用至荷斯坦奶牛泌乳基因调控分析[11]。将-30 d、-15 d、+1 d、15 d、30 d、60 d、120 d、240 d和300 d的乳腺样品进行表达谱分析发现,6 382个差异性表达基因(DEG)
参与泌乳循环,并将数据分析以-30 d作为参照标准,比较各个时期差异性表达基因的变化(图2)。当提高差异性阈值(〉200%)时,显现出的数据可以进一步显示出在调控过程中发挥主要作用的差异性表达基因。结果表明,在60 d和120 d时差异性表达基因的数量最多,大约包含3 500个。分析整个泌乳循环周期,下调基因的数量明显多于上调基因,与泌乳相关的差异性表达基因在-15-1 d和120-240 d期间的变化最大,且在+1 d、60-120 d时DEG的数量达到巅峰值。但提高差异性表达基因的阈值时,上调基因占据一定优势,且60 d和120 d时DEG的数量最多,推测此时的变化可能与泌乳量的增加有关。比较妊娠期和退化期,-15-1 d期间下调基因的数量多于上调基因,120-240 d期间上调基因占据数量上的优势。但其中变化较大的差异性表达基因的情况却与之相反,即妊娠期变化较大的多为上调基因,退化期大多是下调基因。因此,推断后者的变化可能与泌乳减少有关。
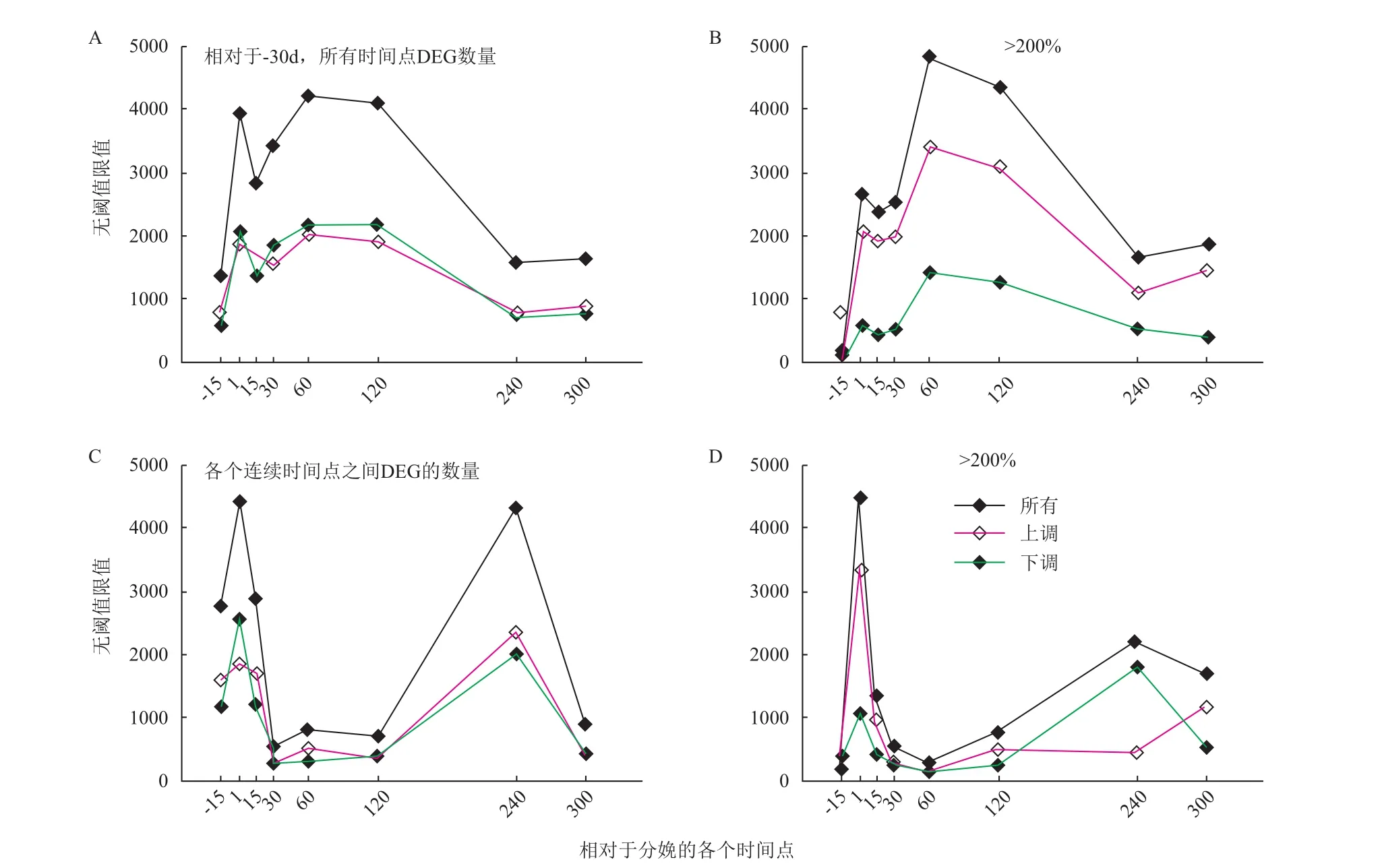
图2 以妊娠期-30 d和各自前一时间点为对照的差异性表达基因的数量
2 乳腺发育各时期的转录组学研究
2.1 妊娠期
妊娠期乳腺组织发生了很多变化,为分娩后的泌乳做好了组织学上的准备,其中表皮细胞迅速增殖是最明显的变化,乳房体积逐渐从总体积的10%增长至分娩前的90%。从转录组学的角度分析,妊娠期要在泌乳期之前做好与产奶相关的分子水平准备,大部分有关乳汁合成和分泌的基因在泌乳前已开始转录。因此,一些研究者猜测妊娠期后期的基因转录是妊娠期向泌乳期过渡的调节开关,是分娩后产奶的必要条件。根据基因芯片数据分析的结果[10],乳腺组织在该阶段开始大量转录与蛋白质合成、转运、定位和分泌相关的基因,同时一些通过乳腺表皮细胞进入乳汁的离子的转运系统也发生上
调,如铁离子和锌离子;下调的基因主要有:⑴脂肪酸氧化酶基因在妊娠后期发生下调;⑵血管生长有关基因抑制(但实际上乳腺在这一阶段生长发育,即血管变大。因此一些GO分析的结果可能无法应用于乳腺发育);⑶阴离子转运下调,尤其是妊娠后期磷酸盐的下调最严重。
2.2 泌乳期
泌乳期是乳腺发挥功能即分泌乳汁的主要时期,乳腺在此阶段十分活跃,乳房会分泌大量的乳汁。但根据芯片分析结果,大部分基因在这个过程发生下调,只有少数进行上调。合成和组装脂肪酸是乳腺在泌乳期的主要功能之一,因此同妊娠期一样,参与脂肪酸氧化的酶在该阶段被抑制。但意外的是,参与蛋白质转运和代谢(合成和分解)的相关基因在泌乳期逐渐衰减甚至被抑制,这也间接证明乳腺组织在妊娠期已经为泌乳期产奶做好了转录组水平的准备。例如,在泌乳期,乳汁中含有一些有关免疫的物质,但分析发现与免疫相关的基因在该时期也开始下调且进入抑制状态,这说明免疫相关基因已经在妊娠后期完成转录。总的来讲,泌乳期大部分基因处于下调状态,与主要功能相关的基因已在妊娠期完成转录。
2.3 退化期
当乳房进入退化期时,妊娠期过程中一些活跃的基因重新启动,包括蛋白质转运等功能在内的基因占据主导优势(大约占85%),只有少部分参与生物合成和新陈代谢的功能基因下调。退化期乳腺的主要功能是分解代谢,大部分有关蛋白质分解代谢的基因逐渐上调,此过程可以满足氧化应激反应和保护乳腺细胞或其周围细胞免受外界伤害。随着蛋白质分解代谢的增强,ATP合成和水解也逐渐加快。为满足乳腺组织对ATP的需求,有关质子转运的相关基因在该时期进行上调。此外,为了维持组织内的离子环境,阳离子的转运功能在泌乳期过后恢复。以上这些基因的上调将有助于促进乳成分退化及间隙小分子的转换。同时,退化期泌乳相关基因逐渐恢复至妊娠期,将为下一次泌乳循环做准备。
3 乳汁主要成分合成的调节网络
3.1 乳蛋白
有关乳蛋白合成的研究已取得很大进展。Massiom等[12]对参与胰岛素信号传导、雷帕霉素靶蛋白(Mammalian target of rapamycin,mTOR)、腺苷酸活化蛋白激酶(AMPK)、Jak2-Stat5信号通路及氨基酸和葡萄糖转运受体的44个基因进行qPCR分析。结果表明,氨基酸和葡萄糖转运受体、胰岛素信号传导通路的相关成分及mTOR信号通路相关基因在泌乳期间发生明显上调,而核糖体组件RPL22出现下调,其中胰岛素和mTOR在调控乳蛋白合成过程中发挥关键作用。胰岛素在过去被认为是乳蛋白合成的间接调控者[13],但Herbein 等[14]研究发现,牛血液中胰岛素在分娩后明显减少,且在泌乳前两个月一直保持这种低水平。直到最近的研究表明,胰岛素是牛和鼠乳蛋白合成的关键调控者[15,16]。对于所有哺乳动物,mTOR信号通路调控蛋白质合成,尤其在转录水平[17]。mTOR通路受支链氨基酸调控,特别是亮氨酸(Leu)[18]。乳腺组织中氨基酸不仅可以调控蛋白质编码基因的转录,其转运效率也是限制乳汁蛋白质合成的重要影响因素[19]。除了氨基酸之外,葡萄糖对调控乳汁其它成分的合成也很重要[20]。Jak2-Stat5信号通路是调控啮齿类动物乳蛋白合成的主要方式[21],但与啮齿类动物相比,该通路对奶牛乳蛋白合成过程中的影响力明显较弱[22]。AMPK主要参与乳脂的合成[23],目前尚无研究表明其在蛋白质合成过程中的重要性。对参与乳蛋白合成调控的基因进行分析发现,蛋白质的合成需要氨基酸、胰岛素、葡萄糖和mTOR通路等相互作用,形成一个整体的调控网络。
3.2 乳脂
目前,有关乳脂合成分子机制的研究并不很完善,大部分的研究从mRNA水平进行分析。乳腺组织合成乳脂的分子生物学过程及主要相关基因如下:血液中获取脂肪酸(LPL,CD36)、细胞内脂肪酸交换(FABP3)、长链(ACSL1)和短链(ACSS2)脂肪酸胞内激活、脂肪酸从头合成(ACACA,FASN)、去饱和(SCD,FADS1)、三酰甘油合成(AGPAT6,
GPAM,LPIN1)、脂滴形成(BTNIA1,XDH)、酮体利用(BDH1)以及转录调控(INSIG1,PPARG,PPARGCIA),这些基因交汇在一起,组成一个庞大的乳脂合成网络,过去的研究将SREBP1作为大鼠乳脂合成的核心调控者[24]。但Massimo等[25]利用qPCR技术分析与奶牛乳脂合成相关基因表达变化情况,分析相关基因在泌乳期间的转录调节方式,并根据分析的结果建立了一个有关乳脂合成的调控网络,描绘了乳脂合成的调控机制,认为SREBF1、SREBF2和PPARG是奶牛乳脂合成过程的主要调节基因,其中PPARG在这个网络中扮演多重角色,例如协调PPARGC1A和INSIG1之间的相互作用及控制SREBF1的表达。
3.3 乳糖
乳糖是乳汁的主要组成部分,其在乳汁中可通过调节渗透压控制乳的体积。目前有关乳腺组织乳糖合成调控网络的研究并不是很多,研究的重点大多倾向于葡萄糖对乳糖合成的影响。作为乳糖合成所需的前体物,葡萄糖的吸收是控制乳糖合成的最直接的方式[26]。葡萄糖主要通过两种截然不同的方式穿过磷脂双分子层进入乳腺细胞:葡萄糖转运蛋白(GLUT)和Na+/葡萄糖泵。目前,研究者大都从葡萄糖吸收水平分析乳糖合成调控网络,有关乳糖合成的其它调控网络仍需要进一步探索。
4 乳腺泌乳期信号传导网络
很多信号通路参与泌乳期分子调控,其中Hedgehog 信号通路、Jak-STAT信号通路和TGF-beta信号通路对整个泌乳周期影响最大,发挥促进作用,即正调控乳腺泌乳;但细胞黏附分子(CAMs)、mTOR信号通路和Notch信号通路在泌乳期时处于抑制状态。
Hedgehog和TGF-beta信号通路参与乳腺干细胞自我更新,当它们解除控制时会发生乳腺癌[27]。Hedgehog信号通路主要参与啮齿类动物泌乳,产奶时被激活,诱导产奶相关基因的表达[28]。有关TGF-beta通路在乳腺组织中调控机理的研究并不完善,研究者发现该通路在抑制乳腺细胞增殖的同时,也可促进细胞凋亡[29];除此之外,科学家猜测TGF-beta通路在乳腺组织中的作用与在人类肠细胞中的生物学效应相似[30];因此,TGF-beta通路可能在整个细胞周期循环过程中发挥关键的调控作用[31]。另外一个正调控乳腺泌乳的通路即Jak-STAT通路,可以直接调控非反刍类产奶动物乳蛋白基因的表达,但对于反刍类动物尤其是奶牛,该通路并不能直接通过Jak或Stat基因的变化进行调节,而是由其下游的效应器直接调控[12]。
mTOR(Mammalian target of rapamycin 雷帕霉素靶蛋白)信号通路参与调控乳蛋白合成,主要被氨基酸、胰岛素和生长因子调控激活,当能量或营养不足时易被抑制。mTOR信号通路对细胞生理学的调控起关键作用[32],其大量组件参与调控蛋白质的合成,如启动子和延伸因子。mTOR信号通路虽然在乳蛋白合成过程中发挥重要作用,但只有很少一部分基因在转录水平起调控作用,该通路大部分基因处于抑制状态。有关Notch信号通路的抑制机理是一个新奇的发现,其主要参与细胞发育、机体器官的细胞进入组织、细胞增殖和凋亡[33]。Callahan等[34]通过人工激活Notch信号发现,该通路也可以抑制乳腺分支和泌乳,同时参与乳腺细胞生长。CAMs主要参与维持乳腺表皮细胞的结构[2],其在泌乳期的抑制现象无法利用KEGG分析。但当进一步放大有关数据时,该“抑制”会被逐渐展现,结果发现其主要由一些与免疫系统相关的细胞黏附分子引起,同时有一些紧密连接分子作出感应。
5 小结
有关乳腺泌乳分子生物学方面的研究逐渐增多,有很大的研究空间和发展潜力,也取得了很多科研成果。与此同时,随着人们生活水平的提高和对奶制品认识的加强,奶产品将逐渐作为“第二粮食”进入我们的餐桌;人们对乳制品需求量增加的同时,也越来越关注它的质量,这种市场需求为乳腺泌乳生物学相关科学研究奠定了很好的商业基础。我国在该领域尚属起步阶段,过去几十年的研究倾向于从组织学和激素层面分析乳腺的生理功能,很少从分子层面进行解析。因此,国内的研究者应逐渐将现代分子生物学、转录组学、蛋白质组学等技术应用到产奶家畜的研究中,建立起我国产奶家畜泌乳相关基因的调控网络,阐明家畜泌乳调控机理,从
分子层面解读家畜泌乳的分子机制,为国内产奶家畜的遗传育种研究奠定分子基础。
[1] Capuco AV, Ellis SE, Hale SA, et al. Lactation persistency:insights from mammary cell proliferation studies[J]. J Anim Sci, 2003, 81(Suppl. 3):18-31.
[2] Alford D, Taylor-Papadimitriou J. Cell adhesion molecules in the normal and cancerous mammary gland[J]. Journal of Mammary Gland Biology and Neoplasia, 1996, 1(2):207-218.
[3] Meyer MJ, Capuco AV, Ross DA, et al. Developmental and nutritional regulation of the prepubertal bovine mammary gland:II. Epithelial cell proliferation, parenchymal accretion rate, and all ometric growth[J]. J Dairy Sci, 2006, 89(11):4298-4304.
[4] Loor JJ, Cohick WS. ASAS centennial paper:Lactation biology for the twenty-first century[J]. J Anim Sci, 2009, 87(2):813-824.
[5] Bionaz M, Periasamy K, Rodriguez-Zas SL, et al. Old and new stories:revelations from functional analysis of the bovine mammary transcriptome during the lactation cycle[J]. PLOS ONE, 2012, 7(3):e33268.
[6] 李庆章等.乳腺发育与泌乳生物学[M].北京:科学出版社, 2009.
[7] 侯晓明, 李庆章, 黄田英. 泌乳期奶牛乳腺基因表达谱研究[J].中国科学:生命科学, 2010(3):231-238.
[8] Naylor S, Culbertson AW, Valentine SJ. Towards a systems level analysis of health and nutrition[J]. Current Opinion in Biotechnology, 2008, 19(2):100-109.
[9] Rudolph MC, McManaman JL, Hunter L, et al. Functional development of the mammary gland:use of expression profiling and trajectory clustering to reveal changes in gene expression during pregnancy, lactation, and involution[J]. J Mammary Gland Biol Neoplasia, 2003, 8(3):287-307.
[10] Lemay DG, Neville MC, Rudolph MC, et al. Gene regulatory networks in lactation:identification of global principles using bioinformatics[J]. BMC Syst Biol, 2007, 1:56.
[11] Bionaz M, Periasamy K, Rodriguez-Zas SL, et al. A novel dynamic impact approach(DIA)for functional analysis of timecourse omics studies:validation using the bovine mammary transcriptome[J]. PloS One, 2012, 7(3):e32455.
[12] Bionaz M, Loor JJ. Gene networks driving bovine mammary protein synthesis during the lactation cycle[J]. Bioinform Biol Insights, 2011, 5:83-98.
[13] Neville MC, McFadden TB, Forsyth I. Hormonal regulation of mammary differentiation and milk secretion[J]. J Mammary Gland Biol Neoplasia, 2002, 7(1):49-66.
[14] Herbein JH, Aiello RJ, Eckler LI, et al. Glucagon, insulin, growth hormone, and glucose concentrations in blood plasma of lactating dairy cows[J]. J Dairy Sci, 1985, 68(2):320-325.
[15] Menzies KK, Lefèvre C, Macmillan KL, et al. Insulin regulates milk protein synthesis at multiple levels in the bovine mammary gland[J]. Functional & Integrative Genomics, 2009, 9(2):197-217.
[16] Menzies KK, Lee HJ, Lefèvre C, et al. Insulin, a key regulator of hormone responsive milk protein synthesis during lactogenesis in murine mammary explants[J]. Functional & Integrative Genomics, 2010, 10(1):87-95.
[17] Wang X, Proud CG. The mTOR pathway in the control of protein synthesis[J]. Physiology, 2006, 21(5):362-369.
[18] Moshel Y, Rhoads RE, Barash I. Role of amino acids in translational mechanisms governing milk protein synthesis in murine and ruminant mammary epithelial cells[J]. Journal of Cellular Biochemistry, 2006, 98(3):685-700.
[19] Kimball SR, Jefferson LS. New functions for amino acids:effects on gene transcription and translation[J]. The American Journal of Clinical Nutrition, 2006, 83(2):500S-507S.
[20] Reynolds CK, Harmon DL, Cecava MJ. Absorption and delivery of nutrients for milk protein synthesis by portal-drained viscera[J]. Journal of Dairy Science, 1994, 77(9):2787-2808.
[21] Wakao H, Gouilleux F, Groner B. Mammary gland factor(MGF)is a novel member of the cytokine regulated transcription factor gene family and confers the prolactin response[J]. The EMBO Journal, 1994, 13(9):2182.
[22] Wheeler TT, Broadhurst MK, Sadowski HB, et al. Stat5 phosphorylation status and DNA-binding activity in the bovine and murine mammary glands[J]. Molecular and Cellular Endocrinology, 2001, 176(1):39-48.
[23] McFadden JW, Corl BA. Activation of AMP-activated protein kinase(AMPK)inhibits fatty acid synthesis in bovine mammary epithelial cells[J]. Biochemical and Biophysical Research Communications, 2009, 390(3):388-393.
[24] Anderson SM, Rudolph MC, McManaman JL, et al. Secretory activation in the mammary gland:it’s not just about milk protein synthesis[J]. Breast Cancer Res, 2007, 9:204-217.
[25] Bionaz M, Loor JJ. Gene networks driving bovine milk fat synthesis during the lactation cycle[J]. BMC Genomics, 2008, 9(1):366.
[26] Zhao FQ, Keating AF. Expression and regulation of glucose transporters in the bovine mammary gland[J]. Journal of Dairy Science, 2007, 90:E76-E86.
[27] Woodward WA, Chen MS, Behbod F, et al. On mammary stem cells[J]. Journal of Cell Science, 2005, 118(16):3585-3594.
[28] Lewis MT, Veltmaat JM. Next stop, the twilight zone:hedgehog network regulation of mammary gland development[J]. Journal of Mammary Gland Biology and Neoplasia, 2004, 9(2):165-181.
[29] Zarzynska J, Motyl T. Apoptosis and autophagy in involuting bovine mammary gland[J]. J Physiol Pharmacol, 2008, 59(Suppl 9):275-288.
[30] Purup S, Vestergaard M, O Pedersen L, Sejrsen K. Biological activity of bovine milk on proliferation of human intestinal cells[J]. J Dairy Res, 2007, 74(1):58-65.
[31] Liu F. Receptor-regulated Smads in TGF-beta signaling[J]. Frontiers in Bioscience:a Journal and Virtual Library, 2003, 8:s1280-303.
[32] Wang X, Proud CG. The mTOR pathway in the control of protein synthesis[J]. Physiology(Bethesda), 2006, 21:362-369.
[33] Fortini ME. Notch signaling:the core pathway and its posttranslational regulation[J]. Dev Cell, 2009, 16(5):633-647.
[34] Callahan R, Egan SE. Notch signaling in mammary development and oncogenesis[J]. J Mammary Gland Biol Neoplasia, 2004, 9(2):145-163.
(责任编辑 狄艳红)
Molecular Mechanism of Lactation and Gene Regulation Networks
Ren Honghui Wang Yong Jiang Mingfeng
(1. Key Laboratory of Animal Genetics & Breeding, State Ethnic Affairs Commission and Ministry of Education, Chengdu 610041;2. Life Science and Technology of Southwest University for Nationalities,Chengdu 610041)
In order to improve the production performance of animals, that understand better the function and regulation mechanisms of the mammary gland become very necessory. Early studies tended to analyze the physiological function of mammary gland from histology and hormone levels. With the development of molecular biology techniques, high-throughput technologies such as gene chip to study mammary gland and lactation biology has entered a new stage of development. In this review, we presented several aspects in molecular biology of lactation biology, such as gene networks about each stage were revealed, synthetic regulation network about the main composition of milk, signaling pathways involved in mammary gland lactation cycle.
Mammary gland Lactation Molecular mechanism Regulation networks Signal pathway
2013-07-30
国家自然科学基金项目(31172198),国家科技支撑计划课题(2012BAD13B06),四川省青年科技基金(09ZQ026-011),教育部科学技术研究重点项目(201265),中央高校科研项目(12ZYXS74)
任洪辉,男,硕士研究生,研究方向:动物遗传育种与繁殖;E-mail:337513362@qq.com
江明锋,男,博士,教授,研究方向:高原家畜生理基因组、高原生物基因资源的开发与利用;E-mail:mingfengjiang@vip.sina.com

