Fe3+离子修饰的金纳米粒子对DNA的高效吸附
付 娆
(东北电力大学化学工程学院,吉林吉林132012)
金纳米粒子可以与多种类型生物分子发生相互作用[1-4],DNA吸附在粒子表面后,在生物医学和生物分析中得到了广泛的应用[5-10]。以往工作都是利用静电作用实现DNA的吸附[11-19],但是在一些复杂体系中DNA在粒子表面的吸附机制并不十分明确,而且在吸附过程中往往会由于非特异性吸附而引入一些其他小分子杂质,而对后续的研究工作产生了不利影响。要解决这些问题,就要明确DNA与粒子之间的作用。研究发现,将金属离子加入到DNA溶液中后,金属离子能够通过与DNA分子中的磷酸根络合而引起DNA分子链的卷曲[20],而这种配位作用往往要大于静电作用。这为我们改善粒子对DNA的吸附提供了新的思路。我们可以利用这种配位作用将DNA最大程度地吸附到粒子表面,而且这种配位作用对盐浓度不敏感并具有很好的选择性,可以避免对其他杂质的非特异性吸附。另外,利用DNA的惰性部位磷酸根进行反应可以充分保留DNA的活性部位,使DNA可以更好地进行后续反应。因此,在本章中,我们研究了表面修饰了Fe3+离子的金纳米粒子对DNA的吸附和脱附行为。利用Fe3+离子与DNA分子中磷酸根的配位作用可以实现在低pH条件下DNA分子在金纳米粒子表面的高效吸附。这种基于配位作用的吸附效率要高于盐桥作用,而且不会受到溶液中盐浓度的影响。
1 实验部分
1.1 试剂与药品
鲑鱼精 DNA(BR),谷胱甘肽(>98%,C10H17N3O6S,FW 307.33),氯金酸(99.9+%,HAuCl4·3H2O,FW 393.8),柠檬酸钠(99.9+%,HAuCl4·3H2O,FW 393.8),硫酸镍(≥99%,NiSO4·7H2O,FW 280.86),三氯化铁(>99%,FeCl3·6H2O,FW 270.21),氯化钠(AR,NaCl,FW 58.44)购买于 Sigma-Aldrich 公司。金溶胶的pH值通过NaOH(0.1 mol/L)和HCl(0.1 mol/L)水溶液来调节。实验中所有溶液均用电阻率为18 MΩ·cm的高纯水配制。
1.2 测试仪器
紫外吸收测试采用型号为Cary-100紫外吸收光谱仪。动力学光散射测试采用Brookhaven仪器公司出品的型号为BI-90Plus的粒度分析仪,散射角度为90°。透射电子显微镜(TEM)为Hitachi H-8100Ⅳ型电子显微镜;红外光谱利用Perkin-ElmerSpectrum One FTIR光谱仪测试,将金纳米粒子样品滴加在CaF2薄片中间,置于持续通气的样品室中。离心机为德国Sigma3K-30型离心机。pH计为北京Sartorius公司PB-20型pH计。实验所有图片采用DiMAGE Z2数码相机采集。所有实验在室温下进行,温度为25±2℃。
1.3 金纳米粒子的表面修饰
用柠檬酸还原氯金酸的方法合成金纳米粒子[21]。250 mL三颈瓶中加入95 mL,0.48 mmol/L的氯金酸,在400 r/min搅拌下加热至沸腾。沸腾30S后,快速加入5 mL,1.94 mmol/L柠檬酸钠溶液,保持沸腾状态反应1 5min后关闭热源,继续搅拌至室温。如此制得金溶胶浓度为290 μmol/L,pH值为6.5左右。透射电镜测试金纳米晶的粒径为17.9±1 nm。动态光散射数据显示金纳米晶的多分散性为4.8%。紫外吸收光谱给出最大吸收峰位在518 nm左右。将金纳米粒子以8 000 r/min分,离心30 min后重新分散于高纯水中,以达到提纯的目的。先用0.1 mol/L的NaOH将离心提纯后的金粒子的pH值调到9.0,然后将60 μmol/L的谷胱甘肽溶液加入到金溶胶中。反应2.5 h后,通过离心提纯的方法去除溶胶当中过剩的谷胱甘肽,最终得到浓度为3.17×10-9mol/L的谷胱甘肽修饰的金纳米粒子溶胶,其pH值为6.4左右。用0.1 mol/L的NaOH分别将金溶胶调至不同的pH值(3.0-9.0),然后加入50 μmol/L的Fe3+离子。测试前,样品放置5 min,每种样品测试至少重复5次。
1.4 金粒子螯合Fe3+离子后对DNA的吸附和脱附过程
将100 μL,1000 μg/mL的鲑鱼精DNA水溶液加入表面修饰Fe3+离子的金纳米粒子水溶液中,DNA和金纳米粒子的混合溶液在pH3.0-9.0下室温摇床振荡1 h。离心后取上清液,测试上清中剩余的DNA浓度[22,23]。金纳米粒子对DNA的吸附效率由公式ηad=(OD0-ODL)/OD0计算得到,其中吸光度OD0代表混合溶液中初始DNA的浓度,吸光度ODL代表离心后上清液中DNA的浓度。测量DNA的脱附效率时,把在pH3.0的混合溶液中吸附DNA达到饱和的金纳米粒子离心分离后分散在不同pH值的水溶液中,摇床振荡1 h后离心,测试上清液中的DNA浓度。金纳米粒子对DNA的脱附效率由公式ηde=ODR/OD0-ODL计算得到,其中吸光度ODR代表从纳米粒子上脱附下来的DNA的浓度。用NaOH(0.1 mol/L)和HCl(0.1 mol/L)的水溶液来调节金溶胶的pH值。将金纳米粒子对DNA吸附和脱附的量与金粒子的质量做比值,得到对DNA的绝对吸附和脱附量(mg/mg)。
2 结果与讨论
2.1 修饰Fe3+离子前后金粒子对DNA吸附量的变化规律
将修饰Fe3+离子前后金纳米粒子对DNA吸附量的变化规律进行了对比,如图1所示。在pH3.0-9.0的条件下,未修饰Fe3+离子的金纳米粒子对DNA的吸附量始终在0.5 mg/mg左右,随着pH条件变化不大。而对于Fe3+离子修饰的金粒子,其对DNA的吸附过程与未修饰Fe3+离子的粒子是完全不同的。在pH3.0时,修饰Fe3+离子的金纳米粒子对DNA的吸附量达到1.96 mg/mg,要远远高于未修饰Fe3+离子的粒子对DNA的吸附量0.52 mg/mg。随着pH升高到大于4.0时,修饰Fe3+离子的粒子对DNA的吸附量迅速下降,甚至低于未修饰Fe3+离子的粒子的吸附量0.4 mg/mg左右。当pH为9.0时,修饰Fe3+离子的金纳米粒子对DNA的吸附量几乎为零。修饰Fe3+离子的金纳米粒子这种随pH变化而产生的对DNA吸附量的差异是由Fe3+离子修饰到粒子表面上引起的。
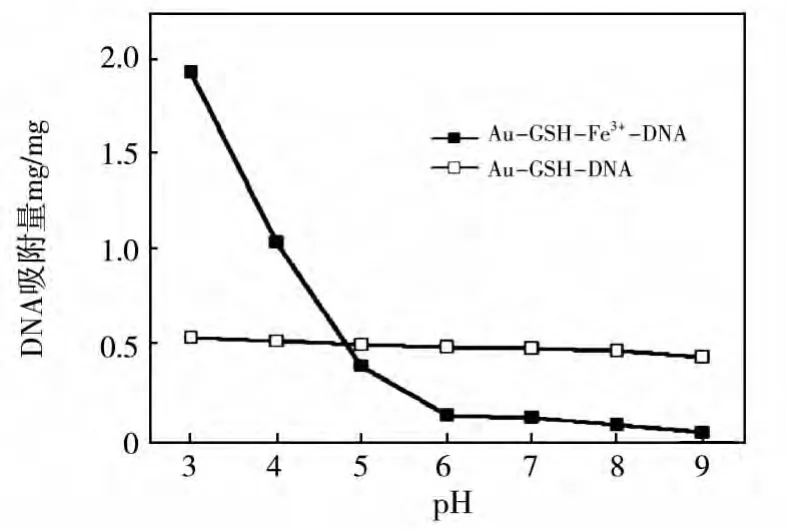
图1 螯合Fe3+离子前后的金纳米粒子对DNA吸附能力随pH条件的变化(DNA浓度为0.1 mg/ml)
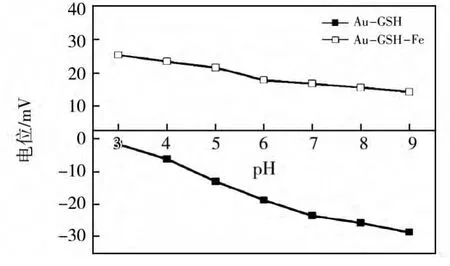
图2 螯合Fe3+离子前后的金纳米粒子表面电位随pH的变化
2.2 修饰Fe3+离子前后金纳米粒子表面电位的变化规律
金纳米粒子与DNA之间的作用通常被认为是静电相互作用,主要是由粒子表面性质决定的。我们对修饰Fe3+离子前后两种粒子表面的Zeta电位进行了测量,来考察粒子表面电荷随pH变化的规律。如图2所示,随着pH值升高,两种粒子表面的电位都逐渐下降,未修饰Fe3+离子的粒子的电位从-1.81下降到-28.57 mV,修饰Fe3+离子的粒子表面电位从27.38下降到了8.11 mV。而两种粒子对DNA的吸附量也随着pH值升高而逐渐下降。对于未修饰Fe3+离子的粒子来说,粒子表面一直带负电,由于静电排斥作用,使DNA不能通过静电作用被大量吸附到粒子表面。但是,粒子通过与DNA之间弱的氢键作用[24]及谷胱甘肽分子中含有的一部分NH3+的静电作用而使粒子对DNA少量的吸附。在pH3.0时变化较大的原因是由于此时处在表面谷胱甘肽分子中的COO-的pK点附近,表面电荷变化较大。对于表面修饰了Fe3+离子的粒子,从pH3.0到9.0粒子表面一直带正电,但是吸附效率却变化明显,当pH升高到大于4.0时,修饰Fe3+离子的粒子对DNA的吸附量迅速下降,甚至低于未修饰Fe3+离子的粒子。这说明两种粒子对DNA的吸附机制完全不同,除了静电作用外,一定存在另一种作用力使用DNA被吸附到粒子表面。
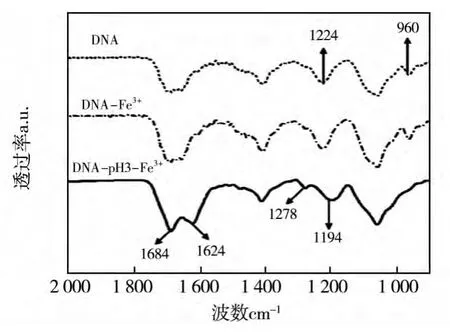
图3 DNA及其在pH6.0和pH3.0条件下加入Fe3+离子后的红外光谱
2.3 粒子表面Fe3+离子与DNA间配位作用的验证
我们通过红外测试来进一步理解粒子表面和DNA之间的相互作用,结果如图3所示。在pH7.0条件下加入Fe3+离子后,红外光谱与未加前比较,并没有发生变化。当在pH3.0时的DNA溶液中加入Fe3+离子后,红外光谱发生了明显的变化。960 cm-1处的峰位代表的是磷酸根的对称伸缩振动[25],加入Fe3+离子后,此处峰消失。1 600-1740 cm-1代表了P-O伸缩振动和弯曲振动的合频谱带[25],加入Fe3+离子后,此处峰位裂解为1 624和1 684 cm-1两个峰。1 224 cm-1是P-O的伸缩振动吸收峰[25],加入Fe3+离子后裂解为1 194和1 278 cm-1。这些数据变化说明磷酸根与Fe3+离子之间发生了配位相互作用。即DNA通过与Fe3+离子之间配位相互作用吸附到金纳米粒子表面。
2.4 盐浓度对吸附效率的影响
我们在确定Fe3+离子与磷酸根之间存在着配位相互作用后,又考察了在不同pH条件下盐浓度变化对两种粒子吸附DNA效率的影响。图4为在两个特定的pH值下(3.0和8.0),不同粒子对DNA吸附过程中盐浓度影响的比较。在具体的实验中,每个反应的溶液温度、DNA的初始浓度和加入硅粒子的浓度均保持一致。在pH3.0时,对于修饰了Fe3+离子的粒子来说,盐浓度的变化并未对吸附效率产生影响,随着盐浓度的增加,粒子对DNA的吸附效率一直保持在98%以上,这说明盐浓度的变化不会影响配位作用。而对于未修饰Fe3+离子的金纳米粒子来说,当盐浓度从0 mol/L增加到0.2 mol/L时,粒子对DNA的吸附率略有增加,从28%增加到32%。此时,粒子对DNA的吸附主要是由静电吸引力或粒子表面同DNA分子间的氢键作用主导的,对盐的敏感性不高。比较该pH条件下,两种粒子的吸附效率随盐浓度的变化规律,可以看出,即使在较高的盐浓度下,配位作用的吸附效率也要远远高于静电作用。这说明,不论在低盐度还是高盐度下,利用配位作用吸附DNA都可以达到很高的吸附效率。
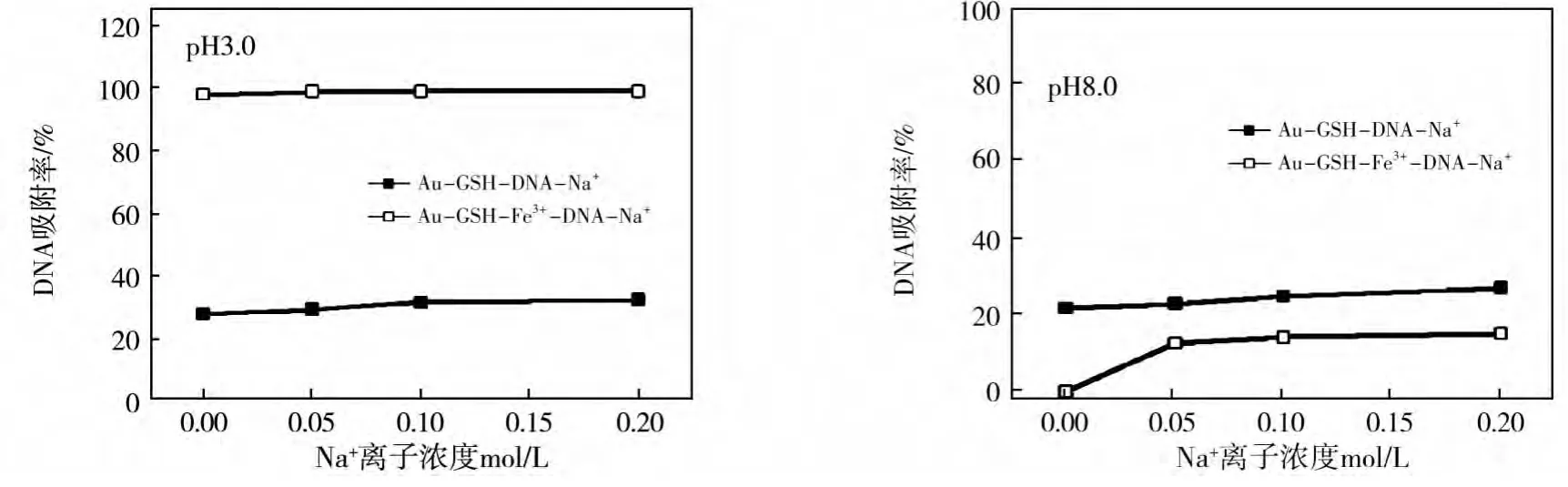
图4 在pH 3.0和8.0条件下,粒子修饰Fe3+离子前后对DNA的吸附效率随盐浓度的变化
在pH8.0时,对于修饰了Fe3+离子的粒子,加入盐后,其吸附效率从0.04%提高到了15%,但是仍然低于未修饰Fe3+离子的粒子。随着盐浓度的增加,吸附效率并没有明显提高。此时,粒子对DNA的吸附是由直接静电力主导的,对盐浓度不敏感。在此pH条件下对于未修饰Fe3+离子的粒子,其吸附DNA的效率从21.56%提高到了26.77%,提高的幅度不大。此时的吸附主要是静电力或粒子表面同DNA分子间的氢键作用来主导的,对盐浓度不敏感。
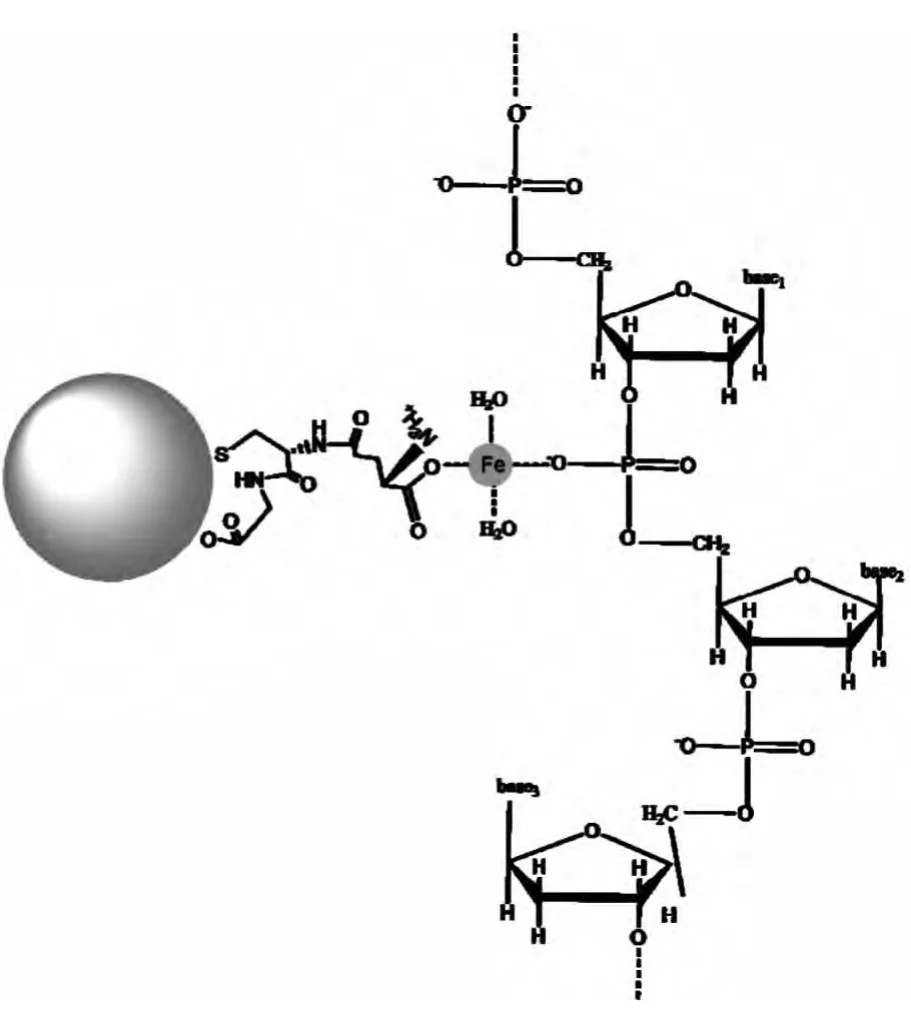
图5 DNA在螯合Fe3+离子的金纳米粒子表面的吸附机制
2.5 螯合Fe3+离子的金纳米粒子对DNA的吸附机制
综合前面的分析,我们总结出了修饰Fe3+离子的金纳米粒子在pH3.0达到最高吸附效率时对DNA的吸附机制,如图5所示。在pH3.0时,对于修饰Fe3+离子后的金纳米粒子,配位作用在吸附过程中占据主导地位,吸附效率达到了98%以上,远远高于未修饰Fe3+离子的粒子的吸附率,并且不受盐浓度的影响。
作为对比,还研究了表面修饰Ni2+离子的金纳米粒子对DNA的吸附行为。图6(A)对比了不同pH条件下,表面修饰Fe3+和Ni2+离子的粒子对DNA吸附量的变化规律。对于表面修饰Ni2+离子的粒子,在pH3.0时,粒子对DNA的吸附量达到了最大值0.61 mg/mg,但是远远低于同样条件下修饰Fe3+离子的粒子对DNA的吸附量。随着pH值升高,吸附量略有下降,但是下降程度並不大。
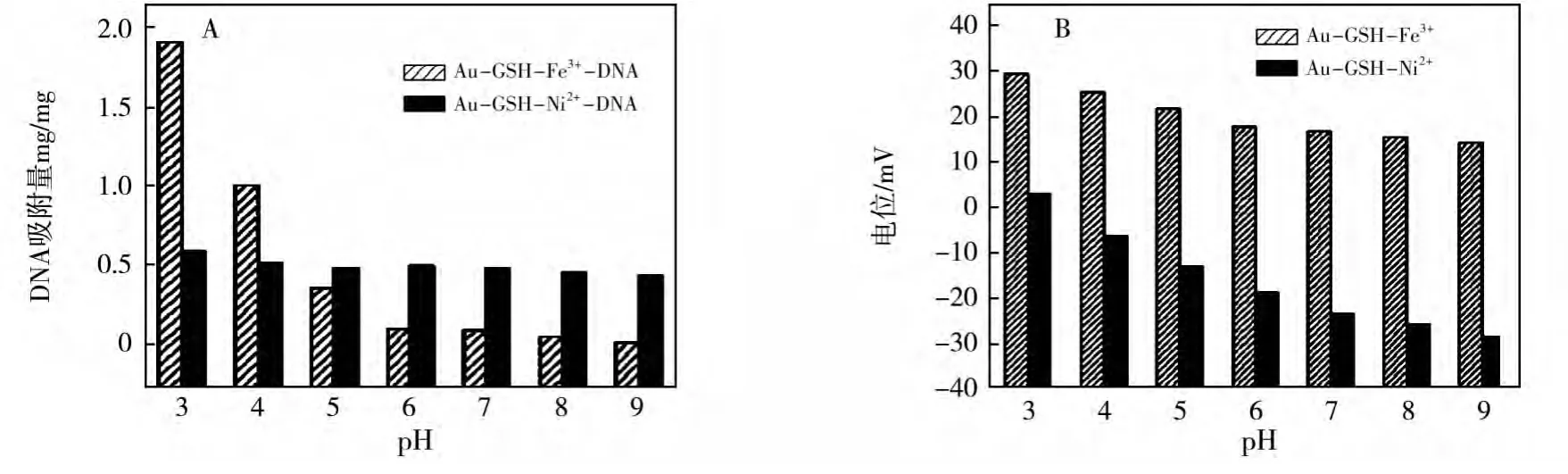
图6 表面修饰不同离子的金粒子对DNA吸附量(A)和表面电位(B)随pH变化的比较
在pH<5.0时,表面修饰Fe3+的粒子对DNA的吸附量要远高于Ni2+离子修饰的粒子。因为Ni2+离子(=2.1)[26]与磷酸根离子的结合能力要低于Fe3+离子(=8.3)[26]。因此,Ni2+离子与磷酸根离子之间的配位作用很弱,使得通过配位作用吸附到粒子表面的DNA的数量很少,因此Ni2+离子修饰的粒子不能通过配位相互作用实现对DNA的高效吸附。Ni2+修饰的粒子对DNA吸附的原理应该与未修饰的金纳米粒子对DNA的吸附类似,是通过静电作用或氢键相互作用实现对DNA的吸附,因此其吸附量要远低于修饰Fe3+的粒子。当pH>5.0时,表面修饰Ni2+离子的粒子对DNA的吸附量与pH<5.0时差不多。因为Ni2+离子与氢氧根离子的作用不是很强(=4.9)[26],仍然有部分Ni2+离子能够与磷酸根离子发生配位相互作用,但是配位能力比较弱,因此只有少量DNA通过配位作用吸附到了粒子表面上。与表面未修饰金属离子的粒子对DNA的吸附原理相似,粒子表面的NH3+与DNA的静电作用及氢键作用使得Ni2+离子修饰的粒子对DNA的吸附量与pH<5.0时差不多。但是要高于Fe3+离子修饰的粒子。我们比较了两种金属离子修饰的粒子的表面电位,如图6(B)所示,表面电位的变化规律与前面未修饰金属离子时的粒子一致,产生的原因也类似。
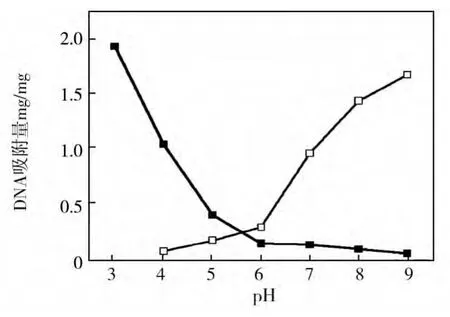
图7 螯合Fe3+离子的金粒子对DNA的吸附(■)和脱附(□)能力随pH值的变化
2.6 螯合Fe3+离子的金粒子对DNA吸附和脱附行为的变化规律
图7给出了在pH3.0-9.0条件下Fe3+离子修饰的金纳米粒子对DNA的吸附和脱附行为的变化规律。随着pH值的升高,粒子对DNA的吸附量逐渐减少。在pH3.0时,粒子对DNA的吸附量达到最高值为1.93 mg/mg。当pH值下降到6.0时,DNA的吸附量下降到0.12 mg/mg,当pH升高到9.0时,吸附量几乎为零,说明DNA几乎不能吸附到粒子表面。我们又对pH3.0时已经达到最大吸附DNA量的粒子进行脱附实验。从图7中可以看出,DNA的脱附曲线与吸附曲线几乎呈现镜像关系,当pH值从9.0下降到4.0时,脱附量从1.67 mg/mg下降到0.05 mg/mg。由此我们可以知道,对于Fe3+离子修饰的金纳米粒子来说,最佳吸附点是pH3.0,最佳脱附点是pH9.0。
3 结 论
文章在谷胱甘肽功能化的金纳米粒子表面螯合Fe3+离子后,研究了不同pH及盐浓度条件下,粒子对DNA分子的吸附及脱附行为。经过紫外、电位、红外等测试手段论证了粒子在不同pH条件下对DNA分子的吸附机制及影响因素,并确定了最佳吸附条件。粒子表面修饰Fe3+离子后,在低pH(<4.0)的条件下,通过Fe3+离子与DNA分子中磷酸根的配位相互作用,实现了粒子对DNA的高效吸附。这种吸附是不受溶液中的盐浓度影响的。以这种配位作用为基础的吸附效率要远远高于静电作用。这种DNA与金属离子的配位作用为高效提取DNA提供了一种新的技术。我们可以把一种能够与磷酸根离子发生强配位作用的离子修饰到粒子表面,通过控制一定的pH条件,来排除溶液中氢氧根离子的干扰,以及一些非特异性的吸附[27],以此来达到对DNA的高效特异性吸附[27]。这种方法也可以用于一些盐桥法无法实现的实际应用中,比如从基因工程生产的多肽中去除去DNA杂质而不引入额外的盐。
[1]Brust M,Walker M,Bethell D,Schlffrin D J,WHYMAN R.Synthesis of Thiol-Derivatized Gold Nanoparticles in a 2-Phase Liquid-Liquid System[J].Journal of the Chemical Society Chemical Communications 1994,7:801-802.
[2]Weare W W,Reed S M,Warner M G,Hutchison J E.Improved Synthesis of Small(dCORE-1.5 nm)Phosphine-Stabilized Gold Nanoparticles[J].Journal of the American Chemical Society 2000,122:12890-12891.
[3]Fu R,Zhuang J Q,Wang C,Yang W S.Aggregation of glutathione-functionalized Au nanoparticles induced by Ni2+ions[J].Journal of Nanoparticle Research 2012,14:929-936.
[4]Yang A C,Weng C I.Structural and Dynamic Properties of Water near Monolayer-Protected Gold Clusters with Various Alkanethiol Tail Groups[J].Journal of Physics Chemistry C 2010,114:8697-8709.
[5]Rosl N L,Mlrkin C A.Nanostructures in Biodiagnostics[J].Chemical Reviews 2005,105:1547.
[6]Storhoff J J,Elghanian R,MUCIC R C,MIRKIN C A,LETSINGER R L.One-Pot Colorimetric Differentiation of Polynucleotides with Single Base Imperfections Using Gold Nanoparticle Probes[J].Journal of the American Chemical Society 1998,120:1959.
[7]ELGHANIAN R,STORHOFF J J,MUCIC R C,LETSINGER R L,MIRKIN C A.Selective Colorimetric Detection of Polynucleotides Based on the Distance-Dependent Optical Properties of Gold Nanoparticles[J].Science 1997,277:1078.
[8]LEE J S,HAN M S,MIRKIN C A.Colorimetric Detection of Mercuric Ion(Hg2+)in Aqueous Media using DNA-Functionalized Gold Nanoparticles[J].Angewandte Chemie International Edition 2007,46:4093.
[9]HAN M S,LYTTON-JEAN A K R,MIRKIN C A.A Gold Nanoparticle Based Approach for Screening Triplex DNA Binders[J].Journal of the American Chemical Society 2006,128:4954.
[10]SONG Y J,WEI W L,QU X G.Colorimetric Biosensing Using Smart Materials[J].Advanced Materials 2011,23:4215.
[11]FU R,ZHUANG J Q,WANG C,YANG W S.Adsorption and desorption of DNA on bovine serum albumin modified gold nanoparticles[J].Colloids and surfaces A:physicochemical and engineering aspects 2014,444:326-329.
[12]TIAN H J,HUHMER A F R,LANDERS J P.Evaluation of silica resins for direct and efficient extraction of DNA from complex biological matrices in a miniaturized format[J].Analytical Biochemistry 2000,283:175-191.
[13]魏庆娟,闫永楠,孔波,等.微阵列技术的应用及发展趋势[J].东北电力大学学报,2008(6):17-21.
[14]WU Q,BIENVENUE J M,HASSAN B J.KWOK Y C,GIORDANO B C,NORRIS P M,LANDERS J P,FERRANCE J P.Microchip-based macroporous silica sol-gel monolith for efficient isolation of DNA from clinical samples[J].Analytical Chemistry 2006,78:5704-5710.
[15]BOOM R,SOL C J A,SALIMANS M M M,JANSEN C L,WERTHEIM-VAN DILLEN P M E,NOORDAA J.Rapid and simple method for purification of nucleic acids[J].Journal of Clinical Microbiology 1990,28:459-503.
[16]UMATA M,SUGIYASU K,HASEGAWA T,SHINKAI S,Sol-gel reaction using DNA as a Template:an Attempt toward Transcription of DNA into Inorganic materials[J].Angewandte Chemie International Edition 2004,43:3279-3283.
[17]TENG Z,LI J,YAN F,ZHAO R,YANG W.Highly magnetizable superparamagnetic iron oxide nanoparticles embedded mesoporous silica spheres and their application for efficient recovery of DNA from agarose gel[J].Journal of Materials Chemistry 2009,19:1811-1815.
[18]PHILIPSE A P,VAN BRUGGEN M P B,PATHMAMANOHARAN C,Magnetic silica dispersions:preparation and stability of surface-modified silica particles with a magnetic core[J].Langmuir,1994,10:92-99.
[19]GIRI S,TREWYN B G,STELLMAKER M P,LIN V S Y.Stimuli-responsivecontrolled-release delivery system based on mesoporous silica nanorods capped with magnetic nanopartilces[J].Angewandte Chemie International Edition 2005,117:5166-5172.
[20]LI Y,YAN B,XU X,DENG C,YANG P,SHEN X,ZHANG X.On-column tryptic mapping of proteins using metal-ion-chelated magnetic silica microspheres by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry[J].Rapid Communications in Mass Spectrometry 2007,21:2263-2268.
[21]JI X H,SONG X N,LI J,BAI Y B,YANG W S,PENG X G.Size Control of Gold Nanocrystals in Citrate Reduction:The Third Role of Citrate[J].Journal of the American Chemical Society 2007,129:13939-13948.
[22]FUJIWARA M,YAMAMOTO F,OKAMOTO K,SHIOKAWA K,NOMURA R.Adsorption of Duplex DNA on Mesoporous Silicas:Possibility of Inclusion of DNA into Their Mesopores[J].Analytical Chemistry 2005,77:8138-8145.
[23]LIN S Y,TSAI Y T,CHEN C C,LIN C M,CHEN C H.Two-Step Functionalization of Neutral and Positively Charged Thiols onto Citrate-Stabilized Au Nanoparticles[J].Journal of Physics Chemistry B 2004,108:2134-2139.
[24]HE S J,SONG B,LI D,ZHU C F,QI W P,WEN Y Q,WANG L H,SONG S P,FANG H P,FAN C H.A Graphene Nanoprobe for Rapid,Sensitive,and Multicolor Fluorescent DNA Analysis[J].Advanced Functional Materials 2010,20:453-459.
[25](a)OUAMEUR A A,ARAKAWA H,AHMAD R,NAOUI M,TAJMIR-RIAHI HA.A comparative study of Fe(II)and Fe(III)interactions with DNA duplex:major and minor grooves bindings[J].DNA and Cell Biology 2005,24:394-401;(b)The Sadtler Handbook of Infrared Spectrasadtler stan dard spectra collection.
[26]DEAN J A.Lange’s Handbook of Chemistry,15th ed.,McGraw-Hill Book Co.[M].New York,1999.
[27]JUEWEN L.Adsorption of DNA onto gold nanoparticles and graphene oxide:surfacescience and applications[J].Physical Chemistry Chemical Physics 2012,14:10485-10496.

