大豆再生抑制消减杂交文库的构建
武小霞,孙 晶,苏安玉,李 静,刘 明,张彬彬,李冬梅,李文滨
(1.东北农业大学农学院,大豆生物学教育部重点实验室,哈尔滨 150030;2.东北农业大学资源与环境学院,哈尔滨 150030)
大豆再生抑制消减杂交文库的构建
武小霞1,孙 晶1,苏安玉2,李 静1,刘 明1,张彬彬1,李冬梅1,李文滨1
(1.东北农业大学农学院,大豆生物学教育部重点实验室,哈尔滨 150030;2.东北农业大学资源与环境学院,哈尔滨 150030)
大豆再生受基因型限制,高效稳定再生体系建立难、遗传转化效率低,影响生物技术的应用。为获得与大豆再生相关基因,解析大豆再生基因分子机制,从而建立高效、稳定再生体系,为生物技术在大豆分子育种中的应用奠定基础。文章利用抑制性消减杂交技术(Suppression subtractive hybridization,SSH),以东农50子叶节为材料构建消减杂交cDNA文库,从文库中随机挑取917个阳性克隆,PCR鉴定插入片段大部分集中在100~750 bp,利用BLAST在GenBank数据库进行序列相似性比对,经过同源性比对归并后得到411个高质量ESTs片段,功能分析表明,这些基因与信号转导、葡萄糖、蛋白质大分子生物合成代谢,光、叶形态发生相关;调控细胞凋亡、细胞自身防御、细胞壁分化;参与各种激素及细胞分裂素介导的信号通路。
大豆;抑制消减杂交(SSH);cDNA文库;再生
大豆再生频率低,重复性差,是公认的难转化作物。自Hinchee等[1]和McCabe等[2]分别用农杆菌介导法和基因枪法成功转化大豆获得转基因植株以来,一直无突破性进展。对大豆再生体系发生机理和分子基础及再生过程中起作用基因研究很少,如果能得到控制或调节大豆再生基因并加以研究,有望解决大豆再生能力低难题。
抑制消减杂交技术(SSH)技术是Diatchenko等于1996年结合抑制PCR(Suppression PCR)技术和消减杂交法建立[3]的鉴定和分离组织、细胞中选择性表达基因的技术,是富集和分离差异基因较成熟的方法,以抑制性多聚酶链反应(PCR)为基础的cDNA消减杂交技术[4]。抑制消减杂交技术应用杂交二级动力学原理[5-6],不仅利用消减杂交技术消减富集作用,也利用抑制性PCR达到更高效率富集。能分离高、低丰度差异表达基因,并具有假阳性率低、敏感性高、筛选周期短、效率高、简便易行优点。
SSH技术已广泛应用于国内外动、植物研究领域。刘军等为分离水稻幼穗发育早期特异表达基因,以水稻茎尖分生组织(Shoot apical meristem,SAM)为对照样本、幼穗分生组织(Primary and sec⁃ondary branch primordia,Pb)为目标样本进行抑制消减杂交,获得40个在Pb中能特异表达或增强表达候选基因[7]。郭新红等为获得与渗透胁迫相关基因,以Hoagland溶液培养梭梭幼苗为对照样本,甘露醇处理的梭梭幼苗为目标样本进行抑制消减杂交,得到含约400个阳性克隆的消减文库[8]。Feng等将受精后的棉纤维作为检测子,未受精植株的相同部位为驱动子,构建减cDNA文库,分离出富含脯氨酸的蛋白(RPRs)、阿拉伯半乳聚糖蛋白(AGPs)、棒曲霉素、微管蛋白及脂类迁移蛋白(LTPs)五大家族基因[9]。张雷等从调控12 h胡萝卜体细胞胚cDNA文库中获得与胡萝卜体细胞胚根发育相关基因[10]。李冲等将冬眠马铃薯发芽部位作为检测子,块茎部位作为驱动子构建消减cDNA文库,找出与生长调节相关基因,着重研究生长因子ARF6基因,证实与植物发芽有关[11]。
Low等以泡桐为材料,利用抑制消减杂交技术差减出再生候选基因PKSF1,PKSF1主要是编码亮氨酸拉链转录因子,促进愈伤组织形成[12]。近年来,对体细胞胚和子叶节再生相关基因有过报道,Zubko等在矮牵牛中得到Sho再生候选基因,Sho能促进细胞分裂素途径中关键酶异戊烯基转移酶(IPTs)增加[13]。Komatsuda等通过QTL定位方法获得与不定芽再生有关基因Shd1[14]。Zakizadeh等在玫瑰中分离出再生候选基因RhSERK1-4,该基因与体细胞胚胎发生受体激酶(SERK,the somatic embryogenesis receptor-like kinase)表现出高度的序列相似性[15]。研究发现RhSERK3和RhSERK4与体细胞胚胎发生有关[16]。
在拟南芥中,再生过程调控的研究相对较多,Banno等在拟南芥中克隆出可促进再生ESR2基因,ESR1和ESR2均是具有AP2/EREBP结构域的转录因子[17]。ESR2调控过程报道,认为ESR2通过调控CUC1基因来控制拟南芥再生[18];除ESR基因外,WUSCHEL基因家族通过对细胞分裂素应答元件的调控,控制体细胞胚的发生[19],在大豆中尚无克隆出调控再生相关基因的报道。
大豆组织培养工作在20世纪80年代中、后期才有突破性进展。大豆经器官发生再生植株最早是由Kartha等报道,外植体采用栽培大豆真叶[20]。Barwale等通过体细胞胚成功获得植株再生,大豆体细胞胚胎再生体系获得初步成功[21]。之后对外植体种类、取材时间、诱导激素种类和浓度等能影响大豆体细胞胚胎发生因素进行深入研究。本试验是以大豆东农50子叶节为材料,在细胞分裂素6-BA诱导下,抑制消减杂交出差异基因,对这些基因进行功能分析,以期获得再生相关基因。即从子叶节器官发生途径结合激素对大豆再生途径相关转录因子基因的表达影响,分析再生基因在再生体系中的作用,寻找再生基因并对基因进行克隆和改造,从分子水平上分析基因功能并研究其作用机理,为发掘大豆再生基因,提高大豆再生和遗传转化效率提供理论依据,为提高我国大豆综合产量奠定基础。
1 材料与方法
1.1 材料
东农50由东北农业大学大豆研究所提供,是具有较高再生能力的大豆品种,氯气灭菌后种脐向下接种到萌发培养基(4.43 g MS,3%蔗糖,0.7%琼脂,pH 5.8)中,25℃光照培养箱,16 000 lx白光,长日照(16 h/8 h,光/暗)培养5~7 d后取出无菌苗,去掉种皮,切去大部分下胚轴,只留靠近子叶的3~5 mm下胚轴,将2片子叶从下胚轴中线处切开,除去顶芽和腋芽,得到子叶节外植体。将一部分子叶节外植体接种到添加6-BA的液体培养基(4.43 g MS+2 mg L-16-BA,3%蔗糖,pH 5.8)中,处理1~10 h取样作为SSH tester;另一部分子叶节外植体接种在不添加6-BA的液体培养基中,同样处理1~10 h取样作为SSH driver,液氮速冻,-80℃保存。
1.2 化学药品及试剂
CLONTECH SMARTerTMPCR cDNA Synthesis Kit和 CLONTECH PCR-SelectTMcDNA Subtraction Kit购自CLONTECH公司;Trizol试剂、pMD 18-T、LATaqDNA聚合酶、DL2000 DNA Marker、DL8000 DNA Marker、DL15000 DNA Marker均购自TaKaRa公司;电泳用琼脂糖(Agarose)为西班牙进口试剂;其他各种生化试剂均为国产分析纯;测序由华大基因组(BGI)完成。
1.3 方法
1.3.1 抑制消减杂交文库的构建[22]
选择Trizol试剂法提取大豆子叶节的总RNA。以细胞分裂素6-BA处理的子叶节cDNA为Tester,未经过6-BA处理的子叶节为Driver,参照CLON⁃TECH SMARTTMPCR cDNA Synthesis Kit说明书上的方法,利用SMART技术将Tester和Driver两个样本各小时总RNA混合在一起,反转录合成cDNA,通过LD-PCR选择最佳扩增状况的循环数,确保双链cDNA处于对数扩增阶段。参照CLONTECH PCRSelectTMcDNA Subtraction Kit的说明进行抑制性消减杂交。先将Tester-cDNA用限制性内切酶RsaⅠ酶切,平分成两份分别连接接头1和接头2R,16℃温育过夜后,将连有接头的Tester和Driver分别与RsaⅠ酶切的driver-cDNA进行第1次杂交,98℃温育90 s,68℃杂交8 h,再向第1次消减杂交体系混合物中加入过量新鲜热变性driver cDNA进行第2次杂交,68℃温育过夜,加入200 μL的稀释缓冲液后68℃温育7 min即可。继尔进行第1次和第2次巢式PCR扩增。巢式PCR反应25 μL PCR反应体系如下:杂交样品1 μL,10×PCR buffer 2.5 μL,dNTP Mix(10 mmol·L-1)0.5 μL,上下游Nested PCR Primer各1 μL,LA DNA聚合酶 (5 U·L-1)0.5 μL,无菌水18.5 μL;进行巢式PCR扩增,94℃变性10 s,68℃复性 30 s,72℃延伸1.5 min。
将巢式PCR产物纯化回收后与pMD-18T载体连接,16℃过夜,使用热击法转化大肠杆菌(Esch⁃erichia coli)DH5α感受态细胞,接种于含有X-gal/ IPTG/Amp的固体LB培养基上进行蓝白斑筛选,挑取白色单斑接种于液体LB培养基中,37℃过夜振荡培养,挑取单斑,进行菌液PCR鉴定,引物为巢式PCR引物,Nested PCR Primer1(5′TCGAGCG CCGCCCGGGCAGGT 3′)和Nested PCR Primer2R(5′AGCGTGGTCGCGGCCGAGGT 3′),进行菌液PCR鉴定,PCR反应25 μL混合体系如下:菌液2 μL,10×PCR buffer 2.5 μL,dNTP Mix(10 mmol·L-1)0.5 μL,Primer1和Primer2R引物各0.5 μL,LA DNA聚合酶(5 U·L-1) 0.3 μL,无菌水18.7 μL。进行PCR扩增反应,94℃预变性5 min;30个循环:94℃变性30 s,68℃复性30 s,72℃延伸2 min;72℃终延伸5 min。将鉴定为阳性克隆的菌液按1∶1加30%甘油,于-80℃中保存菌种。
1.3.2 ESTs测序与序列结果分析
将随机挑取的阳性克隆送至北京华大基因测序,EST(Expressed sequence tag)是从一个随机选择cDNA克隆进行5′端和3′端单向测序,获得cD⁃NA的部分序列,代表一个完整基因的一部分,在数据库中其长度从20到7 000 bp不等,平均长度400 bp。EST来源于一定环境下一个组织总mRNA构建的cDNA文库,因此EST也能说明该组织中各基因的表达水平,序列经过去载体、宿主序列重复序列后进行拼接,所得EST片段利用NCBI和PHYTOZOME的BLAST(Basic local alignment aearch tool)比对进行序列相似性分析。
2 结果与分析
2.1 大豆子叶节总RNA的提取
大豆子叶节总RNA经2%琼脂糖凝胶电泳检测,28S、18S和5S条带清晰RNA(见图1)。
2.2 SMART技术合成cDNA
SMART(Switching mechanism at 5 end of the RNA transcript)技术是利用真核mRNA特有的“帽子结构”而扩增得到全长的cDNA,并经过PCR方法得到处于对数期扩增阶段扩增情况最佳的双链cDNA,既不因循环次数过少而丧失过多的低拷贝数基因,又不会因循环次数太多而产生非特异性产物。选取能够代表原有样品中mRNA丰度的cDNA样品。
试验中Tester在第30个循环时显示扩增效率降低,故采用27个循环进行cDNA扩增;而Driver在第27个循环时显示低扩增效率,所以采用24个循环进行cDNA扩增(见图2)。

图1 大豆子叶节总RNA的提取Fig.1 Total RNA extraction from soybean cotyledonary node
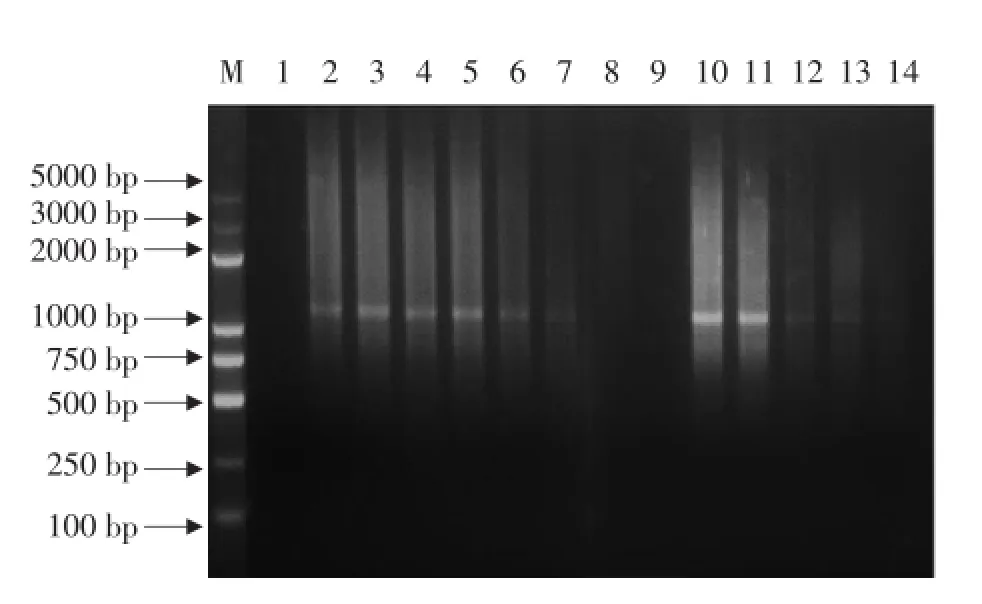
图2 Tester和Driver的cDNA PCR扩增结果Fig.2 Results of cDNA PCR amplification of Tester and Driver
2.3 双链cDNA的RsaⅠ酶切结果
RsaⅠ酶是四碱基内切酶,约256 bp,有1个酶切点,能将双链cDNA酶切成较小的cDNA片段,小片段可降低杂交过程中形成的复杂二级结构对杂交的阻碍作用,提高不同基因检出率,防止长cDNA片段对消减杂交干扰(见图3)。
2.4 消减杂交后两次PCR扩增的结果分析
在杂交液中进行第1次杂交,杂交结束后会形成4种杂交产物:a是均一化差异表达单链Tester cDNA;b是自身退火的Tester cDNA双链;c是异源退火的Tester和Driver cDNA;d是Driver cDNA。将两份第1次杂交样品进行混合,再加入新鲜热变性Driver cDNA在缓冲液中进行第2次杂交,进一步除去共有序列,第1次杂交时在退火中形成均一化连接不同接头的差异表达单链Tester cDNA特殊类型分子e,第2次杂交补平接头末端[23]。加入adapter 1和adapter 2R的部分特异序列进行PCR扩增,第1次PCR扩增时a和d没有引物结合位点故不能进行扩增;b两端连有相同的接头,接头上有反向重复序列抑制PCR也不能进行扩增;而c是单接头,只能进行线性扩增;只有两端连接有不同接头双链DNA分子e才可进行扩增,扩增产物即为目的片段。第2次PCR扩增使接头内侧的引物选择性扩增目的片段,经过两次杂交和两次Nested PCR,目的片段即被消减并富集,得到长度在200~1 400 bp均匀弥散状片段,平均在400~700 bp(见图4)。
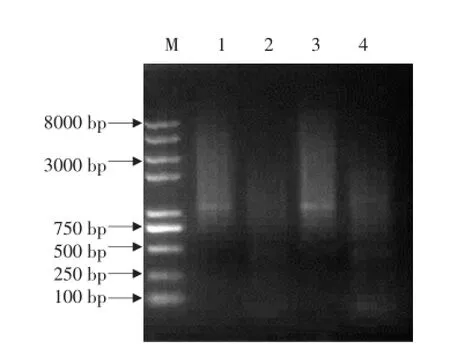
图3 双链cDNA的RsaⅠ酶切结果Fig.3 Results of RsaⅠenzyme cutting double chain cDNA

图4杂交后两次PCR扩增结果Fig.4 Twice PCR products after subtraction
2.5 PCR扩增的消减文库中的插入片段
经SSH消减并富集得到的cDNA片段,转化大肠杆菌DH5α感受态细胞,筛选蓝白班,随机挑取917个克隆,PCR检测表明大多数插入片段长度在100~750 bp(见图5)。
2.6 EST测序与同源性检索结果
随机获得的917个克隆,其中高质量EST 411个,占总数的44.8%。将测序结果到GenBank进行BLASTn和BLASTx比对,基因功能涉及信号转导,葡萄糖、蛋白质大分子生物合成代谢,光、叶形态发生,细胞凋亡、细胞自身防御、细胞壁分化,各种激素及细胞分裂素介导的信号通路,具体见表1、图6。
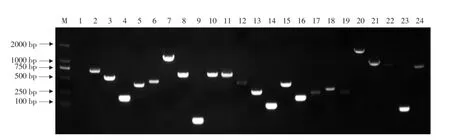
图5 消减文库中插入片段检测Fig.5 Screening of the inserts in of subtractive library

表1 部分ESTs与GenBank已知基因同源性比较结果Table 1 Similarity analysis of ESTs comparing with genes in GenBank
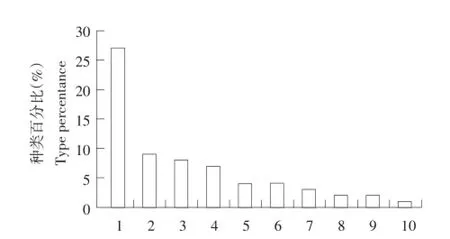
图6ESTs比对结果分类Fig.6 Classification result of the ESTs
3 讨论与结论
本试验采用抑制消减杂交技术,构建细胞分裂素诱导大豆子叶节的差异表达cDNA消减文库,对于研究大豆器官发生再生途径及筛选大豆再生候选基因具有重要意义。对比其他消减杂交文库构建,本试验对试验材料与方法进行优化,以小时为时间点取材,能够更密集消减杂交出有差异变化的基因,随机挑取917个阳性克隆,文库构建足够大,可更全面、准确地挑出有变化基因。
抑制消减杂交方法可弥补其他消减杂交方法缺点和不足,克服分离单双链DNA操作难问题,抑制PCR扩增使差异表达的基因达到均一化,而进行2次抑制PCR能使表达丰度不同差异表达片段有效富集。SSH只用2次消减杂交和2次抑制PCR即可有效获得特异扩增差异表达的目的片段,操作简单快捷、灵敏度高、假阳性频率低。但SSH也存在局限性,需要的初始材料较多,1次只能对2个样品进行杂交,只能分离在Tester中表达而在Driver中不表达的基因[24]。
目前,对植物再生相关基因研究较少,但在模式植物拟南芥、水稻等植物中已有调控再生相关转录因子或基因相关报道。本研究拟通过现代分子生物学技术从根本上提高大豆的再生能力,解决大豆再生体系建立难、转化难问题。关于植物再生相关基因的克隆在国内外报道甚少,而大豆再生相关基因克隆更是未见报道。因此,如果找到控制大豆再生基因,将会对大豆转基因技术产生深远影响。此外,对于大豆再生以及发育分子机制,国内外研究甚少,本研究分析再生相关基因在组织中的表达及外源激素变化对其影响,可为探明大豆再生及发育机制提供理论依据。
本研究目标是寻找调控大豆再生的关键遗传基因,为最终解决大豆再生难、转化难等问题奠定基础。构建大豆器官发生再生途径抑制性消减杂交(SSH)文库,在已获得ESTs序列基础上,将进行大豆再生相关基因克隆和功能分析,在转基因条件下研究再生基因表达及功能和激素信号转导对大豆再生的影响。
对大豆再生抑制消减杂交文库中的ESTs进行基因比对分析发现,细胞分裂素诱导下发生变化的基因按功能可分为,感知各种激素的信号转导通路、影响种子萌发和开花时间、细胞程序性死亡负调控、叶片衰老、花粉管伸长、葡萄糖、蛋白质等大分子生物合成及代谢、光合作用、核糖体、转运蛋白、过氧化氢酶、磷酸转移酶等类别;还发现与冷、热、干旱、高盐、机械损伤胁迫有关的大量基因以及一些未知基因。其中一些基因通过RT-PCR试验结果表明,在受细胞分裂素诱导后差异表达明显;参与激素信号转导途径的基因很有可能与植物再生有相关作用,后续试验将进一步研究。
[1]Hinchee A W,Conner-Ward D V,Newell C A,et al.Production of transgenic soybean plants using Agrobacterium-mediated DNA transfer[J].Nature Biotechnology,1988(6):915-922.
[2]McCabe D E,Swain W F,Martinell B J,et al.Stable transforma⁃tion of soybean(Glycine max)by particle acceleration[J].Nature Biotechnology,1988(6):923-926.
[3]Diatchenko L,Lau Y P,Campbell A P,et al.Suppression subtrac⁃tive hybridization:A method for generating differentially regulat⁃ed or tissue-specific cDNA probes and libraries[J].National Acad Sciences,1996,93(12):6025-6030.
[4]康俊梅,孙彦,杨青川.SSH与cDNA芯片技术的联合及其在植物基因差异表达研究中的应用[J].中国草地学报,2007,29(5): 89-94.
[5] 黄凤兰,胡国富,胡宝忠.抑制性消减杂交在生物基因克隆中的应用[J].生物技术通讯,2004,15(4):396-399.
[6]邵勇,姚珍薇.消减杂交基因克隆技术研究进展[J].检验医学与临床,2005,2(2):71-73.
[7]刘军,袁自强,刘建东,等.应用抑制差减杂交法分离水稻幼穗发育早期特异表达的基因[J].科学通报,2000,45(13):1392-1397.
[8]郭新红,姜聿成,潘晓玲,等.用抑制差减杂交法分离和克隆梭梭幼苗受渗透胁迫诱导相关基因的cDNA片段[J].植物生理学报,2001,27(5):401-406.
[9]Feng J X,Ji S J,Shi Y H,et al.Analysis of five differentially ex⁃pressed gene families in fast elongating cotton fiber[J].Aeta Bioe⁃him Biophys Sin,2004,36(1):51.
[10]张雷,杨志攀,刘一鸣,等.cDNA文库的差异筛选与抑制性减数杂交相结分离胡萝卜体细胞胚根发育相关基因[J].自然科学进展,2002,12(3):261-265.
[11] 李冲,吴耀生.抑制消减杂交(SSH)在植物功能基因研究中的应用[J].国外医药(植物药分册),2005,20(6):242-245.
[12]Low R K,Prakash A,P,Swarup S,et al.A differentially ex⁃pressed bZIP gene is associated with adventitious shoot regenera⁃tion in leaf cultures of Paulownia kawakamii[J].Plant Cell Rep, 2001,20:696-700.
[13]Zubko E,Adams C J,Macháèková I,et al.Activation tagging identifies a gene from Petunia hybrid responsible for the produc⁃tion of active cytokinins in plants[J].The Plant Journal,2002,29 (6):797-808.
[14]Komatsuda T,Annaka T,Oka S,et al.Genetic mapping of a quantitative trait locus(QTL)that enhances the shoot differentia⁃tion rate in Hordeum vulgare L[J].Theoretical and Applied Genet⁃ics,1993,86(6):713-720.
[15]Zakizadeh H,Stummann B M,Lütken H,et al.Isolation and char⁃acterization of four somatic embryogenesis receptor-like kinase (RhSERK)genes from miniature potted rose(Rosa hybridacv.Lin⁃da)[J].Plant Cell,Tissue and Organ Culture,2010,101(3):331-338.
[16]叶兴国,佘茂云,王轲,等.植物组织培养再生相关基因鉴定、克隆和应用研究进展[J].作物学报,2012,38(2):191-201.
[17]Banno H,Ikeda Y,Niu Q W,et al.Overexpression of Arabidopsis ESR1 induces initiation of shoot regeneration[J].The Plant Cell, 2001,13(12):2609-2618.
[18]Ikeda Y,Banno H,Niu Q W,et al.The enhancer of shoot regener⁃ation 2 gene in Arabidopsis regulates cuo-shaped cotyledon 1 at the transcriptional level and controls cotyledon development[J]. Plant and Cell Physiology,2006,47(11):1443-1456.
[19]Leibfried A,Busch W,Stehling S,et al.WUSCHEL controls meri⁃stem function by direct regulation of cytokinin-inducible re⁃sponse regulators[J].Nature,2005,438(7071):1172-1175.
[20]Kartha K K,Pahl K,Leung N L,et al.Plant regeneration from meristems of grain legumes:Soybean,cowpea,peanut,chickpea, and bean[J].Canadian Journal of Botany,1981,59(9):1671-1679.
[21]Barwale U B,Kerns H R,Widholm J M.Plant regeneration from callus cultures of several soybean genotypes via embryogenesis and organogenesis[J].Planta,1986,167:473-481.
[22]赵琳,罗秋兰,杨春亮,等.大豆在暗诱导条件下差异表达cD⁃NA文库的构建及分析[J].大豆科学,2007,26(2):134-140.
[23] 夏启中,张明菊,张献龙,等.抑制差减杂交技术原理及常见操作结果分析[J].黄冈师范学院学报,2005,25(3):30-33.
[24] 任志莹,杨立国,肖军,等.植物基因分离的抑制消减杂交PCR [J].辽宁农业科学,2008,(1):41-43.
Construction of SSH library of regeneration related gene in soybean
It is the world recognized challenge to set up genetic transformation of soybean regeneration system with the problem of low efficiency.In order to establish a highly efficient and stable regeneration system,in this study,cotyledonary node of DN50 were used to construct suppression subtractive cDNA library,and 917 positive clones were screened.The length of most of the insert fragments was focused on 100-750 bp.For further ESTs screening,411 differentially expressed functional ESTs were found and predicted by BLAST(Basic Local Alignment Search Tool)in GenBank.The ESTs were annotated by similarity to orthologs and paralogs detected with Blastn and Blastx.Function analysis showed that genes were annotated by similarity to signal transduction,macromolecules glucose,and protein synthesis and metabolism,light and leaf morphogenesis,regulation of apoptosis,the cell defense,and cell wall differentiation,participate in a variety of hormones and cytokinin-mediated signaling pathways.
soybean;suppression subtractive hybridization(SSH);cDNA library;regeneration
S565.1
A
1005-9369(2014)07-0038-07
时间2014-7-4 17:26:30 [URL]http://www.cnki.net/kcms/detail/23.1391.S.20140707.0843.009.html
武小霞,孙晶,苏安玉,等.大豆再生抑制消减杂交文库的构建[J].东北农业大学学报,2014,45(7):38-44.
Wu Xiaoxia,Sun Jing,Su Anyu,et al.Construction of SSH library of regeneration related gene in soybean[J].Journal of Northeast Agricultural University,2014,45(7):38-44.(in Chinese with English abstract)
2014-01-09
国家自然科学基金资助项目(31071438);黑龙江省自然科学基金项目(ZD201117);黑龙江省教育厅重点项目(12531z001)
武小霞(1971-),女,研究员,博士,博士生导师,研究方向为大豆遗传育种与生物技术应用。E-mail:xxwu2012@126.com
WU Xiaoxia1,SUN Jing1,SU Anyu2,LI Jing1,LIU Ming1,ZHANG Binbin1,LI Dongmei1,LI Wenbin1(1. School of Agriculture,Northeast Agricultural University,Key Laboratory of Soybean Biology in Chinese Ministry of Education,Harbin 150030,China;2.Shool of Resources and Environmental Science,Northeast Agricultural University,Harbin 150030,China)

