河南连翘种群遗传多样性的ISSR分析
汤正辉,祝亚军,谭晓风 ,谭运德
(1. 河南省林业科学研究院,河南 郑州 450008; 2. 中南林业科技大学,湖南 长沙 410004)
河南连翘种群遗传多样性的ISSR分析
汤正辉1,2,祝亚军1,谭晓风2,谭运德1
(1. 河南省林业科学研究院,河南 郑州 450008; 2. 中南林业科技大学,湖南 长沙 410004)
应用ISSR分子标记对分布于河南省不同地区的连翘Forsythia suspensa 6个自然种群进行遗传多样性分析,结果表明,连翘具有较高的遗传多样性。12条引物共得到89个重复性好的位点,根据ISSR扩增结果,利用POPGENE 32软件进行分析,在物种水平上,多态位点百分率为94.38%;总遗传多样性指数H(Nei’s基因多样性指数)和I(Shannon信息多样性指数)分别为0.326 5和0.490 1。种群间遗传分化系数为0.274 3,遗传变异主要来自于种群内部。用NTSYS-pc 2.10e软件,根据遗传一致度构建样本间亲缘关系的UPGMA聚类分析,结果表明,33个样本可划分为2大类群6个亚类。连翘种群间遗传距离与地理距离没有相关性。连翘本身的生物学特性与不同种群所处生境的差异是河南连翘种群现有遗传格局的主要原因。
连翘;遗传多样性;ISSR;聚类分析;河南省
连翘Forsythia suspensa (Thunb.) Vahl 系木犀科Oleaceae连翘属Forsythia Vahl 落叶灌木。别名黄寿丹、黄绶丹、绶带。分布于我国东北、河北、山东、河南、山西、陕西、甘肃等省[1]。河南是全国连翘的主要产区之一,野生连翘主要分布于太行山、崤山、熊耳山、伏牛山等山区的济源、辉县、林州、卢氏、栾川、灵宝、陕县、嵩县、南召等地。连翘喜光,有一定的耐阴性,耐寒,耐干旱、瘠薄,怕涝,不择土壤,抗病虫害能力强[2]。生长在海拔250~2 200 m之间的阳坡或半阳坡的灌丛或林缘。连翘是重要的经济型灌木资源,是造林绿化、干旱半干旱地区植被建设的重要材源,而且具有药用、观赏等重要的经济价值。连翘是常用大宗药材,果实入药,已成为双黄连口服液、银翘解毒片等中药的主要原料。早春开花繁茂,金黄灿烂夺目,适于屋旁、路侧、篱下种植。因根系发达,有固定堤坡的作用,为优良的水土保持灌木[3]。
简单重复序列中间区域DNA标记技术(Intersimple Sequence Repeat,ISSR ) 是 由 Zietkiewicz等提出的建立在PCR反应基础上的一种分子标记技术[4],用于检测简单重复序列(亦称微卫星,Simple Sequence Repeat, SSR)间DNA序列差异。ISSR标记呈孟德尔式遗传,多数为显性标记,技术简单、快速,成本低廉[5]。SSR在双子叶植物与单子叶植物基因组中分布均匀,ISSR技术就是基于SSR而设计的引物来检测2个SSR之间的DNA序列差异,因此能提供更多的信息,更加高效地检测出基因组的多样性。ISSR分子标记技术已在多种植物的种质鉴定、基因定位[6-7]、遗传多样性检测、遗传多样性亲缘关系分析[8-14]等研究方面得到成功的应用。
目前,应用ISSR分子标记技术对野生连翘种群遗传多样性的研究还不多见。本试验采用ISSR分子标记技术对分布于河南省的自然连翘种群进行研究,分析野生连翘种群间的遗传多样性水平和亲缘关系、遗传距离和遗传相似性,旨在为野生连翘遗传育种和构建物理图谱等研究奠定基础。
1 材料和方法
1.1 材 料
试验材料取自河南省连翘主产区的太行山、崤山、伏牛山、熊耳山等地,供试材料地理位置、海拔及生境情况见表1。于2012年8月采集当年生叶片,每样本采集间隔大于500 m,置于-70 ℃冰箱备用。

表1 6个连翘种群的取样地点Table 1 Sampling spots of six nature populations of Forsythia suspensa
1.2 方 法
1.2.1 DNA提取
连翘基因组DNA采用天根生化科技(北京)有限公司提供的植物基因组DNA提取试剂盒提取。使用Beckman DU-640核酸蛋白分析仪测定DNA浓度和纯度,并将浓度稀释为60 ng·μL-1,置于-20 ℃冰箱备用。
1.2.2 引物筛选
引物参照加拿大哥伦比亚大学UBC公司2006年公布的ISSR引物序列,由上海英骏生物技术有限公司合成。通过优化设计出适合连翘ISSR-PCR最佳反应体系,从66条引物中筛选出12条扩增条带清晰、反应稳定的引物用于所有样品的PCR扩增(见表2)。
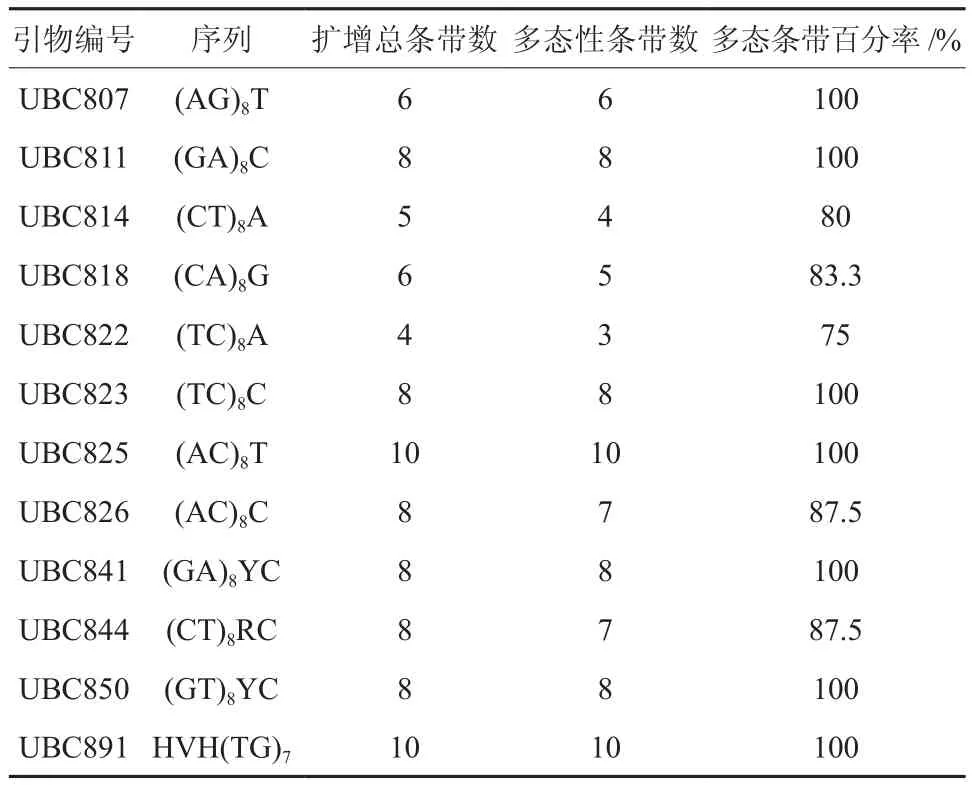
表2 ISSR引物序列及特异性条带数目†Table 2 The 12 ISSR primers and polymorphic bands obtained
1.2.3 PCR扩增
PCR扩增反应使用BIO-RAD Mycycler Thermal Cycler System扩增仪。总体系为25 μL,含 1.2 U Taq DNA聚 合 酶、10×PCR buffer 2.5 μL、1.5 m mol·L-1Mg2+、60 ng 模 版 DNA、0.3 mmol·L-1dNTPs、0.3 μ mol·L-1引物,ddH2O 补齐;最佳退火温度为55 ℃。扩增程序为:94 ℃预变性5 min;94 ℃变性 1 min,55 ℃退火 45 sec,72 ℃延伸2 min,45个循环;72 ℃再延伸5 min;4 ℃保温。100 bp DNA Ladder Marker、琼脂糖、Taq DNA聚合酶均购自宝生物公司的Takara产品。
PCR产物用1.2%琼脂糖凝胶(含SYBR GreenⅠ核酸染料,北京鼎国生物技术有限公司提供)于3 V·cm-1电压下电泳,电泳结果采用BIORAD Gel DocTM XR凝胶成像系统成像。
1.3 数据分析
ISSR为显性标记,电泳图谱的每条带(DNA片段)均为一个分子标记(Marker),同一引物扩增产物中电泳迁移率一致的条带被认为具有同源性。根据各分子标记的迁移率及有无统计所有的二元数据;有带(显性)记作1,无带(隐性)记作0,强带和弱带均赋值为1。对于多态位点,仅在重复实验中能稳定出现的差异带用于数据分析。
得到的原始数据矩阵用于以下分析:(1)用POPGEN 32软件对33份材料进行遗传多样性分析,计算连翘各个种群的多态位点百分率(PPL)、观测等位基因数(A0)、有效等位基因数(Ae)、Nei’s 基因多样性指数(H)、Shannon信息多样性指数(I)、基因分化系数(Gst)、基因流0.5(1-Gst)/Gst、及Nei’s遗传距离和遗传一致度;(2)根据种群遗传一致度和单株间的Nei相似系数,利用NTSYS-pc 2.1软件对连翘种群和单株进行非加权配对算数平均法(UPGMA,unweighted pair group method using arithmetic average)聚类。
2 结 果
2.1 连翘种群的遗传多样性
12条引物用于33个连翘材料的ISSR扩增,共扩增出89个DNA片段,其中多态性片段85个,占总带数的95.5%(表2)。每个引物扩增的DNA带的数目在4~10条,平均达到7.42条,该数值只是对遗传多样性的一个粗略估计。扩增出的DNA条带的大小为300~2 200 bp。引物825扩增的ISSR-PCR产物图谱见图1。

图1 引物UBC825对连翘6个种群的ISSR-PCR扩增Fig. 1 ISSR-PCR bands of 6 natural F. suspensa populations amplifed with primer UBC825
由表3可知,Nei’s基因多样性指数H为0.326 5,Shannon信息多样性指数I为0.490 1。Nei’s指数和Shannon多样性指数是反映条带丰富程度和均匀程度的两个重要指标。依据Nei’s指数计算的各种群遗传多样性数值较据Shannon多样性指数计算的数值偏低,但二者计算的各连翘种群平均遗传多样性的变化趋势一致,依次为ShX>LCh>JY>LZh>LuSh>LSh。6个连翘种群遗传多样性有差异。
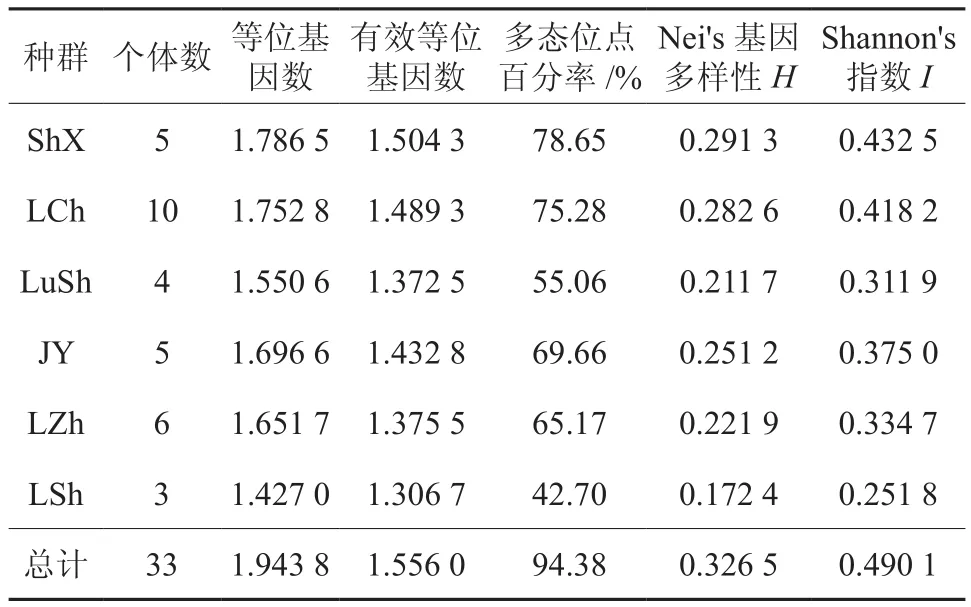
表3 6个连翘种群的遗传多样性参数Table 3 Parameters of genetic diversity in 6 natural F. suspensa populations
2.2 连翘种群遗传变异的分析
分析结果表明,连翘各种群间存在较大的遗传分化。6个连翘群落总的基因多样度Ht为0.328 7,其中种群内的基因多样性Hs为0.238 5;种群间基因流Nm为1.232 3,大于1,表明种群间基因交流较多;种群间的基因分化系数Gst为0.274 3,种群间的遗传变异占了总遗传变异的27.43%,遗传变异主要存在于种群内。
2.3 6个连翘种群的聚类分析
遗传一致度或遗传距离是衡量种群间遗传分化程度的最重要指标。按Nie方法计算种群间的遗传一致度和遗传距离,结果如表4所示,各种群间遗传一致度的变化范围在0.785 9~0.929 5,遗传距离的变化范围在0.073 1~0.241 0。其中JY种群和LCh种群间遗传一致度最高,遗传距离最近;而LuSh种群与LSh种群遗传距离最远,遗传一致度最低。总体看,6个连翘种群间的遗传距离较小,遗传相似度较高。根据UPGMA法进行连翘种群遗传距离聚类分析,图2表明:6个种群共聚成3组:第一组,LCh和JY,分属于伏牛山和太行山南麓;第二组,ShX和LZh,分属于崤山和太行山;第三组,LuSh和LSh,地理位置相距较远,分属于熊耳山和伏牛山东麓。从地理位置分析,6个连翘种群间彼此间隔,每组中遗传距离较近的两种群并未相邻,说明连翘种群间遗传距离与地理位置没有相关性。

表4 连翘6个种群群的Nei遗传一致度和遗传距离†Table 4 Nei’s genetic identity and genetic distance of 6 natural F. suspensa populations

图2 基于Nei’s 遗传距离的连翘种群UPGMA聚类Fig. 2 UPGMA dendrogram for 6 natural F. suspensa populations based on Nei’s genetic distance
基于样品间Nei遗传相似性系数进行UPGMA聚类分析,从图3可知,以阈值0.628为界,可将33份样本分为2个类群,第二大类群可进一步分为5个亚群;而第一大类群仅含ShX4一份样本。有些种群内的连翘个体间遗传相似性很高,如LZh1、LZh2以及LCh4、LCh8等;但各连翘种群彼此交叉,即便在同一样地采样,遗传一致度也会相差较远,如ShX1和ShX4等。这从另一方面证明了不同种群间基因交流多,个体间遗传距离与地理距离无相关性。

图3 基于Nei’s 遗传相似性系数的连翘33株个体的UPGMA聚类分析树状图Fig. 3 UPGMA dendrogram for 33 individuals of F. suspense based on Nei’s genetic similarity coeffcient
3 讨 论
3.1 连翘种群的遗传变异
在物种水平上,6个连翘种群存在着高度的遗传多样性,从表3可以看出,其多态位点百分率(PPL)达到94.38%,说明连翘种群具有较强的适应能力。这与野生连翘抗逆性强,喜光,耐寒,耐干旱、瘠薄,不择土壤等生长习性相一致。由此可见,一个物种或种群遗传多样性水平往往与对环境变化的适应能力成正相关。
用H和I估算6个种群的遗传多样性虽略有差异,但大小顺序一致,都以种群LSh最低。种群LSh地处伏牛山东南边缘的尧山自然风景区,距最近村庄约2 km,受人为干扰严重。在野外调查中发现,距村庄较近的种群常常受到村民采摘连翘果实、砍伐。人为干扰可能会造成种群变小,易发生遗传漂移,使其遗传多样性丧失,形成种群LSh遗传多样性较低的情况。
植物种群自身的遗传基础、繁育体系以及其进化历程,还有自然选择、遗传飘移和基因流等因素都会影响其遗传结构[15]。连翘是多年生灌木,虫媒传粉,而且先花后叶,花量大,是早春重要的蜜源植物,可以获得较多的传粉者,使连翘个体间基因流动较为频繁,提供了基因重组的机会;野生连翘具有无性繁殖和实生繁殖两种繁育方式,确保了种群遗传的稳定性。虽然连翘种群的遗传变异主要存在于种群内,但种群间的遗传变异占了总遗传变异的27.43%。河南连翘分布的地区范围较广阔,分属几个不同的山系,从而造成种群所处生境的温度、水分、光照、土壤等生态因子发生变化,不同种群承受的生态压力导致种群间发生了遗传分化。
3.2 连翘种群的遗传多样性与形态
形态多样性是遗传多样性的表现形式之一。形态学上差异的本质是分子水平上的差异。连翘的遗传多样性与形态性状表现出极高的多样性相一致:植株高矮多样;叶有单叶或一部分形成羽状三出复叶;叶形有卵形、宽卵形或椭圆状卵形;蒴果卵球形,有长果型与短果型之分;另外果实的色泽、果实外表皮疣点的大小、数量以及果皮的厚薄都有区别。
3.3 遗传距离与地理距离
对本次试验连翘种群间的遗传距离和地理距离进行相关性分析,二者之间无相关性。其结果与某些研究结果一致,如李晓东[16]对水杉Metasequoia glyptostroboides、穆立蔷[17]对紫椴Tilia amurensis种群的研究结果均显示遗传距离与地理距离间不存在显著的相关性。但也有相反结论,宾晓芸[18]对金花茶Camellia nitidissima的研究和李群[19]对珍稀濒危植物延龄草Trillium tschonoskii的分析,认为遗传距离和地理距离之间具有一定的相关性。此外还有学者认为遗传距离与地理尺度有关[20-21],大尺度上遗传距离与地理距离相关,而小范围内,如在同一国家的种群间二者无相关性。本试验样本取材虽然地跨河南省4大主要山系,但都属于秦岭山脉东段支脉,因此,本研究中遗传距离与地理距离无相关性可能是由于取样范围较小所致。
本试验首次利用ISSR分子标记技术对河南不同分布区的连翘进行遗传多样性研究,表明连翘种群间存在丰富的遗传多样性,从分子水平揭示了连翘种质广泛而复杂的遗传背景。连翘种群内遗传变异占总变异的72.57%,是变异的主要成分。UPGMA聚类分析结果显示出河南连翘种群间遗传距离与地理距离没有相关性。在育种中可以依据遗传多样性分析和聚类结果进行亲本选择,而不必全部选择,避免了人力物力资源的浪费。本试验为连翘资源可持续性开发利用提供一定的科学依据。
[1] 中国科学院植物研究所.中国高等植物图鉴(第三册) [M].北京:科学出版社, 1983.347.
[2] 白顺江,纪殿荣,黄大庄.树木识别与应用 [M].北京:农村读物出版社,2004.288.
[3] 田朝阳,田国行. 河南野生观赏植物志 [M]. 北京: 科学出版社, 2010. 254.
[4] Zietkiewicz E, Rafalski A, Labuda D. Genome fingerprinting by simple sequence repeat (SSR) –anchored polymerase chain reaction amplifcation [J]. Genomics, 1994, 20: 176-183.
[5] Reddy M P, Sarla N, Siddiq E A. Inter simple sequence repeat(ISSR) polymorphism and its application in plant breeding [J].Euphytica, 2002, 128: 9-17.
[6] Santiago M, Juan P M, Jesus M O. Inter-simple sequence repeats PCR for characterization of closely related grapevine germplasm[J]. Euphytica, 1998, 101: 117-125.
[7] Ratnaparkhe M B, Tekeoglu M, Muehlbauer F J. Inter simple sequence repeat (ISSR) polymorphism are useful for finding markers associated with disease resistance gene clusters [J].Theor Appl Genet, 1998, 97: 515-519.
[8] 张党权, 田 华, 谢耀坚, 等. 桉树4个种遗传多样性的ISSR分析 [J]. 中南林业科技大学学报, 2010, 30 (1): 12-17.
[9] 张党权, 田 华, 谢耀坚, 等. 细叶桉遗传多样性的ISSR分析 [J]. 中南林业科技大学学报, 2009, 29 (5): 91-94.
[10] 闫 女, 王 丹, 高亚卉, 等. 七里峪不同海拔茶条槭种群的遗传多样性 [J]. 林业科学, 2010, 46 (10): 50-55.
[11] 刘国彬, 龚榜初, 赖俊声, 等. 锥栗农家品种的遗传多样性及亲缘关系分析 [J]. 林业科学研究, 2011, 24 (6): 707-712.
[12] 胡绍庆, 邱英雄, 吴光洪, 等. 桂花品种的ISSR-PCR分析[J]. 南京林业大学学报·(自然科学版), 2004, 28 (增刊) (9):71-75.
[13] 原勤勤, 文亚峰, 刘 儒, 等. 枣优良品种亲缘关系的ISSR分析 [J]. 经济林研究, 2012, 30(1): 56-61.
[14] 刘海龙, 马锦林, 张日清, 等. 11份山茶属植物亲缘关系的ISSR分析 [J]. 经济林研究, 2012, 30(4): 87-90.
[15] Ishihama F, Ueno S, Tsumura Y, et al. Gene flow and inbreeding depression inferred from fine-scale genetic structure in an endangered heterostylous perennial, Primula sieboldii [J].Molecular Ecology, 2005, 14(4): 983-990.
[16] 李晓东, 黄宏文, 李建强. 孑遗植物水杉的遗传多样性研究[J]. 生物多样性, 2003, 11 (2): 100-108.
[17] 穆立蔷, 刘赢男. 不同地理分布区紫椴种群的遗传多样性变化 [J]. 植物生态学报, 2007,31(6): 1190-1198.
[18] 宾晓芸, 唐绍清, 周俊亚, 等. 金花茶遗传多样性的ISSR分析 [J]. 武汉植物学研究, 2005, 23(1): 20-26.
[19] 李 群, 肖 猛, 郭 亮, 等. 四川省珍稀濒危植物延龄草遗传多样性分析 [J].北京林业大学学报, 2005, 27 (4): 1-6.
[20] 李 珊, 钱增强, 蔡宇良, 等. 金钱槭和云南金钱槭遗传多样性比较研究 [J]. 植物生态学报, 2005, 29(5): 785-792.
[21] 简曙光, 唐 恬, 张志红,等. 中国银叶树种群及其受威胁原因 [J]. 中山大学学报·自然科学版, 2004, 43(增刊)(6): 91-96.
Genetic diversity study of Forsythia suspensa populations by ISSR in Henan
TANG Zheng-hui1,2, ZHU Ya-jun1, TAN Xiao-feng2, TAN Yun-de1
(1. Henan Academy of Forestry, Zhengzhou 450008, Henan, China; 2. Central South University of Forestry & Technology, Changsha 410004, Hunan, China)
By using inter simple sequence repeat (ISSR) marker technique, the genetic diversity of six natural populations of Forsythia suspensa growing in different regions in Henan province were investigated. The results show that the Forsythia suspense species have higher genetic diversity. 89 bands were amplifed from 6 natural populations by 12 reliable primers. The amplifcation results were analyzed by the software POPGENE 32. At the species level, the percentage of polymorphic loci was 94.38%; Nei’s gene diversity index(H) was 0.3265, Shannon information index (I) was 0.4901; the genetic differentiation coeffcient was 0.2743 which indicated 72.57% of genetic variation existed within populations. A dendrogram showed that the genetic relationship was constructed through an unweighted pair group method (UPGMA) based on the genetic identity and 33 F. suspensa materials were clustered into 2 main groups and 6 subgroups. It was found that the genetic distance and geographic distance were un-correlated, the genetic variation pattern of F. suspense in Henan might be ascribed to heterogeneous habitats and biological characteristic of this species.
Forsythia suspensa; genetic diversity; ISSR; cluster analysis; Henan province
S718.46;Q 949.776.2;Q347
A
1673-923X(2013)08-0032-06
2013-01-21
国家林业公益性行业科研专项(200904024);河南省博士后科研资助 (2011044)
汤正辉 (1973-),男,河南开封市人,博士,主要从事森林培育方面的研究;E-mail: zhenghuitang321@126.com
谭晓风 (1956-),男,湖南茶陵人。博士,教授,博士生导师,主要从事经济林栽培育种与生物技术方面的研究
[本文编校:文凤鸣]

