牦牛及杂种后代犏牛的TSPY基因克隆分析
张利亚,陈智华,钟金城,姜雪鸥,李 娜
(西南民族大学 动物遗传育种学国家民委-教育部重点实验室,四川 成都610041)
牦牛是青藏高原高寒草地牧区畜牧业经济中不可缺少的重要畜种。然而牦牛的生产性能远低于其他牛种,主要表现为生长速度慢,成熟期晚,产乳、产肉量低等。为了提高牦牛的生产性能,研究人员对牦牛和普通牛进行种间杂交,结果杂交一代犏牛产肉量、产奶量均有所提高,表现出显著的杂种优势[1-2]。但由于犏牛雄性不育,限制了杂种优势和牦牛遗传资源的充分利用。
犏牛雄性不育的最终表现是精子发生过程受阻,即公犏牛无法完成减数分裂过程导致不能产生精子。近年来的研究结果显示,Y染色体特异基因在动物精子形成过程中起着重要作用[3-4]。TSPY基因是近年来发现的与精子发生相关的基因,由于该基因在人睾丸中特异性表达,将其命名为TSPY(testis-specific protein Y encoded)[5-6]。TSPY基因是一个多拷贝的重复基因,多数拷贝位于Yp11.2的4A区,少数位于Yq11.23,每个拷贝被插入到DYZ5的一个20 kb串联重复基因簇中,Y染色体长臂和短臂上大小基因簇共存[7-8]。TSPY蛋白是 SET/NAP结构域中的成员,SET/NAP结构域参与细胞周期的调节和细胞分化[9-10]。在人体中,TSPY蛋白结合有丝分裂和减数分裂过程中的细胞周期蛋白B,提高活化的细胞周期蛋白B-CDK1 酶的活性,加快细胞周期G2/M期的转换[11-14],此外小鼠和人的TSPY蛋白可以结合精子细胞细胞质中的组蛋白,作为组蛋白的分子伴侣,在精子发生过程中起着重要作用[15-16]。
Verkaar等[7]和宋大伟[18]克隆了牦牛TSPY基因的部分序列,但目前尚未见牦牛和犏牛TSPY基因编码区全序列的相关报道。鉴于此,本试验采用了PCR扩增、克隆测序获得了牦牛、犏牛TSPY基因编码区全序列,并利用生物信息学方法分析了TSPY基因的序列、结构及与其他物种TSPY基因间的进化关系,以期为进一步研究TSPY基因的功能及其与犏牛雄性不育的关系提供依据。
1 材料与方法
1.1 试验动物及组织样品采集
从四川省红原县龙日种畜场随机选取健康成年牦牛7头,采集耳组织,75%乙醇保存带回实验室,存于-20 ℃的冰箱中备用;从青海省河北乡金科村选取健康犏牛7头,采集睾丸组织,置于液氮中速冻,保存备用。
1.2 主要试剂
基因组提取试剂盒、胶回收试剂盒购于AxyGENE 公司,2×Long Taq PCR MasterMix、DNA Marker 2000购自Tiangen公司,IPTG、X-gal、氨卞青霉素购自大连宝生物(TakaRa)工程有限公司。
1.3 菌株和质粒载体
大肠杆菌E.coliDH5α购于Tiangen公司,pMD19-T克隆载体购自大连宝生物(TakaRa)工程有限公司。
1.4 基因组DNA的提取和检测
采用动物组织基因组DNA提取试剂盒提取牦牛和犏牛基因组DNA。用1.5%的琼脂糖凝胶电泳和紫外分光光度计双重检测 DNA的纯度和浓度,-20 ℃保存备用。
1.5 引物设计、合成
参考Genbank中公布的普通牛TSPY基因DNA序列(登录号为AC_000165.1),应用引物设计软件primer premier 5.0设计两对引物(表1)。
1.6 TSPY基因的PCR扩增
PCR反应在25 μL体系中进行:模板DNA 1 μL,上、下游引物(10 pmol· μL-1)各1 μL,2×Long Taq PCR MasterMix 12.5 μL,ddH2O 9.5 μL。
引物1的PCR扩增按以下程序进行:95 ℃预变性4 min;95 ℃变性30 s,64.5 ℃退火30 s,72 ℃延伸40 s,40个循环;最后72 ℃延伸8 min;4 ℃保存。引物2的PCR扩增按以下程序进行:95 ℃预变性4 min;95 ℃变性30 s,67 ℃退火30 s,72 ℃延伸40 s,35个循环;最后72 ℃延伸8 min;4 ℃保存。产物均用1.5%琼脂糖凝胶电泳进行检测。
1.7 扩增产物的克隆与测序
对牦牛和犏牛TSPY基因PCR产物进行胶回收纯化,回收的目的片段与 pMD19-T载体于16 ℃恒温金属浴中反应连接4 h;将连接产物转化到E.coliDH5α感受态宿主菌中培养;再将菌液涂布在含有X-Gal底物、IPTG诱导物和苄青霉素(Amp )的固体LB平板上,37 ℃培养12~16 h;利用蓝白斑遗传学筛选法筛选;挑取白色单一菌落,接种于LB液体培养基中,37 ℃振荡培养过夜。对鉴定为阳性的克隆进行测序,测序由上海英骏生物技术有限公司完成。

表1 TSPY基因扩增引物及扩增范围Table 1 Primers and the respective size of amplified DNA fragment of the TSPY gene
1.8 数据统计分析
使用DNAMAN 软件对犏牛、牦牛和普通牛的TSPY基因序列进行比对,在线生物软件CpG Island Searcher(http://www.cpgislands.com/)预测CpG岛,核苷酸序列的翻译使用在线物包(http://www.bio-soft.net/sms/),ExPASy Proteomics Server ( http://web.expasy.org/pr- otparam/ ) 在线软件预测氨基酸的理化性质(氨基酸的组成、相对分子生量、理论等电点、蛋白质的亲疏水性及稳定性等),SignalP4.0 程序(http://www.cbs.dtu.dk/services/SignalP)分析可能的信号肽,TMpred Server软件(http://ch.embnet.org/software/TMPRED_form.html)对蛋白进行跨膜螺旋区预测,NCBI conserved domains(http://www.ncbi.nlm.nih.gov/Structure/ cdd/wrpsb.cgi)在线软件分析TSPY蛋白的保守结构域,亚细胞定位预测使用PSORTII(http://psort.nibb.ac.jp/),PBIL NPS@(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_seccons.html)预测蛋白质的二级结构。
2 结果与分析
2.1 牦牛和犏牛TSPY基因的扩增、测序与分析
1.5%琼脂糖凝胶电泳检测发现引物1和2扩增效果良好(图1和图2),克隆测序后,1号扩增片段长度为1 098 bp,2号扩增长度为1 024 bp,与预期结果一致。对序列进行拼接,得到长度为954 bp的编码区序列,此序列包含6个外显子。DNAMAN比对发现牦牛与普通牛TSPY基因序列的一致性分别为98.95%,犏牛与牦牛、普通牛TSPY基因序列的一致性分别为98.95%、99.79%(核酸序列差异性如图2所示),而牦牛、犏牛与人、大鼠的相应序列一致性都仅在50%左右,说明TSPY基因在不同物种之间同源性较低,这与Xue[19]的研究结果相一致。
通过CpG岛在线分析软件搜索发现,牦牛和犏牛TSPY基因编码区序列有 1 个 CpG 岛,包含全部扩增序列1-951,长度为951 bp(图3和图4)。碱基组成分析发现,CpG 岛序列的GC含量分别为58.6%、60.5%,差异主要体现在第二个外显子的CpG 位点上。
2.2 TSPY蛋白的氨基酸序列分析与结构预测
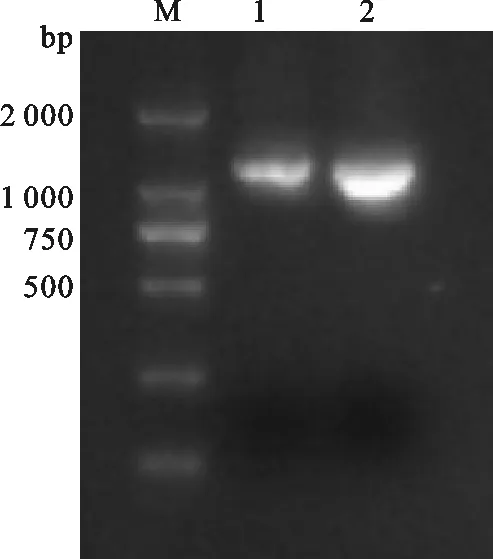
图1牦牛和犏牛TSPY基因的RT-PCR电泳图
M.DNA分子量标记;1.引物1 PCR 产物;2.引物2 PCR产物
Fig.1 RT-PCR of TSPY in yak and cattle-yak
M.DNA molecular weight marker;1.PCR product of primer 1;2.PCR product of primer 2
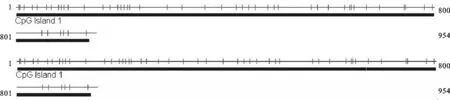
图2 牦牛和犏牛TSPY基因 CpG 岛的预测竖线表示 CpG 位点,下划线表示为 CpG 岛Fig. 2 The result of searching for CpG island of yak and cattle-yak TSPY gene Vertical lines stand for CpG site, underline stands for CpG island
2.2.1 序列分析 牦牛和犏牛TSPY基因均编码含有317个氨基酸残基的TSPY蛋白,使用EMBL-EBI在线比对工具发现牦牛与普通牛TSPY氨基酸序列的一致性为98%,犏牛与牦牛、普通牛TSPY氨基酸一致性分别为98%、99%。结合表2和表3,犏牛TSPY基因与牦牛和普通牛相比,第113位核酸发生了改变(T→C),导致第38位氨基酸发生变化(V→V→A)。
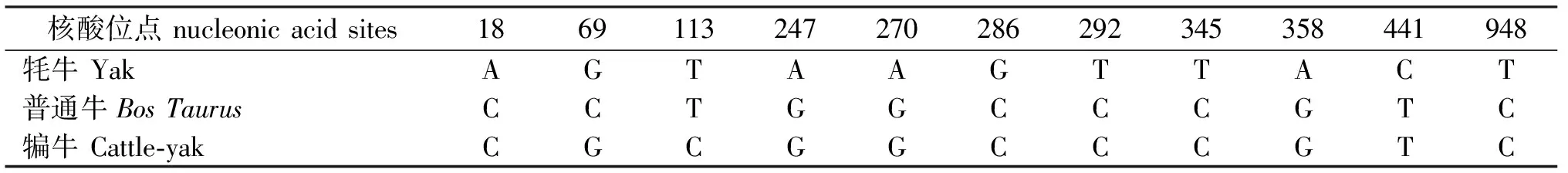
表2 牦牛、普通牛和犏牛TSPY核酸序列的差异性分析Table 2 Difference analysis of nucleonic acid sequences of TSPY from yak,bovine and cattle-yak
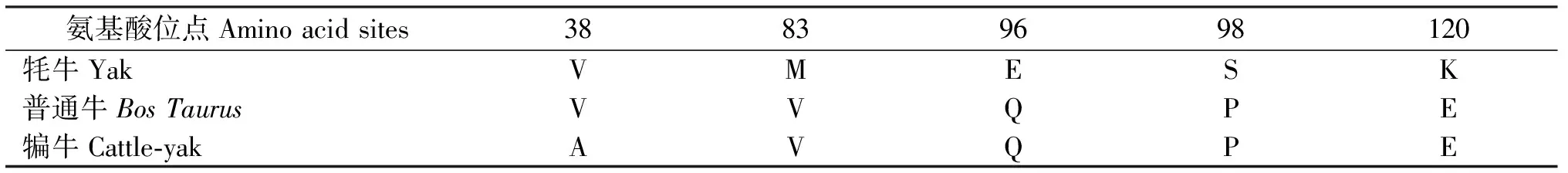
表3 牦牛、普通牛和犏牛TSPY氨基酸序列的差异性分析Table 3 Difference analysis of amino acid sequences of TSPY from yak,Bos Taurus and cattle-yak
2.2.2 理化性质分析 预测牦牛和犏牛的相对分子量分别为36 504 kDa、36 454 kDa,理论等电点分别为为5.39 、5.31。牦牛和犏牛TSPY蛋白的氨基酸序列中Glu的使用频率最高(均为11.4%),不含Pyl和Sec。预测蛋白正电荷18位,基酸残基数(Arg+Lys)分别为43个和42个,负电荷氨基酸残基数(Asp+Glu)均为53个。牦牛具有8个Ser磷酸化位点,犏牛为7个,Thr磷酸化位点和Tyr磷酸化位点二者均为3个和1个,Ser磷酸化位点的不同是由于二者第98位氨基酸序列的差异导致(表3),犏牛失去的催化激酶为周期蛋白依赖性激酶抑制因子,即CKI。蛋白的不稳定系数分别为66.54、64.28(不稳定系数>40为不稳定蛋白,不稳定系数<40为稳定蛋白),说明牦牛和犏牛TSPY蛋白较不稳定。蛋白质氨基酸残基亲疏水性综合GRAVY(grand average of hydropathicity)分别为-0.750、-0.751(疏水性指数> 0为疏水性蛋白,疏水性指数<0为亲水性蛋白),说明蛋白的亲水性较强。牦牛和犏牛TSPY蛋白均不存在信号肽。
2.2.3 亚细胞定位预测 亚细胞定位预测发现牦牛TSPY蛋白主要定位于细胞质(60.9%)和细胞核(30.4%)中,但在细胞骨架(4.3%)和线粒体(4.3%)中也有少量分布;犏牛TSPY蛋白也主要定位于细胞质(65.2%)和细胞核(30.4%)中,在线粒体中有少量分布(4.3%),但却未见细胞骨架中有分布。
2.2.4 保守结构域预测 对牦牛和犏牛TSPY蛋白进行保守结构域预测,发现它与普通牛、人、鼠一样都含有PTZ00007(153-310)超家族和NAP(分别为134-310、141-310)结构域(图5和图6),这两个结构域均与核小体组装有关,其中,NAP蛋白参与组蛋白在细胞核、核小体和染色质中流动[20],影响很多基因的转录,由此推测牦牛和犏牛TSPY可能也有基因的转录有关。
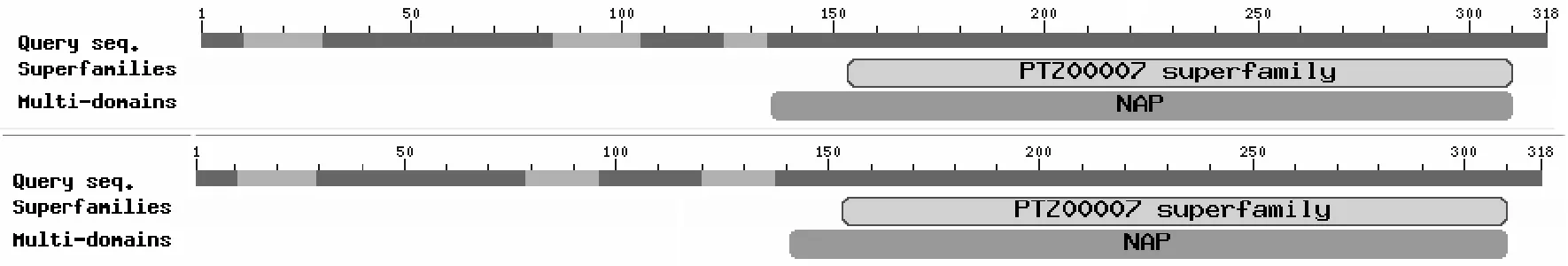
图3 牦牛和犏牛TSPY蛋白序列的保守结构域分析Fig.3 Conserved domains of TSPY protein in yak and cattle-yak
2.2.5 二级结构预测 使用PBIL上的在线软件预测TSPY蛋白的二级结构,表明该蛋白主要含有α-螺旋(h表示)、无规则卷曲(c表示)、β转角(t表示)和伸展链(e表示),其中主要为α-螺旋和无规则卷曲,其余含量相对较少(图4)。对犏牛、牦牛和普通牛的氨基酸序列差异进行分析,发现犏牛第38位氨基酸发生了突变,由V变为A(表3)。犏牛中突变的氨基酸引起了相邻蛋白质二级结构发生变化,由伸展链变为无规则卷曲。
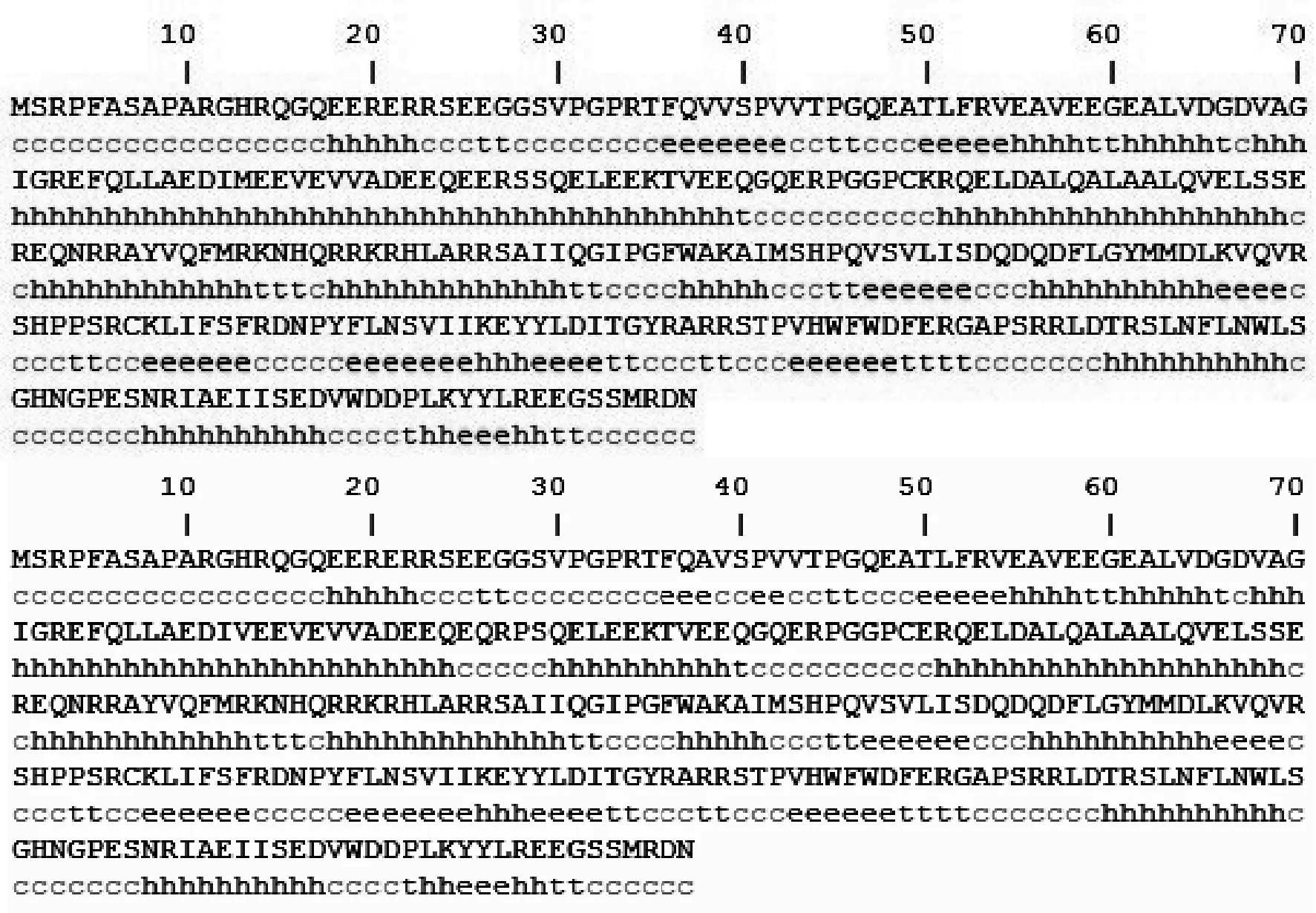
图4 牦牛和犏牛TSPY蛋白质二级结构预测 Fig.4 The second structure of TSPY protein in yak and cattle-yak
3 讨 论
犏牛雄性不育主要是由精子发生过程中减数分裂障碍所引起的。哺乳动物精母细胞减数分裂是精子发生过程中的关键步骤,参与减数分裂调控的基因发生缺失或突变容易引起无精子症和男性不育[21-24]。TSPY被认为为在雄性减数分裂过程中参与了精原细胞和初级精母细胞的调节[25-26]。Veronika Svacinova[28]对人类TSPY基因的研究发现,TSPY基因的突变与男性不育症患者的生育能力受损有关,在几乎一半的男性不育症患者中,该基因第36-42位氨基酸AGAGAGG或缺失或被CAT所替代。在本研究中,克隆了牦牛和犏牛TSPY基因序列。犏牛TSPY基因与牦牛和普通牛相比,第113位核酸位点发生了改变(T→C),导致第38位氨基酸发生变化(V→V→A),并引起蛋白质二级结构相邻构型的变化。更值得注意的是,该突变位点位于TSPY基因的第一外显子区域,而第一个外显子可能会影响精子发生过程[27],因此,作者认为该位点的突变可能与犏牛雄性不育有一定的关系,TSPY基因可能是犏牛雄性不育的候选基因,具体调控机制还需从蛋白质角度进一步验证。
人TSPY基因的多数研究表明,在男性不育症患者中,TSPY重复基因成为全基因组不稳定的热点区域,可导致TSPY可变剪接体表达,正常的基因不再表达,并且拷贝数也发生变化,基因重排[28-30]。Vodicka等[31]发现TSPY基因拷贝数量的增加与不育有关,而数量减少对表型的影响还不清楚,有待于进一步的研究。Giachini等[32]通过临床试验研究发现TSPY基因拷贝数的变化影响精子发生,并且认为低TSPY拷贝数成为雄性不育症的一个新的风险因素。在本试验中,预测到犏牛TSPY蛋白是不稳定蛋白。那么该蛋白是否与人一样,由于不稳定造成TSPY基因拷贝数的变化,进而导致犏牛雄性不育,还有待于进一步的研究。
牦牛和犏牛TSPY基因的编码区有1 个 CpG 岛,包含全部扩增序列,第一外显子区域与精子发生密切相关[27]。本试验中,TSPY基因第一个外显子含有容易被甲基化的CpG岛,在今后的研究中,可通过对牦牛、普通牛和犏牛TSPY基因外显子1的甲基化差异状况来探讨犏牛雄性不育的原因。
4 结 论
本研究首次克隆了牦牛和犏牛TSPY基因编码区序列,并分析其生物信息学,这为进一步研究牦牛和犏牛TSPY基因的功能和作用奠定了基础,为今后研究犏牛雄性不育提供了一定的理论依据。
参考文献:
[1] 钟金城.牦牛遗传与育种[M]. 四川成都:四川科学技术出版社, 1996.
[2] Chen Zhihua, Zhong Jincheng, Hu Ouming, et al. A comparative study on yak,ordinary cattle and their F1 species hybrids[C]∥Proceeding of the International Congress on Yak, 2004.
[3] Krzysztof Ginalski, Leszek Rychlewski, David Baker, et al. Protein structure prediction for the male-specific region of the human Y chromosome[J]. Proceeding of the National Academy of Science of the Untied State of America,2004,101: 2 305-2 310.
[4] 刘振山, 李齐发, 张庆波, 等. 犏牛及其亲本IGF2 基因mRNA表达水平、DNA甲基化状态差异分析[J]. 自然科学进展, 2009, 19(4): 380-385.
[5] Joachim Arnemann, Sibylle Jakubiczka, Sabine Thuring, et al. Cloning and sequence analysis of a human Y-chromosome-dericed,testicular cDNA TSPY[J]. Genomics, 1991, 11(1):108-114.
[6] Shane W Oram, Xing X Liu, Tin-Lap Lee, et al. TSPY potentiates cell proliferation and tumorigenesis by promoting cell cycle progression in HeLa and NIH3T3 cells[J]. BMC Cancer, 2006, 6: 154.
[7] Eberhard Manz, Frank Schnieders, Annette Muller Brechlin, et al. TSPY-related sequences represent a microheterogenous gene family organized as constitutive elements in DYZ5 tandem repeat units on the human Y chromosome[J]. Genomics,1993,17: 726-731.
[8] Werner Schempp, Andreas Binkele, Joachim Arnemann, et al. Comparative mapping of YRRM-and TSPY-related cosmids in man and hominoid apes[J]. Chromosome Research, 1995,3(4):227-234.
[9] Zhonglin Chai, Boris Sarcevic, Amanda Mawson, et al. SET-related cell division autoantigen-1 (CDA1) arrests cell growth[J]. The Journal of Biological Chemistry,2001,276:33 665-33 674.
[10] Nuria Canela, Aina Rodriguez-Vilarrupla, Josep Maria Estanyol, et al. The SET protein regulates G2/M transition by modulating cyclin B-cyclin-dependent kinase 1 activity[J]. The Journal of Biological Chemistry, 2003, 278(2): 1 158-1 164.
[11] Yun-Fai Chris Lau. Expression of a candidate gene for the gonadoblastoma locus in gonadoblastoma and testicular seminoma[J]. Cytogenetiic and Genome Reseach, 2000, 91(1-4): 160-164.
[12] Yun-Fai Chris Lau, Yunmin Li, Tatsuo kido. Gonadoblastoma locus and theTSPYgene on thehuman Y chromosome[J]. Birth Defects Research Part C: Embryo Today,2009,87(1): 114-122.
[13] Stephanie Schubert,Jorg Schmidtke. Transgenic Mouse Studies to Understand the Regulation,Experssion and Function of the Testis-Specific Protein Y-Encoded (TSPY) Gene[J]. Genes, 2010(1):244-262.
[14] Yunmin Li,Yun-Fai Chris Lau. TSPY and its X-encoded homologue interact with cyclin B but exert contrasting functions on cyclin-dependent kinase 1 activities[J]. Oncogene, 2008,27:6 141-6 150.
[15] Cristobal Mezquita. Chromatin composition,structure and function in spermatogenesis[J]. Revisi Revisiones sobre biologia cellular, 1985, 5:1-124.
[16] Irena Ivanovska ,Terry L. Orr-Weaver.Histone modifications and the chromatin scaffold for meiotic chromosome architecture[J]. Cell Cycle, 2006, (18): 2 064-2 071.
[17] Edward L C Verkaar, Carla Zijlstra, Esther M.Vant Veld, et al. Organization and concerted evolution of the ampliconic Y-chromosomalTSPYgenes from cattle[J]. Genomics, 2004, 84(3),468-474.
[18] 宋大伟. 物种的起源与系统发育分析[D]. 南京: 南京农业大学, 2008: 43-50.
[19] Yali Xue,Chris Tyler-Smith. An Exceptional Gene:Evolution of theTSPYGene Family in Humans and Other Great Apes[J]. Genes, 2011(2):36-47.
[20] Tatsco Kido,Yun-Fai Chris Lau. A Cre gene directed by a human TSPY promoter is specific for germ cells and neurons[J]. Genesis, 2005, 42(4):263-275.
[21] Yung-Ming Lin, Ying-Hung Lin, Yen-Ni Teng, et al. Gene-based screening for Y chromosome deletions in taiwanses men presenting with spermatogenic failure[J]. Fertility and Sterility, 2002, 77(5): 897-903.
[22] Yen-Ni Teng, Yung-Ming Lin, Ying-Hung Lin, et al. Association of a single-nucleotide polymorphism of the deleted-in-azoospermia-like gene with susceptibility to spermatogenic failure[J]. The Journal of Clinical Endocrinology Metabolism, 2002, 87(11): 5 258-5 264.
[23] C Marc Luetjens, Eugene Y Xu, Renee A Rejo Pera, et al. Association of meiotic arrest with lack of BOULE protein expression in infertile men[J]. The Journal of Clinical Endocrinology Metabolism, 2004, 89(4):1 926-1 933.
[24] Pao-Lin Kuo, Shan-Tair Wang, Yung-Ming Lin, et al. Expression profiles of the DAZ gene family in human testis with and without tspermatogenic failure[J]. Fertility and Sterility,2004, 81(4):1034-1040.
[25] Frank Schnieders, Thilo Dork, Joachim Arnemann, et al. Testis-specific protein,Y-encoded (TSPY) expression in testicular tissues[J]. Human Molecular Genetics, 1996, 11: 1 801-1 807.
[26] Yun-Fai Chris Lau. Gonadoblastoma testicular and prostate cancers,and the TSPY gene[J]. American Journal of Human Genetics, 1999, 64(4): 921-927.
[27] Veronika Svacinova, Radek Vodicka, Radek Vrtel, et al. Sequence recombination in exon 1 of the TSPY gene in men with impaired fertility[J]. Biomedical Papers, 2011, 155(3): 287-298.
[28] Sapna Vijayakumar, Devon C Hall, Xavier T Reveles, et al. Detection of recurrent copy number loss at Yp11.2 involvingTSPYgene cluster in prostate cancer using array-based comparative genomic hybridization[J]. Cancer Research, 2006, 66: 4 055-4 064.
[29] Sjoerd Repping, Saskia K M van Daalen, Laura G Brown, et al. High mutation rates have driven extensive structural polymorphism among human Y chromosomes[J]. Nature Genetics, 2006,38(4):463-467.
[30] Chris Tyler-Smith. An evolutionary perspective on Y-chromosomal variation and male inferiti-lity[J]. International Journal of Andrology, 2008, 31: 376-382.
[31] Radek Vodicka, Radek Vrtel, Ladislav Dusek, et al.TSPYgene copy number as a potential new risk factor for male infertility[J]. Reprod Biomed Online, 2007, 14(5): 579-587.
[32] Claudia Giachini, Francesca nuti, Daniel J Turner, et al.TSPY1 Copy Number Variation Influences Spermatogenesis and Shows Differences among Y Lineages[J]. The Journal of Clinical Endocrinology Metabolism, 2009, 94(10): 4 016-4 022.

