比较图元向量和点的聚类系数对差异网络的研究
肖碧玉,李先彬,沈良忠,刘文斌
(温州大学物理与电子信息工程学院,浙江温州,325035)
系统生物研究表明:生命体的生长、发育、衰老过程以及疾病的发生与基因之间相互作用的变化密切相关。随着以微阵列技术为代表的各种高通量技术的飞速发展,人们可以同时观察到一个细胞中成千上万个基因的活动状况。如何利用这些数据挖掘出其中潜在变化,对于揭示衰老及疾病的发生发展,具有重要的生物学意义。因此,基于基因表达谱数据的差异分析成为系统生物学的一个研究热点。基因差异表达的分析大致可以分为以下三个层次:
(1)生物学中,有些关键基因表达水平的高低往往是导致疾病产生的关键因素。因此,最早的差异分析主要集中在单个基因表达水平的变化,如Jacob等发现小鼠衰老期各组织的基因表达水平变化存在较大的差异,可归类为神经组织、血管组织、类固醇反应组织三种衰老类型[1]
(2)基因之间往往是通过相互作用来实现某种功能,因而挖掘基因作用关系的变化能够揭示基因(簇)与功能之间的关系。Remondini通过比对基因时间序列网络的度的分布,发现c-myc致癌基因的活性与基因的网络结构有密切关系[2]。Voy等通过研究网络边的变化,确定小鼠受辐射影响的基因簇[3]。Oldham等比对人类和黑猩猩差异网络的拓扑重叠,挖掘与生物驱动进化有关的基因集合[4]。Omar Odiba等在差异网络分析中将网络中介性及中心性结合起来,挖掘出了生物标记(Biomarker)[5]。Zhang基于条件概率提出差异相关子网络(DDN),检测两个差异转录网络的拓扑变化显著性[6]。Southworth等构建基因带权差异网络,挖掘与小鼠衰老密切相关的基因模块[7]。
(3)虽然基因模块往往与某种功能相关,但是这种功能往往需要其他基因模块的协调与配合,基因模块之间关系的变化也往往与功能的变化密切相关。Tesson等提出了DiffCoEx算法,可以同时挖掘基因模块及基因模块间的变化[8]。此外,在差异表达分析方面,Fang研究了二组样本中表达水平高低的变化,提出了三种基因表达谱差异模式及其关系,这种模式特征的研究对于差异模式的挖掘具有重要的指导意义[9-10]在生物网络中,节点往往通过与局部结构作用实现功能,其与局部结构的关系发生变化,可能会影响其功能。因此,如何理解和刻画网络节点的局部结构特征及变化,对于认识生物系统的结构和功能的关系具有非常重要的意义。点的聚类系数是比较经典的网络节点局部结构描述测度,人们利用它做出了许多有意义的结果[16]。2003年,Przulj提出利用图元及图元向量刻画网络中节点邻域关系,并且基于它们研究了生物网络与随机网络的拓扑结构差异[11-12]、癌症相关基因[13]、生物网络的进化树[14]等。本文利用仿真实验的方法分析和比较图元向量和点的聚类系数两个测度的性能,并且分别利用它们设计算法挖掘差异网络中模块化变化的节点簇,最后利用AGEMAP数据库中小鼠的基因表达数据进行实验,分析比较基于图元向量和基于点的聚类系数挖掘小鼠基因时序差异网络的差异模块与小鼠衰老的关系。
1 基本概念
点的聚类系数定义为:
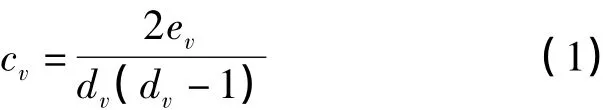
其中dv表示节点v的度,这dv个节点之间实际存在的边数目ev表示节点v及其相邻节点之间的边数。图元就是包含一定节点的非同构子图,其非同构位置唯一标号构成的向量称为图元向量。图1列出了包含2、3、4个节点的图元和其15维图元向量。由于随着图元节点规模的增大,图元向量的规模及计算的复杂度将指数级增加,本文采用图1中的15维的图元向量来研究差异网络。此外,由于标号大的图元向量可能包括标号小的维度信息(如标号14与3,7与2),Milenkovi按照标号大的图元向量包括标号小的图元向量的个数对每一维图元向量定义一个权值 wk(k=0,…,14)。

图1 图元结构Fig.1 Graphlets and orbits
给定两个差异网络 G(V,E)和 G(V,E'),具有相同的节点集不同边集,我们定义节点v∈V的图元向量差异度为:
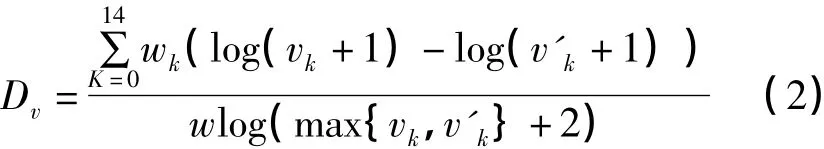
其中vk和 v′k分别表示节点 v在两个差异网络G(V,E)和 G(V,E')中的第 k维图元向量。wk表示图元向量第k维的图元向量的权值,度是反映网络节点邻接结构的一个非常重要的指标,结合图元向量和点的聚类系数两个测度,我们定义节点v∈V的两种局部结构差异度为:
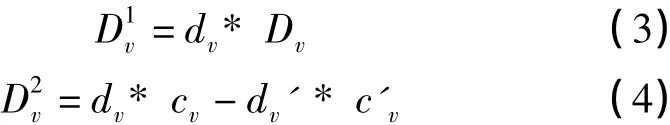
其中dv,d′v分别表示节点v在两个差异网络G(V,E)和 G(V,E')中的度,cv,c′v分别表示节点 v在两个差异网络 G(V,E)和 G(V,E')中的点的聚类系数。直观上,网络中同一个簇的节点变化,应该具有一定的相似性,即对于一个簇中的节点u,v∈V',他们的局部结构差异度可能基本相当。因此,我们给出衡量节点局部结构变化差距的定义如下:

2 比较图元向量和点的聚类系数的性能
为了定量证明图元向量和点的聚类系数两个测度的性能,本文采用计算机产生随机网络作仿真数据,该实验方法已被广泛采用[15-16]本仿真实验产生的每一个随机网络,包含128个节点,每个节点的度p=16,这些点被划分成4个社团,每个点随机的与所在社团内部的pin点相连,与其它的pout=p-pin个点相连。显然,随着pout的增大,网络中的社团结构越来越模糊;反之,社团结构则越来越明显。一共产生了3组随机网络,分别对应 pout=1、5、8,每组共100个网络。为了验证比较图元向量和点的聚类系数的性能,我们对这每一组的100个网络随机加(减)10、20、30、40、50、60、70、80、90、100 条边进行分析。
日前,由于实验平台、实验技术等的限制,生物数据还存在一定的误差,这对差异信息的研究带来了一定的困难,所以,现在人们主要是对较明显的差异信息进行研究,而往往把较小的变化当做噪声处理。本实验以加/减少量10(1%)的边仿真噪声,以加/减较多的边100(10%)仿真差异变化,用噪声鲁棒性衡量测度容忍噪声的能力,也就是计算在有噪声干扰下测度度量时的变化量;用差异灵敏性度量测度发现差异变化的能力,也就是计算发生差异变化时测度发生的变化量。
为了分析和比较图元向量、点的聚类系数的噪声鲁棒性和差异灵敏度,我们分别对3类网络加(减)10条边和100条边,各节点的点的聚类系数和图元向量的变化量做统计(见图2)。图中右侧3个图中,3类网络中加(减)100条边时图元向量的变化量都高于加(减)10条边时的变化,图元向量测度能在容忍噪声的同时,有效提取差异信息,而图中左侧3类网络中点的聚类系数测度则不然。可见,相对点的聚类系数,图元向量的差异灵敏度和噪声鲁棒性比较好。

图2 比较点的聚类系数和图元向量的噪声鲁棒性、差异灵敏度Fig.2 The comparison of robustness and difference sensitivity of clustering coefficient and orbits under various noises
点的聚类系数(图元向量)在各类网络中的噪声鲁棒性、差异灵敏度是否相同呢?我们统计3类网络加(减)10条边、100条边时,各节点的点聚类系数、图元向量变化(见图3)。图3右侧两个图中,在加(减)10条边的噪声鲁棒性研究,和加(减)100条边的差异灵敏性分析,图元向量在3类网络(Pout=1、5、8)中都相似。从图3左侧两个图中不难看出,在加(减)10条边的噪声鲁棒性研究,和加(减)100条边的差异灵敏性分析,各节点的点聚类系数的变化,在Pout=1的网络的噪声鲁棒性最差、差灵敏度最好,在Pout=5的网络噪声鲁棒性次之、差灵敏度也次之,在Pout=8的网络噪声鲁棒性最好、差灵敏度最差,点聚类系数在3类网络(Pout=1、5、8)中的噪声鲁棒性和差异灵敏度不同。相对点的聚类系数,图元向量的适用性较强,应用范围较广。
我们统计 3 类网络加(减)边 10、20、30、40、50、60、70、80、90、100 条,局部结构变化的节点的数量(见图4)。分析图4不难发现,在3类网络中,各种加(减)边时,图元向量变化的节点几乎是整个网络128个节点,而点的聚类系数变化的节点则随加(减)边的数量的增加而增多,可见,相对点的聚类系数,图元向量更能细致反应节点局部结构的影响。
综上可得,相对点的聚类系数,图元向量具更好的噪声鲁棒性和差异灵敏度,有较强的适用能力,但是在时间复杂性方面较差。点的聚类系数和图元向量各具优缺点,我们可以根据需要酌情选择。
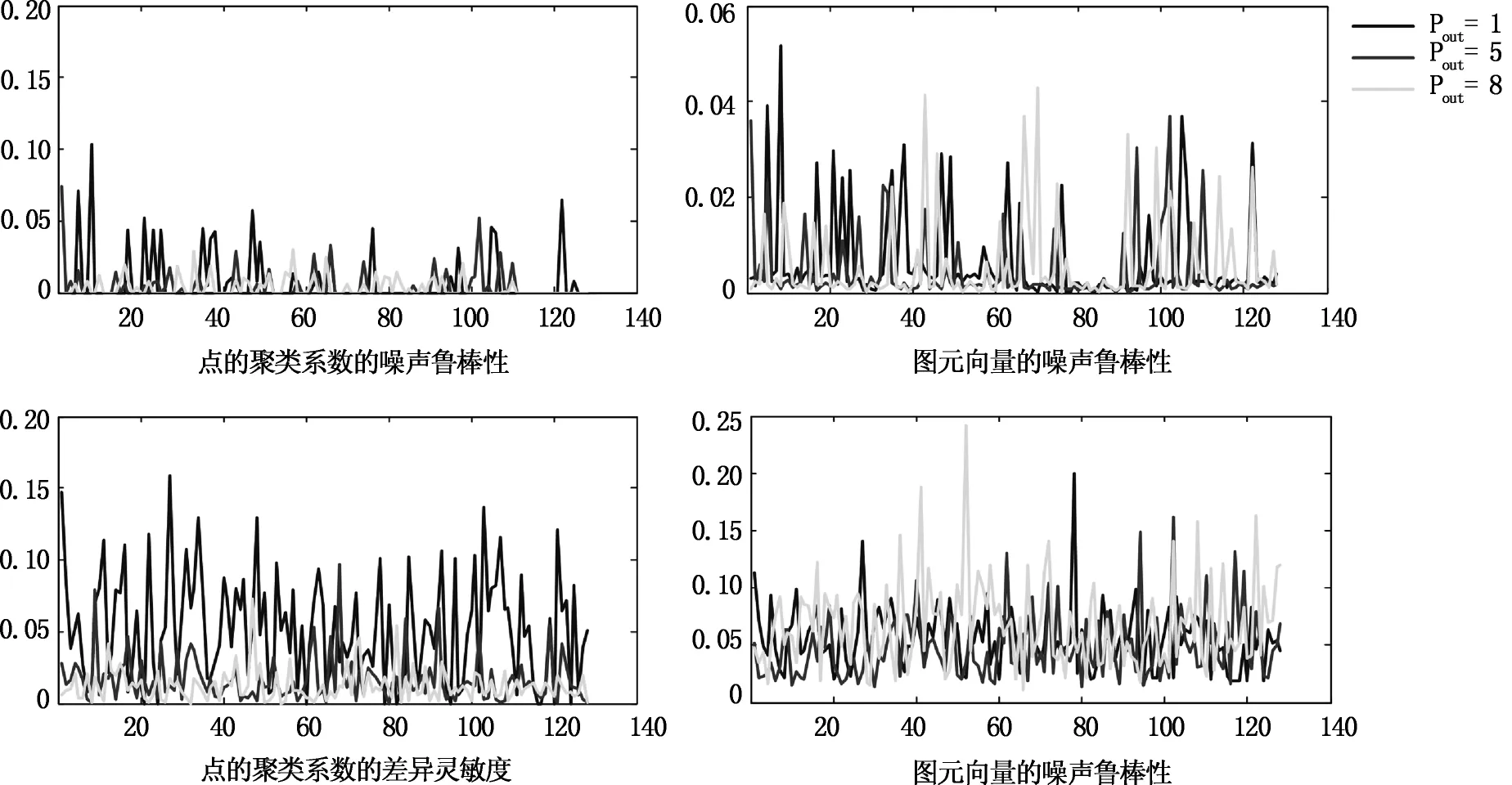
图3 比较点的聚类系数和图元向量的适用性Fig.3 The comparison of clustering coefficient and orbits by using

图4 比较点的聚类系数和图元向量变化的节点数Fig.4 The comparison of clustering coefficient and orbits with the number of nodes Changed adding or cutting
3 实验
3.1 材料
本文生物数据来自AGEMAP数据库(http://cmgm.stanford.edu/~ kimlab/aging_mouse/),其中包括 C57BL6小鼠16个组织的1、6、16、24个月的8 932个基因表达数据,由于其中4个组织数据存在较大噪声和不完整性,本文使用Adrenal Glands(1)、Cerebellum(2)、Cerebrum(3)、Eye(4)、Gonads(5)、Heart(6)、Hippocampus(7)、kidney(8)、Lung(9)、Muscle(10)、Spinal Cord(11)、Thymus(12))等 12 个组织的数据。通常认为16月至24月为小鼠的衰老期,我们利用这两个月基因表达数据研究差异簇与小鼠衰老的关系。共表达网络是一个无向图,每个节点表示一个基因,每条边表示两个基因的共表达关系。共表达关系通过pearson相关系数r确定

然后将Pearson系数r转化另一变量
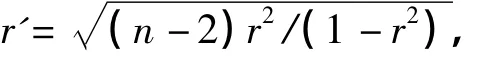
其中,n表示计算这两个基因相关系数时所用的数据点的个数。转化后的r'值服从自由度为n-2的t分布。如果两个基因之间的r'值大于设定的p-value对应的t分布表,则就在这两个基因之间加一条边;否则在这两个基因之间就不加边。除Eye组织的p-value取值为1E-04,Spinal Cord组织为1E-07、其他各组织均取5E-06构建网络。David数据库可以对小鼠基因簇的功能进行分析,如果一个基因簇有50%以上的基因显著的共享一个或多个GO项,通常认为该基因簇具有生物学意义。本文GO项的P-value取0.05。Southworth等验证了401条与小鼠衰老相关的GO项[8],因此,同其分析一个基因簇显著富集的GO项就可以判断其是否与衰老有关。下面我们通过图元向量和点的聚类系数二种测度,分别设计算法挖掘16和24月的基因表达数据中变化的基因簇,并对其功能进行分析。
3.2 挖掘差异基因簇
基于图元向量挖掘差异基因簇,首先,提取局部结构发生变化的差异基因;在16月(24月)基因共表达网络中,取Dv1大于等于5提取16月(24月)的变化的基因。然后,根据提取出的基因多少和D1v分布,利用Luv分别在16月和24月基因共表达网络中进行K-means聚类,最后,对聚类簇进行GO注释和分析。基于点的聚类系数挖掘差异基因簇方法类似。
3.3 实验结果分析
基于图元向量和点的聚类系数挖掘差异基因簇的实验结果如图5。从图中可以看出,基于图元向量和点的聚类系数挖掘的差异基因簇,几乎每个基因簇都显著的富集一个或多个GO项,并且大部分的基因簇都与衰老相关。二种方法挖掘聚类簇显著富集的GO项几乎相似,主要相似GO项见表1,这些功能的差异与衰老具有密切关系。有些基因簇按照Southworth验证的401条衰老GO项与衰老无关,这是由于目前有关衰老相关的GO项还不完善,这方面的研究工作还在不断进行中,如Chunxiao Fu[17]、Jamie L.Barger[18]等也验证其它一些与小鼠衰老有关的GO项。

表1 二种方法挖掘的相似GO项Table 1 Similar GO terms from two algorithms
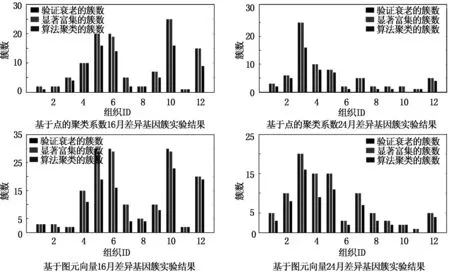
图5 基于图元向量和点的聚类系数的实验结果Fig.5 Experimental results of the two algorithms based on Graphlet and Clustering Coefficient
二种方法取相同的阈值提取差异基因,同组织中基于图元向量提取的差异基因和基于点的聚类系数提取的差异基因大部分相同,且基于图元向量提取的差异基因相对较多(见表2)。
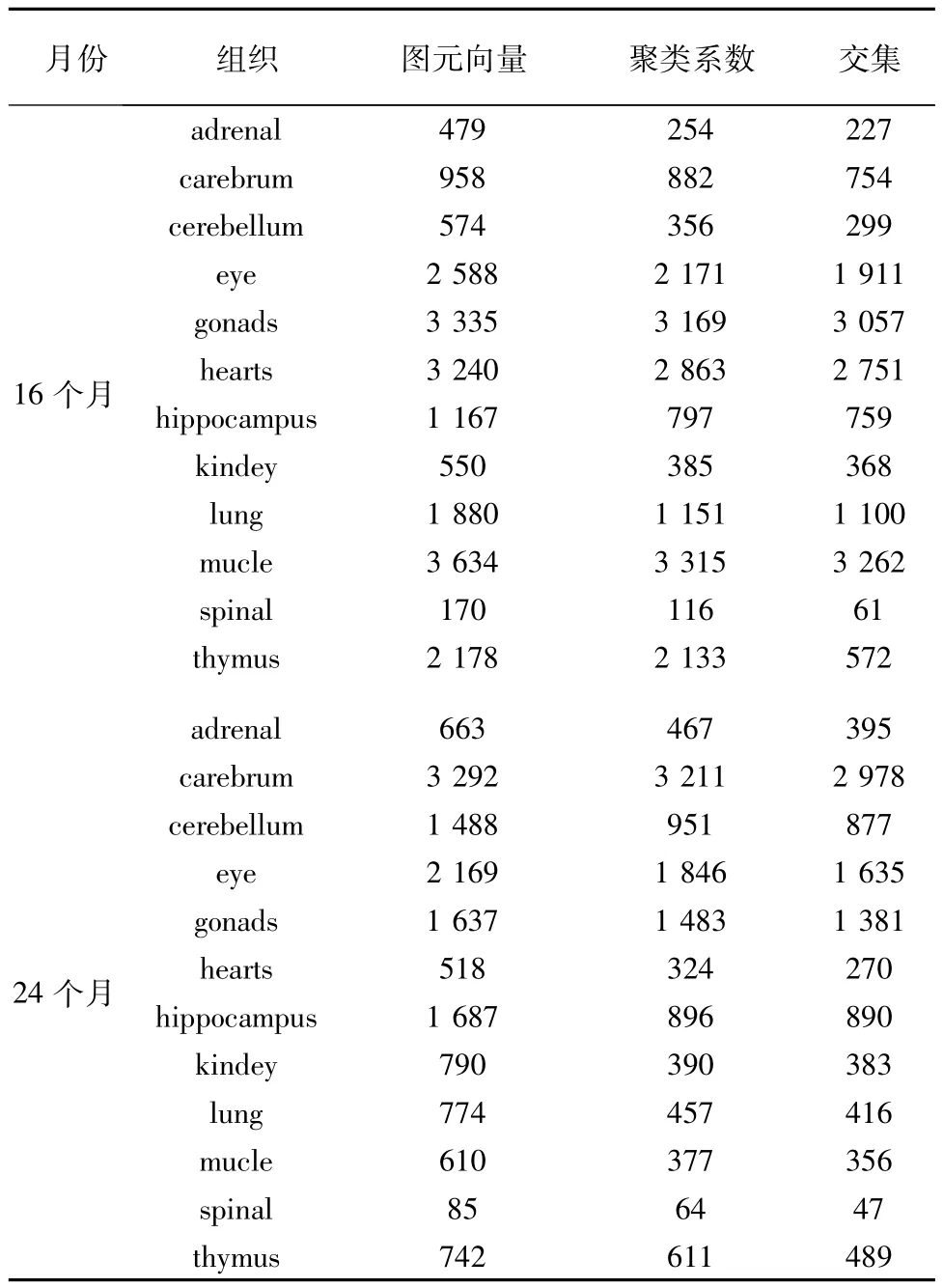
表2 比较各组织基于图元向量和点聚类系数提取的基因数Table 2 Compareing the number of genes mined by the two algorithms
这与前面图元向量和点的聚类系数的仿真实验结论一致,图元向量更能反映局部结构的信息。相对点的聚类系数,图元向量的噪声鲁棒性和差异灵敏度较好,而基于图元向量和点的聚类系数的实验却得到了相似较好的实验结果,从而可知AGEMAP数据库提供的数据质量较高,而这点在其他的实验中也得到证明[1,7]。此外,我们发现基于图元向量和点的聚类系数挖掘的有些基因簇在David数据库中不能注释到与衰老相关的 GO项,但是在AGEMAP数据库中发现它们具有这些功能。如Thymus组织利用基于图元向量挖掘的一个基因簇(其中包括196个基因),在David数据库中注释衰老GO:0005488~binding项的富集度为52.6%,显著性为0.027。此簇中的Mm.20935基因不含GO:0005488~binding项功能,但在AGEMAP提供的数据中则显示此组织基因 Mm.20935具有 GO:0005488~binding功能。另外,Thymus组织利用基于点的聚类系数挖掘的一个基因簇(其中包括90个基因)在David数据库中注释衰老GO:0005488~binding项的富集度为60%,显著性为0.013。此簇中的 Mm.383175基因不含 GO:0005488~binding项功能,但在AGEMAP提供的数据中则显示此组织基因Mm.383175具有GO:0005488~binding功能。因此,本文那些与衰老无关的基因(簇)存在潜在的小鼠衰老研究价值。
4 小结
生物分子往往是通过与局部结构相互作用发挥功能,其与局部结构关系发生变化可能引起其功能变化,所以基于局部结构测度进行差异研究对于认识生命的进化、发育、衰老过程及疾病的产生等生物问题具有重要的意义。本文首先分析和比较了图元向量和点的聚类系数两个测度的性能,并基于图元向量和点的聚类系数分别设计了挖掘模块化变化簇的算法。利用AGEMAP数据库中小鼠12个组织的基因表达数据进行实验,本文设计的二个算法挖掘的差异簇都显著的富集于一些GO条目,而且其中大部分都与衰老有关。
图元向量和点的聚类系数作为刻画网络局部拓扑结构的二种参数,各具优缺点,在差异分析中具有广阔的应用前景。本文是基于局部结构变化挖掘模块变化基因簇,而基于模块基因簇识别变化模块基因簇将是本文后续工作。
References)
[1] Jacob M.Zahn,Suresh Poosala,Art.B Owen.AGEM AP:A Gene Expression Database for Aging in Mice[J].PLOS Genetics,2007,3(11):e201.
[2] Remondini D,O,Connell B,Intrator N,Sedivy JM,Neretti N,Castellani GC,Cooper LN.Targeting c-Myc-activated genes with a correlation method:Detection of global changes in large gene expression network dynamics[J].Proc Natl Acad Sci U S A,2005,102(19):6902 -6906.
[3] Voy BH,Scharff JA,Perkins AD,Saxton AM,Borate B,Chesler EJ,Branstetter LK,Langston MA.Extracting gene networks for low-dose radiation using graph theoretical algorithms[J].PLoS computational biology,2006,2(7):757-768.
[4] Oldham MichaelC,Horvath Steve,Geschwind DanielH.Conserva-tion and evolution of gene coexpression networks in human and chimpanzee brains[J].Proceedings of the National Academy of Sciences,2006,103(47):17973 -17978.
[5] Omar Odibat. RankingDifferentialGenesin Co-expression Networks[J].Proceedings of the 2nd ACM Conference on Bioinformatics,Computational Biology and Biomedicine,2011,350-354.
[6] Zhang B,Li H,Riggins RB,zhan M,Xuan J,Zhang Z,Hoffman EP,Charke R,Wang Y.Differential dependency network analysis to identify condition-specific topological changes in biological networks[J].Bioinformatics,2009,25(4):526 - 532.
[7] Lucinda K.Southworth,Art B.Owen,Stuart K.Kim.Aging Mice Show a Decreasing Correlation of Gene Expression within Genetic Modules[J].PLOS Genetics,2009,(5):e1000776.
[8] Bruno M Tesson,Rainer Breitling and Ritsert C Jansen.Diffcoex:a simple and sensitive method to find differentially coexpressed gene modules[J].BMC Bioinformatics,2010,11(1):497.
[9] Gang Fang,Gaurav Pandey.Mining Low-Support Discriminative Patterns from Dense and High-Dimensional Data.Knowledge and Data Engineering[J].IEEE Transactions,2012,24(2):279 -294.
[10] Gang Fang,Rui Kuang,Gauraw Pandey,Michael Steinbach,Chad L.Myers,Vipin Kumar.Subspace differential coexpression analysis:Problem Definition and a General Approach[J].Pacific Symposium on Biocomputing,2010,15:145 -156.
[11] Natasa Przulj.Biological network comparison using graphlet degree distribution[J].Cancer Inform,2008,6:257 -273.
[12] Przulj N,Corneil D G,Jurisica I.Modeling Interactome:Scale-Free or Geometric[J].Bioinformatics,2004,20(18):3508 -3515.
[13] TijanaMilenkovicand NatasaPrzulj. UncoveringBiological Network Function via Graphlet Degree Signatures[J].Cancer Inform,2008,6:257-273.
[14] Oleksii Kuchaiev,Tijana Milenkovic,Vesna Memisevic,Wayne Hayes,Natasa Przulj.Topological network alignment uncovers biological function and phylogeny[J].Journal of the Royal Society Interface,2011,5(17):1341 -1354.
[15] Yang B.,Liu D.Y.,Liu J.M..Complex Network Clustering Algorithms[J].Journal of Software,2009,20(1):54 - 66.
[16] Radicchi F.,Castellano C.,Cecconi F.Defining and identifying communities in networks[J].PNAS,2004,101(9):2658 -2663.
[17] Fu Chunxiao,Hickey Morgen,Morrison Melissa,McCarter Roger,Han Eun-Soo.Tissue specific and non-specific changes in gene expression by aging and by early stage CR[J].Mechanisms of Ageing and Development,2006,(127):905 -916.
[18] Barger Jamie L,Kayo Tsuyoshi,Vann James M,Arias Edward B,Wang Jelai,Hacker Timothy A,Wang Ying,Raederstorff Daniel,Morrow Jason D,Leeuwenburgh Christiaan,Allison David B,Saupe Kurt W,Cartee Gregory D,Weindruch Richard,Prolla Tomas A.A Low Dose ofDietary ResveratrolPartially Mimics Caloric Restriction and Retards Aging Parameters in Mice[J].PLoS ONE,2008,(3):e2264.

