DNA 条形码快速鉴定广州常见菊花蚜虫
汪珍春,王小兰,郑毅胜,周伯春,姜立云
(1.广州大学生命科学学院,广州 510006;2.中国科学院动物进化与系统学重点实验室,北京 100101)
菊花Chysantemum morifolium为多年生草本植物,是经长期人工选择培育的名贵观赏花卉,我国的菊花品种有3000种以上(Li,1981)。菊花因清雅高洁,花型优美,颜色绚丽,多被用于花坛、地被、盆花和切花等,是广州地区春节期间主要的上市花卉之一。然而,菊花在生长过程中非常容易受到病虫害的威胁,蚜虫是其重要害虫之一(Wang et al.,2002)。蚜虫不仅危害嫩枝、叶背、花蕾,致使植株矮化、卷叶,甚至死亡(Sauge et al.,2002),其排泄物还易导致煤污病发生,同时也是菊花病毒的主要传播者之一(图1)(Li,1981;Ellis et al.,1998)。蚜虫类Aphidinea 昆虫隶属于昆虫纲Insecta 半翅目Hemiptera,世界已知4700 余种(Remaudière and Remaudière,1997),中国蚜虫类资源丰富,已知种类有1000 余种。蚜虫体型很小,形态特征特化或退化,具有复杂的多型现象,有效的形态鉴别特征较少(张广学和钟铁森,1983)。因此仅依据形态特征的开展的蚜虫物种鉴定工作中的错误鉴定和同物异名现象时有发生,需要具有扎实的专业知识与丰富经验的分类学者才能准确鉴定(Foottit et al.,2008;Lee et al.,2011)。DNA 条形码是应用单一基因片段序列来区分所有物种的快速物种鉴定方法(Hebert et al.,2003),可以对处于各个发育阶段的生物型——卵、幼虫、成虫,或死亡个体的残骸进行鉴定,并且不受性别二态性的影响(Floyd et al.,2009),在蚜虫鉴定中具有显著的优势和应用价值。本研究采集了危害广州市17个常见绿化菊花品种的2属2种蚜虫,桃蚜Myzus persicae(Sulzer)和棉蚜Aphis gossypii Glover,探讨基于线粒体COI基因片段的DNA 条形码在快速鉴定危害广州常见菊花蚜虫中的应用,以帮助植保工作者在菊花种植、抗性品系的筛选和害虫防治等工作中进行蚜虫物种的快速、准确鉴定。

图1 蚜虫对菊花植物的为害Fig.1 Damage on Chrysanthemum plants by aphids
1 材料与方法
1.1 材料
研究用蚜虫样品均采自广州市常见的17个菊花品种,共计43份样品,涉及2属2种(样品信息见表1)。所有蚜虫标本浸泡于75%或95%酒精中,保存于中国科学院动物研究所国家动物博物馆(中国,北京)。其中,95%酒精浸泡标本用于分子实验,75%酒精浸泡标本用于制作玻片标本后的形态鉴定。标本由中国科学院动物研究所鉴定。
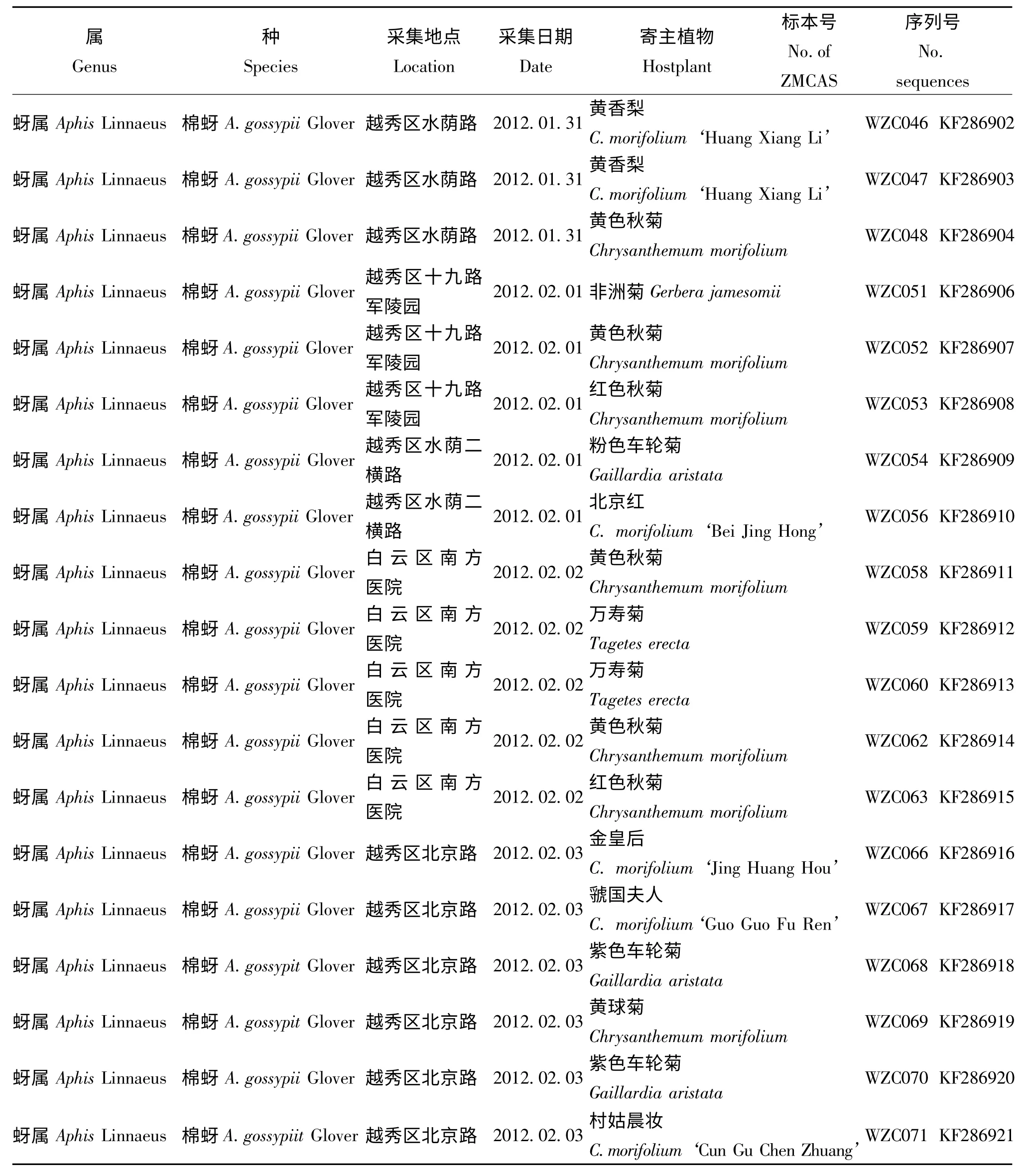
表1 本研究所用蚜虫样品信息Table 1 List of aphid samples used in this study
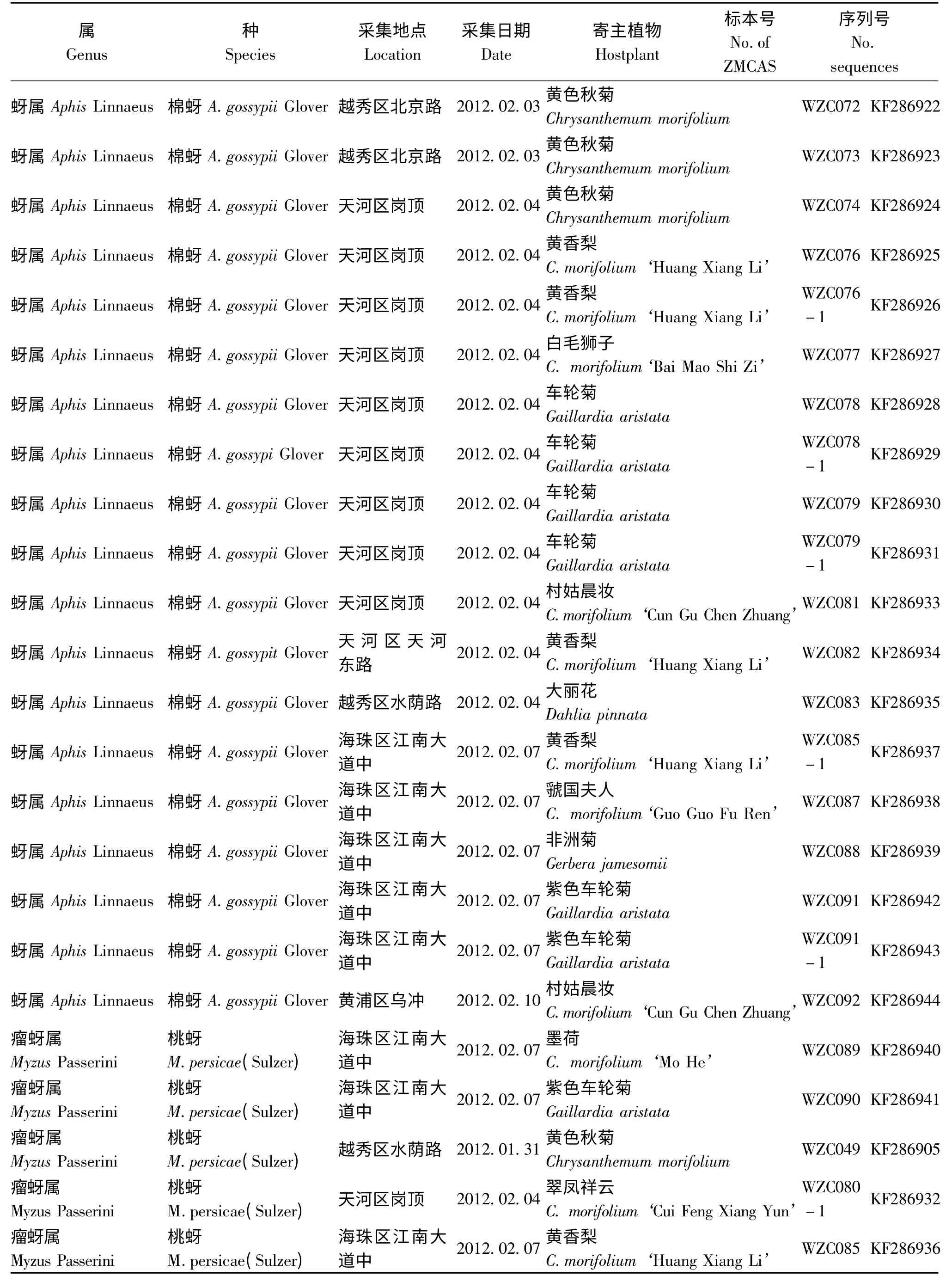
(续上表)
1.2 方法
1.2.1 DNA 粗提、PCR 扩增和测序
DNA 粗提:用TakaRa 公司的MightyAmp DNA Polymerase Ver.2 试剂盒粗提DNA,该试剂盒由MightyAmp DNA Polymerase(1.25 U/μL)和MightyAmp Buffer Ver.2(Mg2+,dNTP plus)组成,可以将血液、动植物组织等生体直接加入到反应液中进行PCR,MightyAmp DNA Polymeras 对粗提样品显示很好的扩增能力。因此,本实验选取浸泡于95%酒精中的体型较大的单个蚜虫虫体,吸干虫体表面酒精后,用dH2O 漂洗三次,浸泡过夜。次日更换dH2O 后,保留留约2μL 的dH2O,用研磨杵将虫体研碎,吸取上清液,即得到单个蚜虫DNA 粗提液。
PCR 引物设计:本研究选用线粒体基因细胞色素氧化酶I 亚基(COI)基因部分序列作为分子标记,其扩增引物为常用引物LEP-F、LEP-R(Foottit et al.,2008),用于扩增COI 基因3'端约700 bp 的片段。引物信息见表2。
PCR扩增:扩增体系为20μL,包括:MightyAmp Buffer Ver.2 10μL,primer1 1μL,primer 2 1μL,DNA 粗提液2μL,MightyAmp DNA Polymerase 0.5μL,dH2O 5.5μL。反应条件为98℃变性2 min,98℃变性10 s,54℃退火15 s,68℃延伸1 min,35个循环。PCR 产物于-20℃保存。
扩增产物检测:取3μL PCR 产物于1%琼脂糖凝胶电泳检测(128 V,10 min)。
扩增产物测序:PCR 产物纯化后,利用ABI 3730xl 96-capillary DNA 分析仪测序(Applied Biosystems,USA,上海美吉生物医药科技有限公司广州分公司)。
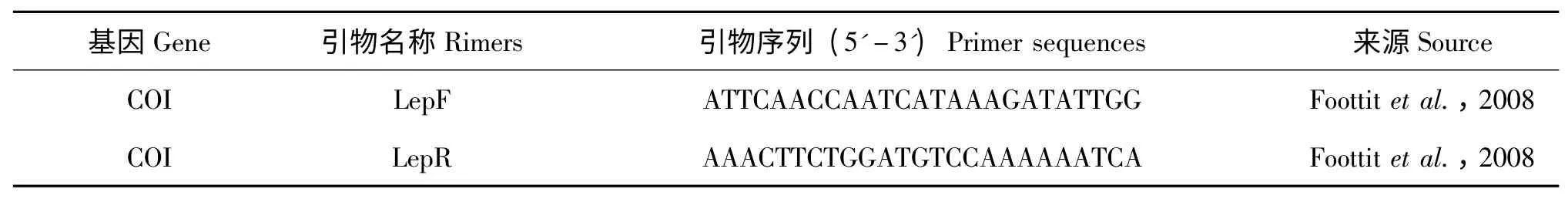
表2 线粒体COI 序列扩增引物信息Table 2 Mitochondrial COI primers used in this study
1.1.2 序列分析和邻接树的构建
通过Seqman 5.01(DNASTAR,Inc.1996)软件对双向测序结果进行拼接、校对、切去引物得到658bp。应用DNAMAN5.2.2(Lynnon Biosoft,Quebec,Canada)软件检验是否能正确翻译蛋白质以确保序列的正确性;使用MEGA 5.0(Tamura et al.,2007)对序列进行多重比对,分析核酸组成;通过DAMBE 程序(Xia and Xie,2001)进行序列饱和性分析,以转换、颠换为纵轴,以TN93 模型(Tamura and Nei,1993)校正的距离为横轴做散点图;基于 K2P 模型(Kimura-2-parameter distance,Kimura,1980),计算序列差异,构建邻接树(Neighbour-Joining tree,NJ tree),并对各分支节点进行Bootsrap 检验(检测1000 次,每次检测随机加样重复100 次);应用EXCEL 软件(Microsoft Office Excel 2003)计算各差异频次,绘制频次图。
2 结果与分析
2.1 COI 序列特征及饱和性分析
实验获得蚜虫2属2种43个序列,经Seqman比对后得到658 bp,编码218个氨基酸。表3 显示不同密码子位点的碱基分布,碱基平均含量为39.8%T,14.3%C,35.3%A,10.6%G。A+T 含量为75.1%,C+G 含量为24.9%,存在明显的A、T 碱基偏好性。其中,有保守位点551个,可变位点62个,简约位点56个,单个突变位点6个。
应用DAMBE 程序对序列进行了饱和性分析,以转换、颠换为纵轴,以TN93 模型校正的距离为横轴做散点图,结果如图2 所示,序列间转换、颠换数随距离增加呈线性增长趋势,未表现出饱和态势,所以658个位点都可被用于后续分析。
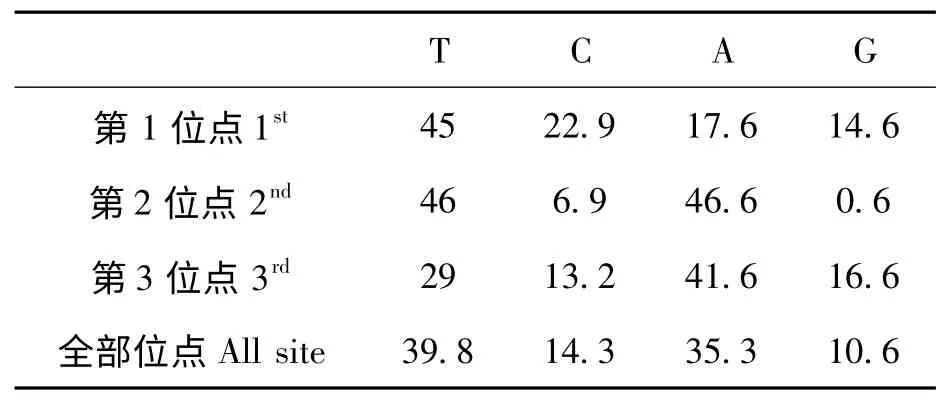
表3 样品中不同密码子位点的碱基含量分布(%)Table 3 The base content for samples and each codon position
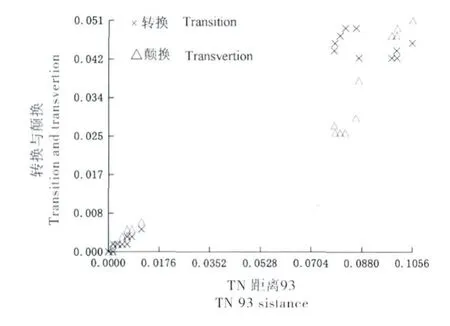
图2 基于658 bp 线粒体基因COI 的序列饱和性分析散点图Fig.2 Saturation plots for the mitochondrial COI sequences with 658bp.
2.2 NJ 树结构分析
基于COI 序列和K2P 模型,使用邻接法分析后得到所有样品的NJ 树(图3)。结果表明,棉蚜A.gossypii Glover和桃蚜M.persicae(Sulzer)的所有样品都能各自聚为一支,并有较高的支持率,分别为棉蚜64%,桃蚜为100%。
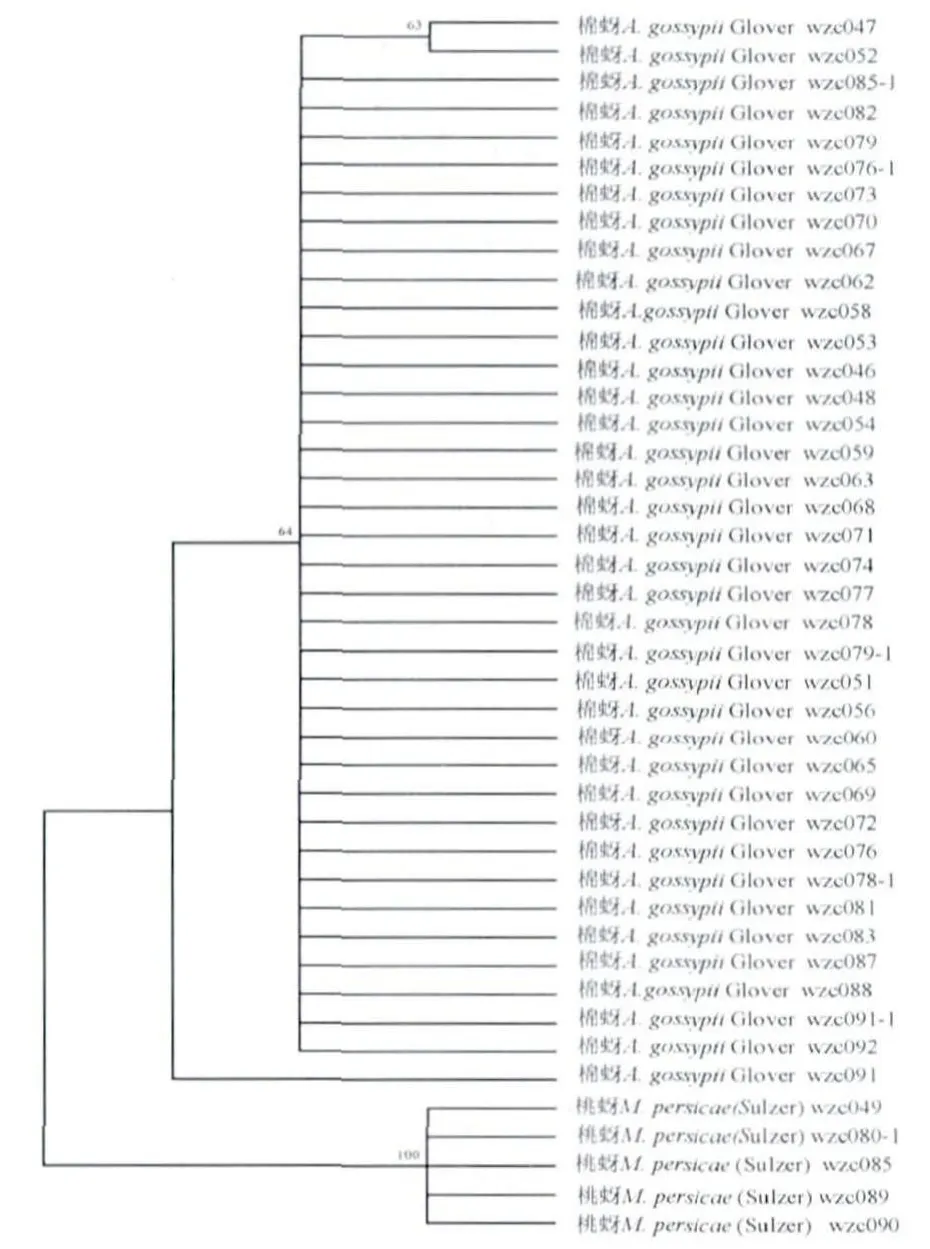
图3 基于COI 序列和K2P 模型构建的NJ 树,自举检验支持率标在分支左侧Fig.3 Neighbour-Joining(NJ)tree based on COI sequences and Kimura's two parameter model with bootstrap percentages shown on left.
2.3 序列差异分析
对实验所得的2种43 条COI 序列差异进行了计算与统计,平均差异为2.10%,标准差为4.00%,差异范围为0.00%~10.3%。主要集中在0.00%~2.50%和10.00%~12.50% 范围内,种内序列差异和种间序列差异之间形成了明显的“Barcoding gap”。种内序列差为0.00%~1.20%,平均差异为0.08%,标准差为0.18%。各物种的种内差异见表4。棉蚜A.gossypii 序列间的差异最大,可达1.20%。种间差异为9.60%~10.30%,平均差异为9.69%,标准差为0.13%。
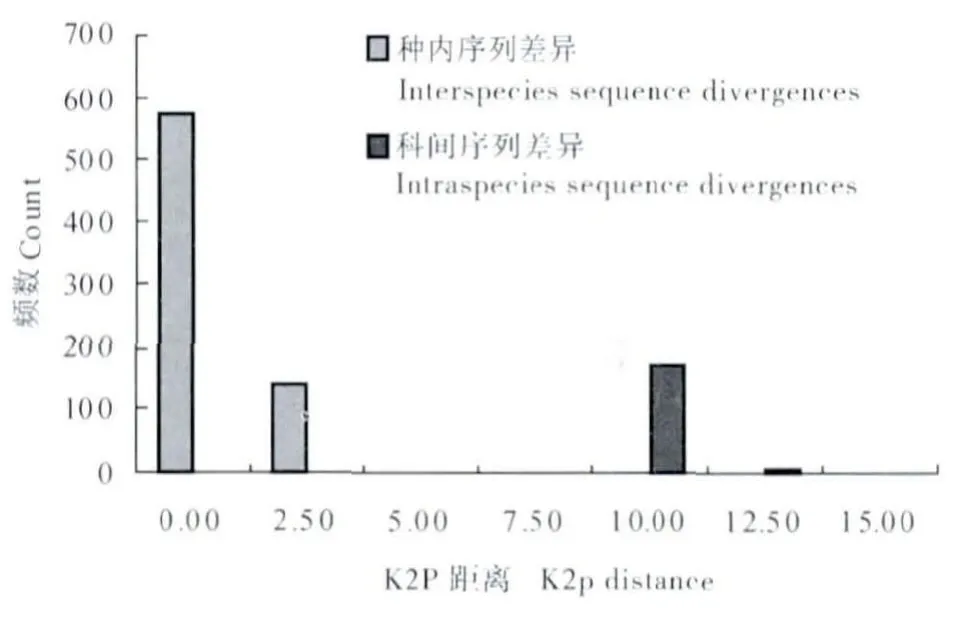
图4 基于线粒体COI 658bp 序列和K2P 距离的种间序列差异频次图Fig.4 Histogram of genetic divergence using Mitochondrial COI 658bp Sequences.Divergences were calculated using Kimura's two parameter(K2P)model.
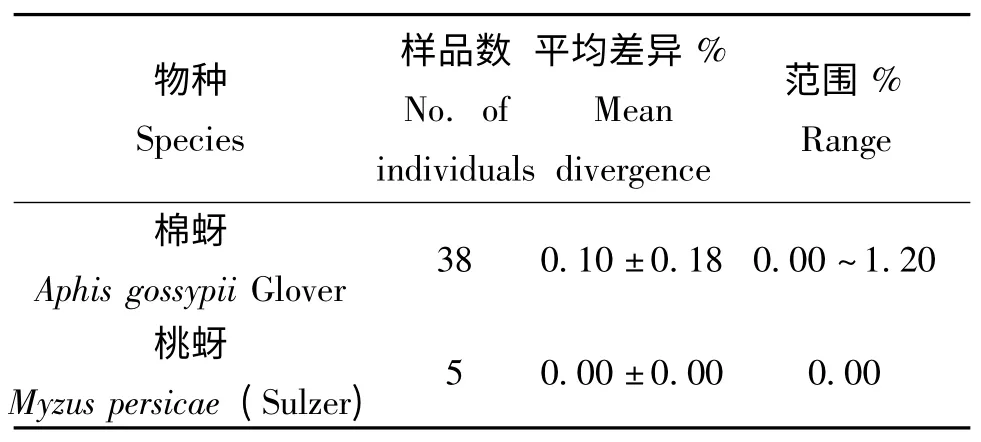
表4 各种种内序列差异Table 4 Mean and range of intraspecific nucleotide divergences
3 结论与讨论
本研究收集了广州市常见绿化菊花17个品种:黄香梨、黄色秋菊、非洲菊、红色秋菊、粉色车轮菊、白毛狮子、北京红、万寿菊、金皇后、紫色车轮菊、黄球菊、虢国夫人、村姑晨妆、车轮菊、大丽花、翠凤祥云、墨荷上的2属2种蚜虫。其中,棉蚜Aphis gossypii Glover 出现在所有的品种上,多数寄生于花瓣,少数寄生于叶背面。桃蚜Myzus persicae(Sulzer)只在墨荷、紫色车轮菊、黄香梨、翠凤祥云、黄色秋菊上发现。花瓣、叶背面都有寄生。
Hebert 等(2003)对包括脊椎动物和无脊椎动物界的线粒体COI 基因序列分析得出结论:除腔肠动物外,98% 物种的种内遗传距离差异为0.00%~2.00%,种间遗传距离差异平均可达11.30%。作为一种新的物种鉴定手段,基于mtDNA COI 基因的条形码技术目前在昆虫鉴定方面有广泛应用,在桔小实蝇(刘慎思等,2012)、对齿小蠹属昆虫(常虹等,2012)、夜蛾科物种(杨聪慧等,2012)、我国常见7种蝗虫(潘程莹等,2006)、我国田间常见25种蓟马(乔玮娜等,2012)以及蚜虫(Foottit et al.,2008;Lee et al.,2011;Wang et al.,2011;温娟等,2013;陈睿等,2013)等类群的研究中均显示出了很高的解决效力。
本研究结果与其他昆虫类群的条形码研究结果一致。采自广州市菊花上的2个蚜虫物种的COI序列存在明显的A、T 碱基偏好性,符合昆虫线粒体基因碱基组成的基本特征(Simon et al.,1994;von Dohlen et al.,2000;Zhang and Qiao,2007a,2007b;Wang et al.,2011;Liu and Beckenbach,1992);种内平均差异为0.08%,最大差异为1.20%,种间平均差异为9.69%,最小差异值达到9.60%,说明线粒体COI 基因作为条形码能有效地区分蚜虫的种类,并且能很好地区分广州地区常见的危害菊花的蚜虫种类。此外,同一个物种的不同地理种群间的差异很小。例如棉蚜的38个样品来自9个不同的地理种群,其序列差异仅为0.10%。来自同一地点,同一寄主的同一器官上的不同样品也得到了很好的区分,例如,编号为wzc090和wzc091 的两个样品(图1A)都来自江南大道中的紫色车轮菊的花瓣,经形态鉴定确认两个样品分别是棉蚜和桃蚜,它们之间的序列差异达到9.80%。采自来自同一地点的同一寄主的不同器官上的两个样品,虽然其体色差异很大,却为同一个物种,例如编号为wzc072(图1B)和wzc 073(图1C)的样品都是采自北京路的黄色秋菊,前者为黑色,寄生于花瓣,后者为墨绿色,寄生于叶背面,但经条形码鉴定都为棉蚜,它们之间的COI 序列差异为0.00。同一品种的菊花在不同地理位置种植,其上寄生的蚜虫物种也会不同,例如,种植在珠江以北地区的天河区,越秀区和白云区的黄香梨上只发现有棉蚜寄生,而种植在珠江以南的海珠区的黄花梨则同时发现有棉蚜和桃蚜寄生。因此,基于COI 基因的DNA 条形码可以帮助解决常见危害菊花的不同种蚜虫易被混淆的问题,同时还可以避免将特征差异较大(例如:体型不等、体色不同)的同种蚜虫鉴定为不同物种的错误,在缺乏蚜虫分类专家指导的前提下,也能够快速准确鉴定危害广州常见菊花的蚜虫物种,极大地提高了植保工作的效率。
References)
Chang H,Hao DJ,Xiao RT,Liu Y,Qian L,An YL,Yang XJ,2012.DNA barcoding based on the mitochondrial COI gene sequences for Ips species(Coleoptera:Scolytidae).Acta Entomologica Sinica,55(9):1075-1081.[常虹,郝德君,肖荣堂,刘勇,钱路,安榆林,杨晓军,2012.基于线粒体COI 基因的齿小蠹属昆虫DNA 条形码研究.昆虫学报,55(9):1075-1081]
Chen R,Jiang LY,Qiao GX,2013.DNA barcoding in rapid identification of aphids on Pinus armandii.Chinese Journal of Applied Entomology,50(1):50-60.[陈睿,姜立云,乔格侠,2013.华山松不同生态位上蚜虫的快速鉴定——基于DNA 条形码.应用昆虫学报,50(1):50-60]
Ellis PR,Pink DAC,Phelps K,Jukes PL,Breeds SE,Pinnegar AE,1998.Evaluation of a core collection of Brassica oleracea accessions for resistance to Brevicoryne brassicae,the cabbage aphid.Euphytica,103(2):149-160.
Floyd RM,Wilson JJ,Hebert PDN,2009.DNA barcodes and insect biodiversity.In:Foottit RG,Adler PH,eds.Insect Biodiversity:Science and Society.Oxford:Wiley-Blackwell.417-432.
Foottit RG,Maw HEL,von Dohlen CD,Hebert PDN,2008.Species identification of aphids(Insecta:Hemiptera:Aphididae)through DNA barcodes.Mol.Ecol.Res.,8:1189-1201.
Hebert PDN,Cywinska A,Ball SL,de Waard JR,2003.Biological identifications through DNA barcodes.Proc.R.Soc.Lond.B,270:313-321.
Lee W,Kim H,Lim J,Choi HR,Kim Y,Kim YS,Ji JY,Foottit RG,Lee S,2011.Barcoding aphids(Hemiptera:Aphididae)of the Korean Peninsula:updating the global data set.Mol.Ecol.Resour,11:32-37.
Liu H,Beckenbach AT,1992.Evolution of the mitochondrial cytochrome oxidase II gene among 10 orders of insects.Molecular Phylogenetics and Evolution,1:41-52.
Liu SS,Zhang GF,Wu Q,Zhang AB,Wang JJ,Wan FH,2012.Establishment and application of DNA barcoding technology for identifi-cation of the immature and adult debris of Bactrocera dorsalis(Hendel)(Diptera:Tephritidae).Acta Entomologica Sinica,55(3):336-343.[刘慎思,张桂芬,武强,张爱兵,王进军,万方浩,2012.桔小实蝇幼体及成虫残体DNA条形码识别技术的建立与应用.昆虫学报,55(3):336-343]
Li HJ,1981.Chysanthemums in China.Nanjing:Jiangsu Science and Technology Press.59-63.
Pan CY,Hu J,Zhang X,Huang Y,2006.The DNA barcoding application of mtDNA COI genes in seven species of Catantopidae(Orthoptera).Entomotaxonomia,28(2):103-110.[潘 程莹,胡婧,张霞,黄原.斑腿蝗科Catantopidae 七种蝗虫线粒体COI 基因的DNA 条形码研究.昆虫分类学报,28(2):103-110]
Qiao WN,Wan FH,Zhang AB,Min L,Zhang GF,2012.Application of DNA barcoding technology for species identification of common thrips(Insecta:Thysanoptera)in China.Acta Entomologica Sinica,55(3):344-356.[乔玮娜,万方浩,张爱兵,闵亮,张桂芬,2012.DNA 条形码技术在田间常见蓟马种类识别中的应用.昆虫学报,55(3):344-356]
Remaudière G,Remaudière M,1997.Catalogue of the World's Aphididae.Paris:Homoptera Aphidoidea.Institut National de la Recherche Agronomique.1-473.
Sauge MH,Lacroze JP,Poёssel JL,Pascal T,Kervella J,2002.Induced resistance by Myzus persicae in the peach cultivar‘Rubira'.Entomologia Experimentalis et Applicata,102(1):29-37.
Simon C,Frati F,Beckenbach A,Crespi B,Liu H,Flook P,1994.Evolution,weighting,and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers.Ann.Ent.Soc.Am.,87:651-701.
Tamura K,Nei M,1993.Estimation of the number nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees.Mol.Biol.Evol.,10:512-526.
Tamura K,Dudley J,Nei M,Kumar S,2007.MEGA4:Molecular Evolutionary Genetics Analysis(MEGA)software version 4.0.Mol.Biol.Evol.,24:1596-1599.
Von Dohlen CD,Moran NA,2000.Molecular data support a rapid radiation of aphids in the Cretaceous and multiple origins of host alternation.Biol.J.Linn.Soc.,71:689-717.
Wang J,Hu HL,Zhang CL,Hu YB,Liu MF,2002.Integrated control of main diseases and insects of Dendranthema morifolium.Chinese Journal of Applied Ecology,13(4):444-448.
Wang JF,Jiang LY,Qiao GX,2011.Use of a mitochondrial COI sequence to identify species of the subtribe Aphidina(Hemiptera,Aphididae).ZooKeys,122:1-17.
Wen J,Chen R,Jiang LY,Qiao GX,2013.Rapid identification of aphids on flowering plants of Rosaceae in Beijing based on DNA barcoding.Chinese Journal of Applied Entomology,50(1):29-40.[温娟,陈睿,姜立云,乔格侠,2013.基于DNA 条形码对北京地区蔷薇科花卉上蚜虫的快速鉴定.应用昆虫学报,50(1):29-40]
Xia X,Xie Z,2001.DAMBE:Data analysis in molecular biology and evolution.J.Hered.,92:371-373.
Yang CH,Han HL,Chi MY,Jin Q,Wu CS,Zhu CD,Zhang AB,2012.Species identification of Noctuidae moths(Insecta:Lepidoptera)from Baihuashan,Beijing,China with DNA barcoding.Acta Entomologica Sinica,55(9):1082-1092.[杨聪慧,韩辉林,迟美妍,金倩,武春生,朱朝东,张爱兵,2012.DNA 条形码技术在北京百花山地区夜蛾科物种鉴定中的应用.昆虫学报,55(9):1082-1092]
Zhang GX,Zhong TS,1983.Economic Insect Fauna of China Fasc.25.Homoptera:Aphidinea.Beijing:Science Press.1-387.[张广学,钟铁森,1983.中国经济昆虫志,第25 册,同翅目,蚜虫类(一).北京:科学出版社.1-387]
Zhang HC,Qiao GX,2007a.Molecular phylogeny of Fordini(Hemiptera:Aphididae:Pemphiginae)inferred from nuclear gene EF-1α and mitochondrial gene COI.Bull.Entomol.Res.,97:379-386.
Zhang HC,Qiao GX,2007b.Systematic status of genus Formosaphis Takahashi and evolution of galls based on the molecular phylogeny of Pemphigini(Hemiptera:Aphididae:Eriosomatinae).Syst.Entomol.,32:690-699.

