新疆哈萨克绵羊MHCⅠ类基因第二、三外显子序列多态性分析
刘志方,吴长新,张辉,李蕊,郭吉星,肖红冉,陈创夫
(石河子大学动物科技学院,石河子832003)
主要组织相容性复合体(major histocompatibility complex,MHC)是Snell等在研究小鼠的移植排斥反应时发现的,是由染色体上紧密连锁、高度多态的基因位点组成的一个遗传区域[1]。绵羊的MHC基因位于20号染色体上的q15~q23[2],是识别自身与非自身抗原免疫反应的多基因家族,在免疫应答的启动和调节中发挥着十分重要的作用[3],与动物感染性疾病特别是病毒、细菌和寄生虫的抵抗和易感性相关[4]。根据MHC抗原结构和功能的不同可将MHC分为classⅠ、classⅡ和classⅢ三个区域,其编码产物称为绵羊白细胞抗原(OLA)或绵羊淋巴细胞抗原[5]。其中Ⅰ类基因编码的产物称为MHCⅠ类分子,是一种由α链和β链非共价结合组成的细胞表面糖蛋白,能识别结合内源性的抗原肽,并将其递呈给CD8+T细胞,从而激发机体发生免疫反应,与动物的抗病能力有较强的相关性[6]。其中α链的α1和α2区域即由第二和第三外显子编码的区域是与抗原结合的区域,也是多态性最集中的区域,可能影响抗原结合区的空间结构或通过影响T细胞受体的特异性识别等方式影响抗原递呈过程的识别与被识别的特异性[4],因可能与动物疾病的抵抗及生产性能有一定的联系而成为抗病育种研究的热点。
有研究证明对某一毒力已知的病毒株的敏感程度取决于绵羊的品种,称为品种敏感性[7],同一品种绵羊的不同个体对于同一种病原的易感性也不同,这可能与绵羊MHCⅠ类分子对特定病原不同的递呈能力相关。本文利用PCR和测序的方法对新疆哈萨克绵羊MHCⅠ类基因第二、三外显子的序列多态性进行了检测与分析,旨在为以后研究绵羊MHCⅠ类分子对抗原的递呈功能及抗病育种工作提供数据基础。
1 材料与方法
1.1 材料
血液基因组提取试剂盒购自康为世纪生物公司;pMD-18-T载体购自 TaKaRa公司、LA Taq酶购自宝生物工程(大连)有限公司、dNTP、DNA凝胶回收试剂盒、DNA分子量标准购自北京天根生化科技有限公司;大肠杆菌DH5α为本实验室保存。其它试剂均为国产分析纯产品。
1.2 方法
1.2.1 采样
从新疆伊犁地区随机选择健康哈萨克绵羊36只。每只绵羊颈静脉无菌采取肝素钠抗凝血5mL,低温运输,-20℃保存。
1.2.2 引物设计
根据Gene Bank中绵羊MHC全基因组序列(登录号为FJ985870),选取包含第二、三外显子及部分侧翼的内含子部分序列,用Primer 5.0软件设计引物:
上游引物为:5′-ACCGCCTCCATCTCATTG-3′;
下游引物为:5′-TGTCTTGTACAGAATCTG AAAA-3′。
引物由上海生物工程有限公司合成。
1.2.3 血液基因组DNA的提取
用血液基因组小提试剂盒提取基因组DNA,用分光光度仪和琼脂糖凝胶电泳对其质量和浓度进行检测,提取的基因组DNA在-20℃保存。
1.2.4 MHCⅠ类基因第二、三外显子的扩增及纯化
以提取的血液基因组DNA为模板,PCR反应条件为:95℃预变性5min;94℃变性30s、65℃退火40s,72℃ 延伸1min30s,35个循环;最后延伸72℃7min。PCR扩增产物经1%琼脂糖凝胶电泳检测。
1.2.5 MHCⅠ类基因第二、三外显子的克隆及测序
将回收后的PCR产物与pMD-18-T载体连接,并转化至大肠杆菌DH5α感受态细胞,经过氨苄抗性筛选挑取单克隆,经菌液PCR鉴定后每个样品挑选2~3个菌液PCR检测阳性克隆菌,摇床37℃培养过夜后送上海生物工程有限公司测序。
1.2.6 序列分析
测序所得的核苷酸序列经GenBank中BLAST比对,验证序列的正确性。核苷酸和氨基酸序列的统计分析利用 MEGA 5.0和 DNA SP4.0软件完成。MEGA软件中Kimura双参数法(Gamma)法计算序列之间的遗传距离(标准误),同时还计算序列的转换(transition)和颠换(transversion)数(率)。序列中的同义(dS)和非同义(dN)替换相对比例通过 MEGA软件中用Jukes-Cantor(1969)校正 Nei Gojobori(1986)方法计算。利用 MEGA软件中基于 Nucleotide:Jukes-Cantor模型的邻近法(neighbor joining,NJ)构建系统发生树(自举检验重复1000次)。
2 结果
2.1 MHCⅠ类基因第二、三外显子的PCR扩增
提取的DNA的260/280为1.8左右,浓度为22.6~104.5ng/μL。以样品的基因组 DNA 为模版,PCR扩增产物经1%琼脂糖凝胶电泳检测,扩增产物为1074bp(图1),与预期结果一致。其中包括内含子1的部分序列、外显子2序列270bp、内含子2序列212bp、外显子3序列276bp及内含子3的部分序列。
2.2 pMD18-T-MHCⅠ重组质粒的鉴定
PCR产物回收纯化后连接pMD18-T载体,转化DH5α感受态细胞,经氨苄抗性筛选,菌液PCR后电泳检测,观察到1条大小为1074bp的目的条带(图2),与预期大小一致。阳性克隆测序结果与已知序列长度完全一致,重组质粒命名为pMD18-T-MHCⅠ。

图1 MHC I类基因第二、三外显子PCR扩增Fig.1PCR amplification of the exon2and 3of MHC I gene
2.3 测序结果分析
2.3.1 测序获得的基因个数及频率
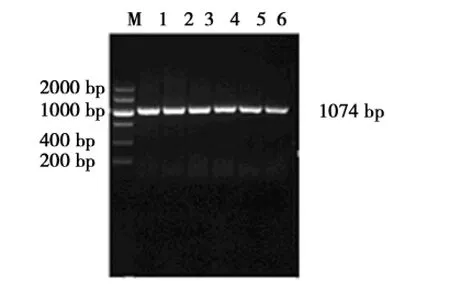
图2 pMD18-T-MHCⅠ PCR鉴定结果Fig.2PCR identification of pMD18-T-MHCⅠ
共获得89个阳性克隆,其中第二外显子序列16个、第三外显子序列15个,各序列的频率见表1、2。

表1 MHCⅠ类基因第二外显子序列基因频率Tab.1Gene frequencies of MHCⅠexon 2

表2 MHCⅠ基因第三外显子序列基因频率Tab.2Gene frequencies of MHCⅠ exon 3
2.3.2 序列之间的遗传距离分析
从表3、4中可以看出,第二外显子遗传距离为0.005~0.063;第三外显子遗传距离为0.005~0.063。
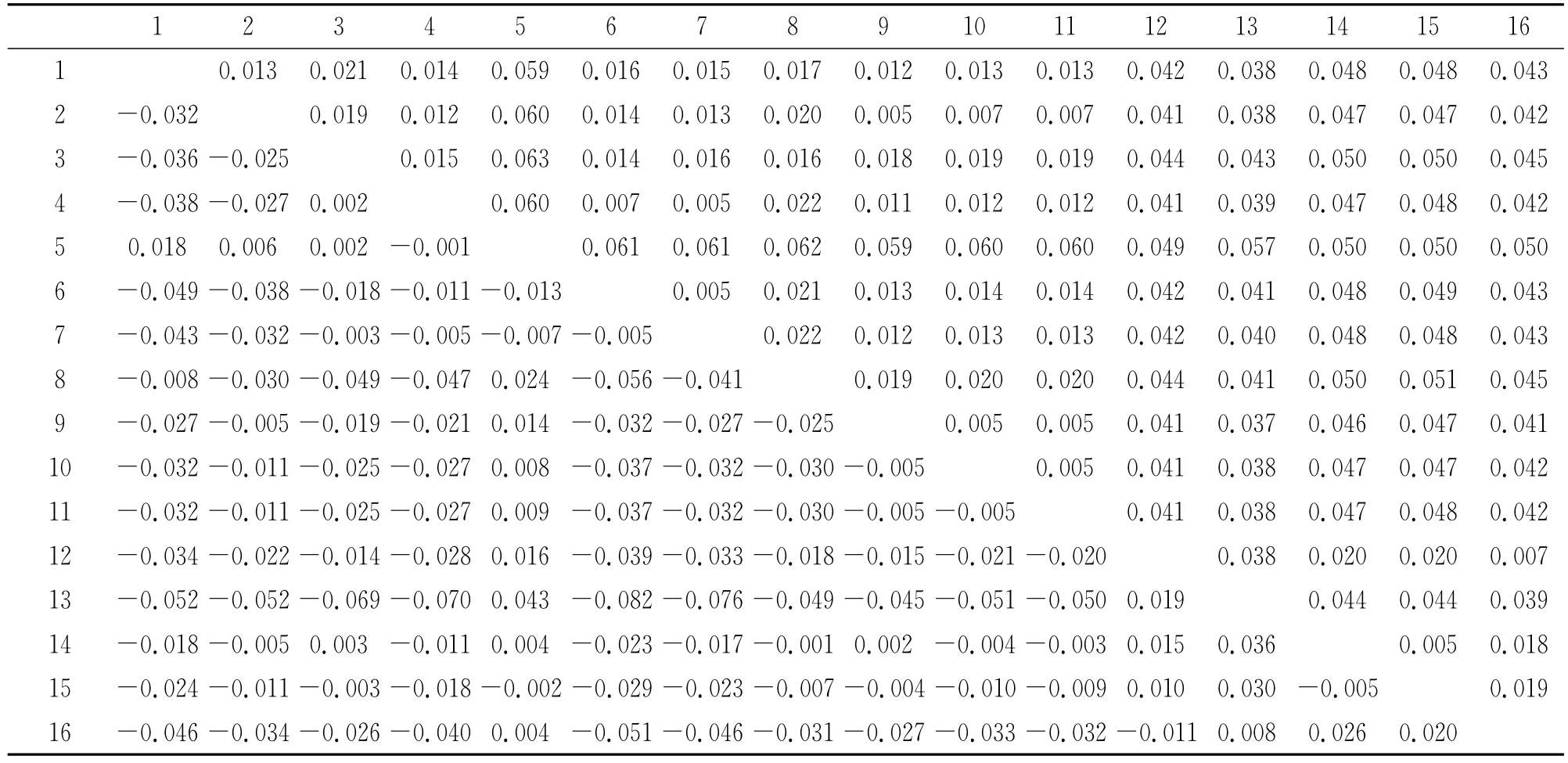
表3 第二外显子序列的遗传距离及标准误Tab.3Genetic distances and their standard errors among exon 2sequences

表4 第三外显子序列的遗传距离及标准误Tab.4Genetic distances and their standard errors among exon 3sequences
2.3.3 序列核苷酸转换、颠换分析
第二外显子碱基组成存在一定的不平衡性,4种碱基A、T、C、G的平均含量分别为19.26%、14.86%、33.98%和31.90%,GC(65.88%)含量明显高于AT(34.12%)含量。核苷酸之间存在着转换和颠换现象,转换、颠换之比R=1.494。(K1=3.698(purines),k2=2.737(pyrimidines);R=[A*G*k1+T*C*K2]/[(A+G)*(T+C)],说明第二外显子变异种存在转换偏倚现象。第三外显子4种碱基 A、T、C、G平均含量分别为19.54%、16.26%、35.07%和29.13%,GC(64.20%)含量也明显高于AT(35.80%)含量。R=0.65,(K1=1.153,k2=1.732)(表5、6)。

表5 用极大似然估计模型计算的第二外显子核苷酸替代概率Tab.5Maximum composite likehihood estimate of the pattern of nucleotide in exon 2

表6 用极大似然估计模型计算的第三外显子核苷酸替代概率Tab.6Maximum composite likehihood estimate of the pattern of nucleotide in exon 3
2.3.4 核苷酸变异位点分析
由表7可见,外显子二、三都有较多的多态位点,较高的核苷酸差异平均值及核苷酸多样性。
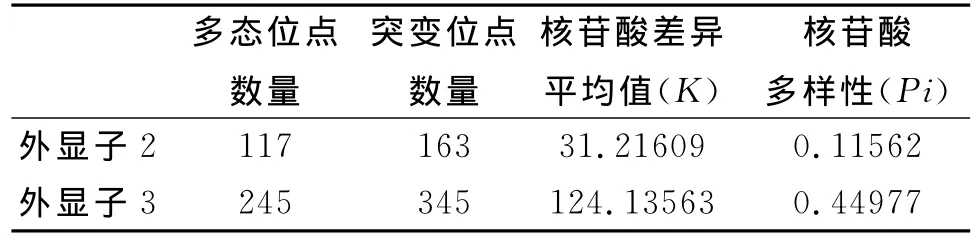
表7 外显子二、三的多态位点数、突变位点数、核苷酸差异平均值及多样性Tab.7The nucleotide polymorphic positions,Mutant position,Kand Pi value
2.3.5 自然选择性检验
由表8中可见,第二外显子的同义替换率略高于非同义替换率,但第三外显子的非同义置换率却略高于同义置换率,说明第二外显子经过了阴性选择作用,而第三外显子经过了阳性选择作用。

表8 dN和dS替换率、dN/dS比率和P值Tab.8Non-synonymous substitution(dN)rates,synonymous substitution(dS),dN/dS,and Pvalues
2.3.6 系统发生分析
图3、4中以大写字母H开头的序列为测得的序列,其余序列为数据库中牛MHCⅠ类基因的序列。图3显示哈萨克绵羊第二外显子序列之间的同源性较高,聚类为3个大的分支:其中序列3、8、6、9、10、11、7、4、1、2和序列13聚为一类,与序列12、14、15、16之间的遗传距离较远,但和牛的序列FJ952946.1有较近的遗传距离;序列5与牛的序列中BC112510.1和BC151687.1有较近的遗传距离,却与测得的其它序列的遗传距离较远;图4显示哈萨克绵羊第三外显子的15个序列与牛的序列之间的差异较大,2个物种序列各聚为一类,没有交叉出现。

图3 MHCⅠ基因第二外显子进化树Fig.3Phylogenetic analysis based on MHCⅠexon2
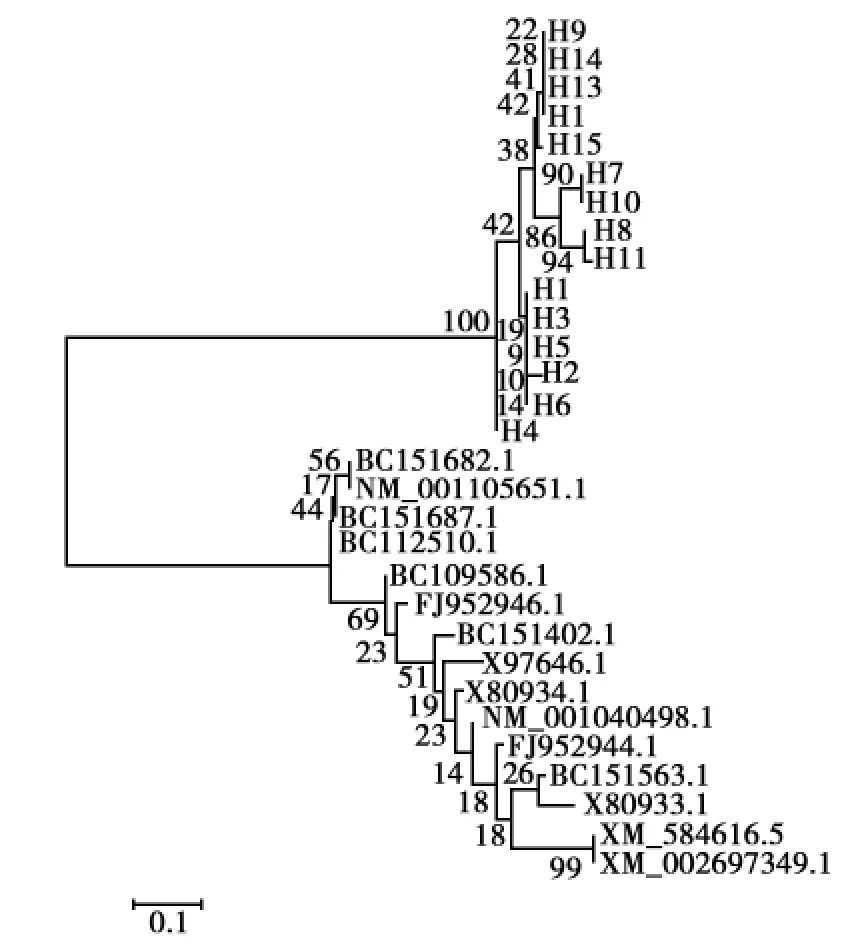
图4 MHCⅠ基因第三外显子进化树Fig.4Phylogenetic analysis based on MHCⅠ exon 3
3 讨论
MHC基因区域的多态性已经在很多脊椎动物中发现,并主要由自然选择来维持[8],由长期进化过程中等位基因的积累,融合与扩增形成,产生多态性的机制主要是突变和基因转换[9]。绵羊MHCⅠ类分子在免疫反应、疾病抵抗、易感性和发病过程[10]以及自身免疫方面都发挥着重要的作用,尤其是与动物的抗病性和易感性存在着强相关关系[11]。不同品系绵羊抗病特征可能与其体内与免疫反应相关基因存在着密切关系,而用生物学的方法研究绵羊的MHC基因可以为绵羊的抗病育种提供科学依据[12]。对于绵羊MHC基因的多态性和与感染性疾病的抗性和易感性方面已经有大量的研究:Li RY等[13]研究证明多浪羊、哈萨克羊和中国美利奴羊的MHC-DRB1基因第二外显子多态性和棘球蚴病抗性之间存在关联;Amaia Larruskain等[14]研究表明绵羊MHCII类DRBⅠ基因的多态性对于梅迪威斯纳病和肺腺炎病毒病的病例进程有影响;Stear等[15]研究表明绵羊MHC与线虫的抗性有关联;贾斌等[16]研究表明多浪羊和中国美利奴羊 MHCDRB1基因多态性与包虫病的遗传易感性相关。但是,目前对于绵羊MHC多态性的研究多集中在绵羊MHC II类基因,且大多数关于DRB基因座,对MHC I类多态性研究的较少,Keith等[17]对4个单体型的MHCⅠ类基因的基因组学和蛋白组学分析结果显示,绵羊MHCI类基因可能最多含有8个基因座。
本实验对新疆147团的48只美利奴羊的MHCⅠ基因的第二、三外显子的多态性进行了分析,这些多态位点与MHCⅠ类分子与内源性抗原的递呈有关,从而与疾病的抗性相关联;尽管羊MHCⅠ类基因之间的同源性较高,但是有时序列能与牛的序列成簇。这些成簇的牛和羊的序列说明它们可能在牛和羊分化前的共同祖先中就存在这样的基因,也就是所谓的跨物种假设[18];病原体的识别也为MHCⅠ类基因的进化提供了选择压力,牛和羊具有相似的序列可能说明了针对共同的病原体的特定免疫反应的存在[19]。本研究结果可为进一步研究MHCⅠ类分子的多态性与疾病抗性之间的关系提供了数据基础,也为抗原表位疫苗的研发做了前期工作。
[1]陶金陵,陈明辉,孙延星,等.新疆4个绵羊群体 MHCDRB1基因外显子2多态性的遗传分析[J].石河子大学学报:自然科学版,2010,28(3):315-318.
[2]Hickford J G,Zhou H,Slow S,et al.Diversity of the ovine DQA2gene[J].Anim Sci,2004,82:1553-1563.
[3]魏丽君,石国庆,王晓申,等.四个绵羊品种 MHC-DRB3基因外显子2的多态性分析[J].中国畜牧兽医,2007,4(7):63-66.
[4]Jianfeng Gao,Ka Liu,Haibo Liu,et al.A complete DNA sequence map of the ovine Major Histocompatibitity Complex[J].BMC Genomics,2010,11:466-473.
[5]Dukkipati V S R,Blair H T,Garrick D J,et al.‘Ovar-MHC’-ovine major histocompatibility complex:structure and gene polymorphisms[J].Genetics and Molecular Research,2006,5(4):581-608.
[6]Despoina Miltiadou,Ballingall K T,Ellis S A,et al.Haplotype characterization of transcribed ovine major histocompatibility complex(MHC)class I genes[J].Immunogenetics,2005,57:499-509.
[7]邓普辉,简子健,邱家祥,等.新疆绵羊群体对绵羊慢病毒的品种敏感性研究[J].中国兽医杂志,2004,14(12):26-27.
[8]Angela Bahr,Anthony Wilson B.The evolution of MHC diversity:evidence of intralocus gene conversion and recombination in a single-locus system[J].Gene,2012,497(1):52-57.
[9]刘秀,胡江,罗玉柱.藏绵羊基因OLA-DQA2第2外显子多态性分析[J].中国农业科学,2009,42(8):2930-2936.
[10]邹毅辉,李鑫,李峰,等.绵羊 MHC区段BAC克隆探针的制备[J].石河子大学学报:自然科学版,2010,28(6):700-702.
[11]Dukkipati,V S R,Blair H T,Garrick D J,et al.Ovar-Mhc-ovine major histocompatibility complex:Role in genetic resistance to diseases[J].New Zealand Veterinary Journal,2006,54(4):153-160.
[12]李鑫,杨晓亮,高剑峰,等.中国美利奴羊外周免疫器官cDNA文库的构建及鉴定[J].石河子大学学报:自然科学版,2010,28(5):529-533.
[13]Li R Y,Hui W Q,Jia B,et al.The relationship between MHC -DRB1gene second exon polymorphism and hydatidosis resistance of Chinese Merino(Sinkiang Junken type),Kazakh and Duolang sheep[J].Parasite,2011,18(2):163-169.
[14]Amaia Larruskain,Esmeralda,Bernardino Moreno,et al.MHC class II DRB1gene polymorphism in the pathogenesis of Maedi-Visna and pulmonary adenocarcinoma viral disease in sheep[J].Immunogenetics,2010,62:75-83.
[15]Stear M J,Belch A,Donskow-Schmelter K,et.al.Detection of genes with moderate effects on disease resistance using ovine mhc and resistance to nematodes an example[J].Veterinary Immunology and Immunopathology,2007,120:3-9.
[16]贾斌,陈明辉,刘宏坤,等.多浪羊和中国美利奴羊MHC-DRB1基因多态性与包虫病的遗传易感性[J].中国人兽共患病学报,2007,23(10):1004-1012.
[17]Ballingall K T,Miltiadou D,Meckeever D J,et al.Genetic and proteomic analysis of the MHC class I repertoire from four ovine haplotypes[J].Immunogenetics,2008,60:177-184.
[18] Klein J.Origin of major histocompatibility complex polymorphism:the trans-species hypothesis[J].Hum Immunol,1987,19:155-162.
[19]Zhou H,Hickford J H.Allelic polymorphism in the ovine DQA1gene[J].Journal of Animal Science,2004,82:8-16.

