花生AhFAD8基因的克隆、表达及活性分析
高继国,惠识瑶,,耿丽丽,张 蕊,孙长坡,张 杰
(1.东北农业大学生命科学学院,哈尔滨 150030;2.中国农业科学院植物保护研究所植物病虫害生物学国家重点实验室,北京 100193;3.国家粮食局科学研究院,北京 100037)
多不饱和脂肪酸(Polyunsaturated fatty acids,PUFAs)指含有两个或两个以上双键且碳链长为16~22个碳原子的直链脂肪酸[1-2]。根据多不饱和脂肪酸分子甲基端起第一个不饱和双键所连结的碳原子在碳链中的位置不同,将PUFAs分为ω-3和ω-6两类[3-4]。α-亚麻酸(Alpha-linolenic acid,ALA)是ω-3多不饱和脂肪酸中的一种,对促进人体健康具有重要作用,尤其可以促进大脑发育[5-6]。ALA是对人体有益的长链不饱和脂肪酸二十碳五烯酸(Eicosapentaenoic acid,EPA)和二十二碳六烯酸(Docosahexenoic acid,DHA)的合成前体物质,ALA通过对同一种必需酶的竞争消耗,抑制ω-6脂肪酸的延伸从而达到平衡人体内ω-3与ω-6脂肪酸水平的功效[7-9]。但是,由于人体内缺乏必要的脱氢酶,不能合成ALA和亚油酸(Linoleic acid,LA)此类基本的脂肪酸,所以只能从饮食中摄取相应的不饱和脂肪酸[10]。
脂肪酸脱氢酶(Fatty acid desaturase,FAD)是催化脂肪酸链特定位置形成双键和产生不饱和脂肪酸的酶类。植物脂肪酸脱氢酶主要有ω-3(FAD3、FAD7、FAD8)和ω-6(FAD2、FAD6)两大类[11-12],其中FAD2和FAD 3在内质网中,FAD6、FAD 7和FAD 8在脂质中[13-14]。FAD8属于ω-3脂肪酸脱氢酶,是ALA合成的关键酶[15-17],此酶在LA(18∶2)脂肪酸链的距甲基端的第三个碳原子处引入双键生成ALA(18∶3)[18]。
花生(Arachis hypogaea L.)是世界第四大油料作物,花生籽粒含油量约占种子干重的44.27%~53.86%;而其储藏油脂中80%左右为不饱和脂肪酸(其中油酸36%~67%,亚油酸15%~43%)[19]。目前,花生ω-6脂肪酸脱氢酶基因已被克隆,且已成功将AhFAD2A和AhFAD2B转入大肠杆菌和酿酒酵母中,诱导表达并分析其脱氢酶活性。但是,对于花生ω-3多不饱和脂肪酸代谢合成以及相关酶的研究还未见报道。牛丽红等成功克隆拟南芥和莱茵衣藻中的ω-3脂肪酸脱氢酶基因,在毕赤酵母中诱导表达,并且分析其生物活性[20-21]。
本研究生在物信息学分析基础上,利用电子克隆结合RT-PCR方法克隆花生ω-3脂肪酸脱氢酶基因(FAD8),构建ω-3脂肪酸脱氢酶的真核表达载体,在毕赤酵母中表达,分析其生物学功能,为花生不饱和脂肪酸代谢的研究奠定理论基础。
1 材料与方法
1.1 材料
1.1.1 植物材料
花生(Arachis hypogaea L.)品种为白沙 1016,由山东省花生研究所提供。
1.1.2 菌株与质粒
大肠杆菌(Escherichia coil)DH5α、毕赤酵母(Pichia pastoris)GS115、pPIC3.5K酵母游离型穿梭表达载体由本实验室保存,T载体pMD19-T购自TaKaRa公司,RNAsimple Total RNA试剂盒购自Tiangen公司。
1.1.3 试剂和培养基
LB液体培养基:NaCl 1%,Tryptone 1%,Yeast extract 0.5%,pH 7.0,0.1 MPa灭菌20 min。固体LB培养基:在液体LB培养基中加入琼脂,浓度为1.3%。毕赤酵母YDB基础培养基、RDB筛选培养基及BMGY和BMMY诱导表达培养基等配置方法参照Invitrogen公司毕赤酵母操作说明进行。
抗生素:氨苄青霉素100 mg·mL-1,卡那霉素100 mg·mL-1,-20 ℃保存备用。
Taq DNA聚合酶、Ex Taq DNA聚合酶、限制性内切酶及T4DNA连接酶等购自TaKaRa公司;引物由上海生工生物工程公司合成;2×Taq Mix PCR扩增试剂购自北京博迈得生物技术有限公司;DNA回收试剂盒、质粒小量提取试剂盒等购自Axygen公司;Trizol试剂购自Invitrogen公司;其他均为市售国产、进口分析纯试剂。
1.2 方法
1.2.1 ω-3脂肪酸脱氢酶基因的电子克隆
采用DNAMAN软件比对GenBank中下载的9种植物ω-3脂肪酸脱氢酶基因序列,选取保守片段作为种子序列,利用BioEdit软件在本实验室花生不同时期的转录组测序数据库中调取同源性较高的序列片段,结合Peanut DB花生数据库(http://www.bioinfolab.org/txid3818v1)(世界上第一个花生数据库网站,由本实验室完成花生转录组测序,与美国Miami University Dr Liang Chun实验室合作建立)中对于花生ω-3脂肪酸代谢途径中相关酶的序列信息,得到候选序列。根据NCBI、EBI、GenBank、EXPACY和Soflberry等网站提供的各种在线生物信息学软件进行综合分析预测,并与同类植物已克隆的ω-3脱氢酶进行比较分析,克隆出花生ω-3脂肪酸脱氢酶基因全长序列。引物由上海Sangon合成:
AhFAD85f:5'TACGTAACCATGGCAACATGG GTCTTATC 3'
AhFAD83r:5'GCGGCCGCTTAAATTGATGTA GAAGAGCC 3'
1.2.2 AhFAD8基因全长cDNA序列的获得
花生未成熟种子总RNA的提取参照Trizol说明书进行,使用RNAsimple Total RNA Kit纯化分离mRNA。取1 μg所提总RNA为模板,Oligo(dT)为反转录引物,70℃保温4 min后立即冰浴,然后分别加入 5×primescript buffer 4 μL,dNTP,RNase inhibitor,M-MLV各0.5 μL,加DEPC处理水至20 μL,42℃反应90 min,合成第一链cDNA。以AhFAD85f、AhFAD83r为引物,以反转录产物作为模板进行PCR扩增,扩增条件:94℃预变性5 min;94℃变性1 min,58℃ 30 s,72℃延伸30 s,35个循环;最后72℃延伸10 min。
1.2.3 AhFAD8基因克隆、测序
将PCR产物回收并克隆到pMD19-T载体中,并转化大肠杆菌DH5α,蓝白斑筛选和酶切鉴定阳性克隆,挑取正确的阳性克隆进行测序。序列测定由北京天一辉远生物科技有限公司完成。
1.2.4 AhFAD8基因表达载体的构建
利用酶Sna B I和NotⅠ将目的基因片段从pMDFAD8上切下插入pPIC3.5K,得到重组表达载体。大肠杆菌感受态细胞的制备、载体构建、转化和重组质粒的筛选和鉴定均参见文献[19]。
1.2.5 毕赤酵母细胞的转化及阳性克隆的鉴定
用限制性内切酶Sna B I分别对pPIC3.5K及重组载体进行线性化处理,然后采用电穿孔法转化毕赤酵母GS115感受态细胞。电转化条件参见文献[20],取上清液为模板,以AhFAD85f和AhFAD83r为引物进行PCR鉴定,检验目的基因是否已整合到毕赤酵母基因组DNA中。从RDB培养基上挑取生长较好的转化子,进一步转接到G418浓度分别为1.0、2.0、4.0 mg·mL-1的平板上进行筛选,得到高拷贝转化子。
1.2.6 酵母工程菌的表达
分别挑取单阳性克隆,接种于装有BMGY培养基中,28℃,220 r·min-1培养至OD600=3.0;收集菌体,菌体重悬于BMMY培养基中,甲醇终浓度为0.5%,28℃,220 r·min-1诱导表达;每24 h向培养基中添加100%甲醇至其终浓度为0.5%;诱导72 h后,离心收集菌体,菌体用无菌水洗涤3次。
1.2.7 重组基因表达产物样品的制备
重组表达产物样品的制备方法详见文献[20]。
1.2.8 重组基因表达产物样品气相色谱(GC)分析
采用Agilent 6890N气相色谱仪,色谱柱为VF-23MS,规格为30 m×0.25 mm×0.25 μm,分流比100∶1,进样口温度260℃,检测器温度260℃,程序升温:110℃3 min,以4℃·min-1的速度上升至220℃保温15 min。
2 结果与分析
2.1 花生FAD8基因的cDNA克隆与序列分析
扩增得到花生FAD8基因的cDNA,电泳检测到一条约1.4 kp的条带(图略),测序结果表明该cDNA片段含有1 368 bp,由此推测该基因编码456个氨基酸,与大豆(Glycine max)FAD8(NM0012516 80.1)序列相似性最高,为88%;与大豆等10个物种的FAD8氨基酸序列作多重比对,建立11个物种的FAD8脱氢酶的系统发育树如图1所示,结果表明花生FAD8与(Glycine max)和豇豆(Vigna uiculata chloroplast)进化关系最近,而与毕赤酵母(Pichia pastoris)进化关系最远。
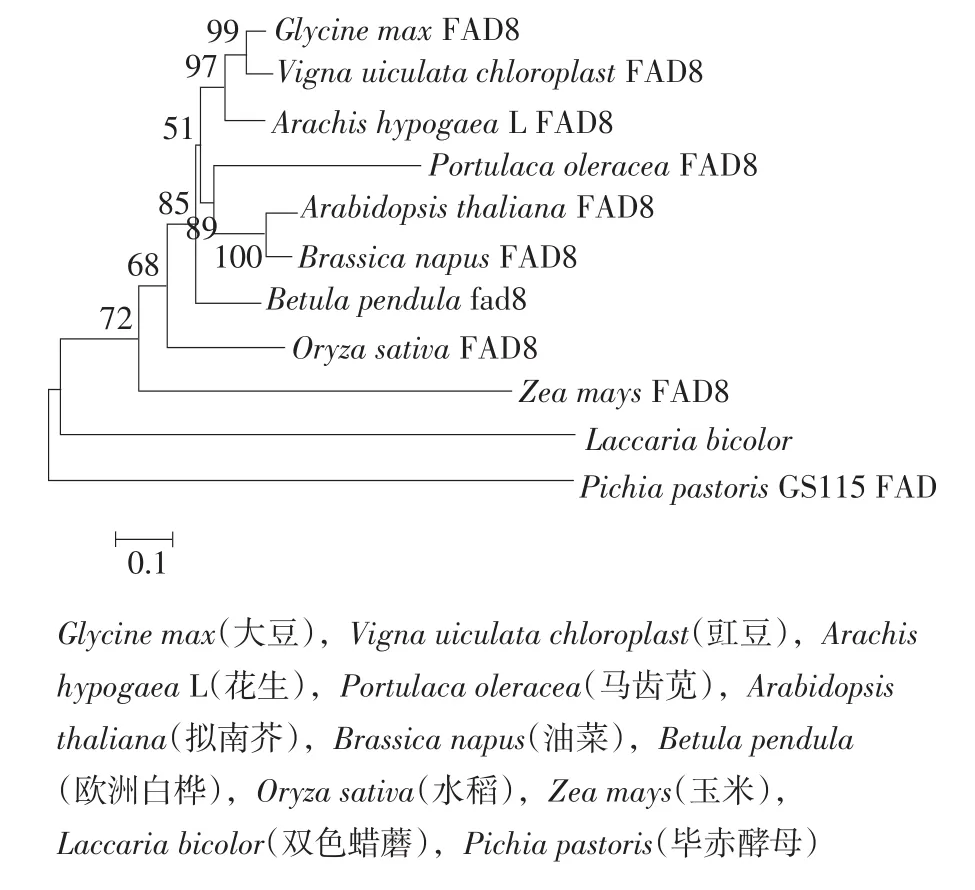
图1 11种ω-3脱氢酶的分子系统发育Fig.1 Molecular phylogenetic tree of 11 kinds of ω-3 FADs
2.2 表达载体的构建及阳性克隆的鉴定
构建表达载体获得重组质粒p3.5K8(见图2),挑取阳性克隆,酶切鉴定显示,p3.5K8经Sna BⅠ和NotⅠ双酶切分别产生9.3 kb及1.4 kb两条带,经Sna BⅠ单酶切仅有10.7 kb一条带;空载体pPIC3.5K经同样酶切仅有9.3 kb一条带(见图3),说明AhFAD8基因已成功克隆到pPIC3.5K中。
2.3 AhFAD8基因在毕赤酵母中的表达
以引物AhFAD85f与AhFAD83r对转化子进行鉴定,结果表明目的基因已整合入毕赤酵母基因组中。RT-PCR检测结果说明AhFAD8基因在毕赤酵母中能够正常转录(见图4)。
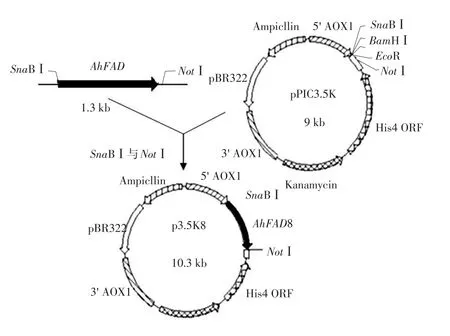
图2 p3.5K8质粒的构建Fig.2 Construction of p3.5K8 plasmid
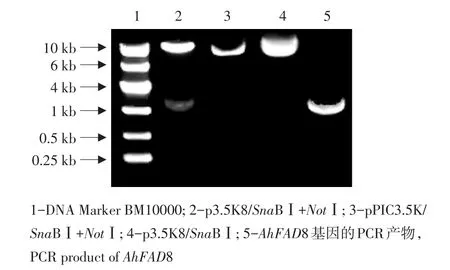
图3 重组质粒p3.5K8的酶切及PCR分析Fig.3 Restrictive digestion and PCR analysis of recombinant plasmid p3.5K8
2.4 花生FAD8脱氢酶的活性分析
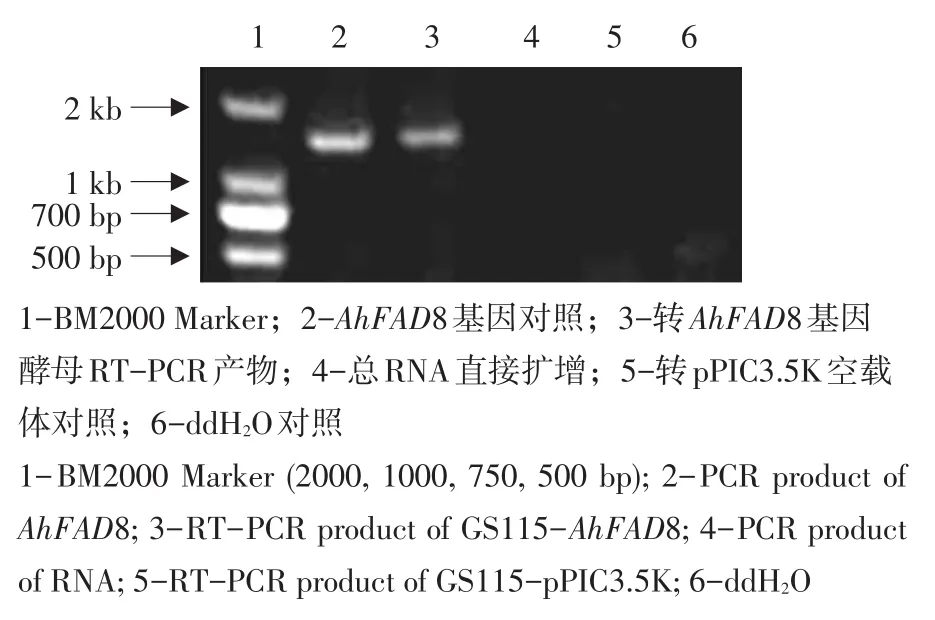
图4 转脱氢酶基因毕赤酵母RT-PCR检测结果Fig.4 RT-PCR Result of Pichia pastoris transformants with desaturase genes
毕赤酵母工程菌经细胞破碎、提取、对总脂肪酸进行甲酯化后,经GC检测,对图谱进行分析,得到毕赤酵母每种具体的脂肪酸的物质的量含量,并依照去饱和率计算4个转化子亚油酸的去饱和率及平均去饱和率,饱和率(%)=产物含量/(产物含量+底物含量)×100%,结果见表1,结果显示转AhFAD8的毕赤酵母比转空载体pPIC3.5K的毕赤酵母亚油酸的平均去饱和率高。如图5所示,图5A为转空载体对照,图5B为转化含AhFAD8基因的毕赤酵母中亚油酸的平均去饱和率,可见转入AhFAD8基因后亚油酸的平均去饱和率升高25.5%,证明在毕赤酵母中表达的花生FAD8脱氢酶基因具有脱氢活性。

表1 毕赤酵母中亚油酸的去饱和率Table1 Desaturation rate of LA in Pichia pastoris
3 讨论与结论
本研究通过多种植物以及毕赤酵母的ω-3脱氢酶基因序列比对分析得到FAD8脱氢酶基因的保守区,利用本实验室花生不同时期的转录组数据以及Peanut BD数据库的信息,结合RT-PCR方法,首次克隆花生AhFAD8脱氢酶全长cDNA,并在毕赤酵母中成功表达该基因,通过不饱和脂肪酸分析证实该基因表达产物具有一定的ω-3脱氢酶活性,为花生以及其他油料作物脂肪酸代谢合成的相关基因研究创造条件。
本研究采用外源蛋白表达系统为巴斯德毕赤酵母(Pichia pastoris),克服酿酒酵母(Saccharomyces cerevisiae)分泌效率低、表达菌株不够稳定、表达质粒易丢失等缺陷[22-24]。其本身含有丰富的油酸和亚油酸,可在不加其他底物的情况下,直接用于脱氢酶基因的生物学功能的验证[25-27]。通过对脂肪酸脱氢酶的真核表达,摸索出利用毕赤酵母的真核表达的技术路线,为其他脱氢酶基因的真核异源表达提供技术支持。
本研究中AhFAD8基因在毕赤酵母中表达,亚油酸去饱和率由25.9%上升到32.5%,上升25.5%,证实此酶能异源表达,而且具有一定脱氢酶活性。但是,毕赤酵母中ω-3不饱和脂肪酸合成代谢途径与植物有一定区别,缺少植物中特有的调控机制,从而影响脂肪酸脱氢酶的表达及活性,后续研究拟将该脱氢酶基因转入模式植物拟南芥,并研究其生物活性。
[1]Petrie J R,Singh S P.Expanding the docosahexaenoic acid food web for sustainable production:Engineering lower plant pathways into higher plants[J].Aob Plants,2011(11):1-11.
[2]Geleijnse J M,Goede J D,Brouwer I A.Alpha-linolenic acid:Is it essential to cardiovascularhealth[J].Curr Atheroscler Rep,2010(12):359-367.
[3]Yazawa H,Iwahashi H,Kamisaka Y,et al.Heterologous production of dihomo-linolenic acid in Saccharomyces cerevisiae[J].Applied and Environmental Microbiology,2007,73:6965-6971.
[4]Wu Q,Liu T,Liu H.Unsaturated fatty acid:metabolism,synthesis and gene regulation[J].African Journal of Biotechnology,2009(8):1782-1785.
[5]Senadheera S D,Turchini G M,Thanuthong T,et al.Effects of dietary α-linolenic acid(18:3n-3)/linoleic acid(18:2n-6)ratio on fatty acid metabolism in murray cod(Maccullochella peelii peelii)[J].Agric Food Chem,2011,59(3):1020-1030.
[6]Andre V,Lagunas B,Collados R,et al.The GmFAD7 gene family from soybean:identification of novel genes and tissue-specific conformations of the FAD7 enzyme involved in desaturase activity[J].Journal of Experimental Botany,2010,61:3371-3384.
[7]Torres-Franklin M,Repellin A,Huynh V,et al.Omega-3 fatty acid desaturase(FAD3,FAD7,FAD8)gene expression and linolenic acid content in cowpea leaves submitted to drought and after rehydration[J].Environmental and Experimental Botany,2009,65:162-169
[8]Ward O P,Singh A.Omega-3/6 fatty acids:Alternative sources of production[J].Process Biochemistry,2005,40:3627-3652.
[9]Kishore K,Sinha S K,Kumar R,et al.Isolation and characterization of microsomal ω-6-desaturase gene from soybean[J].Indian Journal of Experimental Biology,2007,45:390-397.
[10]Andreu V,Collados R,Pilar S,et al.In situ molecular identification of the plastid ω-3 fatty acid desaturase FAD7 from soybean:evidence of thylakoid membranelocalization[J].Plant Physiology,2007,145:1336-1344.
[11]Pha A T,Lee J D,Shannon J G,et al.Mutant alleles of FAD2-1A and FAD2-1B combine to produce soybeans with the high oleic acid seed oil trait[J].BMC Plant Biology,2010(10):195-217.
[12]Knoll J E,Ramos M L,Zeng Y,et al.Tilling for allergen reduction and improvement of quality traits in peanut(Arachis hypogaea L.)[J].BMC Plant Biology,2011,11:81-93.
[13]Chi X Y,Yang Q L,Pan L J.Isolation and characterization of fatty acid desaturase genes from peanut(Arachis hypogaea L)[J].Plant Cell Rep,2011,30:1393-1404.
[14]吴静,杨利敏,施振声,等.ω-3多聚不饱和脂肪酸脱氢酶的真核表达及鉴定[J].中国生物工程杂志,2007,27(10):44-47
[15]Yu S L,Pan L J,Yang Q L,et al.Comparison of the Δ12 fatty acid desaturase gene between high-oleic and normal-oleic peanut genotypes[J].Journal of Genetics and Genomics,2008,35:679-685.
[16]Kim O Y,Lim H H,Yang L I,et al.Fatty acid desaturase(FADS)gene polymorphisms and insulin resistance in association with serum phospholipid polyunsaturated fatty acid composition in healthy Korean men:Cross-sectional study[J].Nutrition and Metabolism,2011(8):24-35.
[17]潘丽娟,禹山林,杨庆利,等.花生△12-脂肪酸脱氢酶基因的克隆及序列分析[J].花生学报,2007,36(3):5-10.
[18]周丽侠,唐桂英,陈高,等.花生AhFAD2基因的多态性及其与籽粒油酸/亚油酸比值间的相关性[J].作物学报,2011,37(3):415-423.
[19]Sambook J,Russell D W.Molecular cloning:A laboratory manual[M].New York:Cold Spring Harbor Laboratory Press,2001.
[20]陈文波,李卫国.两种鲤鱼胰凝乳蛋白酶原基因的电子克隆及分析[J].生物技术通报,2011(1):124-129.
[21]牛丽红,耿丽丽,张蕊,等.莱茵衣藻ω-3脂肪酸脱氢酶基因在毕赤酵母中的表达与活性分析[J].生物技术通报,2011(12):96-101.
[22]Yin D M,Cui D Q,Jia B.Construction of a high-efficient expression vector of Δ12 fatty acid desaturase in peanut and its prokaryotical expression[J].Journal of Genetics and Genomics,2007,34:81-88.
[23]Liu X Q,Lin F,Wang L,et al.The in silico map-based cloning of Pi36,a rice coiled-coil-nucleotide-binding site-leucine-rich repeat gene that confers race-specific resistance to the blast fungus[J].the Genetics Society of America,2007,176:2541-2549.
[24]Asada H,Uemura T,Yurugi-Kobayashi T,et al.Evaluation of the Pichia pastoris expressionsystem for the production of GPCRs for structural analysis[J].Microbial Cell Factories,2011(10):24-36.
[25]Salamin K,Sriranganadane D,Lechenne B,et al.Secretion of an endogenous subtilisin by Pichia pastoris strains GS115 and KM71[J].Applied and Environmental Microbiology,2010(7):4269-4276.
[26]张洪涛,单雷,全先庆,等.花生△12-脂肪酸脱氢酶基因AhFAD2B在酿酒酵母中的表达及功能分析[J].花生学报,2006,935(1):1-7.
[27]Hao W J,Li G Q,Xu M Y,et al.Induction and expression of T4 lysozyme gene in Pichia pastoris[J].Journal of Chinese Pharmaceutical Sciences,2007,16:33-37.
[28]Jiang Z B,Gao B,Ren R,et al.Efficient display of active lipase LipB52 with a Pichia pastoris cellsurface display system and comparison with the LipB52 displayedon Saccharomyces cerevisiae cell surface[J].BMC Biotechnology,2008(8):4-11.
[29]殷冬梅,崔党群.花生油酸脱氢酶基因在酿酒酵母中的高效表达[J].植物生理学通讯,2007,43(4):697-700.

