129株新型隐球菌基因型及感染人群研究
戚建明,陈江汉,杨 军
2.第二军医大学长征医院。
1999至2006年,国内期刊上发表了224篇与新型隐球菌病相关的论文,涉及到2 850例感染新型隐球菌的患者,仅69例(2.4%)与艾滋病有关[1-6]。现对我国129株新型隐球菌临床分离株进行流行病学、分子生物学、实验动物学分析,以明确我国新型隐球菌的主要感染人群。
材料与方法
一、菌株来源
129株新型隐球菌临床株从不同的新型隐球菌病患者脑脊液中分离获得,这些患者来自于我国中东部地区的18个省、直辖市,菌株保存在上海长征医院皮肤科新型隐球菌专业实验室,以“长征”加数字命名(如长征114,长征186),所有菌株接种到L-刀豆氨酸-甘氨酸-溴百里酚蓝培养基上,通过培养显色区分新型隐球菌和格特隐球菌种属[7]。标准株148(VNⅠ血清型A)、标准株63(VNB血清型A)、标准株626(VNⅡ血清型A)、标准株628(VNⅢ血清型D)、标准株629(VNⅣ血清型AD)和标准株179(VGⅠ血清型B)、标准株178(VGⅡ血清型B)、标准株161(VGⅢ血清型B)、标准株779(VGⅣ血清型C)和标准株99(血清型A)则由美国国立卫生研究院 Kwon-Chung教授提供[8]。在研究过程中,所有菌株均25℃保存在YEPD培养基中(1%酵母抽提物,2%蛋白胨,2%葡萄糖)。
二、方法
(一)血清分型和MAT分型 血清分型采用Cryptocheck试剂盒(latron,日本东京),MAT分型采用 Kwon-Chung等[9]、Lengeler等[10]报道的显色杂交法进行。上述方法仍不能明确的,则以MAT等位基因为引物行PCR分析。
(二)DNA提取 按照 Wang等[11]的氯化苄法操作,4℃保存抽提出的DNA。
(三)基因分型 基因分型方法包括M13微卫星DNA引物PCR指纹图谱和多位点序列分型(MLST)DNA序列分析。M13的引物序列为:5′-GAGGGTGGCGGTTCT-3′[12],MLST 涉及位于新型隐球菌7个不同染色体上的8个不同基因,分别是 CAP10、GPD、IGS1、LAC1、PLB1、SOD1、TEF1和 URE1[13]。
PCR反应体系:反应总体积50μL,模板DNA 50~100 ng,上下游引物各 0.5 mmol/L,1.0U DNAzymeⅡ聚合酶,1.5mmol/L 氯化镁,200 mmol/L dNTP;反应条件:94℃变性1 min;95℃2 min,57 ℃ 1 min,72 ℃ 3 min,40个循环,72 ℃7 min。纯化采用 QIAquick PCR Purification试剂盒(Qiagen,Valencia,CA,USA),采用凝胶电泳确认扩增产物。
(四)毒力研究 随机选取3株来自中国的新型隐球菌临床株(长征114、长征186和长征193),3株血清A型标准株(标准株99、标准株148和标准株626)进行毒力比较,将上述菌株配成含5×107cells的悬液,分别滴鼻感染9周龄雌性BALB/c小鼠,监测生存状况。
(五)无明显免疫缺陷定义 已经明确的新型隐球菌感染危险因素有:HIV感染、恶性肿瘤、肝硬化、器官移植、终末期肾衰竭、自身免疫性疾病、糖尿病、特发性CD4+T细胞减少症、结节病、长期使用皮质类固醇激素或其他免疫疗法等[14],无上述任何易感因素人群定义为无明显免疫缺陷人群。
结 果
一、感染人群
129株新型隐球菌临床株是1980年至2006年从上海长征医院诊治过的不同患者脑脊液中分离到,大部分患者主要表现出头痛、恶心、呕吐、发热等中枢神经系统感染症状,部分患者有不同程度意识障碍。患者经腰椎穿剌后,取脑脊液标本送上海长征医院皮肤科新型隐球菌专业实验室,取离心后底部脑脊液,经墨汁染色后镜检,主要观察菌株的形状及荚膜情况,同时行乳胶凝集试验,再取适量脑脊液于YEPD培养基上25℃培养,培养时间为1个月,在此时间段内生长的株菌,再行墨汁染色镜检、乳胶凝集试验、咖啡酸试验、尿素酶试验、37℃生长试验,以培养后的检查结果为准,本次研究采用分子生物学的方法对129株临床株再次确认无误。这些新型隐球菌患者来自中国中东部18个省、直辖市。2002年之前,每例新型隐球菌病患者常规行细胞免疫和体液免疫检测,有异常者均行HIV血清学检测,2002年以后,所有的新型隐球菌感染者均检测相关免疫学指标,包括HIV血清学、CD4+细胞水平等。
依据患者的临床资料及无明显免疫缺陷的定义,129株临床株中,70.5%(91/129)分离自无明显免疫缺陷的患者,8.5%(11/129)分离自艾滋病患者,20.9%(27/129)分离自有其他基础疾病的非HIV感染患者。129株临床株均为MATα型,其中120株为血清A型新型隐球菌,9株为血清B型格特隐球菌。120株血清A型新型隐球菌来自中国中东部16个省、直辖市,84株(70.0%)分离自无明显免疫缺陷患者,27株(22.5%)分离自除HIV外的其他危险因素的患者,只有9株(7.5%)分离自艾滋病患者;9株血清B型格特隐球菌,1株来自山东,1株来自上海,5株来自浙江省的桉树种植区,其他均来自气候暖和的中国东南部。9株血清B型格特隐球菌中,2株分离自艾滋病患者,7株分离自无明显免疫缺陷患者。
二、基因分型
120株血清A型新型隐球菌临床株具有一致的M13指纹图谱,将其中的12株与各标准株进行对比,其主要条带与VNⅠ型标准株相同,但次要条带不同于已知VNⅠ各亚型,属于一种新的亚型,我们将其命名为VNⅠc型(图1)。
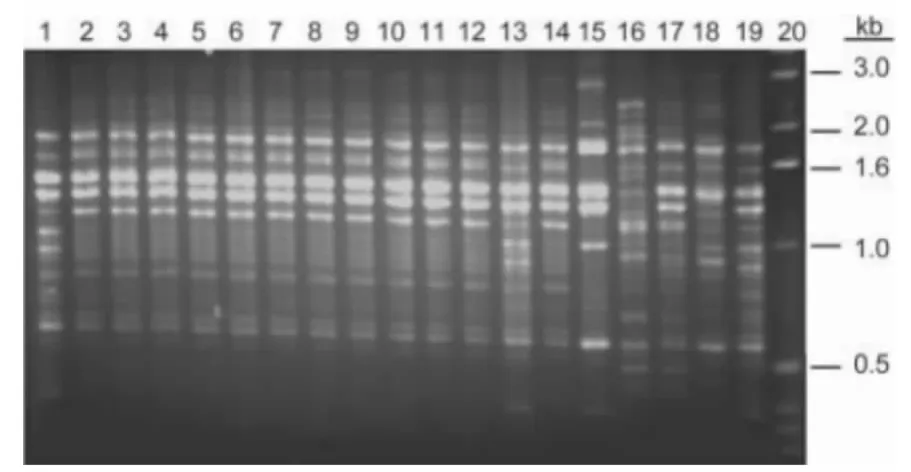
图1 12株我国新型隐球菌临床株和标准株的M13分析DNA指纹图谱Figure 1 DNA fingerprint patterns of 12 Cryptococcus neoformans clinical strains from China and the reference strains
在同其他菌株M13指纹图谱比对的过程中,我们发现来自美国 (美国8、美国48和美国5-36-17)、日本(日本1086和日本1088)、意大利743和马拉维212i的7株新型隐球菌也呈现出VNⅠc指纹图谱的特点(图2)。对1株大陆血清A型临床株和上述7株菌株进行8个基因(CAP10、GPD、LAC1、PLB1、SOD1、IGS1、TEF1和 URE1)MLST,分析发现,明显区别于VNⅠ标准株148。
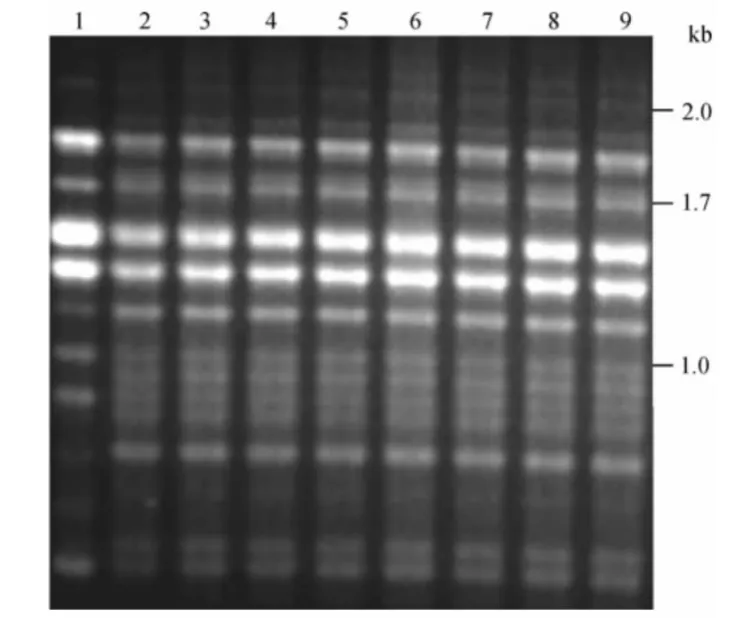
图2 8株VNⅠc型同VNⅠ型标准株148指纹图谱比较Figure 2 DNA fingerprint patterns of 8 VNⅠc Cryptococcus neoformans strains and the WM148 reference strains
三、毒力研究
毒力分析结果显示3株随机选择的中国新型隐球菌毒力低于标准株99,但明显强于VNⅠ和VNⅡ标准株(图3)。
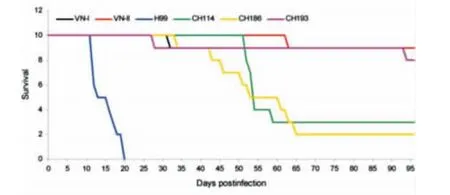
图3 新型隐球菌毒力研究结果Figure 3 Virulence research of Cryptococcus neoformans strains
讨 论
此次研究分析了我国新型隐球菌临床株流行病学特征,与巴西、澳大利亚、美国等其他国家血清A型新型隐球菌的基因多样性相比显示[15-16],我国120株血清A型临床株显示出明显的遗传同质性,这点在M13指纹图谱和MLST结果均有体现。印度有研究者报道57株新型隐球菌临床株遗传多样性与中国相似[17],但有1株是血清D型。
值得注意的是,尽管我国已确诊相当多数量的艾滋病患者,但我国新型隐球菌病患者中,大部分无免疫缺陷或其他基础疾病,我国大陆129株临床株感染者中,只有11株(8.5%)分离自艾滋患者,91株(70.5%)分离自无明显免疫缺陷患者,这与欧美超过80%、非洲超过69%隐球菌病感染者为艾滋病患者的报道显著不同[18-20]。同样的情况在澳大利亚、新西兰、印度也有报道,在澳大利亚和新西兰[21],多数新型隐球菌病患者为非艾滋病患者,但有明显免疫功能低下,这点与此次研究有所不同。印度报道41%新型隐球菌患者为HIV感染者[17],但未提及其他59%的非HIV感染患者中是否有其他基础疾病。
在此研究中,我们认为70.5%新型隐球菌病患者无明显免疫缺陷,基因因素可能在这些无明显免疫缺陷的新型隐球菌病患者感染过程中扮演了重要角色,120株血清A型新型隐球菌临床株具有一致的M13指纹图谱,其主要条带同VNⅠ型标准株相同,但次要条带不同于已知的VNⅠ各亚型,属于一种新的亚型,我们将其命名为VNⅠc型,MLST分析也显示,我国VNⅠc亚型临床株和前述7株菌株明显区别于VNⅠ标准株 WM148,这7株VNⅠc株来自4个大洲[13],这表明VNⅠc型在世界范围内广泛分布,不是中国特有的,但这7株中是否大部分也分离自无明显免疫缺陷者就难以追寻了。
一般情况下,无明显的免疫缺陷或相关基础疾病人群隐球菌病的发生率较低[14]。在美国,加利福尼亚州每年免疫功能正常者的隐球菌病发病率为0.2/10万[22],佐治亚州的亚特兰大为0.9/10万,阿拉巴马州为0.93/10万[19]。此次我们只分析了129株新型隐球菌临床株,因此难以确定VNⅠc基因亚型新型隐球菌在我国分布是否最广泛,如对环境株大量分离并进行类似的基因分型,将有利于更深入地了解VNⅠc型在中国的分布,进一步探索VNⅠc型新型隐球菌在无明显免疫缺陷人群感染过程中的意义。
东南亚分布最广的格特隐球菌基因型是VGⅠ,此次从我国东南部分离到的格特隐球菌主要为VGⅠ型再次证明以上结论。此次研究中格特隐球菌菌株只有9株,数量较少,但其在艾滋病相关隐球菌感染者中占的比例高于预期,国外学者报道,除非洲某些地区外,格特隐球菌很难从艾滋病患者身上分离到[14],澳大利亚格特隐球菌分布广泛,但从新型隐球病合并艾滋病患者身上分离到的临床株中,47株中只有1株是格特隐球菌[21]。
在我们前期研究中,分离到1株尿素酶阴性的B-4578临床株,在小鼠身上进行的毒力研究显示,其与标准株99的高致病性相当[23],而B-4578临床株也是VNⅠc型,故推测我国VNⅠc型可能具有与标准株99相似的高致病性。小鼠毒力研究显示,3株来自中国不同省份VNⅠc型临床株的致病性比VNⅠ更高,但低于标准株99,这表明不同的VNⅠc型菌株对动物致病性存在明显差异,新型隐球菌对人类和实验动物是否有相同程度的致病性目前尚不明确,本研究结果与美国12株在动物和人体的致病性无明显的相关性[24],在BALB/c小鼠身上,美国12株是致命性最强的血清D型菌株之一,但是它只引起人类局部慢性感染,并不扩散,经两性霉素B治疗后基本无传播或复发。
有学者研究发现,澳大利亚的原住民新型隐球菌感染率高于白种人[25],洛杉矶也曾报道西班牙裔美国人新型隐球菌感染率是白人的2倍[26],此次129株新型隐球菌临床株均来自中国大陆患者,是否存在类似上述的种族差异有待进一步研究。流行病学分析显示我国大部分临床株分离自非艾滋病患者,可能与分离到菌株的患者来源也有一定的关系,虽有部分新型隐菌病患者来自河南、云南、新疆等艾滋病发病率较高省份,大部分患者来自艾滋病感染率较低的省市。此外,在我国,患者一旦查出HIV血清阳性,将很快隔离,国内外对艾滋病患者的处理方式差异是否为感染人群差异的重要原因,我们下一步将开展更深入的研究,进一步明确。
综上所述,此研究揭示了我国新型隐球菌惊人的遗传同质性,血清A型新型隐球菌主要基因型为VNⅠc,血清B型格特隐球菌主要基因型为VGⅠ,且我国无明显免疫缺陷人群新型隐球菌高发病率与国外报道形成明显的对比,基因亚型的不同可能是导致我国与其他国家地区新型隐球菌感染人群差异的重要因素。
[1] 黄艮彬,张志坚,慕容慎行,等.32例隐球菌性脑膜炎临床分析[J].临床神经病学杂志,2001,15(6):371-372.
[2] 吴成翰,王华燕,杨渤生,等.46例隐球菌性脑膜炎病人的疗效分析[J].中华神经科杂志,2002,35(2):102.
[3] 朱利平,潘孝彰,石尧忠,等.艾滋病合并隐球菌性脑膜炎、隐球菌性败血症一例[J].中华传染病杂志,2004,22(5):301.
[4] 郭爱华,胡学强.新型隐球菌颅内感染101例临床特点及诊断[J].中华神经科杂志,2005,38(7):445-447.
[5] 顾菊林,温海,陈江汉,等.隐球菌性脑膜炎39例临床分析[J].临床皮肤科杂志,2005,34(12):801-803.
[6] 陈江汉,温海,吴建华,等.两性霉素B治疗隐球菌性脑膜脑炎的不良反应及其处理[J].第二军医大学学报,2005,26(4):456-457.
[7] Kwon-Chung KJ,Polacheck I,Bennett JE.Improved diagnostic medium for separation of Cryptococcus neoformans var.neoformans(serotypes A and D)and Cryptococcus neoformans var.gattii(serotypes B and C)[J].J Clin Microbiol,1982,15(3):535-537.
[8] Perfect JR,Lang SD,Durack DT.Chronic cryptococcal meningitis:a new experimental model in rabbits[J].Am J Pathol,1980,101(1):177-194.
[9] Kwon-Chung KJ,Bennett JE,Rhodes JC.Taxonomic studies on Filobasidiella species and their anamorphs[J].Antonie Van Leeuwenhoek,1982,48(1):25-38.
[10] Lengeler KB,Cox GM,Heitman J.Serotype AD strains of Cryptococcus neoformans are diploid or aneuploid and are heterozygous at the mating-type locus[J].Infect Immun,2001,69(1):115-122.
[11] Wang S,Miao X,Zhao W,et al.Genetic diversity and population structure among strains of the entomopathogenic fungus,Beauveria bassiana,as revealed by inter-simple sequence repeats(ISSR)[J].Mycol Res,2005,109(Pt 12):1364-1372.
[12] Cogliati M,Allaria M,Tortorano AM,et al.Genotyping Cryptococcus neoformans var.neoformans with specific primers designed from PCR-fingerprinting bands sequenced using a modified PCR-based strategy[J].Med Mycol,2000,38(2):97-103.
[13] Litvintseva AP,Thakur R,Vilgalys R,et al.Multilocus sequence typing reveals three genetic subpopulations of Cryptococcus neoformans var.grubii(serotype A),including a unique population in Botswana[J].Genetics,2006,172(4):2223-2238.
[14] Kwon-Chung KJ,Bennett JE.Medical mycology[M].Philadelphia:Lea &Febiger;1992.
[15] Meyer W,Marszewska K,Amirmostofian M,et al.Molecular typing of global isolates of Cryptococcus neoformans var.neoformans by polymerase chain reaction fingerprinting and randomly amplified polymorphic DNA-apilot study to standardize techniques on which to base a detailed epidemiological survey[J].Electrophoresis,1999,20(8):1790-1799.
[16] Barreto de Oliveira MT,Boekhaut T,Theelen B,et al.Cryptococcus neoformans shows a remarkable genotypic diversity in Brazil[J].J Clin Microbiol,2004,42(3):1356-1359.
[17] Jain N,Wickes BL,Keller SM,et al.Molecular epidemiology of clinical Cryptococcus neoformans strains from India[J].J Clin Microbiol,2005,43(11):5733-5742.
[18] Dromer F,Mathoulin S,Dupont B,et al.Epidemiology ofcryptococcosis in France:a9 year survey (1985-1993).French Cryptococcosis Study Group[J].Clin Infect Dis,1996,23(1):82-90.
[19] Hajjeh RA,Conn LA,Stephen DS,et al.Cryptococcosis:population-based multistate active surveillance and risk factors in human immunodeficiency virus-infected persons.Cryptococcal Active Surveillance Group[J].J Infect Dis,1999,179(2):449-454.
[20] Moosa MY,Coovadia YM.Cryptococcal meningitis in Durban,South Africa:a comparison of clinical features,laboratory findings,and outcome for human immunodeficiency virus(HIV)-positive and HIV-negative patients[J].Clin Infect Dis,1997,24(2):131-134.
[21] Chen S,Sorrell T,Nimmo G,et al.Epidemiology and hostand variety-dependent characteristics of infection due to Cryptococcus neoformans in Australia and New Zealand[J].Clin Infect Dis,2000,31(2):499-508.
[22] Friedman GD.The rarity of cryptococcosis in northern California:the 10-year experience of a large defined population[J].Am J Epidemiol,1983,117(2):230-234.
[23] Varma A,Wu S,Guo N,et al.Identification of a novel gene URE2that functionally complements a urease negative clinical strain of Cryptococcus neoformans[J].Microbiology,2006,152(Pt 12):3723-3731.
[24] Kwon-Chung KJ,Hill WB.Sexuality and pathogenicity of Filobasidiella neoformans(Cryptococcus neoformans)[M]//Vanbreuseghem R,DeVroey C,editors.Sexuality and pathogenicity of fungi.New York:Masson;1981:243-250.
[25] Lo D.Cryptococcosis in the Northern Territory[J].Med J Aust,1976,2:825-828.
[26] Sorvillo F,Beall G,Turner PA,et al.Incidence and factors associated with extrapulmonary cryptococcosis among persons with HIV infection in Los Angeles County[J].AIDS,1997,11(5):673-679.

