稠油厌氧降解产甲烷菌群的组成及产甲烷特性
顾贵洲,张 强,刘春爽,胡恒宇,赵东风
(1.中国石油大学(华东)化学工程学院,山东 青岛266555;2.辽宁石油化工大学环境与生物工程学院,辽宁 抚顺113001)
经历二次和三次采油之后,许多老油气田面临废弃,尤其是一些稠油甚至超稠油油田,无论技术如何更新,仍有50%以上的原油无法回收,而油田一旦废弃,这些储量将永远失去[1]。残余低品位稠油微生物气化技术是近几年来国际上正在探索的延长油藏开发寿命的新技术,它是利用产甲烷微生物菌群在厌氧环境下将残余稠油转化为天然气,然后以天然气形式开采或作为战略性资源就地储备[2]。作为国内外研究热点,石油烃生物气化的可行性已经在实验室条件下得到证实[3-5]。研究表明[6-9],原 油 厌 氧 降 解 产 甲 烷 过 程 需 要由不同功能菌群共同参与、协同作用才能完成,这种协同作用主要分为2个阶段:(1)降解阶段,即石油烃降解为小分子有机物;(2)产气阶段,即小分子物质最终转化成甲烷等气体。虽然国内外对石油烃厌氧降解产甲烷研究较多[10-12],但原油气化开采一般针对经历过三采后的老油田,油品更稠、胶质与沥青质含量更高,开采难度更大,因而目前国际上以稠油为气化对象的还比较少。因此,富集获得高效稠油降解产甲烷混合菌群是残余低品位稠油微生物气化技术的关键。
作者从胜利油田某区块油井采出水中筛选获得一组高效稠油降解产甲烷混合菌群,考察混合菌群的组成,并探讨其产甲烷特性,以期为残余低品位稠油微生物气化奠定基础。
1 实验
1.1 样品来源、仪器与培养基
实验样品来源于胜利油田某区块油井采出水,该区块地质信息见表1。采出液通过采样井的井口阀门收集到10L采样桶中,待采样桶完全被油水混合物充满后,密封,快速送回实验室4℃下保存。
通用突变检测系统,美国DCode;凝胶成像系统,美国伯乐;Allegra 25R型高速冷冻离心机,美国Beckman;GC-3800型气相色谱仪,美国 Varian;OIL-510型全自动红外分光测油仪,北京华夏科创仪器技术有限公司。
无机盐培养基(g):K2HPO45.0,KH2PO45.0,NH4Cl 5.0,NaCl 10,MgCl22.0,CaCl20.1,H2O 1000mL,pH值7.0~7.5。
1.2 混合菌群的富集
以油田采出水为接种物富集培养,接种量按20%(体积分数)接种到厌氧瓶中,加入2mL原油,每个样品设置3个平行实验,于55℃生化培养箱暗室培养380d。
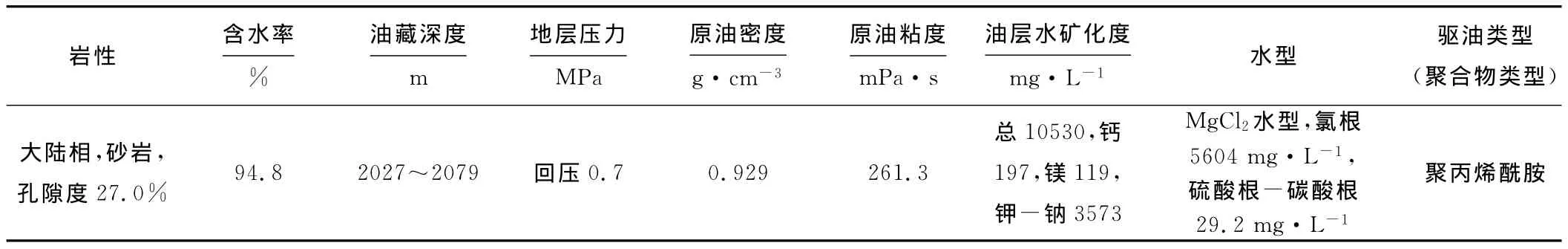
表1采样区块地质信息Tab.1 Geological information of sampling block
1.3 单菌株分离
运用亨盖特厌氧滚管技术[13,14]对混合菌群进行单菌株分离。具体方法为:在无机盐培养基中补加2%琼脂粉,分装于厌氧试管(4.5mL培养基/管),灭菌。置厌氧试管于沸水浴至琼脂完全熔化。转到55℃的水浴锅中,补加Na2S·9H2O至终浓度为0.03%(质量浓度);补加 NaHCO3至终浓度为0.2%(质量浓度)。滚管,将滚好的所有厌氧试管静置2h以上,使培养基表面的液体被充分吸收。接种后于55℃暗箱培养。
1.4 菌群结构分析
使用Tiangen细菌基因组DNA提取试剂盒(天根生化科技有限公司)提取混合菌群总DNA。以细菌16SrDNA V3 区 的 引 物 341F (5′-CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGGCCTACGGGAGGCAGCAG-3′)和 534R (5′-ATTACCGCGGCTGCTGG-3′)对 提 取 的 DNA 进 行PCR扩增,PCR反应条件:94℃预变性5min;94℃变性1min,55℃退火1min,72℃延伸1min,30个循环;最后72℃延伸10min。以Biorad Dcode系统对PCR产物进行DGGE分析,聚丙烯酰胺胶质量浓度为8%,变性剂浓度梯度为40%~60%。电泳完毕后,将胶置于含1mg·L-1EB的1×TAE缓冲溶液中染色,将染色后的凝胶用凝胶成像系统拍摄图像。将待测序条带切下,按前述PCR体系与条件进行扩增,送上海生工生物工程公司纯化测序,测序结果输入GenBank进行比对分析。
1.5 甲烷含量测定
用气相色谱仪测定富集厌氧瓶顶空气体中甲烷含量。进样体积为0.5mL,采用面积归一法定量。色谱条件如下:Al2O3色谱柱(50m×0.53mm×20μm);柱温从40℃开始,2min升至100℃,再以10℃·min-1的速率升至170℃,保持25min;进样口温度为100℃;FID检测器温度为150℃,载气为高纯氦气。
1.6 含油量测定
用全自动红外分光测油仪测定含油量。将《水质、石油类和动植物油的测定-红外光度法》(GB/T 16488-1996)稍作改进。培养基中的原油经混合菌降解后变为微小絮体,萃取液通过铺有无水Na2SO4的玻璃砂芯漏斗脱水时极易堵塞,因此采用循环水式真空泵将萃取液抽入抽滤瓶中。同时,为了保证脱水效果,将无水Na2SO4的厚度由标准中的10mm增至15 mm。
2 结果与讨论
2.1 稠油降解产甲烷菌群的组成
胜利油田某区块油井采出水在55℃下经过380d富集培养,获得一组稠油降解产甲烷混合菌群,命名为SL-7。通过亨盖特厌氧滚管技术从该菌群中分离获得2株可分离培养单菌,分别记为SL-7-1、SL-7-2。分别提取混合菌群SL-7与2株单菌的DNA同时进行DGGE分析,比较单菌在混合菌群中的位置,结果见图1。
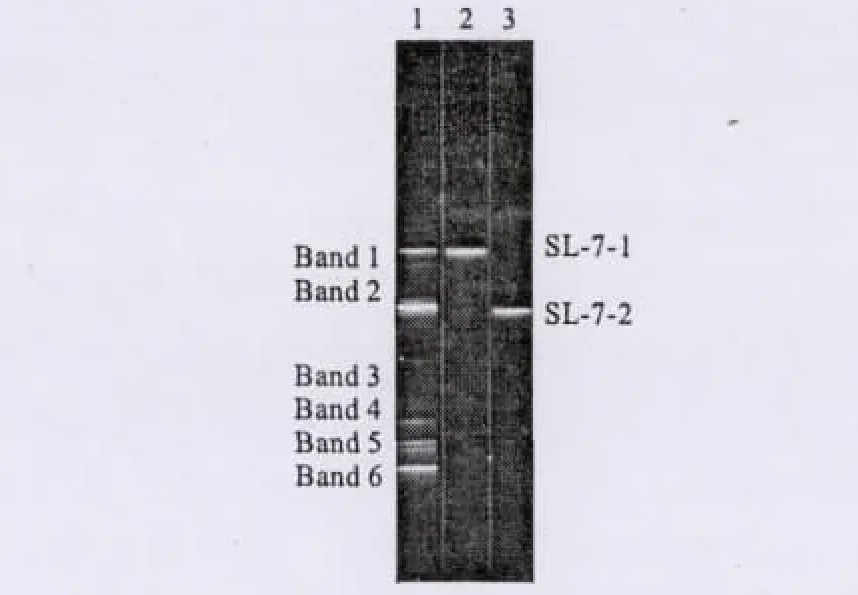
图1 混合菌群SL-7与单菌株的DGGE图谱Fig.1 The DGGE analysis of the microbial consortium SL-7and two single strains
由图1可知,混合菌群SL-7由6个主要的单菌株组成,并且2株降解单菌条带均能与SL-7中的条带相对应,SL-7-1对应Band 1,SL-7-2对应 Band 2。对主要条带进行测序分析,确定了这些菌株的可能分类,结果见表2。

表2混合菌群SL-7DGGE条带序列分析Tab.2 Sequence analysis of the microbial consortium SL-7from dominant DGGE bands
PCR-DGGE技术可以直观地反映微生物的群落结构[15],检测到不易分离培养的优势菌株,揭示混合菌群的结构组成。由表2可知,混合菌群SL-7包含4株不可培养细菌(Band 3、Band 4、Band 5、Band 6)和2株已分离获得的降解单菌(Band 1、Band 2)。单菌株SL-7-1与来自Firmiucutes(厚壁菌门)的Clostridium sp.XB90同源性为98%。研究表明,该类细菌能够厌氧降解烃类化合物或者利用烃降解的中间产物产生甲烷菌可以利用的前体物质[16]。单菌株SL-7-2与来自β-Proteobacteria(β-变性菌门)的 Achromobacter sp.BPZ11同源性为97%。
SL-7对稠油的降解可能是微生物降解和其中多株单菌共同代谢、共同作用的结果。油田采出水中存在着经自然选择优化过的混合菌群,其中不同菌株的功能和作用通过长期的演化稳定下来,在混合菌群SL-7中形成了一种较为稳定的细菌群落结构,一些不可分离培养的菌株(如Band 3、Band 4、Band 5、Band 6所代表的菌株)可能对稠油的降解起着重要的作用,但在目前培养条件下无法分离得到。因此,直接利用混合菌群来降解稠油,可以避免这些无法分离培养的关键菌株的丢失[17]。混合菌群SL-7将稠油中难以直接利用的大分子烃类降解为小分子烃类物质,完成稠油厌氧降解产甲烷的第一个阶段,为下一步产气阶段做准备。
2.2 稠油降解产甲烷菌群的产甲烷特性
采用气相色谱法测定富集厌氧瓶顶空气体,以面积归一法定量,结果如图2所示。
由图2可知,55℃下,混合菌群SL-7在380d的培养过程产生大量的甲烷气体。培养30d时就有少量的甲烷气体产生,随着培养时间的延长,甲烷产生量逐渐增加,培养270d时甲烷产生量达到最大值,为1110μmol,之后甲烷产生量保持相对稳定。混合菌群SL-7的产甲烷规律与微生物的生长周期相关,符合其生长代谢规律。
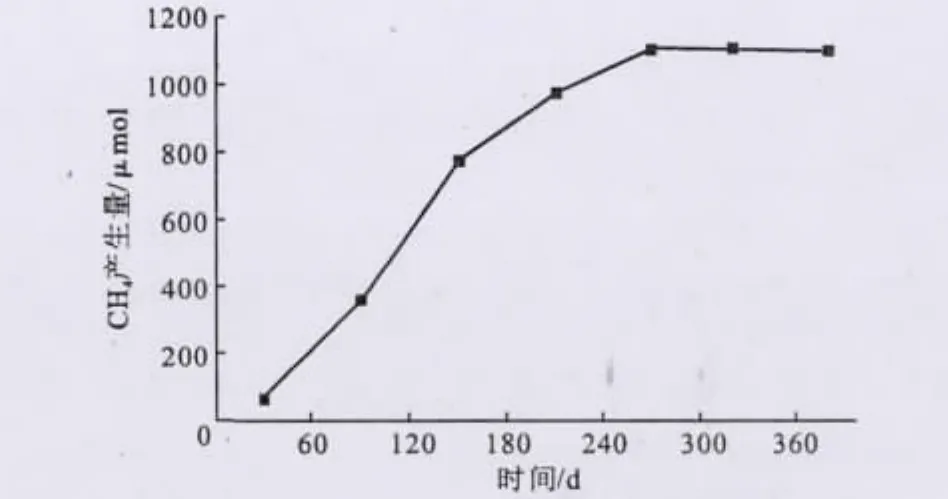
图2 混合菌群SL-7的产甲烷量Fig.2 Methane production of the microbial consortium SL-7
混合菌群SL-7培养270d所产混合气的气相色谱见图3。
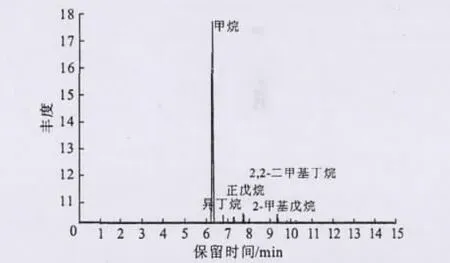
图3 混合菌群SL-7培养270d所产混合气Fig.3 Mixed gas produced by microbial consortium SL-7cultured for 270d
由图3可知,混合菌群SL-7在富集培养270d后生成的有机气体中,甲烷含量最高,占95.2%;检测到的其它气体有异丁烷、正戊烷、2,2-二甲基丁烷、2-甲基戊烷等,共占4.8%;此时混合菌群SL-7对稠油的降解率达到30.6%。
3 结论
(1)胜利油田某区块油井采出水在55℃下经过380d富集培养获得一组稠油降解产甲烷混合菌群SL-7,经过DGGE分析发现,该菌群由6株主要的单菌组成,其中2株单菌可分离培养,分别来自Firmiucutes(厚壁菌门)和β-Proteobacteria(β-变性菌门),另外4株单菌为不可分离培养的菌株。
(2)混合菌群SL-7经过270d培养,甲烷产生量达到最大值,为1110μmol;所产有机气体中甲烷含量最高,占95.2%,其它气体(异丁烷、正戊烷、2,2-二甲基丁烷、2-甲基戊烷等)占4.8%;此时混合菌群SL-7对稠油的降解率达到30.6%。
[1]孙焕泉.强基固本 开拓创新 确保油气硬稳定 再创开发新水平[J].油气地质与采收率,2007,4(2):1-6.
[2]汪卫东,王静,耿雪丽,等.储层残余油生物气化技术现状与展望[J].石油地质与工程,2012,26(1):78-81.
[3]Zengler K,Richnow H H,Rossello M R,et al.Methane formation from long-chain alkanes by anaerobic microorganisms[J].Nature,1999,401(6750):266-269.
[4]Anderson R T,Lovley D R.Hexadecane decay by methanogenesis[J].Nature,2000,404(13):722-723.
[5]Jones D M,Head I M,Gray N D,et al.Crude-oil biodegradation via methanogenesis in subsurface petroleum reservoirs[J].Nature,2008,451(7175):176-180.
[6]Li H,Yang S Z,Mu B Z,et al.Molecular analysis of bacterial community structure in a continental high-temperature and waterflooded petroleum reservoir[J].FEMS Microbiology Letters,2006,257(1):92-98.
[7]Milkov A V.Secondary microbial origin of gas in giant cenomanian pools of western siberia[C].Abstract for AAPG Annual Convention and Exhibition,Denver,Colorado,2009:7-10.
[8]包木太,牟伯中,王修林.采油微生物代谢产物分析[J].油田化学,2002,19(2):187-191.
[9]陈硕,李辉,杨世忠,等.PCR-DGGE用于检测油田产出液中烃降解菌的多样性[J].微生物学杂志,2010,30(2):1-6.
[10]Hendrickx B,Dejonghe W,Faber F,et al.PCR-DGGE Method to assess the diversity of BTEX mono-oxygenase genes at contaminated sites[J].FEMS Microbiology Ecology,2006,55(2):262-273.
[11]Hallmann C,Schwark L,Grice K.Community dynamics of anaerobic bacteria in deep petroleum reservoirs[J].Nature Geoscience,2008,1(9):588-591.
[12]Pham V D,Hnatow L L,Zhang S,et al.Characterizing microbial diversity in production water from an Alaskan mesothermic petroleum reservoir with two independent molecular methods[J].Environmental Microbiology,2009,11(1):176-187.
[13]Balch W E,Wolfe R S.New approach to the cultivation of methanogenic bacteria:2-Mercaptoethanesulfonic acid(HS-CoM)-dependent growth of Methanobacterium ruminantiumin a pressureized atmosphere[J].Applied and Environmental Microbiology,1976,32(6):781-791.
[14]李平兰,张篪.利用亨盖特厌氧滚管技术检测双歧杆菌制品中的活菌数[J].食品科学,1999,20(2):68-69.
[15]Muyzer G.DGGE/TGGE A method for identifying genes from natural ecosystems[J].Current Opinion in Microbiology,1999,2(3):317-322.
[16]Kunapuli U,Lueders T,Meekenstoek R U.The use of stable isotope probing to identify key iron-reducing microorganisms involved in anaerobic benzene degradation[J].The ISME Journal,2007,1(7):643-653.
[17]李政,赵朝成,张云波,等.16种EPA-PAHs复合污染土壤的菌群修复[J].中国石油大学学报(自然科学版),2012,36(1):175-181.

