微生物全基因组范围内的最小基因组研究进展
刘晓艳,闵勇,张薇,王开梅,万中义,江爱兵,张光阳,程贤亮,杨自文
(1.国家生物农药工程技术研究中心,湖北省生物农药工程研究中心,湖北省农业科学院,武汉,430064;2.湖北生物科技职业学院)
微生物全基因组范围内的最小基因组研究进展
刘晓艳1,闵勇1,张薇2,王开梅1,万中义1,江爱兵1,张光阳1,程贤亮1,杨自文1
(1.国家生物农药工程技术研究中心,湖北省生物农药工程研究中心,湖北省农业科学院,武汉,430064;2.湖北生物科技职业学院)
最小基因组是当前合成生物学研究的热点,从全基因组代谢网络研究入手,介绍了最小基因组研究的必要性和技术方法,并对其在工业微生物分子育种中的应用进行分析,综合阐述了最小基因组研究的发展前景。
微生物;全基因组;最小基因组;分子育种
1 基因组代谢网络
如今,伴随着高通量测序技术的应用和费用的降低,全基因组测序技术也越来越受欢迎。通过基因组测序,对基因进行功能注释,把基因编码蛋白所催化的生化反应称为一个代谢网络,通过计算机转化为数学模型,再用试验数据加以验证,提出假设,能够对生物体以及生物现象作出预测,是合成生物学研究中的一个重要的研究手段[1]。
2 研究最小基因组的必要性
最小基因组是指在最合适的条件下维持细胞生长繁殖所必需的最少基因[2],优势小基因组菌株是指含有特殊功能基因/簇的最小基因组菌株。通过对菌株基因组的减缩,可以达到如下目的:①可以提高菌株的代谢效率、降低代谢冗余;②基因组越小,微生物生长速度越快;③增加目标产物合成量,消除副产物[3~5],解决目前发酵产业中存在的发酵过程有副产物、底物转化率偏低、生长周期过长、增效因子产量较低和菌株不稳定等问题。
3 最小基因组研究内容和手段
3.1 菌株全基因组测序
截止到2011年7月1日,据Genomes Online Database数据库报道:6 891株微生物完成全基因组测序,其中包含细菌2 780株、真核微生物121株、古菌107株、病毒2 697株、质粒1 186个。越来越多的微生物开始进行全基因组测序[6~13]。
3.2 必需基因的确定
必需基因的确定主要有3种方法:
①利用生物信息学预测必需基因[14~17]第一步,利用在线数据库(Database of Essential Genes,DEG,http://tubic.tju.edu.cn/deg/)进行分析。将目标菌株的全基因组序列和数据库中同类菌株的基因组数据进行对比,如果遗传相似性大于50%,则可能为必需基因。
第二步,利用在线分析软件(http://tubic.tju.edu. cn/GC-Profile/)分析基因组岛。分析结果将全基因组分成N+1个区域,N+1个区域的 (G+C)%值与全基因组的(G+C)%值进行比对,相差较大的可能是基因组岛,即可能通过水平转移获得的基因,可以删除。
第三步,利用在线分析软件(http://tubic.tju.edu. cn/Ori-Finder/)分析复制原点,确定oriC的位置和大小,复制原点侧翼序列需要保留。
②确立菌株核心编码区,确定必需基因 通过选择同源关系较远的同属菌株全基因组序列,确定保守核心编码区。
③通过计算机构建模型 将代谢途径与数学符号进行对应,从而构建出全基因组代谢模型[18]。
4 微生物最小基因组缩小常用的技术手段
对微生物基因组进行定点无痕修饰是目前基因组缩小常用的技术手段,主要包括:①传统同源重组;②利用λ-Red系统的同源重组;③利用Cre/ loxP切除系统和Tn转座系统[19~22]。
4.1 大肠杆菌基因组的缩短
大肠杆菌是生物领域被研究得最透彻的模型生物之一,其代谢网络也被研究得很清楚,因此被用于科研领域、医药研究、发酵工业等,生产重组蛋白、氨基酸或者其他化学品。2001年,Mizoguchi等[23]利用2个连续的λ-Red介导的同源重组(图1)对Escherichia coliK12菌株进行了基因组删除,突变株MGF-01苏氨酸的合成能力是野生株的2倍。
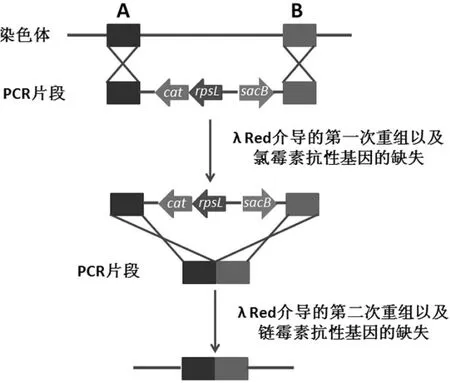
图1 大肠杆菌大片段敲除技术的原理示意[23]

图2 枯草芽孢杆菌大片段敲除技术的原理示意[24]
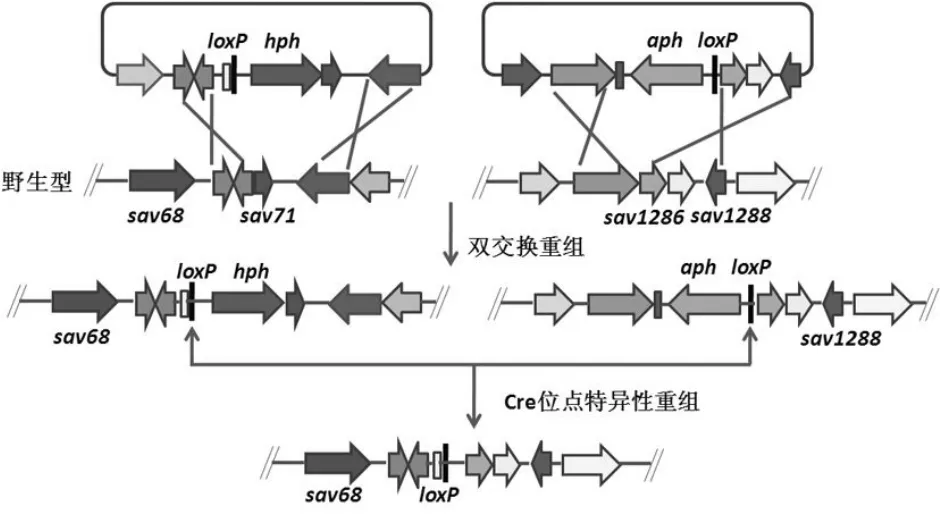
图3 链霉菌大片段敲除技术的原理示意[26]
4.2 枯草芽孢杆菌基因组的缩短
枯草芽孢杆菌是一种在土壤中广泛存在的革兰氏阳性菌,同苏云金芽孢杆菌、炭疽芽孢杆菌和蜡样芽孢杆菌归类于日蜡样芽孢杆菌群。因为该菌可以产生很多具有良好生物活性的次级代谢物,所以被研究得很广泛。Takuya等[24]2008年通过RecA介导的同源重组(图 2)对枯草芽孢杆菌的基因组进行了缩短, 得到了一系列突变株。 突变株MGB874由于缺少0.87 Mb的基因组序列,表现出良好的特性,该突变株MGB874发酵液中的纤维素酶和蛋白酶活性均比野生型细孢高出约1.7倍和2.5倍,同时还能延长蛋白酶的生产期。
4.3 链霉菌基因组的缩短
链霉菌是一种应用较广的工业微生物,能够合成较丰富的次级代谢产物,如抗生素和一些有应用价值的化合物[25]。Murakami等[26]2007年利用CreloxP的位点特异性重组对链霉菌的基因组进行了缩短,获得了一系列的突变体(图 3)。当使用的培养基为链霉菌合成阿佛菌素的最佳培养基时,链霉素和头孢霉素C在突变体菌株中的产量均高于原宿主菌的产量。
5 小结
目前优势小基因组研究是合成生物学领域研究的热点。通过基因组缩小研究,能够构建一个“小且单纯”的宿主,来维持外源基因的稳定性,以便于大规模培养。这样的优势宿主在重组蛋白表达、基因治疗或疫苗载体的制备,以及药用化合物和大宗化学品的制备等方面具有重要应用价值和前景。
[1]房柯池,王晶.全基因组范围代谢网络的构建和最小基因组研究[J].生命科学,2011,23(9):853-859.
[2]张柳燕,常素华,王晶.从首个合成细胞看合成生物学的现状与发展[J].科学通报,2010,55(36):3 477-3 488.
[3]王媛媛,郭文斌,宋存江.工业微生物菌种改造的新方法——优势小基因组生产菌的构建[J].微生物学报,2012,52(3):286-294.
[4]Koonin E V.How many genes can make a cell:The minimal gene set concept[J].Annual Review of Genomics and Human Genetics,2000(1):99-116.
[5]方宏清,陈惠鹏.大肠杆菌基因组减小研究进展[J].生物技术通讯,2009,20(4):564-567.
[6]宋存江.合成生物学技术在功能发酵制品生产菌种改造中的应用[C]//2012年第五届全国微生物遗传学学术研讨会论文摘要集.北京:中国知网,2012:11-12.
[7]Cello J,Paul A V,Wimmer E.Chemical synthesis of poliovirus cDNA:Generation of infectious virus in the absence of natural tem-plate[J].Science,2002,297(5 583): 1 016-1 018.
[8]Smith H O,Hutchison C A,Pfannkoch C,et al.Generating a syntheticgenome by whole genome assembly:phiX174 bacteriophage from synthetic oligonucleotides[J].Proc Natl Acad Sci USA,2003,100:15 440-15 445.
[9]Gibson D G,Benders G A,Andrews-Pfannkoch C,et al. Complete chemical synthesis,assembly,and cloning of a Mycoplasma genitalium genome[J].Science,2008,319 (5 867):1 215-1 220.
[10]Pósfai G,Plunkett G,Fehér T,et al.Emergent properties of reduced-genomeEscherichia coli[J].Science,2006, 312(5 776):1 044-1 046.
[11]Hashimoto M,Ichimura T,Mizoguchi H,et al.Cell size and nucleoid organization of engineeredEscherichia coli cells with a reduced genome[J].Molecular Microbiology, 2005,55(1):137-149.
[12]Mizoguchi H,Mori H,Fujio T.Escherichia coliminimum genome factory[J].Biotechnology and Applied Biochemistry,2007,46:157-167.
[13]Geng W,Cao M,Song C,et al.Complete genome sequence ofBacillus amyloliquefaciens LL3,which exhibits glutamic acid-independent production of polyγ-glutamic acid[J].Journal of Bacteriology,2011,193 (13):3 393-3 394.
[14]Mushegian A R,Koonin E V.A minimal gene set for cellular life derived by comparison of complete bacterial genomes[J].Proceedings of the National Academy of Science of the United states of America,1996,93:10 268-10 273.
[15]Koonin E V.Comparative genomics,minimal gene-sets
and the last universal common ancestor[J].Natural Reviews Microbiology,2003(1):127-136.
[16]Gao F,Zhang C T.Ori-Finder:A web-based system for findingoriCsin unannotated bacterial genomes[J].BMC Bioinformatics,2008(9):79-84.
[17]Gao F,Zhang C T.GC-Profile:a web-based tool for visualizing and analyzing the variation of GC content in genomic sequences[J].Nucleic Acids Research,2006,34: 686-691.
[18]陈琦,王卓,魏冬青.代谢网络流分析进展及应用[J].科学通报,2010,55(14):1 302-1 309.
[19]Fehér T,Papp B,Pal C,et al.Systematic genome reductions:Theoretical and experimental approaches[J].Chemical Reviews,2007,107:3 498-3 513.
[20]Zhang L,Chang S,Wang J.How to make a minimal genome for synthetic minimal cell[J].Protein and Cell, 2010(1):427-434.
[21]Baba T,Ara T,Hasegawa M,et al.Construction ofEscherichia coliK-12 in-frame,single-gene knockout mutants:The Keio collection[J].Molecular System Biology, 2006(2):1-5.
[22]Lee S Y.Systems biology and biotechnology ofEscherichia coli[M].Daejeon:Republic of Korea(South Korea), 2009:19-40.
[23]Mizoguchi H,Mori H,Fujio T.Escherichia coliminimum genome factory[J].Biotechnology and Applied Biochemistry,2007,46:157-167.
[24]Takuya M,Ryosuke K,Keiji E,et al.Enhanced recombinant protein productivity by genome reduction inBacillus subtilis[J].DNA Research,2008,15:73-81.
[25]Mamoru K,Takuma U,Satoshi O,et al.Genome-minimizedStreptomyceshost for the heterologous expression of secondary metabolism[J].Proceedings of the National A-cademy of Sciences,2009,107(6):2 646-2 651.
[26]Murakami K,Tao E,Ito Y,et al.Large scale deletions in theSaccharomyces cerevisiaegenome create strains with altered regulation of carbon metabolism[J].Applied Microbiology Biotechnology,2007,75:589-597.
Advances in Minimal Genome Research in Microbial Whole Genome
LIU Xiaoyan1,MIN Yong1,ZHANG Wei2,WANG Kaimei1,WAN Zhongyi1,JIANG Aibing1, ZHANG Guangyang1,CHENG Xianliang1,YANG Ziwen1
(1.National Biopesticide Engineering Research Center,Hubei Biopesticide Engineering Research Center, Hubei Academy of Agricultural Science,Wuhan 430064;2.Hubei Vocational College of Bio-technology)
Minimal genome has become the current research focus of synthetic biology.In this paper,we introduced the necessity and technical methods to carry out the research of minimal genome,and analyzed its application in industrial microbial molecular breeding,in addition,we discussed the development prospect of minimal genome research.
Microbe;Whole genome;Minimal genome;Molecular breeding
Q933;Q344+.13
A
1001-3547(2013)24-0009-03
10.3865/j.issn.1001-3547.2013.24.003
刘晓艳(1979-),女,博士,副研究员,主要从事植物病理学及生物农药分子生物学研究,电话:027-59101919,E-mail:xiaoyanliu6613@163.com
杨自文,通信作者,电话:027-59101919,E-mail:lky666888@126.com
2013-10-17

