狂犬病毒糖蛋白及其中和性抗体研究进展
常 亮, 刘晓志, 高 健
华北制药集团新药研究开发有限责任公司,抗体药物研制国家重点实验室,石家庄050015
狂犬病毒糖蛋白及其中和性抗体研究进展
常 亮, 刘晓志, 高 健
华北制药集团新药研究开发有限责任公司,抗体药物研制国家重点实验室,石家庄050015
狂犬病是一种由狂犬病毒引发的高致命人兽共患传染病,狂犬病毒糖蛋白作为唯一暴露于包膜外的病毒蛋白可有效诱生中和性抗体。本文介绍了狂犬病毒及其糖蛋白结构与功能,全面综述了狂犬病毒中和抗体研究进展及其作用机制,为中和抗体的深入研究提供理论基础和研发策略。
狂犬病毒;糖蛋白;中和抗体;作用机制
狂犬病是由狂犬病毒(rabies virus,RV)引发的一种高致命人兽共患传染病[1],呈全球性分布,目前全世界87个国家有狂犬病报道。据世界卫生组织(World Health Organization,WHO)统计,全球每年有5万多人死于狂犬病,约有1 000万人需要接受狂犬病毒的暴露后预防。狂犬病毒主要通过动物咬伤传播,在哺乳动物中枢神经系统中大量复制并破坏神经元细胞,尚无有效的治疗措施,WHO推荐采用疫苗结合抗病毒免疫球蛋白(rabies immune globulin,RIG)的方法进行狂犬病暴露后预防[2,3]。目前应用于暴露后预防的RIG为马抗狂犬病毒免疫球蛋白(equine rabies immune globulin,ERIG)和人抗狂犬病毒免疫球蛋白(human rabies immune globulin,HRIG),前者副反应严重,后者价格昂贵且有潜在的病原威胁。近年来随着基因工程抗体技术的不断发展,获得了大量具有临床应用潜力的抗狂犬病毒单克隆抗体(Mabs),单抗特异性高、安全性好、生产成本低,大量实验表明单抗可作为RIG的替代品用于狂犬病暴露后预防。目前报道的抗狂犬病毒单克隆抗体除少部分识别核蛋白外[4],大部分均针对病毒表面抗原——糖蛋白。本文主要针对狂犬病毒糖蛋白特异的中和性抗体最新进展进行综述。
1 狂犬病毒概述
1.1 狂犬病毒的分类地位与结构特征
狂犬病毒(rabie virus,RABV)属于弹状病毒科(Rhabdoviridae)狂犬病毒属(Lyssavirus)。弹状病毒在自然界中分布广泛,从动物到植物均是弹状病毒的感染宿主,家族成员超过150多种,除狂犬病毒外,还包括口炎病毒属水泡性口炎病毒(vesicular stomatitis virus,VSV)、Novirhabdovirus病毒属的病毒性出血性败血病病毒(viral hemor⁃rhagic septicemia virus,VHSV)和传染性造血组织坏死病毒(infectious hematopoietic necrosis virus),其中可感染哺乳动物的主要是狂犬病毒属和口炎病毒属。狂犬病毒呈子弹状结构(长170~180 nm、直径75~80 nm),由核衣壳和包膜组成,核衣壳包含与核蛋白N紧密结合的长约12 kb的单股、不分节负链RNA基因组、RNA依赖的RNA聚合酶L和P蛋白,包膜为双层脂膜,其上分布有400多个由糖蛋白G组成的三聚跨膜棘突,包膜与核衣壳间为病毒基质蛋白M。大量试验证实,由唯一的狂犬病毒表面抗原糖蛋白决定病毒的免疫原性,能诱生中和抗体,保护动物抵抗强毒株攻击,是制备疫苗的重要成分[5]。核蛋白与磷蛋白可诱导细胞免疫,N主要诱导活化辅助T淋巴细胞(TH),而P可以有效地诱导TH细胞和Tc细胞,并表现相应的免疫反应性。对这些蛋白在不同毒株间的系统进化分析结果表明,其氨基酸序列保守性顺序为:N>L>M>G>P[6]。
1.2 狂犬病毒的分型
初期根据血清学分析和抗狂犬病毒单克隆抗体分析,将狂犬病毒属分成4个血清型:血清Ⅰ型(狂犬病毒RABV)、血清Ⅱ型(拉各斯蝙蝠病毒LBV)、血清Ⅲ型(莫科拉病毒MOKV)和血清Ⅳ型(杜文海格病毒 DUVV)[7]。1993年,Bourhy等[8]根据核蛋白基因N端500个核苷酸的相似率,在原有分类基础上将狂犬病毒属分为6个基因型,其中基因型1型~4型与血清型分型相同,增加了两株欧洲蝙蝠狂犬病毒(European bat virus,EBL),分别为基因5型(EBL1)和基因6型(EBL2)[8]。1996年7月,澳大利亚发现果蝠体内的澳大利亚蝙蝠狂犬病毒(Australia bat virus, ABL),被定为基因7型[9]。狂犬病毒基因分型研究进一步将7个基因型分为2个进化组[6,10],基因型1、4、5、6和7为一组,基因型2和3为另一组,同组病毒的糖蛋白膜外区同源性至少为74%,且病毒保护抗体可与同组内其他病毒产生交叉反应,不同组病毒间膜外区同源性则低于64.5%,且不能产生交叉免疫保护。最近几年,不断有新的病毒基因型被发现,从Eurasian bat中分离出了4种新的狂犬病毒:Aravan virus(ARAV)、Khujand virus(KHUV)、Irkut virus(IRKV)和West Caucasian bat virus(WCBV)[11]。在肯尼亚蝙蝠体内分离出的Shimoni bat virus也是一种尚待分类的新基因型[12]。基因分型和抗原性分析均支持将其列为新的基因型。此外,几种在蚊子上发现的病毒与Lyssavirus也有不少相似性[13],但有待最后确认。
2 狂犬糖蛋白结构及功能
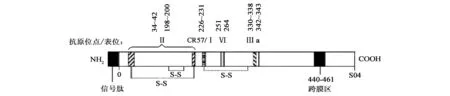
图1 狂犬糖蛋白的结构示意[18]Fig.1 Schematic diagram of domain and neutralizing epitope of glycoprotein[18].
1.2 狂犬糖蛋白结构及中和表位
狂犬病毒糖蛋白(rabies virus glycoprotein,RVG)是唯一暴露于病毒包膜外的病毒蛋白,属于Ⅰ类跨膜糖蛋白,成熟糖蛋白以三聚棘突(长8.3 nm)形式位于狂犬病毒双层脂膜上,在病毒的细胞粘附、膜融合及轴浆运输过程中发挥重要作用。成熟糖蛋白由505个氨基酸组成,分成3个区域(图1):膜外区1~439位氨基酸,参与识别受体与膜融合反应,其空间结构通过3对二硫键(C35⁃C207、C169⁃C189、C228⁃C283)维持;跨膜区440~461位氨基酸,由22个氨基酸组成连续性疏水区,与糖蛋白的病毒脂膜固定有关;膜内区462~505位氨基酸,位于病毒包膜内表面,提供基质蛋白和核蛋白作用位点,对三聚棘突的稳定性具有一定影响。
抗体逃逸株点突变分析结果表明,糖蛋白上至少存在3个主要中和抗体结合位点:抗原位点Ⅰ~Ⅲ,此外,还有一些次要线性表位[14~17]。其中抗原位点Ⅰ位于218~240位氨基酸区域,是一个次要线性中和表位,其最小结合区为KLCGVL(226~231位),核心残基为K⁃CGV⁃[18]。Ⅱ号位点是由第34~42位和第198~200位氨基酸两个位点通过二硫键C35⁃C207形成的一个保守性较高的空间表位[19],其中34~42位、147位、184位和198~200位氨基酸至关重要[20]。Ⅲ号位点位于330~357位氨基酸,也是一个空间表位,其中333~338位氨基酸区段是中和抗体主要结合部位,位于342~343位的a表位是次要的中和位点[14],第330位、333位、336位及338位氨基酸是关键残基[21]。Ⅱ号和Ⅲ号抗原位点是主要病毒中和表位,两位点在空间上十分接近,其中一些关键残基突变可对病毒的毒力、宿主范围或传播速度产生影响。实验发现,糖蛋白上260~267位(LHDFRSDE)为一线性中和表位[16],单独或与T细胞表位联用可在小鼠体内诱生抗狂犬抗体,有效抵抗病毒攻击。
近年来随着分子生物学及计算生物学的发展,发现了一些新的糖蛋白中和表位。Yang等[22]利用犬抗狂犬病毒免疫球蛋白对随机12肽库进行筛选,经序列比对发现获得的球蛋白特异识别肽段与已报道的狂犬糖蛋白及核蛋白中和表位高度匹配,其中30%的肽段与Ⅱ号、Ⅲ号表位重叠,此外还发现一个之前没有报道过的糖蛋白中和表位基序RYDD⁃W⁃T(126~141位氨基酸),该研究表明对于狂犬病毒中和表位仍有继续研究的空间。
2.2 糖蛋白介导的受体识别
RVG在病毒进入宿主细胞的过程中起主要作用,包括介导病毒与细胞受体的结合、病毒的内吞及病毒质膜与内吞体膜的融合等。一些膜分子被证实参与糖蛋白结合,主要有烟碱型乙酰胆碱受体(nicotinic acetylcholine receptor,NAchR)、神经营养因子受体(p75NTR)和神经细胞黏附分子(NCAM)等,此外还有一些磷脂及神经节苷脂也被发现具有狂犬病毒受体的功能。狂犬病毒受体由于种类多样目前仍存在争议,推测其受体多样性的可能原因是病毒在哺乳动物神经系统感染、转运过程中不同阶段需要不同的受体发挥作用。
2.2.1 烟碱型乙酰胆碱受体NachR NAchR是第一个被发现的可能的狂犬病毒受体[23],主要分布于突触后肌细胞膜。狂犬病毒糖蛋白和蛇毒箭毒样神经毒蛋白序列同源性分析以及两种蛋白合成肽与NAchR结合活性试验表明,NAchR主要识别糖蛋白173~204位氨基酸区域[24]。然而有些实验发现,缺乏乙酰胆碱受体的细胞系仍可被狂犬病毒感染,另外预先经蛇毒处理也不能降低这些细胞被感染的比例[25],表明病毒的侵入过程涉及其他受体分子。
2.2.2 神经营养因子受体p75NTR p75NTR是1型、6型狂犬病毒受体[26],参与病毒的内吞及逆轴浆运输。转染神经营养因子受体可使狂犬病毒耐受细胞株BSR重新变成病毒易感型。
2.2.3 神经细胞黏附分子NCAM 通过不同细胞株的比较观测确定了另一种候选受体——神经细胞黏附分子 NCAM[27]。在病毒感染过程中,NCAM聚集于神经肌肉接头(NMJ)的突触前膜,与RV在神经系统内的转运相关。细胞的病毒易感性由NCAM分子决定,阻断NCAM可降低病毒感染比例,转染NCAM可提高细胞的病毒易感性。体内实验发现,相比野生型小鼠,缺乏NCAM的小鼠受病毒感染后的死亡进程明显滞后[27]。
2.3 糖蛋白介导的膜融合
RVG介导病毒与宿主细胞的膜融合过程。病毒融合蛋白根据分子结构可分为三类。Ⅰ类融合蛋白包括正粘病毒、副粘病毒、反转录病毒及丝状病毒的糖蛋白等。流感病毒血凝素为典型的Ⅰ类融合蛋白,其特征在于前体蛋白剪切后形成的三聚棘突结构及发夹样后融合构象[28]。Ⅱ类融合蛋白(包括黄病毒属的E蛋白和甲病毒属的E1蛋白等)通常以二聚体形式存在,不会被蛋白酶剪切,而与之连接的其他病毒膜蛋白在融合过程中往往会被剪切[29]。这两类蛋白在融合机制上具有共同点,即体内酸性环境可诱导包膜二聚体解聚重排形成三聚体,融合肽位于多聚体分子的一端,尽管结构不同,但这两类融合蛋白在此过程中均倾向于形成一个较为稳定的后融合态,向后融合态不可逆的构象转变提供了融合过程所需的能量。隶属于弹状病毒科的狂犬病毒糖蛋白属于Ⅲ类融合蛋白。对水泡性口炎病毒(vesicular stomatitis virus,VSV)糖蛋白晶体结构的解析揭示了弹状病毒糖蛋白的融合机制与前两者均不相同[30]。电镜及单克隆抗体、酶切实验结果均表明,在低pH诱导的膜融合过程中,糖蛋白存在三种不同的pH依赖的构象[31]:中性环境下为天然构象(native,天然态),pH 6.7以下为活化构象(activated,活化态),pH 6.4以下为非活化构象(inactive,失活态)(图2)。电镜研究结果表明,G蛋白处于失活态的时间更长,且失活态G蛋白比天然态G蛋白的蛋白酶敏感性更高,pH诱导的变构作用破坏了天然构象G蛋白上的几个主要的构象表位(如Ⅱ号表位),导致非活化构象糖蛋白与天然态糖蛋白的形态和免疫原性均有较大的差异;活化态G蛋白疏水性较强,可触发病毒聚集,与天然态在形态和免疫原性上均十分相似,其稳定性依赖于pH和温度[32]。许多试验表明G蛋白的不同构象之间存在一个pH依赖的动态平衡。特异识别失活态的单克隆抗体在中性条件下长时间孵育也可与病毒颗粒结合,使该平衡向失活态偏移[33]。同样识别天然态的单克隆抗体也可以使膜融合最适pH条件下的平衡向天然态发生移动[34]。电镜结果显示在37℃、pH>6.4的环境中孵育2 h的病毒颗粒表面同时存在天然态和失活态G蛋白,在pH达到6.7时两种状态的G蛋白数量相当[32]。G蛋白这种结构变化的可逆性对于其在分泌系统中的转运十分必要。高尔基体中pH接近6.2,后高尔基体中pH更低,这意味着G蛋白在分泌过程中无法处于天然态,实验也表明在分泌小泡中存在失活态G蛋白,这可以避免不必要的融合且可以保证其在质膜表面重折叠形成天然态。由于结构转变的可逆性,此过程产生的能量十分有限,破坏靶膜与形成初始融合孔所需的能量需要至少13~19个G蛋白三聚体的形成来提供[34]。
3 狂犬病毒中和性抗体研究进展

图2 狂犬糖蛋白依赖pH的构象变化[31]Fig.2 pH⁃dependent conformation change of rabies glycoprotein[31].
3.1 糖蛋白特异的人源中和抗体研究
1978年,Wiktor等[35]应用杂交瘤技术制备了抗狂犬病毒单克隆抗体,并证明其能够保护动物抵抗致死性狂犬病毒的攻击,开启了抗狂犬病毒抗体研究的大门。随着分子免疫学和抗体工程技术的发展,特别是噬菌体抗体库技术的出现,使大量获得人源基因工程抗体成为可能,克服了人抗鼠抗体反应(human anti⁃mouse antibody,HAMA)。Dietzschold等[36]获得了9种能够识别狂犬病毒G蛋白、N蛋白和M蛋白的单克隆抗体,但其中针对G蛋白的Mab57具有中和多种狂犬病毒的能力。随后 Prosniak等[37]和 Jones等[38]将 Mab57进行了一系列改造,获得的抗体CR57识别G蛋白Ⅰ号表位的KLCGVL位点(1型狂犬病毒中保守性高达99%),可保护多种毒株的攻击。Kramer等[39]构建了人源抗狂犬病毒单链噬菌体抗体库,筛选到一株针对狂犬病毒糖蛋白Ⅲ号表位的单克隆抗体CR4098,该抗体逃逸株的Ⅲ号表位存在N336D突变,该突变在1型狂犬病毒的发生率为4%。Goudsmit等[40]将 CR57和CR4098联合使用,可中和26种典型的街毒株,再与疫苗联用可有效保护暴露24 h后的叙利亚仓鼠,临床结果表明两者联用可作为安全有效的RIG替代品用于暴露后预防[41]。Sloan等[42]利用携带人免疫球蛋白基因的转基因小鼠获得了另一株针对狂犬病毒糖蛋白Ⅲ号表位的人源单抗17C7。Houimel和Dellagi[43]基于抗狂犬人源Fab噬菌体库,筛选到6株Fab抗体,其中3株特异识别Ⅲ号表位并显示了一定的CVS⁃11中和能力。Wang等[44]报道了另一株针对糖蛋白Ⅲ号和a表位的中和抗体,该抗体结合336和346位点,该表位虽然保守性较差,但突变株中和实验结果表明1型狂犬病毒中336和346位点存在的各种氨基酸组合均可被该抗体有效识别。
我国在狂犬单抗领域的研究也十分深入。赵小玲等[45]基于抗狂犬病毒人源单链抗体核糖体库,筛选到几株针对狂犬糖蛋白的单链抗体。华北制药集团研发的人源狂犬抗体NM57已进入Ⅰ期临床试验阶段[46],并与中国疾病控制中心病毒所合作开发了针对保守性更高的Ⅱ号表位的中和抗体[47]。Sun等[47]基于高滴度狂犬病毒抗体的疫苗注射者外周血淋巴细胞构建噬菌体人源抗体库,成功筛选到11株针对狂犬病毒颗粒aG和CTN株的Fab抗体,利用昆虫/杆状病毒系统表达出其中5株全抗,实验结果表明这些抗体具有明显的体外病毒中和活性,所识别表位相互重叠且均针对狂犬病毒糖蛋白Ⅱ号表位(在狂犬病毒属1型进化亚组病毒中高度保守),该表位与某些细胞受体识别表位有部分重叠,提示中和机制可能与阻碍病毒的细胞黏附有关。
3.2 抗体的病毒中和机制研究
尽管目前对于狂犬病毒致病机制及其预防已进行了深入研究,且筛选到大量具有临床应用前景的抗体,然而由于宿主对狂犬病毒攻击的抵抗依赖于包括中和抗体、抗原递呈细胞、T淋巴细胞和干扰素等免疫系统各组成部分之间复杂的相互作用,这种相互作用至今仍无法完全阐明,所以对抗体中和机制的研究仍相对滞后,并没有能够从分子水平上将中和表位与病毒致病机制联系起来。抗病毒抗体的可能作用机制主要有:①阻断病毒糖蛋白与靶细胞受体或胞膜间的相互作用;②介导病毒交联以减少感染“单位”数量;③介导天然免疫系统对病毒颗粒的攻击;④介导抗体依赖的细胞毒作用;⑤活化补体系统。
Dietzschold等[48]早在1987年就对狂犬病毒特异抗体作用机制进行过探索,研究发现除Fc依赖的病毒清除机制外,抗体的中和机制与抗体类型及识别表位密切相关。电镜试验结果表明中和状态下,3个棘突平均可结合1~2个IgG分子,一个IgM分子则可结合9到10个棘突[49]。Burton等[50]发现抗病毒抗体的相似作用机制,达到63%中和率所需IgG分子数N与病毒颗粒表面积A呈线性关系(N=0.0033×A),应用该公式推算中和一个狂犬病毒颗粒需225个IgG抗体分子。Irie等[51]全面比较了两株糖蛋白特异的单克隆抗体#1⁃46⁃12和#7⁃1⁃9的病毒中和能力的差异,发现两株抗体的病毒结合能力相近,亲和力均为1.7×10-10mol/L左右,平衡态单颗粒结合数分别为707和770,大概为病毒表面抗原数量(约1 350个G蛋白[52],即约445个棘突结构)的一半。抗体中和活性分析结果表明,两株抗体均可通过阻断病毒的细胞黏附来实现中和作用,而两者中和滴度差别显著,前者高于后者200多倍,中和所需最少抗体数目也有较大差别(#1⁃46⁃12数量≤20,而#7⁃1⁃9数量≥250)[51]。抗体逃逸株分析结果表明,两株抗体识别表位不同,#1⁃46⁃12识别一个高度保守的空间表位,而#7⁃1⁃9识别G蛋白中间区域的一个线性表位,F263L会导致结合活性的完全丧失[51]。综合最小抗体结合数推测:超过250个#7⁃1⁃9分子可以达到对三聚棘突60%的覆盖率,可能是通过位阻效应实现对病毒粘附的阻断作用;而仅需不到20个#1⁃46⁃12分子即可实现病毒的有效中和,说明两者的中和机制存在较大差异。对#1⁃46⁃12作用机制的进一步研究发现,该抗体逃逸株可分为两类[53]:一类识别表位存在T36P突变(R⁃31株),无抗体结合活性;另一类表位发生S39T突变(R⁃61株),具有正常的抗体结合活性,与野生型G蛋白不同的是其N37位点发生额外的糖基化。在突变株中引入少量野生型G蛋白后,R⁃61株重新恢复了对抗体的敏感性,而R⁃31仍对#1⁃46⁃12具有耐受性[54],这一结果表明#1⁃46⁃12并不是通过遮蔽G蛋白受体结合位点来直接阻断病毒粘附,而可能通过结合来诱导糖蛋白发生变构使之丧失细胞受体结合能力,这种诱变作用具有抗体结合活性依赖的多米诺效应,N37位点上的寡糖链可以一定程度上抑制#1⁃46⁃12的初始诱变但无法抑制多米诺级联的变构过程。该研究表明狂犬中和机制十分复杂,涉及糖基化、变构等分子水平层面的过程,对抗体作用机制的阐明需要进一步分子水平的糖蛋白结构研究。
4 展望
尽管国内外对于狂犬病毒,尤其是其表面抗原——狂犬病毒糖蛋白及其中和表位开展了十分广泛的研究,筛选获得了大量具有临床应用前景的治疗型抗体,但对这些抗体的中和机制的研究仍相对滞后,无法阐明中和抗体的抗病毒机制。对狂犬病毒糖蛋白及其中和性抗体结构的进一步研究有利于从分子水平上阐明中和抗体对病毒致病过程的抑制机制,对于抗病毒抗体的研究具有重要意义,同时有利于阐明病毒表面抗原与病毒致病机制的相关性,对于病毒中和表位的研究具有指导意义。
[1] 俞永新(主编).狂犬病和狂犬病疫苗[M].北京:中国医药科技出版社,2001.
[2] Centers for Disease Control and Prevention.Human rabies prevention——United States,1999.Recommendations of the advisory committee on immunization practices(ACIP)[Z].MMWR Recomm.Rep.,1999,48:1-21.
[3] Ray K,Embleton M J,Jailkhani B L,et al.Selection of single chain variable fragments(scFv)against the glycoprotein antigen of the rabies virus from a human synthetic scFv phage display library and their fusion with the Fc region of human IgG1[J].Clin.Exp.Immunol.,2001,125:94-101.
[4] Lv X J,Ma X J,Wang L H,et al..Preparation and initial application of a monoclonal antibody specific for a newly discovered conserved linear epitope of rabies virus nucleoprotein[J].Biomed.Environ.Sci.,2012;25(1):98-103.
[5] Taylor J,Trimarchi C,Weinberg R,et al..Efficacy studies on a canarypox⁃rabies recombinant virus[J].Vaccine,1991,9(3):190-193.
[6] Delmas O,Holmes E C,Talbi C,et al..Genomic diversity and evolution of the lyssaviruses[J].PLoS One,2008,3(4):e2057.
[7] WHO Expert Committee on Rabies.WHO Technical Report Series,No.824[R].Geneva:WHO,1992.
[8] Bourhy H,Kissi B,Tordo N.Molecular diversity of the Lyssavirus genus[J].Virology,1993,194(1):70-81.
[9] Gould A R,Hyatt A D,Lunt R,et al..Characterization of a novel lyssavirus isolated from Pteropid bats in Australia[J].Virus Res.,1998,54(2):165-187.
[10] de Mattos C A,de Mattos CC,Rupprecht CE.Rhabdoviruses[A].In:Knipe DM,Howley PM,Griffin D E,et al.Fields Virology[M].(4th ed).Philadelphia:Lippincott Williams&Wilkins,2001,1245-1257.
[11] Hanlon C A,Kuzmin I V,Blanton J D,et al..Efficacy of rabies biologics against new lyssaviruses from Eurasia[J].Virus Res.,2005,111(1):44-54.
[12] Kuzmin I V,Mayer A E,Niezgoda M,et al..Shimoni bat virus,a new representative of the Lyssavirus genus[J].Virus Res.,2010,149(2):197-210.
[13] Smith DW,Speers D J,Mackenzie J S.The viruses of Australia and the risk to tourists[J].Travel Med.Infect.Dis.,2011,9(3):113-125.
[14] Benmansour A,Gore M.Characterization of a new virus⁃neutralizing epitope that denotes a sequential determinant on the rabies virus glycoprotein[J].J.Gen.Virol.,1989,70:291-298.
[15] Dietzschold B,Gore M,Marchadier D,et al..Structural and immunological characterization of a linear virus⁃neutralizing epitope of the rabies virus glycoprotein and its possible use in a synthetic vaccine[J].J.Virol.,1990,64(8):3804-3809.
[16] van der Heijden RW, Langedijk J P, Groen J, et al..Structural and functional studies on a unique linear neutralizing antigenic site(G5)of the rabies virusglycoprotein[J].J.Gen.Virol.,1993,74(Pt8):1539-1545.
[17] Luo T R,Minamoto N,Ito H,et al..A virus⁃neutralizing epitope on the glycoprotein of rabies virus that contains Trp251 is a linear epitope[J].Virus Res.,1997,51(1):35-41.
[18] Marissen W E,Kramer R A,Rice A,et al..Novel rabies virus⁃neutralizing epitope recognized by human monoclonal antibody:fine mapping and escape mutant analysis[J].J.Virol.,2005,79(8):4672-4678.
[19] Benmansour A,Leblois H,Coulon P,et al..Antigenicity of rabies virus glycoprotein[J].J.Virol.,1991,65(8):4198 -4203.
[20] Prehaud C,Coulon P,LaFay F,et al..Antigenic siteⅡof the rabies virus glycoprotein:structure and role in viral virulence[J].J.Virol.,1988,62(1):1-7.
[21] Seif I,Coulon P,Rollin P E,et al..Rabies virulence:effect on pathogenicity and sequence characterization of rabies virus mutations affecting antigenic siteⅢof the glycoprotein[J].J.Virol.,1985,53(3):926-934.
[22] Yang L,Cen J,Xue Q,et al.Identification of rabies virus mimotopes screened from a phage display peptide library with purified dog anti⁃rabies virus serum IgG[J].Virus Res.,2013,174(1-2):47-51.
[23] Lentz T L, Burrage T G, Smith A L, et al..Is the acetylcholine receptor a rabies virus receptor[J]?Science,1982,215(4529):182-184.
[24] Lentz T L, Hawrot E, Wilson P T.Synthetic peptides corresponding to sequences of snake venom neurotoxins and rabies virus glycoprotein bind to the nicotinic acetylcholine receptor[J].Proteins,1987,2(4):298-307.
[25] Reagan K J,Wunner W H.Rabies virus interaction with various cell⁃lines is independent of the acetylcholine⁃receptor[J].Arch.Virol.,1985,84(3-4):277-282.
[26] Warrell M J,Warrell D A.Rabies and other lyssavirus diseases[J].Lancet,2004,363(9413):959-969.
[27] Thoulouze M I,Lafage M,Schachner M,et al..The neural cell adhesion molecule is a receptor for rabies virus[J].J.Virol.,1998,72(9):7181-7190.
[28] Wilson IA,Skehel J J, Wiley D C.Structure of the haemagglutinin membrane glycoprotein of influenza virus at 3Å resolution[J].Nature,1981,289(5796):366-373.
[29] Kielian M.ClassⅡ virus membrane fusion proteins[J].Virology,2006,344(1):38-47.
[30] Sun X,Belouzard S,Whittaker G R.Molecular architecture of the bipartite fusion loops of vesicular stomatitis virus glycoprotein G,a classⅢ viral fusion protein[J].J.Biol.Chem.,2008,283(10):6418-6427.
[31] Gaudin Y,Tuffereau C,Durrer P,et al.Rabies virus⁃induced membrane fusion[J].Mol.Membr.Biol.,1999,16(1):21 -31.
[32] Gaudin Y,Ruigrok R W,Knossow M,et al.Low⁃pH conformational changes of rabies virus glycoprotein and their role in membrane fusion[J].J.Virol.,1993,67(3):1365 -1372.
[33] Raux H,Coulon P,Lafay F,et al..Monoclonal antibodies which recognize the acidic configuration of the rabies glycoprotein at the surface of the virion can be neutralizing[J].Virology,1995,210(2):400-408.
[34] Roche S, Gaudin Y.Characterization of the equilibrium between the native and fusion⁃inactive conformation of rabies virus glycoprotein indicates that the fusion complex ismade of several trimers[J].Virology,2002,297(1):128-135.
[35] Wiktor T J,Koprowski H.Monoclonal antibodies against rabies virus produced by somatic cell hybridization:detection of antigenic variants[J].Proc.Natl.Acad.Sci.USA,1978,75(8):3938-3942.
[36] Dietzschold B, Gore M, Casali P, et al.. Biological characterization of human monoclonal antibodies to rabies virus[J].J.Virol.,1990,64(6):3087-3090.
[37] Prosniak M,Faber M,Hanlon C A,et al..Development of a cocktail of recombinant⁃expressed human rabies virus neutralizingmonoclonal antibodies for postexposure prophylaxis of rabies[J].J.Infect.Dis.,2003,188(1):53-56.
[38] Jones D,Kroos N,Anema R,et al..High⁃level expression of recombinant IgG in the human cell line per.c6[J].Biotechnol.Prog.,2003,19(1):163-168.
[39] Kramer R A,Marissen W E,Goudsmit J,et al..The human antibody repertoire specific for rabies virus glycoprotein as selected from immune libraries[J].Eur.J.Immunol.,2005,35(7):2131-2145.
[40] Goudsmit J,Marissen W E,Weldon W C,et al..Comparison of an anti⁃rabies human monoclonal antibody combination with human polyclonal anti⁃rabies immune globulin[J].J.Infect.Dis.,2006,193(6):796-801.
[41] Bakker A B, Python C, Kissling C J, et al.. First administration to humans of a monoclonal antibody cocktail against rabies virus: safety,tolerability, and neutralizing activity[J].Vaccine,2008,26(47):5922-5927.
[42] Sloan SE,Hanlon C,Weldon W,et al.Identification and characterization of a human monoclonal antibody that potently neutralizes a broad panel of rabies virus isolates[J].Vaccine,2007,25(15):2800-2810.
[43] Houimel M,Dellagi K.Isolation and characterization of human neutralizing antibodies to rabies virus derived from a recombinant immune antibody library[J].J.Virol.Methods,2009,161(2):205-215.
[44] Wang Y,Rowley K J,Booth B J,et al..G glycoprotein amino acid residues required for human monoclonal antibody RAB1 neutralization are conserved in rabies virus street isolates[J].Antiviral Res.,2011,91(2):187-194.
[45] Zhao X L,Chen W Q,Yang ZH,et al..Selection and affinity maturation of human antibodies against rabies virus from a scFv gene library using ribosome display[J].J.Biotechnol.,2009,144(4):253-258.
[46] 常亮,刘晓志,赵伟,等.利用表面等离子体共振技术检测重组抗体NM57免疫原性[J].药学学报,2013,48(4):532-535.
[47] Sun L,Chen Z,Yu L,et al..Generation and characterization of neutralizing human recombinant antibodies against antigenic siteⅡ of rabies virus glycoprotein[J].Appl.Microbiol.Biotechnol.,2012,96:357-366.
[48] Dietzschold B,Tollis M,Lafon M,et al.Mechanisms of rabies virus neutralization by glycoprotein⁃specific monoclonal antibodies[J].Virology,1987,161(1):29-36.
[49] Flamand A,Raux H,Gaudin Y,et al..Mechanisms of rabies virus neutralization[J].Virology,1993,194(1):302-313.
[50] Burton D R,Saphire E O,Parren P W H I.A model for neutralization of viruses based on antibody coating of the virion surface[J].Curr.TopicsMicrobiol.Immunol.,2001,260:109 -143.
[51] Irie T,Kawai A.Studies on the different conditions for rabies virus neutralization bymonoclonal antibodies#1⁃46⁃12 and#7⁃1⁃9[J].J.Gen.Virol.,2002,83(Pt12):3045-3053.
[52] Dietzschold B,Cox JH,Schneider L G..Rabies virus strains:a comparison study by polypeptide analysis of vaccine strains with different pathogenic patterns[J].Virology,1979,98:63 -75.
[53] Irie T,Matsuda Y,Honda Y,et al..Studies on the escape mutants of rabies virus which are resistant to the neutralization by a highly conserved conformational epitope⁃speci c monoclonal antibody g1⁃46⁃12[J].Microbiol.Immunol.,2002,47:449-461.
[54] Irie T,Kawai A.Further studies on the mechanism of rabies virus neutralization by a viral glycoprotein⁃specific monoclonal antibody,#1⁃46⁃12[J].Microbiol.Immunol.,2005,49(8):721-731.
Progress in Rabies Virus Glycoprotein and Neutralizing Antibody
CHANG Liang,LIU Xiao⁃zhi,GAO Jian
State Key Laboratory of Antibody Research and Development,New Drug Research and Development Company Ltd.,North China Pharmaceutical Corporation,Shijiazhuang 050051,China
Rabies is a fatal zoonosis caused by rabies virus(RV).Rabies virus glycoprotein(RVG),the unique viral protein exposed on the surface of viral envelope,can induce the production of rabies neutralizing antibodies.This review focuses on recent advances in rabies virus,the structure and function of glycoprotein,progress of neutralizing antibodies and their action mechanism,which can provide theoretical foundations and development strategies for further study of neutralizing antibodies.
rabies virus;glycoprotein;neutralizing antibody;mechanism
10.3969/j.issn.2095⁃2341.2013.05.02
2013⁃05⁃02;接受日期:2013⁃06⁃14
国家975项目“生物活性分子结构和功能的关系研究”(2012CB724500)资助。
常 亮,工程师,博士,研究方向为生化与分子生物学。E⁃mail:changliang1981@gmail.com

