川西高原牦牛酸奶子乳酸菌遗传多样性及系统发育研究
田 鸿,蒲 彪,张小平,*
(1.四川农业大学资源环境学院,四川 成都 611134;2.四川农业大学食品学院,四川 雅安 625014)
川西高原牦牛酸奶子乳酸菌遗传多样性及系统发育研究
田 鸿1,蒲 彪2,张小平1,*
(1.四川农业大学资源环境学院,四川 成都 611134;2.四川农业大学食品学院,四川 雅安 625014)
采用16S rDNA基因的限制性酶切片段长度多态性分析(16S rDNA-PCR-RFLP)和16S~23S rDNA基因间区(ISR)的酶切多态性分析(ISR-PCR-RFLP)技术,研究分离自川西高原牦牛酸奶子的81株乳酸菌的遗传多样性,并结合16S rDNA序列分析技术研究16S rDNA-PCR-RFLP的各类群代表菌株的系统发育关系。结果表明:供试菌株共有23种16S rDNA遗传型,供试菌株按61%相似系数可分为4个类群,按81%相似系数,可分为16个类群,结合序列分析结果,乳杆菌属(Lactobacillus)、肠球菌属(Enterococcus)细菌为主要类群;16S~23S rDNA基因的ISR扩增产物的酶切分析表明,供试菌株共有19种ISR遗传型,按60%相似系数,供试菌株可分为4个类群,按80%相似系数,供试菌株可划分为14个类群。
牦牛酸奶子;乳酸菌;相似水平;系统发育
近年来,发酵型酸奶已经成为乳品市场的重要产品,产量年增长40%以上[1]。由于国内自然发酵乳品中乳酸菌资源的收集、发酵特性、遗传特性、系统发育关系研究、菌种鉴定以及菌种安全性研究等还比较欠缺,国内主要乳品企业生产酸奶以直接购买国外直投式发酵剂为主,与此同时,随着小型酸奶机走入家庭,小包装酸奶发酵剂必将成为商业发酵剂的重要产品,但是,目前采用自有知识产权菌株组配的发酵剂鲜见报道,因此亟需对国内天然发酵乳品中乳酸菌资源进行深入系统的研究。我国地域辽阔,民族众多,藏族、维吾尔族、蒙古族等民族有制作和食用自然发酵酸奶的传统,有研究表明这些自然发酵酸奶中含有丰富的乳酸菌资源[2-8],研究开发这些资源对推动国内乳业可持续发展具有十分重要的战略意义。
川西高原甘孜、阿坝地区海拔高,紫外线辐射强,牦牛奶酸奶子是生活在该区域藏民族的主食之一。酸奶子风味独特,低温发酵,说明川西高原牦牛酸奶子中乳酸菌具有某些特点。鲁妮等[7]对酸奶子样品中的乳酸菌进行了分离和初步鉴定;敖晓琳等[9-10]研究了分离菌株的发酵性能并初步筛选出一些优良单株;严以兰等[11]对分离自该区域的几株优良单菌株的酸碱、胆盐等耐受性进行了研究,初步筛选出几株抗逆性较好的菌株。通过这些工作,收集了该区域的乳酸菌资源,初步筛选出一些优良单菌株。但该区域乳酸菌分离株的遗传多样性和系统发育关系研究尚未见报道。
本实验采用16S rDNA基因的限制性酶切片段长度多态性分析(16S rDNA-PCR-RFLP)和16S~23S rDNA基因间区(ISR)的酶切长度多态性分析(ISR-PCR-RFLP)技术研究分离自川西高原地区藏民族的牦牛奶酸奶子的81株乳酸菌的遗传多样性和系统发育关系,为进一步开发此区域乳酸菌资源打下基础。
1 材料与方法
1.1 菌株与试剂
81株分离株和11株参比菌株见表1,由四川农业大学微生物学实验室于-70℃保存。DNA提取和保存采用北京天根生化科技有限公司的DNA提取试剂盒中的试剂。酶切实验采用BBI公司的5种限制性内切酶(HindⅢ、MspⅠ、HinfⅠ、HindⅢ和TaqⅠ),酶切体系和酶切反应按产品说明书进行。
1.2 菌株总DNA提取
供试菌株先在MRS培养基上连续划线活化两代,活化后接种于5mL液体MRS培养基,42℃培养16h,12000r/min离心收集菌体,STE缓冲液(pH8.0)洗涤菌体3次,37℃条件下采用溶菌酶处理直至细胞壁溶解,用细菌DNA提取试剂盒提取菌株总DNA,TE缓冲液(pH7.4)悬浮,分装小离心管,2%琼脂糖凝胶水平电泳检测提取的总DNA,-20℃保存备用,作为PCR扩增模版。
1.3 16S rDNA基因PCR-RFLP分析
以菌株总DNA为模板,选用双向引物P1和P6扩增供试菌株的16S rDNA基因。正向引物P1:5′-CGAGAGTTTGATCCTGGCTCAGAACGAACGCT-3′;反向引物P6:5′-CGT ACGGCTACCTTGTTACGACTTCACC CC-3′,50μL扩增体系。反应条件:92℃、3min;94℃、1min,58℃、1min,72℃、2min,30个循环;72℃、8min。进行含溴乙锭(EB)的2%琼脂糖凝胶水平电泳,电压120V,时间40min,检测扩增产物长度是否符合要求。
按实验用酶的使用说明酶切16S rDNA基因扩增产物,酶切结束,进行溴乙锭EB的2%琼脂糖凝胶水平电泳,电压120V,时间3h,凝胶成像系统成像,统计各菌株酶切条带数。采用NTSYSpc21软件对酶切结果进行聚类分析。
1.4 16S rDNA基因的序列分析
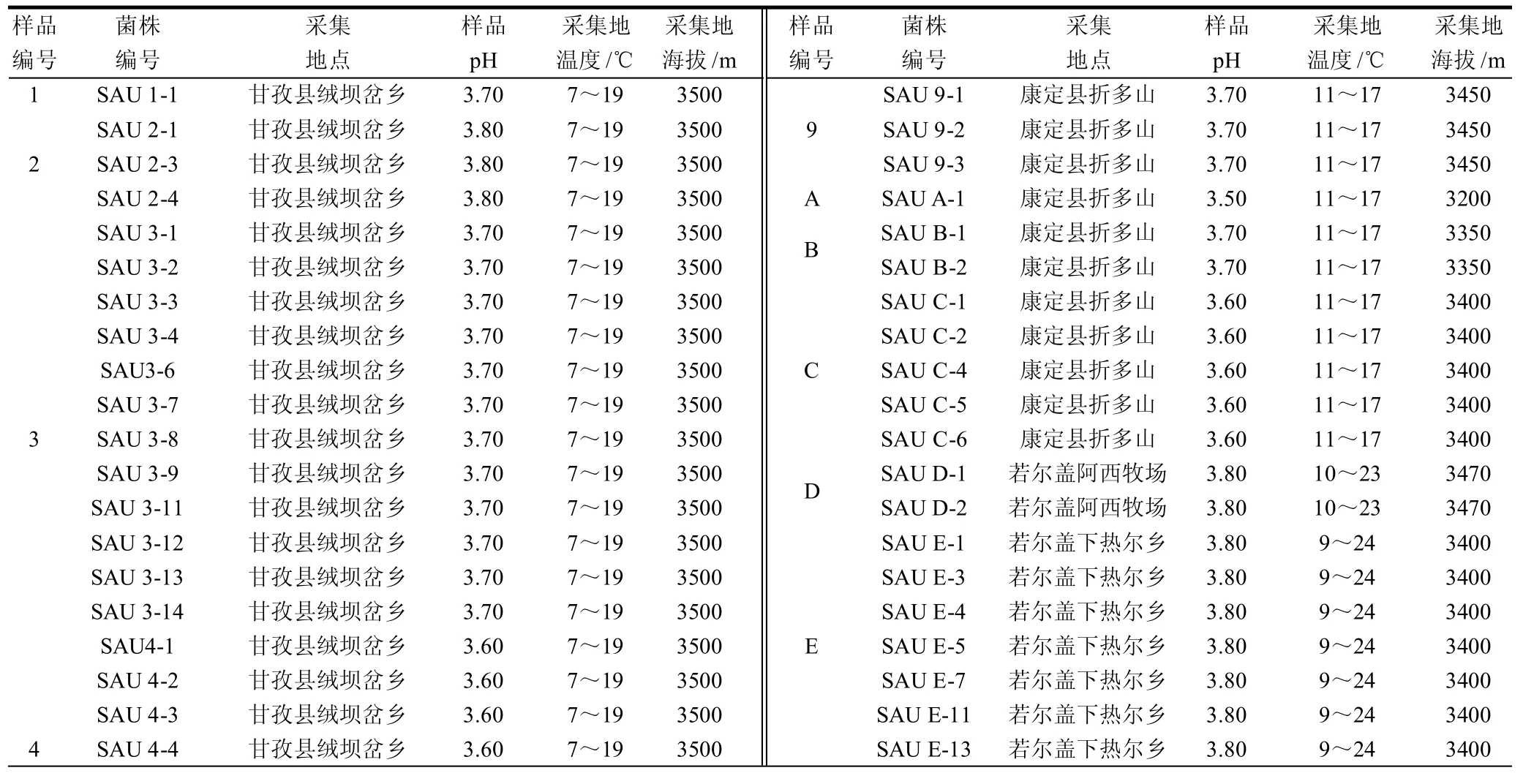
表1 供试菌株及来源Table 1 The tested strains and the sources of collected samples
根据16S rDNA基因酶切聚类结果,选取各类群中心菌株16S rDNA基因扩增产物送上海英骏生物技术有限公司测序,测序结果提交NCBI(http://www.ncbi.nlm.nih. gov)的GenBank,取得登录号,Blast搜寻与各测序序列高一致度序列,查找高一致度序列典型菌株的16S rDNA基因序列,采用MAGA4.1软件包中的ClustalW1.6子程序对测序序列和典型菌株16S rDNA基因序列进行多序列匹配排列,排列结果采用邻位连接(Neighbor-Joining)法计算序列间遗传距离,构建测序序列的系统发育树,同时计算测序序列和GenBank中高一致度序列的相似度。

续表1
1.5 16S~23S的ISR-PCR-RFLP分析
以菌株总DNA为模板,采用双向引物PHr和p23SR01扩增供试菌株的ISR,正向引物PHr:5′-TGCGGCTGGATCACCTCCTT-3′,反向引物p23SR01:5′-GGCTGCTTCTAAGCCAAC-3′,扩增体系50μL。反应条件:94℃、5min;94℃、1min,55℃、1min;72℃、1min,35个循环;72℃、6min。进行含EB的2%琼脂糖凝胶水平电泳,电压120V,时间40min,检测扩增产物是否符合1000kb大小要求。
按实验用酶的使用说明酶切ISR扩增产物,酶切结束,进行含EB的2%琼脂糖凝胶水平电泳,电压120V,时间2.5h,凝胶成像系统成像,统计各菌株酶切条带数。采用NTSYSpc21软件对酶切结果进行聚类分析。
2 结果与分析
2.1 16S rDNA基因的PCR-RFLP分析

图1 4种酶切16S rDNA基因扩增产物具有较丰富酶切条带的限制性内切酶的部分酶切结果Fig.1 Fingerprinting of 16S rDNA PCR products digested by 4 restriction enzymes
5个限制性内切酶酶切扩增产物电泳图谱显示不同限制性内切酶所反应的遗传信息量不相同,4种限制性内切酶(HaeⅢ、MspI、HinfI)酶切条带多(图1),尤以MspI酶切条带最多,所反映的遗传信息量最丰富,MspI是本实验中揭示乳酸菌遗传多样性的最佳内切酶。
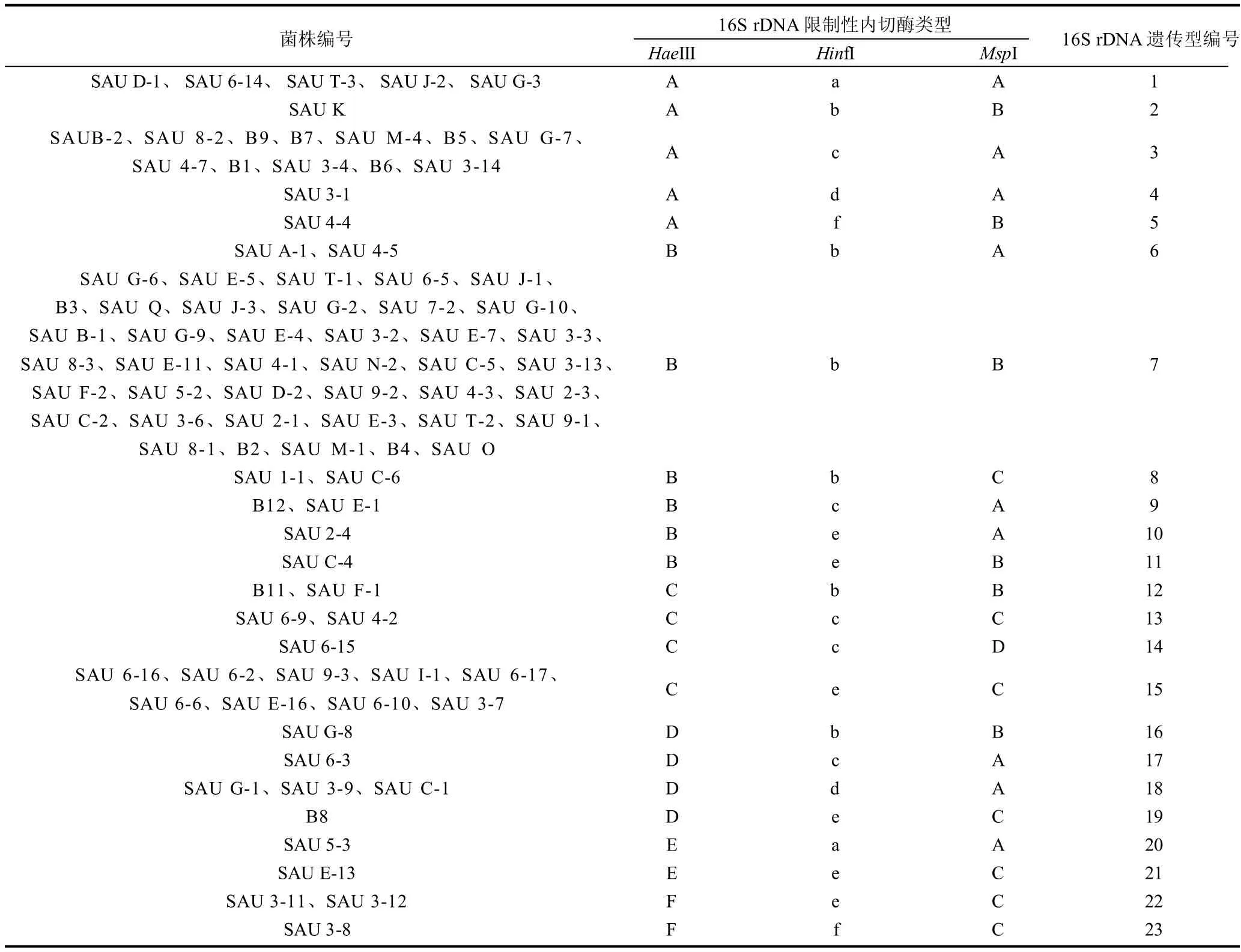
表2 供试菌株16S rDNA扩增产物的酶切图谱类型Table 2 16S rDNA genotype patterns of PCR products digested by restriction enzymes
由表2可见,供试菌株有23种16S rDNA基因的遗传型,其中第3、7、15种遗传型是主要遗传型,第7种遗传型共40株,占供试菌株的43.4%。16S rDNA基因酶切聚类结果见图2,如果按相似系数61%划分类群,可以划分为4个类群,其中,参考菌株B12 单独聚群,4个16S rDNA主要遗传型(编号1、3、7、15),在按61%相似系数的聚类中,编号1、3和15的3个主要遗传型16S rDNA菌株是第Ⅰ类群的主要菌株;编号为7的遗传型菌株全部为第Ⅳ群菌株。如果按81%的相似系数,供试菌株可分为16个类群。
2.2 基于16S rDNA基因序列分析
根据图2聚类结果,将主要类群代表菌株的16S rDNA基因进行测序,测序结果提交NCBI的GenBank,取得索引号,分别是SAU E-5(FJ462691)、SAU F-2 (GU187337)、SAU C-1(GU177634)、SAU 3-2 (GU177629)、SAU J-2(FJ462690)、SAU G-10 (GU177632)、SAU 3-12(GU177628)、SAU 2-4 (GU139738)、SAU E-16(GU177633)。通过构建测序序列的系统发育树(图3),并计算测序序列与典型菌株16S rDNA基因序列的相似度。
由图3可见,代表菌株集中于两个属,即乳杆菌属(Lactobacillus)和肠球菌属(Enterococcus)。其中菌株SAU E-5、SAU F-2、SAU C-1、SAU 3-2、SAU J-2和SAU G-10为乳杆菌属菌株,分布于按61%相似水平的Ⅰ和Ⅳ群中,其余菌株(SAU 3-12、SAU 2-4、SAU E-16)为肠球菌属菌株,分布于图2的第Ⅱ群中。系统发育树中菌株SAU E-5和Lactobacillus pentosusJCM1558T处于同一分支,相似系数为98.5%,表明与典型菌株的亲缘关系较近,可能为Lactobacilluspentosus中的一个菌株。SAU F-2与短小乳杆菌Lactobacillus brevisATCC14869T处于同一分支,相似系数为97.6%;菌株SAU 3-2、SAU C-1与Lactobacillus caseiATCC334T处于同一分支,SAU 3-2与Lactobacillus caseiATCC334T的相似系数为97.8%,表明SAU 3-2和Lactobacillus caseiATCC334T亲缘关系较近,SAU C-1与Lac-tobacillus caseiATCC334T为94.9%,小于97.5%,表明二者亲缘关系较远;菌株SAU J-2与Lactobacillus curvatusDSM20010T(AJ270591)处于同一分支,相似系数为95.6%,小于97.5%,二者亲缘关系较远;菌株SAU G-10与Lactobacillus fermentumATCC14931T处于同一分支,相似系数为98.2%,说明二者有较近的亲缘关系。菌株SAU 3-12与Enterococcus faecalisLMG7937T处于同一分支,相似系数仅为92.4%,与典型菌株16S rDNA基因序列的相似系数小于97.5%,表明与典型菌株的亲缘关系较远。菌株SAU 2-4、SAU E-16和Enterococcus duransDSM20633T处于同一分支,但SAU 2-4与Enterococcus duransDSM20633T相似系数仅为93.7%, SAU E-16与Enterococcus duransDSM20633T的相似系数也不高,仅为93.9%,说明两个菌株与EnterococcusduransDSM20633T亲缘关系较远。上述研究尽管采用典型菌株的16S rDNA基因序列为参比序列,但典型菌株的选择可能对序列分析有影响,也有可能有新种。要准确确定菌株的系统发育关系还需要结合其他方法,如RecA基因[12-13]、dnaA及gyrB基因[14]、rpoA基因[15]等特殊基因的序列分析结果才能最终确定菌株的系统发育关系。

图2 供试菌株基于16S rDNA基因扩增产物的酶切结果的聚类分析Fig.2 Dendrogram of the tested strains based on 16S rDNA-PCR-RFLP

图3 基于16S rDNA基因序列分析的主要类群代表菌株系统发育树Fig.3 Dendrogram of the representative strains based on 16S rDNA gene sequence analysis
2.3 供试菌株ISR扩增产物的酶切分析
2.3.1 16S~23S 的ISR酶切分析

图4 3种酶切ISR扩增产物具有较丰富酶切条带的限制性内切酶的部分酶切结果Fig.4 Fingerprinting of ISR-PCR products digested by 3 restriction enzymes

图5 基于16S~23S rDNA基因的ISR扩增产物酶切结果的供试菌株聚类分析Fig.5 Dendrogram of the tested strains based on ISR-PCR-RFLP
采用5种限制性内切酶进行酶切反应,发现3种限制性内切酶(MspI、HinfI、TaqI)酶切条带较多(图4),揭示的供试菌株遗传信息丰富,能反应出供试菌株的遗传多样性,尤其是以HinfI酶切条带最多,所反映的遗传信息量最丰富。
依据ISR扩增产物的5种酶切图谱,选择酶切条带丰富的3种酶切图谱,对酶切结果进行聚类分析,结果见表3和图5。由图5可见,如果按60%的相似系数,供试菌株可分为4个类群,如果按80%的相似系数,供试菌株可分为14个类群。表3中,供试菌株共有19个ISR遗传类型,主要类型为编号1、2和4的遗传类型,编号1的遗传类型共有13株,编号2有43株,编号4有8株。编号1的遗传群的菌株大部分聚类于按60%相似系数聚类的第I大群,编号2的遗传群的菌株全部聚类于按60%相似系数聚类的第Ⅱ大群,编号4的遗传群的菌株大部分聚类于按60%相似系数聚类的第Ⅳ群中,说明供试菌株中第Ⅱ遗传群是主要遗传群。
一般认为,和16S rDNA相比,ISR具有更高的变异性,通过ISR的序列(IGS)分析,更能反映菌株的变异情况,比较适合种以内菌株的遗传多样性研究。本实验中,ISR的酶切结果遗传类型较少,可能与所选择的内切酶的种类和数量有关,进一步的研究应对内切酶进行筛选以及对ISR进行IGS序列分析才能得到更准确的结果。
3 结论与讨论
16S rDAN基因分析技术和16S~23S rDNA ISR分析技术是细菌多相分类广泛使用的方法,被广泛用于乳酸菌的分类鉴定[16],马春燕[17]、王英[18]等采用16S rDNA序列分析技术分别对哈萨克族传统发酵乳酪、新疆酸马奶中乳酸菌进行了鉴定。本实验采用16S rDNA酶切聚类和序列分析技术以及16S~23S rDNA的 ISR扩增产物的酶切分析技术初步研究了分离自川西高原牧区牦牛酸奶子中乳酸菌的遗传多样性,结果表明,分离菌株主要为乳杆菌属(Lactobacillus)和肠球菌属(Enterococcus)细菌;16S rDNA扩增产物的酶切分析,MspI酶切条带最丰富,供试菌株共有23种16S rDNA基因型,显示分离菌株有比较丰富的遗传多样性,这将为筛选优良菌株提供较丰富的材料。16S~23S rDNA的ISR扩增产物的酶切分析,HinfI酶切条带最丰富,供试菌株共有19种ISR遗传型。16S rDNA基因和ISR的酶切聚类分析结果都表明川西高原牦牛酸奶子中具有比较丰富的乳酸菌资源。
自然发酵的牦牛酸奶子是川西北藏民的主食之一,由于其特殊的奶源,高海拔、强紫外线环境,较低的酸奶子发酵温度,独特的风味,显示出发酵过程中乳酸菌类群的特殊性。由于本实验的采样点仅在甘孜、阿坝两州的部分地区,采样时间也比较集中,后续工作需要在整个生产季节的不同时间,更广泛的采集样品,收集更多的菌株,在目前研究基础上,进一步采用其他方法,如API数值分类,全细胞水溶蛋白分析,特殊基因(RecA基因和RecB基因等)的序列分析等方法,对分离菌株进行更深入研究,以便对该区域牦牛酸奶子中乳酸菌的生物多样性进行更全面的了解,为其优良菌株的筛选和鉴定提供更丰富的材料。

表3 供试菌株转录间区(ISR)扩增产物酶切图谱类型Table 3 ISR genotype patterns of the PCR products digested by restriction enzymes
[1] 张和平. 我国乳品工业乳酸菌资源的开发利用现状及发展趋势[J]. 农产食品科技, 2008, 2(3): 3-6.
[2] 李少英, 乌尼, 李培锋, 等. 内蒙古牧区乳与乳制品中乳酸菌资源及其生态分布[J]. 生态学杂志, 2006, 25(12): 1495-1499.
[3] 段宇珩, 谈重芳, 王雁, 等. 萍牦牛乳曲拉中微生物及乳酸菌特性及菌群构成的研究[J]. 中国乳品工业, 2008, 36(4): 27-30.
[4] 肖仔君, 杨汝德, 黄国清. 西藏雪莲中乳酸菌的分离鉴定及抑菌活性的研究[J]. 现代食品科技, 2009, 25(3): 249-251.
[5] 刘芳, 都立辉, 杜鹏, 等. 内蒙古酸马奶中乳酸菌多样性的研究[J].食品科学, 2008, 29(2): 218-224.
[6] 赵胜娟, 赵暄, 罗红霞, 等. 新疆传统乳制品中乳酸菌的分离鉴定[J].中国乳品工业, 2007, 35(11): 8-11.
[7] 鲁妮, 张小平, 敖晓琳. 川西牧区自然发酵酸乳中几株优良乳酸菌的分离及鉴定[J]. 四川农业大学学报, 2005, 23(1): 43-47.
[8] 张宜凤, 杨洋, 李启明, 等. 云南传统乳产品中乳酸菌的菌群分析[J].乳业科学与技术, 2009, 32(3): 113-135.
[9] 敖晓琳, 张小平, 李诚. 川西高原牧区自然发酵酸乳中优良乳酸菌性能测定及应用[J]. 乳业科学与技术, 2005, 27(2): 56-60.
[10] 敖晓琳, 鲁妮, 张小平. 川西高原牧区自然发酵酸乳中优良酸奶发酵剂菌种的筛选[J]. 中国乳品工业, 2005, 33(7): 23-26.
[11] 严以兰, 张小平. 几株优良乳酸菌耐受特性研究[J]. 安徽农业科学, 2007, 35(18): 5507-5511.
[12] EHRMANN M A, FREIDING S, VOGEL R F.Leuconostoc palmaesp. nov., a novel lactic acid bacterium isolated from palm wine[J]. Int J Syst Evol Microbiol, 2009, 59: 943-947.
[13] WATANABE K, FUJIMOTO J, TOMII Y, et al.Lactobacillus kisonensissp. nov.,Lactobacillus otakiensis sp. nov.,Lactobacillus rapisp. nov. andLactobacillus sunkiisp. nov., heterofermentative species isolated from sunki, a traditional Japanese pickle[J]. Int J Syst Evol Microbiol, 2009, 59: 754-760.
[14] CHELO I M, ZE-ZE L, TENREIRO R, et al. Congruence of evolutionary relationships inside theLeuconostoc-Oenococcus-Weissellaclade assessed by phylogenetic analysis of the 16S rRNA gene,dnaA,gyrB,rpoCanddnaK[J]. Int J Syst Evol Microbiol, 2007, 57: 276-286.
[15] NASER S M, DAWYNDT P, HOSTE B, et al. Identification of lactobacilli bypheSandrpoAgene sequence analyses[J]. Int J Syst Evol Microbiol, 2007, 57: 2777-2789.
[16] VANDAMME P, POT B, GILLIS M, et al. Polyphasic taxonomy, a consensus approach to bacterial systematics[J]. Microbiol Rev, 1996, 60 (2): 407-438.
[17] 马春燕, 新华·那比, 刘红梅. 哈萨克族传统发酵乳酪中发酵菌的分离鉴定[J]. 中国乳品工业, 2010, 38(4): 7-9.
[18] 王英, 周剑忠, 陈晓红, 等. 新疆酸马奶中优良乳酸菌的分离鉴定及发酵特性分析[J]. 江西农业大学学报, 2009, 31(5): 807-811.
Genetic Diversity and Phylogeny of Lactic Acid Bacterial Strains Isolated from Yak Yoghurt in Western Sichuan Platea
TIAN Hong1,PU Biao2,ZHANG Xiao-ping1,*
(1. College of Resource and Enviroment, Sichuan Agricultural University, Chengdu 611134, China;2. College of Food Science, Sichuan Agricultural University, Ya,an 625014, China)
The genetic diversity and the phylogeny of 81 lactic acid bacterial strains isolated from yak yogurt samples collected from Western Sichuan Plateau were studied by using 16S rDNA-PCR-RFLP, ISR-PCR-RFLP techniques, and 16S rDNA gene sequence analysis. The results showed totally 23 16S rDNA genotypes were detected and the tested strains could be divided into four taxa on the basis of 61% similarity, and 16 taxa on the basis of 81% similarity. Sequence analysis of 16S rDNA genes showed that genusLactobacillusand genusEnterococcuswere two major genera in all tested strains. ISR-PCR-RFLP analysis also revealed 19 ISR genotypes, and the tested strains could be divided into four taxa on the basis of 60% similarity, and 14 taxa on the basis of 80% similarity.
yak yoghurt;lactic acid bacteria;similarity level;phylogeny
TS252.1
A
1002-6630(2012)05-0170-07
2011-01-22
四川省科技厅科技支撑计划项目(2010NZ0045)
田鸿(1966—),男,讲师,博士,研究方向为农业及资源微生物。E-mail:tianhongmicrobiol@yahoo.com.cn
*通信作者:张小平(1962—),男,教授,博士,研究方向为农业及资源微生物。E-mail:aumdwsb@sicau.edu.cn

