深海热液喷口金属硫化物中甲烷菌的多样性研究
王淑芳, 陈 月 钱媛媛
(1. 淮海工学院 海洋学院, 江苏 连云港 222005; 2. 淮海工学院 江苏省海洋生物技术重点建设实验室, 江苏 连云港 222005)
深海热液喷口金属硫化物中甲烷菌的多样性研究
王淑芳1,2, 陈 月1, 钱媛媛1
(1. 淮海工学院 海洋学院, 江苏 连云港 222005; 2. 淮海工学院 江苏省海洋生物技术重点建设实验室, 江苏 连云港 222005)
对两个来自太平洋洋脊胡安·德富卡的两个热液烟囱的金属硫化物4136-2和4148-B1进行甲烷菌甲基辅酶M还原酶的编码基因mcrA的序列扩增, 构建克隆文库并进行分子进化分析。结果表明在这个甲烷富集的热液喷口周围含有丰富的甲烷产生菌, 没有任何甲烷氧化菌存在。两个硫化物样品的甲烷产生菌种类完全不同。在4136-2硫化物中的甲烷产生菌都与热液口的高温环境有关系, 主要属于甲烷球菌目的甲烷暖球菌(Methanocaldococcus), 少部分属于甲烷火菌目甲烷嗜高热菌属的坎氏甲烷嗜高热菌(Methanopyrus kandleri)。与这两个属的可分离菌株的氨基酸同源性为89%~97%, 核苷酸同源性高达92%~100%。4148-B1硫化物中发现的一类疑似甲基辅酶M还原酶的编码序列, 它们与已知的甲烷菌mcrA序列核苷酸同源性为69%~72%, 氨基酸同源性仅为43%~47%。这可能是由于4148-B1来自于正在喷发的超高温热液喷口相关。由于与已发表的甲烷菌克隆子或菌株同源性较低, 有可能是热液口特有的以前未发现的甲烷菌。
胡安·德富卡; 金属硫化物; 甲烷产生菌; mcrA
20世纪70年代, 科学家在东太平洋深海区发现的海底热液口及其周围生机盎然的生物群。这是生命科学史上一次具有深远意义的重大发现, 也是人类认识自然的一大进步。研究热液口环境下的微生物群落结构对探索生命起源具有重要的基础研究意义。
在太平洋洋脊胡安·德富卡(Juan de Fuca Ridge,JDF)热液场 Endeavour部分, 甲烷浓度高达 1.8~3.4 mmol/L, 二氧化碳浓度高达11.6~18.2 mmol/L, 氢气浓度高达 50 mmol/L[1]。远远高于 EPR、SJdF和Guanymas的热液口。在东太平洋样脊一次爆发过程中,“A”烟囱口中的氢气从 25 mmol/kg升高到 45 mmol/kg, 甲烷的浓度从 0.13 mmol/kg升高到 0.19 mmol/kg[2]。Gorda热液喷发后的甲烷浓度达到背景海水的16倍, 浓度从2 nmol/L到100 nmol/L, 与其他热液场稳定的热液口环境中的浓度相似[3]。热液流体一般含有2~20 mmol/kg的二氧化碳。另外在非常热的流体中还检测到约为 300 mmol/kg的二氧化碳[4,5]。丰富的甲烷含量为甲烷氧化菌的生命活动提供必要的物质能量, 丰富的氢和二氧化碳为甲烷产生菌的生命活动提供必要的物质和能量。生物可以利用这些气体作为能源维持生长和代谢活动[6]。
Endeavour 热液系统最重要的特点是热液流体具有高浓度的甲烷和氨根离子[7]。热液流体中甲烷的碳同位素组成最高而且出现在海底系统中没有沉积物覆盖的位置, 同时这些同位素数据与高达背景值900倍的甲烷含量相呼应, 说明Endeavour热液场海底有丰富的甲烷产生菌[8]。能够利用二氧化碳产生甲烷的超嗜热的甲烷菌已经从东太平洋脊(EPR)13°N,21°N[9-11]和胡安·德富卡热液场的 Main Endeavour附近的热液烟囱中分离得到[12]。本文采用甲烷菌的甲基辅酶M还原酶α亚单位编码基因(mcrA)作为分子标记检测甲烷产生古菌和甲烷氧化古菌在胡安·德富卡热液烟囱的金属硫化物4148-B1及4136-2中甲烷循环中发挥的作用。
1 材料与方法
1.1 菌株、培养基与试剂
大肠杆菌 (Escherichia coli) DH5α 为本实验室保存; pMD18-T Vector购自宝生物工程(大连)有限公司(TaKaRa Biotechnology (Dalian) Co., Ltd.); 氨苄青霉素 (Amp)购自南京创瑞公司; PCR引物由上海英俊生物技术有限公司合成; DNA回收试剂盒购自上海生工公司的; 培养基配制参照 Maniatis等[13]的分子克隆方法。
1.2 采样点特征描述及金属硫化物岩石样品的采集
本试验所用样品是2005年秋季, 由中国和美国两国科学家在胡安·德富卡洋脊(Juan de Fuca Ridge或JDF)热液区由Atlantis/Alvin科考船采集的热液烟囱样品。采样喷口位点如图1所示。样品4148是在Main Endeavour热液场(MEF)采集的一个正在喷发的烟囱体, 可以清晰地观察到热液通道的内中外各层, 以及各层矿物不同的颜色, 中心温度估计超过350℃。烟囱结构在超净工作台把烟囱体从烟囱通道到外壁分成三层, 4148-B1是烟囱的最外层, 估计温度在 100℃。硫化物样品 4136-2采自 MEF北面的Clam Bed热液场。Clam Bed热液场是一个大的热液弥散流活动场。这里观察到有微生物席、螃蟹和管状蠕虫的存在, 但没有发现以前这里的标志性生物贻贝(Clam), 探测器显示此处最高温度是29.2℃。采集的硫化物样品迅速冷却, 放入-20℃冰箱保存直到进行DNA提取。在保存过程中没有发现样品有外观的变化。
1.3 金属硫化物样品中总DNA的提取
硫化物岩石样品用已灭菌的凿子和小刀从大的样品上分离下来, 用研钵研磨。DNA的提取方法参照zhou等[14]的方法。30 mL的离心管加入5g样品和13.5 mL DNA提取液(100 mmol/L Tris-HCl [pH 8.0], 100 mmol/L EDTA钠盐[pH8.0], 100 mmol/L磷酸钠 [pH 8.0], 1.5 mol/L NaCl, 1% CTAB)和 100µL蛋白酶K(10 g/L)在37℃温浴30 min。再加入1.5 mL 20% SDS, 在65℃温浴2 h, 温浴过程中每隔15~20 min混匀一次。温浴结束离心并收集上清, 加入氯仿/异戊醇(24:1)进行抽提两次。收集上清, 加入 2/3体积的异丙醇进行 DNA沉淀, 离心得到的沉淀就是DNA的粗提物。用1%的琼脂糖检测提取的总DNA的大小。粗提物用上海生工的UNIQ-10柱式PCR产物回收试剂盒纯化。纯化后的DNA放入-80℃冰箱保存。
1.4 mcrA基因PCR扩增
mcrA扩增参照前人的方法[15]用通用引物 ME1(5′-GCM ATG CAR ATH GGW ATG TC-3′)和 ME2(5′-TCA TKG CRT AGT TDG GRT AGT-3′)扩增环境基因组的mcr基因。反映程序如下: 94℃变性 3 min;再接94℃40 s, 49℃1 min, 72℃1 min, 35个循环; 最后72℃延伸10 min。扩增反应体系:DNA (约20~50 ng/μL) 1 μL, Taq 聚合酶 Buffer(10×)5μL,dNTP(20 mmol/L) 4 μL,引物(10 pmol/uL)各 1 μL, Mg2+(25 mmol/L)3~5 μL, Taq 酶(1U/μL) 3μL, 补水至 50 μL。扩增2~3管重复, 产物用1%的琼脂糖凝胶电泳检测并回收。
1.5 mcrA基因的文库构建及阳性菌筛选
对PCR产物进行胶回收以纯化mcrA基因片段,然后用pMD18-T克隆试剂盒(TaKaRa)按照操作说明连接到pMD18-T载体并转化到DH5α大肠杆菌感受态细胞,从而构建 mcrA基因的克隆文库。37℃培养12~16 h后, 用 pMD18-T载体的通用引物 UPI(5′-GGAAACAGCTATGACCATG-3′)和 UPII(5′-TTGGGTAACGCCAGGGT-3′)进行菌落 PCR, 检测插入片段大小。检测到插入片段在960 bp左右的克隆子为阳性。随机挑选不同数目的克隆子进行测序。测序数目根据多样性指数的计算结果进行, 以文库的库容(Coverage)[16]达到85%以上为标准。
1.6 mcrA阳性克隆子测序及序列分析
引物M13+/-的PCR产物用Applied Biosystems 3730xl 测序仪进行序列核苷酸测序。核苷酸序列用DNAMAN 6.0蛋白翻译功能翻译成氨基酸序列。氨基酸序列和核苷酸序列在 NCBI上的网上数据库进行同源序列比对, 查找同源性最高的克隆子和菌株并对所有氨基酸序列用ClustalX 1.83进行比对。最终截取78个序列的173个氨基酸用Mega 3.1(http://www.megasoftware.net/index.html)进行分子进化树的构建, 并以1000次的步长和临位法进行计算。Dotur[17](http://www.plantpath.wisc.edu/fac/joh/dotur.html)用来做稀释曲线和Chao1非参数丰度度评估及西普森指数(Simpson)和香农指数(Shanoon index)。库容(Coverage)用Good[16]的计算公式进行手工计算。最小分类操作单元(OTUs)用来定义不同的克隆子群,本文以古菌mcrA氨基酸大于等于 98%作为分类最小分类操作单元的标准。

图1 Endeavour段Main Endeavour热液场采样喷口和样品图Fig. 1 Hydrothermal field vent and sample of the endeavour segment main endeavour
2 结果
口的两个金属硫化物岩石中提取的环境总 DNA进行PCR扩增, 得到约760 bp的片段。克隆到DH5α菌株后, 4136-2随机挑选34个阳性克隆子, 4148-B1随机挑选27个阳性克隆子, 将获得的61个克隆子进行测序。
2.1 甲烷菌甲基辅酶M编码基因mcrA的文库构建
采用mcrA的通用引物ME1和ME2对来源于热液
2.2 甲烷菌甲基辅酶 M 编码基因mcrA序列分析及多样性评估
获得的 61个氨基酸序列在 NCBI(http://www.ncbi.nlm.nih.gov/)中进行比对, 搜索最同源的克隆子和可培养菌株, 结果如表1。61个序列中的43个氨基酸序列与甲烷古菌甲烷辅酶 M的α亚基同源, 同源性在 43%~97%之间。同时这 43个序列的核苷酸序列与已发表的甲烷甲基辅酶M的序列的同源性达到69%~100%。另外18个序列最同序列不是甲烷古菌的mcrA蛋白, 说明在扩增过程中出现了比较严重的非特异性扩增。在胡安·德富卡热液口的金属硫化物中甲烷菌都属于广古菌中的甲烷产生菌。4136-2文库中 97.06%的甲烷菌来自在甲烷球菌(Methanococcales), 2.94%甲烷菌来自甲烷嗜高热菌(Methanopyrales)。4148-B1与已知的克隆子及可培养菌株氨基酸同源性只有 43%~47%, 核苷酸同源性为69%~72%, 属于未知的甲烷古菌。
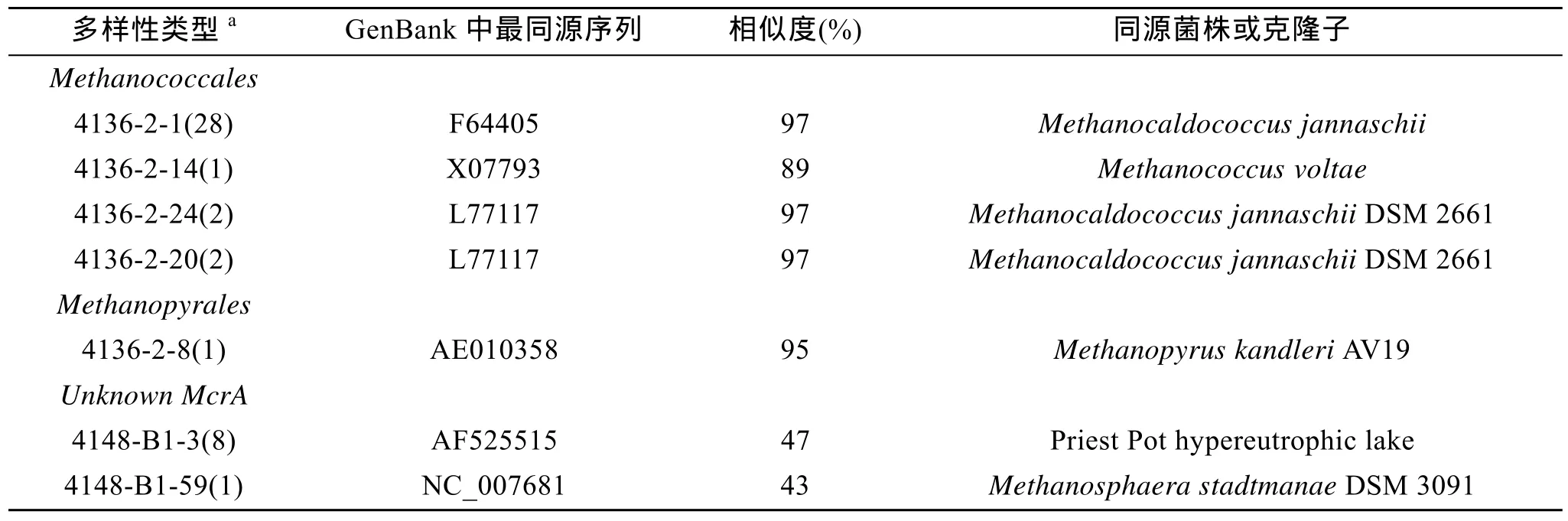
表1 胡安·德富卡热液口甲烷菌mcrA序列NCBI数据库比对分析Tab. 1 Analysis of mcrA sequences from JDF vent
本试验采用自动分析软件 Dotur对两个硫化物岩石中的的甲烷古菌进行多样性评估, 结果如表2。Shannon-Wiener指数, Simpson's指数以及Chao1[18-19]无偏估计, 文库覆盖率等几个指数用来评估构建的文库的多样性, 以评估文库对取样位点甲烷古菌多样性的反应程度。43个mcrA序列以≥98%的氨基酸同源性分为7个OUTs, 两个OTUs来自4148-B1, 5个OTUs来自4136-2。4136-2和4148-B1mcrA基因构建的克隆文库覆盖率分别为 94.1%和 88.9%,Shannon-Wiener指数分别为0.68和0.78, Chao1无偏估计值为5.33和2.00。这表明在胡安·德富卡热液口甲烷菌的多样性并不丰富, 试验中构建的两个文库得到的 7个 OTUs完全可以代表这两个位点的甲烷古菌的多样性分布。比较而言, 4148-B1文库的H’及1/D较4136-2大, 表明4148-B1文库的多样性较4136-2丰富。稀释曲线(Rarefaction curves)分析(图2)支持以上结果。

图 2 热液口金属硫化物岩石中甲烷古菌 mcrA基因多样性的稀释曲线Fig. 2 Rarefaction curves of mcrA gene from vent sulfide rock

表2 胡安·德富卡热液口甲烷菌mcrA基因克隆文库的多样性评估Tab. 2 Measurement of mcrA clone library from JDF vent
2.3 甲烷菌甲基辅酶 M 编码基因mcrA系统进化分析

图3 胡安·德富卡热液口甲烷菌mcrA基因编码的氨基酸的系统发育树Fig. 3 Phylogentic tree of mcrA gene from JDF vent系统发育树采用邻接法构建; 粗体为本研究中获得的甲烷菌mcrA基因的克隆子, 命名为4136-2和4148-B1分别表示克隆子来自于硫化物样品4136-2和4148-B1; 分支上的数值代表经1000次计算后的置信度值; 比例标尺代表氨基酸5%的置换概率The tree was constructed by Neighbor-Joining Method; the bold ones were got in this study, names 4136-2 and 4148-B1 expressing clones from sulfide rock 4136-2 and 4148-B1, respectively; the values on the branches expressing 1000 bootstrap and the scale expressing 5% exchange rate
NCBI比对结果为甲烷古菌mcrA的43个序列,用MAGE 3.1构建分子进化树, 如图3所示。4136-2的mcrA序列与已分离的嗜热菌有最大同源关系, 在89%~97%。与克隆子的同源性较菌株低, 在70%~72%。4136-2硫化物中的甲烷菌群落97.06%来自在甲烷球菌(Methanococcales), 2.94%来自甲烷嗜高热菌(Methanopyrales)。4148-B1序列没有找到氨基酸同源性超过47%的mcrA序列。核苷酸比对可以找到同源性在 69%~72%的同源序列, 说明这些克隆子序列属于甲烷菌的甲基辅酶M的编码基因。多样性类型4136-2-8与Methanopyrus kandleri[20](GenBank序列号: AE010358)有最大同源关系, 同源性为95%。多样性类型 4136-2-14与Methanococcus voltae[21](GenBank序列号: X07793)有最近同源关系,同源性为 89%。与这两个序列同源关系最近的克隆子是来源于酸性泥炭的克隆子 FinME_09[22](GenBank序列号: DQ099689), 同源性分别为71%和70%。4136-2-1与 Methanocaldococcus jannaschii[23](GenBank序列号: F64405)的同源性为 97%。4136-2-20和4136-2-24与Methanocaldococcus jannaschii DSM 2661[23](GenBank 序列号: L77117)有最大同源关系, 同源性分别为93%和97%。与这三个序列最同源的克隆子 novmcr4[24](GenBank序列号:AF525516)来自富营养湖, 与三个序列的同源性在71%~72%间。样品4148-B1的mcrA序列4148-B1-3与来源于富营养湖的克隆子novmcr2[24](GenBank序列号: AF525515)同源关系最近, 为 47%。与之同源关系最近的菌株是嗜热菌 Methanopyrus kandleri AV19[25](GenBank序列号: AM114193)氨基酸同源性为 43%, 核苷酸同源性为 72%。另一类型克隆子4148-B1-59与来源于富含甲烷的水稻土壤的克隆子RC-I[25](GenBank 序 列 号 : NC_009464)及Methanosphaera stadtmanae DSM 3091[26](GenBank序列号: NC_007681)的氨基酸同源性最高, 都是43%。核苷酸比对时, 与RC-I的同源性为69%。
3 分析与讨论
3.1 热液口金属硫化物甲烷菌类型分析
甲烷菌甲基辅酶M编码基因mcrA序列的系统发育树分析已经表明在胡安·德富卡热液场热液口硫化物岩石中甲烷古菌的类型比较丰富, 存在不同类型的甲烷产生菌及一类未知的甲烷菌。温度较低的硫化物4136-2中的甲烷产生菌与已分离培养的甲烷产生菌有最近的同源关系。说明胡安·德富卡热液口与其他热液口一样, 其附近环境中甲烷菌是热液口附近比较常见的菌种。但热液通道外壁的硫化物 4148-B1距离喷口较近, 其中的甲烷菌与已知的菌完全不同。
甲烷产生菌是一类绝对厌氧菌, 是产生甲烷的生物来源[27]。甲烷球菌(Methanococcales)生活在中温、嗜热或极端嗜热的环境中, 通过还原CO2和水生成甲烷产生能量供其活动[28]。胡安·德富卡热液场周围环境含有丰富的二氧化碳和氢气, 为甲烷产生菌的代谢和活动提供物质和能量[6,29-30]。本试验中克隆子数最大的一个多样性类型 4136-2-1(占总数的82.4%)的 97%的同源菌株 Methanocaldococcus jannaschii(GenBank序列号: F64405)最初分离自太平洋EPR热液场的白烟囱附近的沉积物中, 发表在《Science》杂志上。另外一类多样性类型属于甲烷火菌(Methanopyrus)。与本试验中得到的序列最近亲缘关系的甲烷火菌是 Methanopyrus kandleri AV19(GenBank序列号: AE010358)。它是一株超嗜热菌, 分离自加利福尼亚海湾二氧化碳和氢气丰富的黑烟囱中, 与其他产甲烷生物一样还原二氧化碳和水生成甲烷。
3.2 金属硫化物岩石中甲烷菌类型与环境的关系
4136-2和 4148-B1两个金属硫化物样品的甲烷菌甲基辅酶M编码基因mcrA反映的甲烷菌类型反映了胡安·德富卡热液口的甲烷菌的类型和变化。4136-2样品中得到的甲烷菌全部是甲烷产生古菌,且与高温来源的甲烷球菌及甲烷火菌有最近的同源关系。这反映了4136-2样品采集自热液喷口附近, 并且喷口附近含有丰富的二氧化碳和氢气, 这与胡安·德富卡的文献资料报道的相同。4148-B1样品的克隆子虽然与已知的克隆子序列同源性不高, 但也与热液口或水稻田等甲烷丰富的环境相关。其中一类与嗜热菌的核苷酸同源性达到 72%, 氨基酸同源性为47%。由于序列很新, 更多的研究需要继续。
致谢: 本试验是在国家海洋局第三海洋研究所海洋生物遗传资源重点实验室完成。实验期间受到实验室各位老师的指导, 在此表示感谢。
[1] Childress C, Fisher R. The biology of hydrothermal vent animals: physiology, biochemistry, and autotrophic symbioses, Oceanogr[J]. Mar Biol Annu Rev, 1992,30:337-441.
[2] Lilley M D, Olson E J. Methane and hydrogen in active hydrothermal systems[J]. Annu Gold Conf, 2001, 11:3682.
[3] Lilley M D. Real-time mapping of hydrothermal plumes on the endeavour segment of the Juan de Fuca[J]. Eos Trans Am Geophys Union, 1995,76: 420.
[4] Lilley M D, Lupton J E, Damm K L V. Volatiles in the 9±N hydrothermal system: a comparison of 1991 and 1992 data[J]. Eos Trans Am Geophys Union, Fall Meet Suppl,1992. 73(43): 524.
[5] Butterfield D A, Massoth G J. Geochemistry of the North Cleft Segment vent fluids-temporal changes in chlorinity and their possible relation to recent volcanism[J]. J Geophys Res, 1994, 99: 4951-4968.
[6] McCollom T M. Geochemical constraints on primary productivity in submarine hydrothermal vent plumes[J].Deep-Sea Res I, 2000, 47: 85-101.
[7] Lilley M D. Anomalous CH4and NH4+concentrations at an unsedimented mid-ocean-ridge hydrothermal system[J].Nature, 1993, 364: 45-47.
[8] Lilley M D. Anomalous CH4and NH4+concentrations at an unsedimented mid-ocean-ridge hydrothermal system[J].Nature, 1993, 346: 45-47.
[9] Jones W J, Paynter M J B, Gupta R. Characterization of Methanococcus maripaludis sp. nov., a new methanogen isolated from salt marsh sediment[J]. Arch Microbiol,1983,153: 91-97.
[10] Jeanthon C. Methanococcus vulcanius sp. nov., a novel hyperthermophilic methanogen isolated from East Pacific Rise, and identification of Methanococcus sp. DSM 4213T as Methanococcus fervens sp. nov[J]. Int J Syst Bacteriol,1999, 49: 583-589.
[11] Jeanthon C. Methanococcus infernus sp. nov., a novel hyperthermophilic lithotrophic methanogen isolated from a deep-sea hydrothermal vent[J]. Int J Syst Bacteriol, 1998,48: 913-919.
[12] Summit M. Ecology, physiology, and phylogeny of subseafloor thermophiles from mid-ocean ridge environments[M]. Seattle :University of Washington, 2000.
[13] Maniatis T, Fritsch E F, Sambrook J. Molecular Cloning:A Laboratory Manual[M]. New York: Cold Spring Harbour Laboratory Press, 1982: 440-468.
[14] Zhou J, Bruns M A, Tiedje J M. DNA recovery from soils of diverse composition[J]. Appl Environ Microbiol, 1996,62(2): 316-322.
[15] Hales B A. Isolation and identification of methanogen-specific DNA from blanket bog peat by PCR amplification and sequence analysis[J]. Appl Environ Microbiol,1996,62(2): 668-675.
[16] Good I L. The population frequencies of species and the estimation of population parameters[J]. Biometrika, 1953,40: 237-264.
[17] Schloss P D, Handelsman J. Introducing DOTUR, a computer program for defining operational taxonomic units and estimating species richness[J]. Appl Environ Microbiol, 2005, 71(3): 1501-1506.
[18] Chao A. Non-parametric estimation of the number of classes in a population[J]. Scand J Stat, 1984, 11:265-270.
[19] Chao A, Lee S M. Estimationg the number of classes via sample coverage[J]. J Am Stat Assoc, 1992, 87: 210-217.
[20] Slesarev A I. The complete genome of hyperthermophile Methanopyrus kandleri AV19 and monophyly of archaeal methanogens[J]. Proc Natl Acad Sci U S A, 2002, 99(7):4644-4649.
[21] Klein A. Comparative analysis of genes encoding methyl coenzyme M reductase in methanogenic bacteria[J]. Mol Gen Genet, 1988, 213(2-3): 409-420.
[22] Metje M, Frenzel P. Effect of temperature on anaerobic ethanol oxidation and methanogenesis in acidic peat from a northern wetland[J]. Appl Environ Microbiol, 2005,71(12): p. 8191-8200.
[23] Bult C J. Complete genome sequence of the methanogenic archaeon, Methanococcus jannaschii[J]. Science, 1996,273(5278): 1058-1073.
[24] Earl J. Analysis of methanogen diversity in a hypereutrophic lake using PCR-RFLP analysis of mcr sequences[J].Microb Ecol, 2003, 46(2): 270-278.
[25] Erkel C. Genome of rice cluster I archaea——the key methane producers in the rice rhizosphere[J]. Science,2006, 313(5785): 370-372.
[26] Fricke W F. The Genome sequence of methanosphaera stadtmanae reveals why this human intestinal archaeon is restricted to methanol and H2for methane formation and ATP synthesis[J]. J Bacteriol, 2006, 188(2): 642-658.
[27] Singh N. Isolation and characterization of methylotrophic methanogens from anoxic marine sediments in Skan Bay,Alaska: description of Methanococcoides alaskense sp.nov., and emended description of Methanosarcina baltica[J]. Int J Syst Evol Microbiol, 2005,55: 2531-2538.
[28] Euzeby J P, Tindall B J. Nomenclatural type of orders:corrections necessary according to rules 15 and 21a of the bacteriological code (1990 Revision), and designation of appropriate nomenclatural types of classes and subclasses[J]. Int J Syst Evol Microbiol, 2001, 51: 725-727.
[29] McCollom T M, Shock E L. Geochemical constraints on chemolithoautotrophic metabolism by microorganisms in seafloor hydrothermal systems[J]. Geochim Cosmochim Acta, 1997,61(20): 4375-4391.
[30] Delaney J R. The quantum event of oceanic crustal accretion: Impacts of diking at mid-ocean ridges[J]. Science,1998, 281(5374): 222-230.
Studies on the biodiversity of methanogens from hydrothermal vent chimney
WANG Shu-fang1,2, CHEN Yue1, QIAN Yuan-yuan1
(1. Huaihai Institute Technology, Oceanography Institute, Lianyungang 222005, China; 2. Key Laboratory of Marine Biotechnology in Jiangsu Province, Huaihai Institute of Technology, Lianyungang 222005, China)
Jun.,25,2011
juan de fuca (JDF); sulfide; methogen; mcrA
The chimney sulfides (named 4136-2 and 4148-B1) obtained from Juan de Fuca Ridge were used as samples, aiming at mcrA gene amplification, clone library construction and phylogenetic tree analysis. The results showed that there were only CH4production microbes but no CH4oxidation microbes in the samples and the species in the two sulfides were different. The methanogen from 4136-2 sulfide rock were linked to a high temperature environment. These methanogen belong to Methanocaldococcus and Methanopyrus kandleri. The similarities of protein sequences between 4136-2 clones and isolated Methanocaldococcus and Methanopyrus kandleri strains were from 89% to 97%, and The similarities of mucleotide sequences between 4136-2 clones and isolated Methanocaldococcus and Methanopyrus kandleri strains were from 92% to 100%. The mcrA sequences from 4148-B1 sulfide rock had low identities with sequences deposited in GenBank. The similarities of mucleotide sequences identities of 4148-B1 clones and published mcrA sequences were form 69% to 72%, and the similarities of protein sequences were form 43% to 37%. This also linked to its super high temperature environment and may suggest there were un-discovered methanogen living in the vent.
Q93.3
A
1000-3096(2012)06-0031-08
2011-06-25;
2011-12-11
淮海工学院引进人才基金项目(KQ09021)
王淑芳(1976-), 女, 河北安国人, 讲师, 博士。从事微生物资源研究。电话: 0518-85895427, E-mail: wangshufang2001@hotmail.com
(本文编辑:梁德海)

