影响碳青霉烯类抗生素耐药性的VIM型金属β-内酰胺酶关键残基的研究
陈 浩, 石 屹 峰
( 大连工业大学 生物工程学院, 辽宁 大连 116034 )
0 引 言
碳青霉烯类抗生素是自1976年发现并逐步发展起来的一组新型的β-内酰胺类抗生素,其抗菌谱广,对绝大多数革兰氏阳性菌与阴性菌、需氧菌与厌氧菌均具有良好的抗菌作用。现今应用于临床的主要品种有亚胺培南、美罗培南等。碳青霉烯类对细菌产生的各类β-内酰胺酶都很稳定,但是金属β-内酰胺酶对碳青霉烯类耐药[1]。金属β-内酰胺酶(Metallo β-Lactamases, MBLs)是一类活性依赖二价金属阳离子(主要是Zn(Ⅱ))的β-内酰胺酶,根据Ambler等对β-内酰胺酶分子结构分类,MBLs属于B类。该类酶除了能够有效地水解大部分的β-内酰胺类抗生素,还能够水解抗菌谱广的碳青霉烯类抗生素,并且常用的β-内酰胺酶抑制剂(如克拉维酸、舒巴坦、他唑巴坦等)对其无作用。绝大多数获得性MBLs基因位于整合子等可移动的基因元件,可通过结合性质粒、转座子等发生转移,并且整合子通常携带有临床重要的抗性基因,这就造成了MBLs的广泛耐药,成为临床治疗细菌性感染疾病的一个重大难题。自1990年以来,MBLs在世界范围内被广泛发现,主要是IMP、VIM型,还包括SPM、GIM和SIM等多种类型。其中VIM型起初来源于铜绿假单胞菌Pseudomonasaeruginosa,迄今为止已发现19种[2],它的活性依赖于2个Zn(Ⅱ)。VIM型不仅能够水解青霉素类和头孢霉素类抗生素,而且能够水解碳青霉烯类抗生素,临床常用的β-内酰胺酶抑制剂对VIM型也不起任何抑制作用,因此携带MBLs的耐药菌被视为人类面临的紧迫的传染和生物安全问题。作者以VIM型为例,通过使用生物信息学方法的系统研究,找出影响碳青霉烯类抗生素水解催化作用的关键残基,以期为将来合理设计金属β-内酰胺酶抑制剂奠定基础。
1 材料与方法
1.1 VIM型金属β-内酰胺酶的序列及结构
以VIM型金属β-内酰胺酶(MBLs)为研究对象,其氨基酸序列来源于NCBI protein数据库(http://www.ncbi.nlm.nih.gov),结构从PDB数据库搜索得到。
1.2 VIM型金属β-内酰胺酶结构与功能研究
利用clustalw软件对VIM型MBLs氨基酸序列(14种VIM型序列来源列于表1)进行序列比对及同源性分析,找出突变位点,在MEGA3.1软件中生成最终的比对结果并构建系统进化树。使用Pymol软件对蛋白质结构进行观察和分析,并结合动力学数据确定催化功能的关键残基。
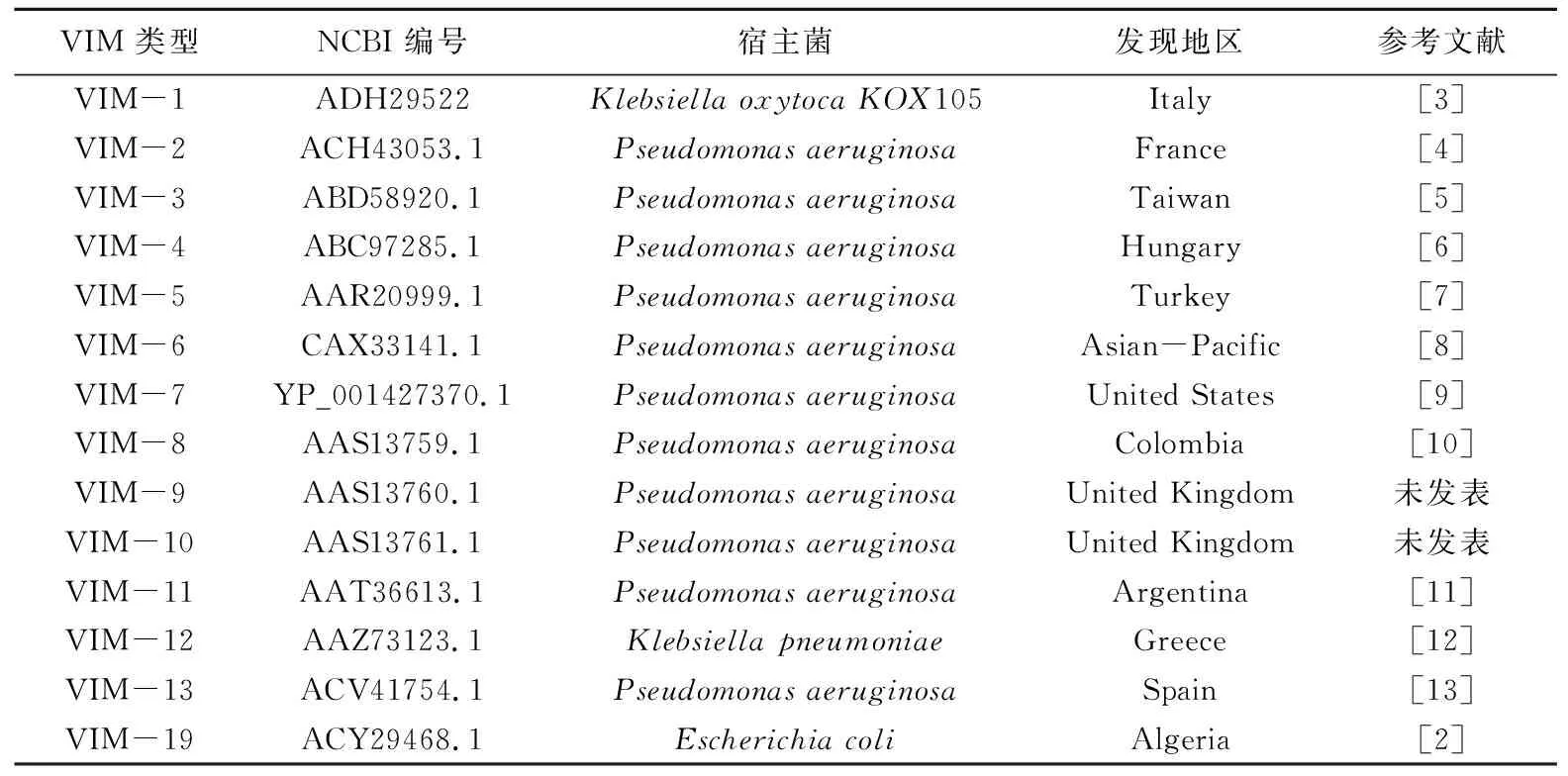
表1 VIM型金属β-内酰胺酶氨基酸序列NCBI编号及宿主和地区来源
2 结果和讨论
2.1 VIM型金属β-内酰胺酶结构和催化机制
所有的MBLs都具有αββα折叠结构,以β折叠为核心,α螺旋环绕在β折叠的周围,活性中心位于两个αβ结构域中间,并依赖金属离子的存在而发挥其活性作用。VIM型金属β-内酰胺酶同样也具有αββα折叠结构,从VIM-2的晶体结构(图1)可以看出该蛋白质结构两半是相似的αβ结构域,该酶必需的Zn(Ⅱ)辅助因子所在的活性中心位于2个αβ结构域交界处浅沟中[14]。
影响碳青霉烯类水解的关键残基的位置由黑点标出,L3和L1环区所在的位置如所指。
VIM型是双锌原子型MBLs以两个Zn(Ⅱ)作为辅助因子, Zn1与3个His (His116、His118、His196)及1个水分子(与Zn2共用)形成了一个暂时的四面体结构,在催化过程中Zn1与Asn233的侧链形成一个氧负离子洞,稳定了该反应的反应中间体, Zn2与Asp120、Cys221、His263结合。两个Zn(Ⅱ)与共用的水分子结合后形成亲核试剂攻击β-内酰胺环的羰基,使β--内酰胺环的C—N键裂解,β-内酰胺类抗生素水解失效[15]。

图1 VIM-2型金属β-内酰胺酶结构(PDB 1KO3)
2.2 VIM型金属β-内酰胺酶序列比对
在酶分子的进化过程当中,一些功能上起重要作用的残基在不同种类但是同源的蛋白质中表现出高度的保守性,这些序列可以很容易地通过多序列比对的方法找到。根据已知的VIM-2晶体结构找出活性中心两个Zn(Ⅱ)结合的配基共有6个 (图1)。根据氨基酸序列比对结果(图2)可以看出,其他13种VIM型活性中心与Zn1和Zn2结合的这6个作为配基氨基酸的位点是高度保守的。由于所有VIM型Asn233的侧链参与形成氧负离子洞[16],所以Asn233也是保守的(图2)。另外,在60~66号位有一个重要的环区——L1[16],在S11与H4之间220~240号位还有一个很重要的环区——L3[17](图1),这两个环区邻近酶活性中心所在的位置, 影响MBLs对底物的水解。

黑框,活性中心保守序列;*,发生替换的关键残基;上方标注数字,BBL序列编号; H,α螺旋;S,β折叠;L,环区
2.3 VIM金属β-内酰胺酶催化碳青霉烯类抗生素水解关键残基
表2列出了已发表的VIM-1、VIM-2、VIM-7、VIM-19、VIM-11的碳青霉烯类的酶动力学参数。可以看出,VIM-2表现出对碳青霉烯类很高的水解效率,水解美罗培南和亚胺培南的kcat/Km分别是VIM-1的10倍和30倍。VIM-2的大部分与VIM-1不同的位点(35,148,215,246,251,257,258,284,287,294,299)都位于远离活性中心的位置,但是有3个残基(223,224,228)的替换在L3环区上(图2)。由于 L3环区邻近酶活性中心所在的位置,这3个残基的替换可能影响底物与酶的结合[17]。VIM-1的224号位是His,而在VIM-2中则是Tyr,它的侧链变长对底物的结合和稳定性有利。VIM-1的228号位是Ser,而VIM-2中则是Arg,其侧链变长伸进活性中心可与底物发生相互作用。所以对于碳青霉烯类酶动力学参数的不同很可能是由于VIM-2在224号位和228号位发生的替换导致增强了对底物的相互作用所引起的。

表2 碳青霉烯类酶动力学参数
VIM-7对碳青霉烯类的水解效率同样高于VIM-1。VIM-7在224号位和228号位发生了与VIM-2相同的替换:His224Tyr,Ser228Arg。Pro68Ser和Tyr218Phe这两个位点的替换仅仅发生在VIM-7中,Pro68是金属β-内酰胺酶中的较为保守的残基,通过与His263(Zn2的配基)在主链上羰基化氧形成氢键。尽管此残基被Ser所取代,可能增加了L1的柔性,但仍能形成氢键[16],不降低对碳青霉烯类的水解。
VIM-11表现出与VIM-2基本相同的水解效率,VIM-11 在223、224、228残基位置发生了与VIM-2相同的替换。
VIM-19对碳青霉烯类的水解效率依然高于VIM-1。VIM-1与VIM-19之间仅有两个位点不同:Asn215Lys和Ser228Arg。Asn215Lys位于远离活性中心的位置, Arg228可导致VIM-19对碳青霉烯类水解效率提高[2]。与VIM-1相比,这个残基的替换与VIM-2、VIM-7、VIM-11中相同。
2.4 VIM型进化树
在VIM型MBLs中VIM-1、VIM-2、VIM-7的基因发现的频率最高[9]。根据现今发现的14种VIM型MBLs序列比对结果生成进化树。根据进化树VIM型被分为3组:VIM-1组包括VIM-4、VIM-5、VIM-19等;VIM-2组包括VIM-3、VIM-6、VIM-8、VIM-9、VIM-10、VIM-11等[19],它们与VIM-1在序列上有高达90%的同源性。VIM-7单独分为一组[19],由于它与其他VIM型的酶有显著的不同,VIM-7与VIM-1氨基酸序列同源性只有77%(图3)。
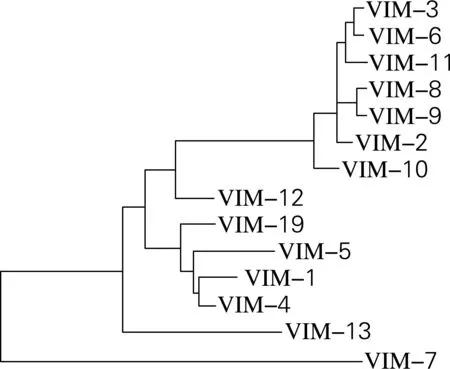
图3 VIM型金属β-内酰胺酶进化树
3 结 论
通过对VIM型MBLs氨基酸序列比对并且结合动力学参数分析其结构和功能的关系,确定了结合两个Zn(Ⅱ)发挥催化作用的保守残基分别是His116、His118、His196和Asp120、Cys221、His263,以及形成氧负离子洞的Asn233,还发现了L3环区上残基改变会影响碳青霉烯类的水解。通过找出影响VIM型金属β-内酰胺酶对碳青霉烯类抗生素水解的这些关键残基,为将来合理设计金属β-内酰胺酶抑制剂奠定基础。
[1] QUEENAN A M, BUSH K. Carbapenemases:the versatile β-lactamases[J]. Clinical Microbiology Reviews, 2007, 20(3):440-458.
[2] RODRIGUEZ-MARTINEZ J M, NORDMANN P, FORTINEAU N, et al. VIM-19, a metallo-β-lactamase with increased carbapenemase activity fromEscherichiacoliandKlebsiellapneumoniae[J]. Antimicrobial Agents and Chemotherapy, 2010, 54(1):471-476.
[3] CARATTOLI A, ASCHBACHER R, MARCH A, et al. Complete nucleotide sequence of the IncN plasmid pKOX105 encoding VIM-1, QnrS1 and SHV-12 proteins inEnterobacteriaceaefrom Bolzano, Italy compared with IncN plasmids encoding KPC enzymes in the USA[J]. Antimicrobial Agents and Chemotherapy, 2010, 65(10):2070-2075.
[4] POIREL L, NAAS T, NICOLAS D, et al. Characterization of VIM-2, a carbapenem-hydrolyzing metallo-β-lactamase and its plasmid- and integron-borne gene from aPseudomonasaeruginosaclinical isolate in France[J]. Antimicrobial Agents and Chemotherapy, 2000, 44(4):891-897.
[5] YAN J, HSUEH P, KO W, et al. Metallo-β-Lactamases in clinicalPseudomonasisolates in Taiwan and identification of VIM-3, a novel variant of the VIM-2 enzyme[J]. Antimicrobial Agents and Chemotherapy, 2001, 45(8):2224-2228.
[6] LIBISCH B, MUZSLAY M, GACS M, et al. Molecular epidemiology of VIM-4 metallo-beta-lactamase-producingPseudomonassp. isolates in Hungary[J]. Antimicrobial Agents and Chemotherapy, 2006, 50 (12):4220-4223.
[7] BAHAR G, MAZZARIOL A, KONCAN R. et al. Detection of VIM-5 metallo-beta-lactamase in aPseudomonasaeruginosaclinical isolate from Turkey[J]. Antimicrobial Agents and Chemotherapy, 2004, 54(1):282-283.
[8] CASTANHEIRA M, BELL J M, TURNIDGE J D, et al. Dissemination and genetic context analysis of bla(VIM-6) amongPseudomonasaeruginosaisolates in Asian-Pacific Nations[J]. Clinical Microbiology and Infection, 2010, 16(2):186-189.
[9] SAMUELSEN Φ, CASTANHEIRA M, WALSH T R, et al. Kinetic characterization of VIM-7, a divergent member of the VIM metallo-β-lactamase family[J]. Antimicrobial Agents and Chemotherapy, 2008, 52(8):2905-2908.
[10] CRESPO M P, WOODFORD N, SINCLAIR A, et al. Outbreak of carbapenem-resistantPseudomonasaeruginosaproducing VIM-8, a novel metallo-beta-lactamase, in a tertiary care center in Cali, Colombia[J]. Joural of Clinical Microbiology, 2004, 42(11):5094-5101.
[11] PASTERAN F, FACCONE D, PETRONI A, et al. Novel variant blaVIM-11of the metallo-β-lactamase blaVIMfamily in a GES-1 extended-spectrum-β-lactamase-producingPseudomonasaeruginosaclinical isolate in Argentina[J]. Antimicrobial Agents and Chemotherapy, 2005, 49(1):474-475.
[12] IKONOMIDIS A, LABROU M, AFKOU Z, et al. First occurrence of anEscherichiacoliclinical isolate producing the VIM-1/VIM-2 hybrid metallo-β-lactamase VIM-12[J]. Antimicrobial Agents and Chemotherapy, 2007, 51(8):3038-3039.
[13] JUAN C, BECEIRO A, GUTIERREZ O, et al. Characterization of the new metallo-β-lactamase VIM-13 and its integron-borne gene from aPseudomonasaeruginosaclinical isolate in Spain[J]. Antimicrobial Agents and Chemotherapy, 2008, 52(10):3589-3596.
[14] CROWDER M W, SPENCER J, VILA A J.Metallo-β-lactamases:novel weaponry for antibiotic resistance in bacteria[J]. Accounts of Chemical Research, 2006, 39(10):721-728.
[15] PAGE M I. Understanding metallo β-lactamases[J]. ASM News, 2002, 68(5):217-221.
[16] MOALI C, ANNE C, LAMOTTE-BRASSEUR J, et al. Analysis of the importance of the metallo-β-lactamase active site loop in substrate binding and catalysis[J]. Chemistry & Biology, 2003, 10:319-329.
[17] DOCQUIER J D, LAMOTTE-BRASSEUR J, GALLENI M, et al. On functional and structural heterogeneity of VIM-type metallo-β-lactamases[J]. Journal of Antimicrobial Chemotherapy, 2003, 51:257-266.
[18] MARCHIARO P, TOMATIS P E, MUSSI M A, et al. Biochemical characterization of metallo-β-lactamase VIM-11 from aPseudomonasaeruginosaclinical strain[J]. Antimicrobial Agents and Chemotherapy, 2008, 52(6):2250-2252.
[19] POURNARAS S, IKONOMIDIS A, TZOUVELEKIS L S, et al. VIM-12, a novel plasmid-mediated metallo-β-lactamase fromKlebsiellapneumoniaethat resembles aVIM-1/VIM-2 hybrid[J]. Antimicrobial Agents and Chemotherapy, 2005, 49(12):5153-5156.

