巨大革耳遗传多样性的ISSR分析*
江玉姬,谢宝贵,刘新锐,邓优锦,陈东兴,朱 坚
(1.福建农林大学食品科学学院,福建 福州 350002;2.福建农林大学菌物研究中心,福建 福州 350002;3.福建农林大学园艺学院,福建 福州 350002)
巨大革耳(Panus giganteus) 又名大杯伞、大杯蕈、大漏斗菌、猪肚菇、笋菇、大杯香菇等,隶属真担子菌门(Basidiomycota)、担子菌纲 (Basidiomycetes)、多孔菌目(Polyporales)、多孔菌科(Polyporaceae)、革耳属 (Panus)[1]。自然环境中夏、秋季节大量群生或丛生于林地腐枝层上,主要分布在我国的河北、山西、黑龙江、青海等地区。从自然界的生长习性和分布可以看出,巨大革耳(猪肚菇)是一种中高温型的美味珍稀食用菌种类,菌肉肥厚,个体较大,味道鲜美,是一种很有发展前途的夏、秋季栽培食用菌[2]。人工栽培多进行熟料袋栽,栽培方法与常规木腐生菌相同。但巨大革耳(猪肚菇)人工栽培历史很短,目前栽培用种主要来源于野生资源的驯化和引种,存在着同种异名,同名异种的现象,给生产和育种带来了混乱产生了严重的后果。本文应用ISSR分子标记对国内不同地区收集或分离的9株巨大革耳进行了分子遗传多样性研究,为巨大革耳菌种管理、遗传资源的保护和充分利用、育种研究工作的开展等提供基础数据。
1 材料与方法
1.1 实验材料
1.1.1 供试菌种
9株供试菌株,由福建食用菌株,质资源保藏管理中心提供,试验编号及其它信息如表1所示。
1.1.2 试剂
20个ISSR引物为上海Sangon生物工程公司生产;PCR kit、500 bp DNA Ladder Marker购自 TaKaRa公司,其它试剂均为国产分析纯。
1.1.3 供试培养基
斜面固体培养基:马铃薯200 g、葡萄糖20 g、琼脂20 g,水1 000 mL,pH值自然。
液体培养基:马铃薯200 g、葡萄糖20 g,水1 000mL,pH值自然。

表1 供试巨大革耳菌株及来源
出菇培养基:棉籽壳85%、麦麸10%、玉米粉3%、碳酸钙或石膏2%,含水量65%~70%,pH6.0。
1.1.4 巨大革耳菌丝体制备
供试菌株在PDA试管斜面培养基上培养7 d~10 d后,转接到新鲜PDA试管斜面培养基上培养至满管,用接种耙耙碎菌块,接入250 mL三角瓶液体培养基中,每个菌株各接2瓶,置于120 r·min-1摇床上26℃恒温培养10 d,用纱布过滤,蒸馏水冲洗干净,滤纸吸干水分,称取1 g菌丝用滤纸包好,保存于-20℃下备用。
1.1.5 DNA提取
取1 g菌丝放入研钵,加入液氮迅速研磨成粉末,转入7 mL的离心管,采用CTAB法提取DNA,-20℃保存备用,具体操作参考文献[3]、[4]。
1.1.6 ISSR扩增
ISSR 反应体系 25 μL:模板 DNA 1.2 ng·μL-1,引物0.4 μmol·L-1,dNTPs 0.2 mmol·L-1,MgCl22 mmol·L-1,Taq DNA聚合酶2 U,PCR缓冲液1×,具体操作参考文献[3]、 [4],将重复性好、强度较高、清晰可辨无争议、能体现菌株间差异的条带确认为标记条带,进行相应的统计和分析。
1.1.7 数据统计与分析
ISSR扩增产物以0、1统计建立ISSR数据库。将相同引物PCR扩增产物中电泳迁移率一致的标记条带确认为同一条带,扩增阳性记录为“1”,扩增阴性记录为“0”,利用SPSS10.0分析软件的Jaccard方法计算样品间的遗传相似性,用类平均法(UPGMA) 对ISSR统计形成“0/1”表型数据矩阵进行聚类分析,生成遗传距离聚类图[5,6]。
1.1.8 出菇实验
栽培出菇实验参考刘斌等[7]的方法。
2 结果与分析
2.1 ISSR结果与分析
从20个引物筛选出4个多态性好的引物(表2),扩增的结果见图1。
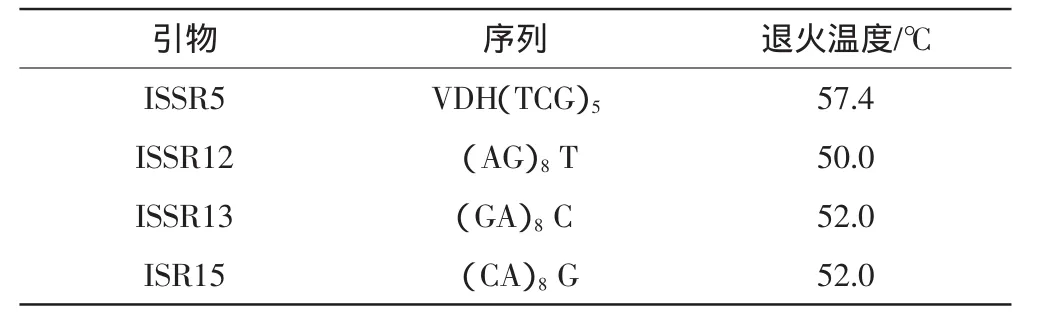
表2 ISSR引物序列

图1-1 引物ISSR5和ISSR12对巨大革耳菌株ISSR-PCR扩增图谱

图1-2引物ISSR13和ISSR15的扩增电泳图
从图1中可知,4个引物对9个菌株扩增的片段大小在1.0 kb~3.0 kb之间,各个引物扩增的位点数分别为7、8、5、7,平均位点数为6.25;平均每个引物产生个5.75个多态性条带。ISSR共扩增出27个位点,多态性条带总共为23,多态率为85.19%。经过软件处理后,建立树状图 (图 2)。

图2 9个菌株的ISSR聚类树状图
从图2可以看出,5号、6号、7号、9号菌株聚为一类,它们的相异系数为0,可能为同种异名;3号、4号菌株及1号、8号菌株各聚为一类,2号菌株单独一类;2号菌株与其它8个菌株的遗传距离很远,3号、4号、1号、8号菌株与5号、6号、7号、9号菌株有一定的遗传距离。当相异系数D为0.20水平上,9个菌株聚为2类:Ι类包括2号菌株;Π类包括1号、3号、4号、5号、6号、7号、8号、9号菌株。2号菌株与其它8个菌株的遗传距离很远,故对9个菌株进行了栽培出菇实验,以观察子实体的形态是否有差异。
2.2 子实体形态分析
根据栽培出菇实验结果表明,C.m0002菌株的子实体(图3)明显不同于其它菌株,酷似多脂鳞伞(黄伞),是同名异种,单独归为Ι类;其它8个菌株均为巨大革耳,子实体的形态较一致,部分菌株的子实体形态见图4、图5和图6,归为第Π类。子实体特征分析的结果与ISSR分类的结果吻合。

图3 C.m002菌株的子实体图

图4 C.m003菌株的子实体图

图5 C.m001菌株的子实体图

图6 C.m007菌株的子实体图
3 讨论
近年来,不断快速发展的DNA分子标记技术越来越多的被应用在研究种质资源的多样性以及分子辅助遗传育种上,显示出了极大的优越性。它不仅能快速的获得分析结果,而且其结果不受外界环境因素和生长发育阶段的影响;所以,分子标记已经成为分子水平的种质资源亲缘关系及检测种质资源多样性和品种鉴定的有效工具[8]。
ISSR是由 Zietkiewi CZ等[9]创建的一种简单重复序列间扩增多态性分子标记。ISSR技术与常规PCR有相同的反应条件,该技术克服了限制性内切酶片段长度多态性RFLP标记、SSR标记和随机扩增多态性DNA标记(RAPD)的技术限制,具有操作简单、引物扩增的结果重现性较好、模板DNA用量少等优点,且不需要知道基因组序列,可以检测基因组许多位点的差异[6-8],已广泛应用于动植物种质鉴定、遗传作图和基因定位,尤其在分子遗传学基础贫乏的作物研究中显示出极大的优势[10,11]。但在食用菌中的应用还不多,在香菇[8]、杏鲍菇[12]、金针菇[13]、松口蘑及花鹅膏菌[11]、天麻[14]、侧耳属[10]等品种的鉴定及亲缘关系研究中的应用已有报道,但用ISSR技术分析巨大革耳菌株间的亲远关系目前没有相关的文献报道。
9个菌株的 ISSR分析中,5号(C.m0005)、6号(C.m0006)、7号 (C.m0007)、9号 (C.m0009) 的遗传距离为零,结合子实体的形态特征可认定它们为同种内的菌株。C.m0003、 C.m0004、 C.m0005、 C.m0006、 C.m0007、 C.m0008、C.m0009七个栽培菌株分别来自武汉、湖北嘉鱼、福州、三明4个不同的地方,其中C.m0008与其它6个菌株有一定的遗传距离,说明有一定的遗传多态性,但不丰富。而C.m0001和C.m0002菌株与其它7个菌株间的遗传距离较远。出菇实验结果表明,C.m0002菌株的子实体似多脂鳞伞(黄伞),是同名异种;其它8个菌株均为巨大革耳。本研究的结果以及ISSR标记在香菇、杏鲍菇、金针菇、侧耳属等的应用,说明ISSR分子标记在食用菌的种质资源遗传多样性、亲缘关系分析中的应用是可信的。本研究结果也将为巨大革耳的生产和育种以及种菌管理提供基础数据。
[1]董洪新,蔡德华,李玉.猪肚菇的研究现状及展望[J].中国食用菌,2010,29(3):3-6.
[2]彭智华.大杯蕈的生物学特性及人工驯化的研究[M].杭州:浙江农业大学,1998.
[3]熊芳,郑闽江,刘新锐,等.鲍鱼菇种质资源SCAR标记的建立及其初步应用[J].中国农学通报,2010,26(11):330-335.
[4]傅俊生,刘新锐,谢宝贵,等.草菇SCAR遗传标记建立及其杂种鉴定应用[J].中国农学通报,2010,26(17):41-46.
[5]Ferriol M,Picó B,Nuez F.Genetic diversity of some accessions of Cucurbita maxima from Spain using RAPD and SRAP markers[J].Genetic Resource and Crop Evolution,2003(50):227-238.
[6]周延清.DNA分子标记技术在植物研究中的应用[M].北京:化学工业出版社,2005.
[7]刘斌,上官舟建,林汝楷.大杯蕈不同菌株栽培品比试验[J].食用菌,2005(5):15-16.
[8]秦莲花,宋春艳,谭琦,等.用ITS和ISSR分子标记技术鉴别香菇生产用种[J].菌物学报,2006,25(1):94-100.
[9]Zietkiewicz E,Rafslski A,Labuda D.Genome fingerprinting by simple sequence repeat(SSR)-anchored polymerase chain reaction amplification[J].Genomics,1994,20(2):176-183.
[10]徐晓波,蒋冬花,李杰,等.应用ISSR分子标记探讨酵母菌种间亲缘关系[J].浙江师范大学学报,2010,33(1):89-94.
[11]边银丙,宋小亚.几种新型DNA分子标记及其在食用菌研究中的应用[J].食用菌学报,2006,13(1):78-81.
[12]冯伟林,蔡为明,金群力,等.ISSR分子标记分析杏鲍菇菌株的遗传并差异研究[J].中国食用菌,2009,28(1):47-49.
[13]宿红艳,王磊,明永飞,等.ISSR分子标记技术在金针菇菌株鉴别中的应用[J].生态学杂志,2008,27(10):1725-1728.
[14]关萍,马丹炜,王贵侠,等.利用ISSR标记对天麻的贵州种群遗传多样性分析[J].北京林业大学学报,2007,29(6):35-40.

