食管鳞癌组织中TIG1基因甲基化及其mRNA表达的研究
童 强, 刘晓波△, 罗和生, 李胜保, 余宗涛, 张吉才, 高 波, 蒙建超
(湖北医药学院附属太和医院 1消化内科,3检验部,湖北 十堰442000;2武汉大学人民医院消化内科,湖北武汉430060)
全世界每年大约有48万食管癌(esophageal carcinoma,EC)新患病例,超过40万人死于该病,主要有2种类型:好发于中上段的食管鳞状细胞癌(esophageal squamous-cell carcinoma,ESCC)及好发于食管下段1/3区域及贲门部的食管腺癌(esophageal adenocarcinoma,EA)[1]。食管癌的发生是遗传学及表观遗传学共同作用的结果,表观遗传学研究的是基因组DNA序列未改变的情况下,基因发生可遗传的改变并导致个体表型变化的学说。DNA甲基化是表观遗传学重要组成部分,不同肿瘤中DNA甲基化模式有差异[2]。对食管癌甲基化模式的探索,可以用于早期诊断、化疗疗效预测、预后判断、复发及转移的评估等[3-5]。
他扎罗汀(tazarotene)可以诱导机体产生他扎罗汀诱导基因(tazarotene-induced gene,TIG)1、2 和3[6]。TIG1 基因又名视黄酸受体应答因子-1(retinoic acid receptor responder-1,RARRES1),集中在人类的3号染色体的短臂,位于3p12和3p13之间,其cDNA可以编码含228个氨基酸的蛋白质,该蛋白N端的胞内区和跨膜区,以及C端胞外区含有糖基化信号[7]。在其它癌组织如胃癌、膀胱癌、前列腺癌[8-10]等中TIG1基因均发生高度甲基化,且甲基化水平明显高于正常组织和良性增生,基因TIG1失表达与其启动子异常甲基化密切相关。
本实验采用甲基化特异性聚合酶链反应(methylationspecific PCR,MSP)法及实时定量RT-PCR(real-time fluorescence quantitative PCR,FQ-PCR)检测食管鳞癌组织、癌旁组织及正常组织中TIG1基因的甲基化状态及其mRNA表达水平,比较不同来源的食管组织中TIG1基因的甲基化水平,分析基因甲基化水平与mRNA表达之间的关系,以期进一步探讨TIG1基因在食管癌发生、发展中的作用。
材料和方法
1 材料
1.1 研究对象 收集湖北医药学院附属太和医院2010年2月~2011年12月间手术切除的食管癌组织43例、癌旁组织(距肿瘤边缘2.5 cm以上)20例及15例收集自胃镜室的食管正常组织,结果均经病理证实,标本在获得后立即放置液氮中保存,研究中所有患者均知情同意。所有肿瘤患者术前未行放化疗治疗,患者年龄38~75岁,平均59.2岁,详细资料见表1。
1.2 主要试剂和仪器 TRIzol reagent(Invitrogen);Tis-饱和酚(北京索莱宝);5-Aza-CdR和亚硫酸氢钠(Sigma);SYBR GreenⅠ染料(Gene);Wizard DNA Clean-Up Systerm纯化试剂盒和Reverse Transcription System A3500(Promega);PCR扩增试剂盒(上海生工);DNA marker(北京天根);蛋白酶K、糖原和鲑精DNA(华美);ND-2000微量核酸定量仪(NanoDrop);台式高速冷冻离心机(日立);ABI PRISM7500(ABI);G-BOX紫外凝胶成像系统(Gene)。
1.3 引物 均由北京赛百盛生物公司合成。TIG1基因甲基化引物[11]:甲基化 M(118 bp)上游引物 5'-GTAGTACGGGCGGGT CGC-3',下游引物 5'-GACATCGCCTCCGCAACG-3';非甲基化 U(134 bp)上游引物 5'-GAGGTAGTATGGGTGGGTTGT-3',下游引物 5'-AATACTAAAATACAACATCACCTCCA-3'。TIG1 mRNA[11](134 bp)上游引物 5'-GAAAAACCCCTTGGAAATAGTCAGC-3',下游引物 5'-AGTGTGACACCTGTGTTGTCATTTCC-3';GAPDH mRNA[12](226 bp)上游引物 5'-GAAGGTGAAGGTCGGAGTC-3',下游引物5'-GAAGATGGTGATGGGATTTC-3'。

表1 食管鳞癌组织中TIG1基因甲基化状态及其相应的临床病理特征Table 1.The methylation status of TIG1 gene in esophageal squamous-cell carcinoma tissues and its correlation with clinicopathological features
2 研究方法及步骤
2.1 DNA甲基化检测
2.1.1 DNA的磺化修饰 以酚/氯仿法提取各食管组织的DNA,并对其行亚硫酸氢盐修饰。修饰步骤参照[13]:检测所提取的DNA浓度,计算4 μg DNA相应体积,用ddH2O补齐至总体积50 μL,100℃加热10 min,立即放入冰块内5 min,加入3.0 mol/L 的 NaOH 5.5 μL,置入 37 ℃水箱中孵化 30 min。向处理后的DNA中加入10 mmol/L的临时配置的氢醌30 μL 和3.0 mol/L 的亚硫酸氢钠 520 μL,轻轻混匀,加入200 μL无菌矿物油覆盖液面,放入55℃的水浴箱内16 h。取出标本,离心后吸出无菌矿物油,将修饰后的DNA用Wizard DNA Clean-Up Systerm纯化体系混匀、过柱,经脱盐、纯化回收,洗脱进50 μL的双蒸水中。向标本中加入3 mol/L NaOH 5.5 μL,于37℃的水浴箱内15 min脱硫化基团,加入10 mol/L NH4Ac 56 μL,静置 5 min 后,加入 5 g/L 糖原、1 μL鲑精DNA、2.5倍体积预冷的无水乙醇沉淀DNA,将标本置入-20℃的冰箱内过夜沉淀。将标本离心,用75%预冷的乙醇洗涤2次,弃上清,得DNA沉淀,经干燥后加入30 μL的双蒸水内溶解,于-80℃的冰箱内冻存备用。
2.1.2 MSP检测TIG1基因的甲基化状态 30 μL的扩增体系:DNA 3 μL,2 mmol/L dNTP 1.5 μL,10 × PCR 缓冲液 3 μL,25 mmol/L MgCl22.4 μL,上、下游引物各1.7 μL,5 U/μL Taq DNA 聚合酶0.3 μL,20 × SYBR Green I 0.5 μL,ddH2O 补齐至30 μL。置于ABI PRISM7500扩增,条件为:95℃ 5 min;然后95℃ 30 s,M引物56℃ 45 s(U引物61℃),72℃ 1 min,40个循环;72 ℃ 5 min。取 10 μL PCR 扩增产物,用2.5%的琼脂糖凝胶进行电泳,用凝胶分析仪拍照分析。
2.2 FQ-PCR法检测TIG1mRNA的表达
2.2.1 RNA提取及cDNA的合成 按照TRIzol reagent说明书提取总RNA,以1 μg(约2 μL)总RNA作为模板,依照逆转录试剂盒A3500说明书配制20 μL的反应体系后放入PCR仪中,反应条件:42℃ 15 min,95℃ 5 min,而后立即放置冰块上备用。
2.2.2 SYBR Green I FQ-PCR 将新合成的 cDNA用无RNA酶的ddH2O稀释至100 μL,行 PCR 扩增,30 μL的扩增体系:2 mmol/L dNTP 2 μL,10 × PCR 缓冲液3 μL,25 mmol/L MgCl22 μL,上、下游引物各 2 μL,5 U/μL Taq 酶 0.5 μL,20× SYBR Green I 0.5μL,稀释后 cDNA 5 μL,灭菌 ddH2O 补齐至30 μL。将反应体系置于ABI PRISM7500内扩增:95℃变性5 min;95 ℃ 30 s,56 ℃ 45 s,72 ℃ 1 min,40个循环,最后一循环72℃时读荧光值;72℃ 5 min,取扩增产物行2.5%琼脂糖凝胶电泳分析,凝胶成像系统拍照分析。
2.3 TIG1 mRNA的相对定量 以GAPDH为内参照,采用实验测定Ct值及扩增效率E,计算TIG1 mRNA的相对定量值:TIG1 mRNA/GAPDH mRNA=(1+EGAPDH)CtGAPDH/[(1+ETIG1)CtTIG1][14]。
3 统计学处理
应用SPSS 13.0统计学软件,基因甲基化数据处理采用χ2检验,当n<40或理论频数<1时采用Fisher's exact test;TIG1 mRNA相对定量结果以中位数(下四分位数,上四分位数)表示,组间差异比较采用 Kruskal-Wallis检验,以 P<0.05为差异有统计学意义。
结 果
1 各食管组织中TIG1基因甲基化状态
43例食管鳞癌组织25.6%(11/43)甲基化阳性,显著高于癌旁组织阳性率5%(1/20)(P<0.05);正常组织中未检测到基因甲基化(0/15),与鳞癌组织比较差异显著(P<0.05)。
2 食管鳞癌组织中TIG1基因甲基化状态与患者临床病理特征关系
如表1所示,性别、年龄、病变部位和分化程度不同的患者TIG1基因甲基化水平差异无统计学意义(P>0.05);T1~2分期组织甲基化阳性率较T3期低,差异有统计学意义(P<0.01),提示该基因甲基化程度与患者TNM分期有关,晚期组织样本甲基化程度高;淋巴结转移阳性组较阴性组阳性甲基化率高,差异有统计学意义(P<0.05),表明组织甲基化与是否伴淋巴结转移有关。食管鳞癌组织M及U扩增产物电泳结果见图1。

Figure 1.Electrophoretogram of TIG1 promoter methylation status in esophageal squamous-cell carcinoma tissues.DW:double-distilled water;M:methylated primer product;U:unmethylated primer product;A:completely methylated sample;B:partially methylated sample;C:unmethylated sample.图1 食管鳞癌组织中TIG1基因甲基化状态电泳图
3 食管鳞癌组织、癌旁组织及正常组织中TIG1 mRNA缺失及相对定量比较
如表2示,鳞癌组织中TIG1 mRNA表达相对定量值显著低于癌旁组织(P<0.05)及正常组织(P<0.01);癌旁组织与正常组织比较差异无统计学意义(P>0.05),表明2组基因表达水平相当;甲基化组显著低于非甲基化组(P<0.01),表明甲基化是导致基因表达缺失重要原因。
讨 论
食管癌是起源于食管黏膜的恶性肿瘤。它以基因及表观遗传学变化为起始,继而多种癌基因激活、抑癌基因失活[15],最终导致功能蛋白的下调和缺失。DNA甲基化是一种重要的表观遗传学机制,主要包括启动子区高甲基化、全部基因组低甲基化以及DNA甲基转移酶异常表达[16],可以参与多种重要的生命过程。包括食管癌在内的多种肿瘤均有不同程度、不同形式的DNA甲基化异常[17]。表观遗传学变化与组织病理学变异水平具有相关性[18]。启动子区CpG岛异常甲基化是可以抑制mRNA的转录,从而导致基因表达下调甚至沉默,进而引发下游事件,导致正常细胞的生长分化调控失常,这一机制与多种肿瘤的形成密切相关[19-20]。
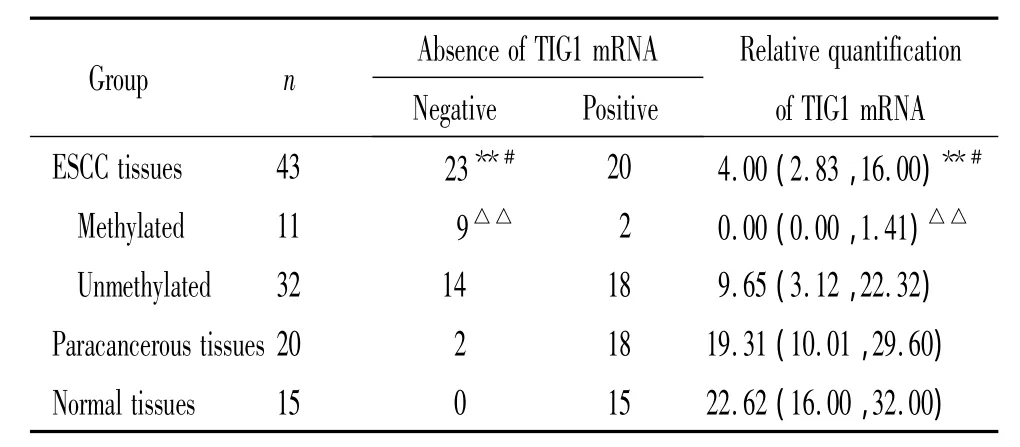
表2 食管鳞癌组织、癌旁组织及正常组织中TIG1 mRNA表达的变化Table 2.The expression level of TIG1 mRNA in esophageal tissues,paracancerous tissues and normol tissues
TIG1蛋白有TIG1A及TIG1B两种异构体,TIG1B可以抑制肿瘤细胞的生长与入侵能力,肿瘤组织中TIG1B的表达水平大多降低,在正常前列腺和结肠组织TIG1两个异构体均有表达,而结肠癌细胞株中表达降低。结肠癌细胞株HCT116经转染表达TIG1异构体使TIG1A和TIG1B均有稳定表达后,其生长显著受抑制,表明TIG1基因及其异构体TIG1A及TIG1B可能为抑癌基因[21]。Kwol等[22]证实鼻咽癌细胞株经转染质粒表达TIG1基因后,较TIG1表达阴性细胞株生长显著抑制(P<0.05),而表达该基因的细胞株敲除TIG1基因后较未敲除的细胞增殖显著增加(P<0.01),提示TIG1基因表达降低可能与鼻咽癌的发生有关。Son等[9]报道晚期胃癌TIG1表达缺失或减少较早、中期肿瘤显著;低分化组织缺失或减少较高、中分化显著。TIG1基因表达缺失的细胞株经去甲基化制剂5-Aza-CdR处理后,TIG1可以恢复表达或原有的表达水平提高,表明TIG1经历频繁的异常甲基化导致表观遗传学失活,它的表达变化与胃癌的进展有关。
本研究采用MSP检测各食管组织TIG1基因甲基化状态,鳞癌组织甲基化率为25.6%(11/43),癌旁组织为5%(1/20),正常组织中未检测到基因甲基化,表明随着组织恶性程度的增加,甲基化率增高。我们还发现鳞癌组织中TIG1基因甲基化多发生在淋巴结转移(P<0.05)和TNM分期晚期(P<0.01)的患者。该结果与Mizuiri等[23]的报道有出入,可能是不同人种患者流行病学及肿瘤发病机制有差异导致,根本原因有待进一步研究证实。不同甲基化水平的基因可以作为预测肿瘤的分期及转移的分子标记物[24],我们推测TIG1基因甲基化不仅导致了TIG1基因的表达异常,而且与食管鳞癌的转移相关,TIG1基因启动子甲基化多发生在晚期肿瘤患者,提示检测组织TIG1基因甲基化状态可以用于患者肿瘤分期及转移的预测因子。
基因表达定量结果显示鳞癌组织基因表达缺失率为53.5%(23/43),11例甲基化的标本中TIG1 mRNA表达缺失率为81.8%(9/11),mRNA表达相对定量显示甲基化组表达量显著低于非甲基化组,表明TIG1基因表达缺失与其异常甲基化有关,异常甲基化导致TIG1表达失活。同时,我们还注意到鳞癌组织中非甲基化组织TIG1基因亦有不同程度的基因表达缺失,可能有可能原因为肿瘤组织中TIG1基因组DNA已发生甲基化,但尚未检测到;另外,基因表达缺失是遗传学及表观遗传学共同作用的结果,考虑DNA甲基化之外的原因引起TIG1基因表达缺失和(或)降低,有待进一步研究明确。本文证实DNA甲基化是导致抑癌基因TIG1表达降低及失活的重要原因,相信随着研究的不断深入,TIG1基因的重要作用以及其表达缺失在食管鳞癌发生、发展中的作用将进一步为人们所认识。
[1] Jemal A,Bray F,Center MM,et al.Global cancer statistics[J].CA Cancer J Clin,2011,61(2):69-90.
[2] Li LW,Yu XY,Li XY,et al.Mechanism of loss of human esophageal cancer-related gene 4(ECRG4)gene expression in esophageal squamous cell carcinoma cell line EC9706[J].Zhonghua Zhong Liu Za Zhi,2011,33(8):570-573.
[3] Gyobu K,Yamashita S,Matsuda Y,et al.Identification and validation of DNA methylation markers to predict lymph node metastasis of esophageal squamous cell carcinomas[J].Ann Surg Oncol,2011,18(4):1185-1194.
[4] Ling ZQ,Zhao Q,Zhou SL,et al.MSH2 promoter hypermethylation in circulating tumor DNA is a valuable predictor of disease-free survival for patients with esophageal squamous cell carcinoma[J].Eur J Surg Oncol,2012,38(4):326-332.
[5] Liu J B,Qiang FL,Dong J,et al.Plasma DNA methylation of Wnt antagonists predicts recurrence of esophageal squamous cell carcinoma[J].World J Gastroenterol,2011,17(44):4917-4921.
[6] Zheng Y,Luo S,Wang G,et al.Downregulation of tazarotene induced gene-2(TIG2)in skin squamous cell carcinoma[J].Eur J Dermatol,2008,18(6):638-641.
[7] Nagpal S,Patel S,Asano AT,et al.Tazarotene-induced gene 1(TIG1),a novel retinoic acid receptor-responsive gene in skin[J].J Invest Dermatol,1996,106(2):269-274.
[8] Vasiljevic'N,Wu K,Brentnall AR,et al.Absolute quantitation of DNA methylation of 28 candidate genes in prostate cancer using pyrosequencing[J].Dis Markers,2011,30(4):151-161.
[9] Son MS,Kang MJ,Park HC,et al.Expression and mutation analysis of TIG1(tazarotene-induced gene 1)in human gastric cancer[J].Oncol Res,2009,17(11-12):571-580.
[10] Esteller M,Guo M,Moreno V,et al.Hypermethylationassociated inactivation of the cellular retinol-bindingprotein 1 gene in human cancer[J].Cancer Res,2002,62(20):5902-5905.
[11] Youssef EM,Chen XQ,Higuchi E,et al.Hypermethylation and silencing of the putative tumor suppressor Tazarotene-induced gene 1 in human cancers[J].Cancer Res,2004,64(7):2411-2417.
[12] Kanai Y,Ushijima S,Kondo Y,et al.DNA methyltransferase expression and DNA methylation of CPG islands and peri-centromeric satellite regions in human colorectal and stomach cancers[J].Int J Cancer,2001,91(2):205-212.
[13] Zhang J,Yu Z.Lü J,et al.Persistent infection of hepatitis B virus is involved in high rate of p16 methylation in hepatocellular carcinoma[J].Mol Carcinog,2006,45(7):530-536.
[14] 余宗涛,高 琼,张吉才,等.肺癌患者PTEN基因启动子甲基化和转录表达的研究[J].中国老年学杂志,2009,29(17):2163-2166.
[15] Zhang XM,Guo MZ.The value of epigenetic markers in esophageal cancer[J].Front Med China,2010,4(4):378-384.
[16] Chen C,Yin N,Yin B,et al.DNA methylation in thoracic neoplasms[J].Cancer Lett,2011,301(1):7-16.
[17] Lima SC,Hernandez-Vargas H,Simao T,et al.Identification of a DNA methylome signature of esophageal squamous cell carcinoma and potential epigenetic biomarkers[J].Epigenetics,2011,6(10):1217-1227.
[18] Jing Z,Zou CL,Li G,et al.Aberrant methylation of T-cadherin gene in human esophageal carcinoma cells[J].Zhonghua Yi Xue Za Zhi,2011,91(37):2653-2656.
[19] 王小韵,杨越波,李小毛,等.CHFR在子宫内膜癌中的表达和甲基化调控[J].中国病理生理杂志,2011,27(9):1746-1750.
[20] 郭艳丽,郭 炜,邝 钢,等.食管鳞状细胞癌中SFRP基因启动子区甲基化状态的检测[J].中国病理生理杂志,2011,27(2):278-283.
[21] Wu CC,Tsai FM,Shyu RY,et al.G protein-coupled receptor kinase 5 mediates Tazarotene-induced gene 1-induced growth suppression of human colon cancer cells[J].BMC Cancer,2011,11:175.
[22] Kwok WK,Pang JC,Lo KW,et al.Role of the RARRES1 gene in nasopharyngeal carcinoma[J].Cancer Genet Cytogenet,2009,194(1):58-64.
[23] Mizuiri H,Yoshida K,Toge T,et al.DNA methylation of genes linked to retinoid signaling in squamous cell carcinoma of the esophagus:DNA methylation of CRBP1 and TIG1 is associated with tumor stage[J].Cancer Sci,2005,96(9):571-577.
[24] Sung CO,Han SY,Kim SH.Low expression of claudin-4 is associated with poor prognosis in esophageal squamous cell carcinoma[J].Ann Surg Oncol,2011,18(1):273-281.

