低温厌氧氨氧化生物滤池群落结构分析
曾涛涛,李 冬,邱文新,曾辉平,刘 涛,张 杰,
(1.哈尔滨工业大学城市水资源与水环境国家重点实验室,150090哈尔滨;2.北京工业大学水质科学与水环境恢复工程北京市重点实验室,100124北京)
与传统硝化-反硝化工艺相比,厌氧氨氧化(anaerobic ammonia oxidation,ANAMMOX)具有需氧量低、污泥产量低和无需外加碳源等优点[1],是目前最简捷的废水生物脱氮途径.厌氧氨氧化的推动者——厌氧氨氧化菌是一群分支很深的浮霉状菌,属于自养型革兰氏阴性细菌.迄今为止,通过分子生物学检测手段已经在不同生态系统中鉴定了5种不同的“Candidatus”Anammox菌属.
目前,ANAMMOX工艺研究主要是针对污泥消化回流液和垃圾渗滤液等高温高氨氮废水方面[2],反应器也以 UASB 类型为主[3],而对于低温厌氧氨氧化生物滤池的研究较少,相应的微生物研究更是缺乏.本课题组已成功启动了上流式厌氧氨氧化生物滤池,脱氮效果良好,并进行了厌氧氨氧化影响因素实验[4-5].与厌氧氨氧化最适温度30~40℃相比,在较低温度下(16℃左右)的厌氧氨氧化研究通常用低温厌氧氨氧化表示[6].本研究对在低温(15.0~16.5℃)稳定运行的两个上流式厌氧氨氧化生物滤池取样,通过扫描电镜(SEM)、变性梯度凝胶电泳技术(DGGE)和克隆测序等方法对微生物群落结构进行分析,探索不同填料生物滤池微生物多样性之间的关系,为促进厌氧氨氧化菌生长、提高反应器效能提供依据.
1 实验
1.1 反应器装置
试验装置为有机玻璃加工而成、相同大小的两个生物滤柱,内径185mm,高度2.0m,有效容积45 L.采用易于接种挂膜的陶粒和火山岩作为填料,其主要性能参数如表1所示.B1柱内装填表面多微孔的陶粒填料,B2柱内装填轻质多孔、表面粗糙的火山岩填料.两个反应器均通过接种厌氧氨氧化污泥启动成功,在自来水中通过添加硫酸铵与亚硝酸钠配置实验用水,使得m(NH+-N)∶m(NO-42-N)约为1∶1.31,以符合厌氧氨氧化反应基质比例要求,氨氮质量浓度约200mg/L.另外添加1.5%的生活污水A/O(厌氧/好氧)除磷工艺的二级处理出水[7].反应器内pH 保持在7.4~8.2,温度为冬季室内自然温度(15.0~16.5℃).

表1 两种填料的主要性能参数
1.2 生物膜样品电镜(SEM)观察
收集B1与B2厌氧氨氧化生物膜0.5 g,加入2.5%(体积分数)的戊二醛,置于4℃冰箱中固定4h;用0.1mol/L,pH为8.0的磷酸缓冲溶液冲洗3次,每次10min;分别用30%,50%,70%,90%(体积分数)的乙醇进行脱水,每次15min,再用无水乙醇脱水3次,每次15min;然后加入体积比为1∶1的100%乙醇和乙酸异戊酯及纯乙酸异戊酯各一次进行置换,每次15min;样品真空干燥后喷金,通过扫描电镜(HITACHIS-4300)观察生物膜形态.
1.3 变性凝胶梯度电泳
1.3.1 总DNA提取
取200mL生物膜水样,12000×g,4℃离心10min,收集沉淀.沉淀加入10mL,0.1mol/L的磷酸缓冲液(PBS,pH8.0)重悬2次.总DNA提取参考文献[8]进行,之后进行纯化回收.
1.3.2 PCR扩增
对大多数细菌16SrRNA基因V3区,采用通用引物BSF338-GC(5’-CGCCCGCCGCGCGCGGCGGGC GGGGCGGGGCACGGGGGG ACTCCTACGGGAGGCAGCAG)(下划线部分为“GC”夹)和 BSR518(ATTACCGCGGCGCTGG)进行扩增[9].对于厌氧氨氧化16S rRNA基因,采用特异性引物 Amx368-GC(5’-CGCCCGCCGCGCGCGGCGGGC GGGGCGGGGCACGGGGGGCCTTTCGGGCATTGCGAA-3’)(下划线部分为“GC”夹)和 Amx820(AAAACCCCTCTACTTAGTGCCC)[10]进行扩增.两种引物PCR反应体系及扩增条件相同.反应体系组成为:DNA模板1.0μL,10×Buffer2.5μL,dNTPs(2.5mmol·L-1)2.0μL,上游引物和下游引物(20μmol·L-1)各0.5μL,ExTaq酶(5U/μL)0.125μL,补水至终体积为25μL.PCR扩增条件为:94℃,5min;94℃,40s,55℃,40s,72℃,1min,35个循环;72℃,10min.PCR产物采用DNA纯化回收试剂盒(天根,中国)纯化回收.
1.3.3 DGGE及其结果分析
利用DGGE电泳对PCR产物进行分离,仪器为D-Code System(Bio-Rad公司),电泳条件为:凝胶变性梯度30%~60%,聚丙烯酰胺质量分数分别为8%(BSF338-GC/BSR518扩增产物)和6%(amx368-GC/amx820扩增产物),电压120V,电泳缓冲液为1×TAE,电泳温度60℃,电泳时间分别为5h(BSF338-GC/BSR518扩增产物)和8h(amx368-GC/amx820扩增产物).电泳结束后,凝胶进行银染[11],通过凝胶成像仪(BioRad,Gel Doc XR)获取图像.
为了解生物滤池反应器中微生物群落结构,通过软件Quantity One 4.6.0(Bio-Rad,USA)对DGGE图谱进行分析,其中微生物群落多样性用Shannon-Weaver指数 (H)表示[12],相关性分析主要分析不同填料反应器内细菌种群的相似性,用Sorenson配对比较相似性系数(Cs)表示.
1.4 微生物系统发育分析
对于细菌及厌氧氨氧化DGGE凝胶上条带进行切胶溶于100μL1×TE中,4℃,16h.以此为模板,相应不带GC夹的BSF338/BSR518及amx368/amx820为引物,扩增细菌及厌氧氨氧化细菌16SrRNA片段.将纯化回收的PCR产物连接到载体pMD19-T(TaKaRa)上,并转化到感受态细胞Escherichia coli DH5α(天根,中国)中.阳性克隆送交上海生工生物公司(中国)进行测序.
获得的序列通过NCBI网站的BLAST工具搜索相近序列,并进行比对.通过MEGA4.1软件,以bootstrap-NJ法构建系统进化树.本研究所测得的ANAMMOX菌16SrRNA序列已提交至GenBank,登录号为JN244671.
2 结果与讨论
2.1 微生物形态
两个反应器启动成功后稳定运行1个月,B1反应器平均总氮去除负荷为0.5kg/(m3·d),去除率为50%;B2平均总氮去除负荷1.6kg/(m3·d),去除率为60%.两个生物滤柱填料表面都形成红色生物膜,这正是anammox菌独特的颜色特征.其中B1反应器生物膜红色比较浅,分布稀疏;B2反应器生物膜红色比较深,分布稠密.生物膜微观结构通过扫描电子显微镜观察,结果见图1.
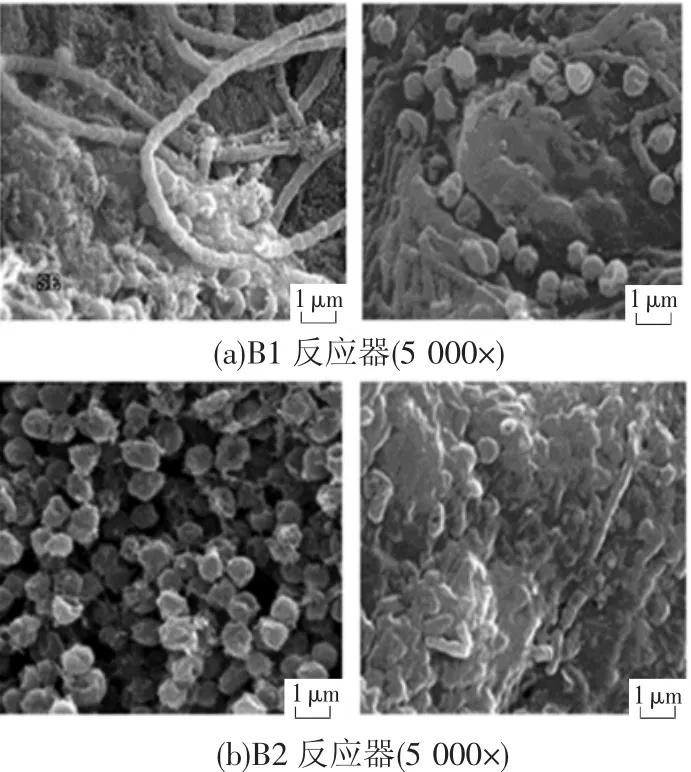
图1 厌氧氨氧化生物膜扫描电镜照片
比较B1和B2扫描电镜结果,发现B1反应器生物膜特点是丝状菌比较多,球形细菌分布密度较低.而B2反应器生物膜特点是以球形细菌为主,存在少量卵状、杆状细菌,没有发现丝状菌的存在.丝状菌一般为异养细菌,比自养菌繁殖速度快[13],本文认为丝状菌的大量繁殖会优先占据反应器内填料表面及其空隙,不利于自养菌anammox菌的生长与附着,因而会影响反应器的脱氮效果.这可能是B1反应器总氮去除负荷(0.5 kg/(m3·d))远低于B2反应器(1.6kg/(m3·d))的原因.已报道的典型厌氧氨氧化细菌形态为球形,直径在0.8~1.1μm[14].两个厌氧氨氧化反应器内都发现这种球形细菌的存在,推测可能是厌氧氨氧化细菌.B2反应器球形细菌分布更为密集,推测原因是火山岩填料比陶粒填料具有更大的空隙率与孔隙率,更有利于这种微生物的富集.
2.2 微生物群落结构
对银染后的DGGE凝胶进行成像,在两条泳道上共观察到12条不同的条带,见图2(a);图2(b)是对DGGE照片经过Quantity One 4.6.0分析后绘制的DGGE图谱示意图,12条横线位置及横线颜色深浅可以简单反映各条带的分布与相对强度.微生物群落多样性指数H1、H2分别为1.258和1.276.图2(c)是ANAMMOX细菌DGGE结果.对图2(a)中12个条带进行切胶、PCR扩增,克隆测序鉴定,结果见表2.

图2 两种厌氧氨氧化生物膜DGGE结果比较
由图2(a)可见,B1和B2中的微生物种类丰度不是很高,这可能与反应器进水采用无机配水有关[15].H2略大于H1,表明B2反应器微生物多样性更高,火山岩填料(B2)比陶粒填料(B1)更有利于微生物富集.B1和B2反应器内细菌种群的相似性为68.1%,表明不同填料ANAMMOX反应器内微生物种类有所差别.DGGE结果中(图2(a)),B1总共有8个条带,B2有10个条带,两个反应器有6个条带在相同位置(6、7、8、9、10 和12),表明这6 种微生物在两个反应器内都存在,分别与Candidatus Kuenenia stuttgartiensis(AF375995)、Ignavibacterium album(AB478415.1)、Hippea maritime(AB072402.1)、Acinetobacter sp.'Acinet.1'(EF409307.1)、Sulfuroxidizing bacterium gps61 (AB266389.1)及Rhodopseudomonas palustris(D12700.1)最相似(表2).两反应器内具有厌氧氨氧化作用的为条带6所代表的Candidatus Kuenenia stuttgartiensis,其他为污水处理系统常见微生物.这表明两个反应器虽然填料不同,但经过较长时间运行后,能富集到同一种厌氧氨氧化细菌.
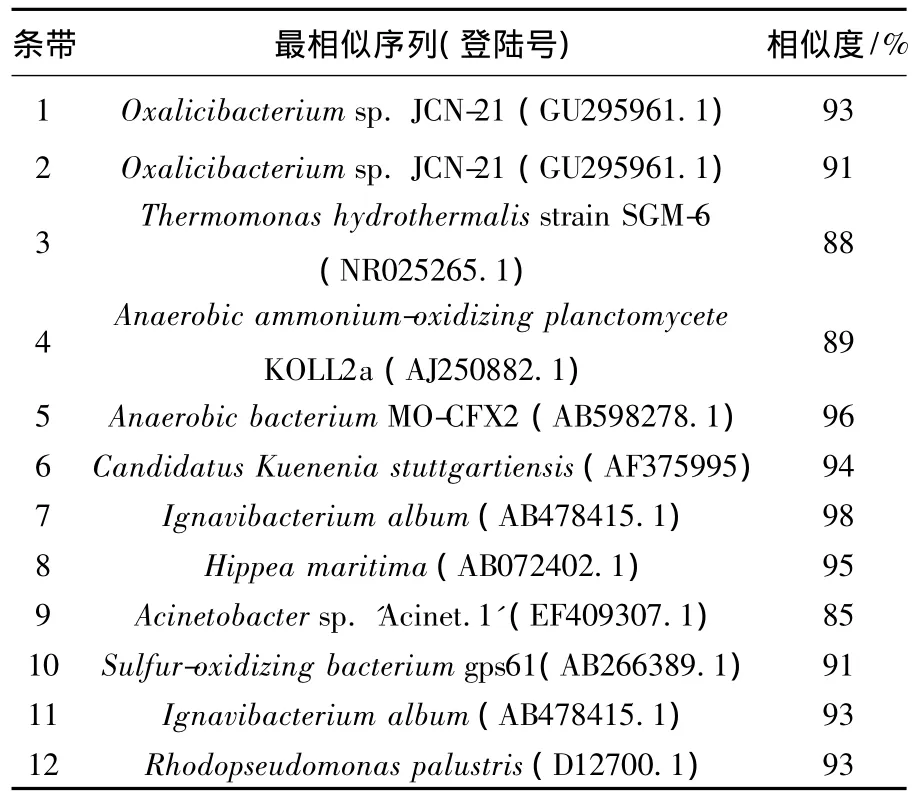
表2 厌氧氨氧化反应器内细菌16S rRNA测序结果
条带5与条带11只出现在B1反应器内,它们与Anaerobic bacterium MO-CFX2(AB598278.1)和Rhodopseudomonas palustris(D12700.1)最相似,相似度分别为96%和93%,表明这两种细菌更容易在陶粒填料生物滤柱(B1)内生长.而条带1、2、3和4仅出现在B2反应器内,它们与Oxalicibacterium sp.JCN-21(GU295961.1)、 Oxalicibacterium sp.JCN-21(GU295961.1)、Thermomonas hydrothermalis strain SGM-6(NR025265.1)和 Anaerobicammonium-oxidizing planctomycete KOLL2a(AJ250882.1)相似度分别为93%、91%、88%及89%,表明这4种细菌更容易在火山岩填料生物滤柱(B2)内生长.两种反应器内存在不同的细菌,这可能与填料性质相关,因为火山岩填料具有更好的孔隙度,因而能富集更多的细菌.
虽然B1和B2生物滤池的填料不同,但在连续运行过程中,二者的操作条件基本相同.图2(c)中两组泳道都只有一个条带且位置相同,表明长时间运行后,两个反应器中存在同一类厌氧氨氧化菌,细菌16S rRNA克隆测序也发现两个反应器内ANAMMOX菌同为Candidatus Kuenenia stuttgartiensis.大多数对于废水处理厌氧氨氧化菌的研究发现,虽然不同反应器内ANAMMOX菌属可能不同,但其种类比较单一,以某一种为主[16],这可能与进水水质及运行工况相关,而Candidatus Kuenenia stuttgartiensis比较容易在生物滤池形式的反应器内出现[14].
2.3 厌氧氨氧化细菌系统发育分析
因为细菌16SrRNAV3区片断较短(约180bp),信息量较少,为了更详尽地了解反应器内ANAMMOX菌种类,对ANAMMOX菌DGGE条带(约480bp)进行切胶回收、PCR重扩增和克隆测序.将该序列与其他相关细菌的16SrRNA序列,通过MEGA4.1软件构建系统发育树,如图3所示.
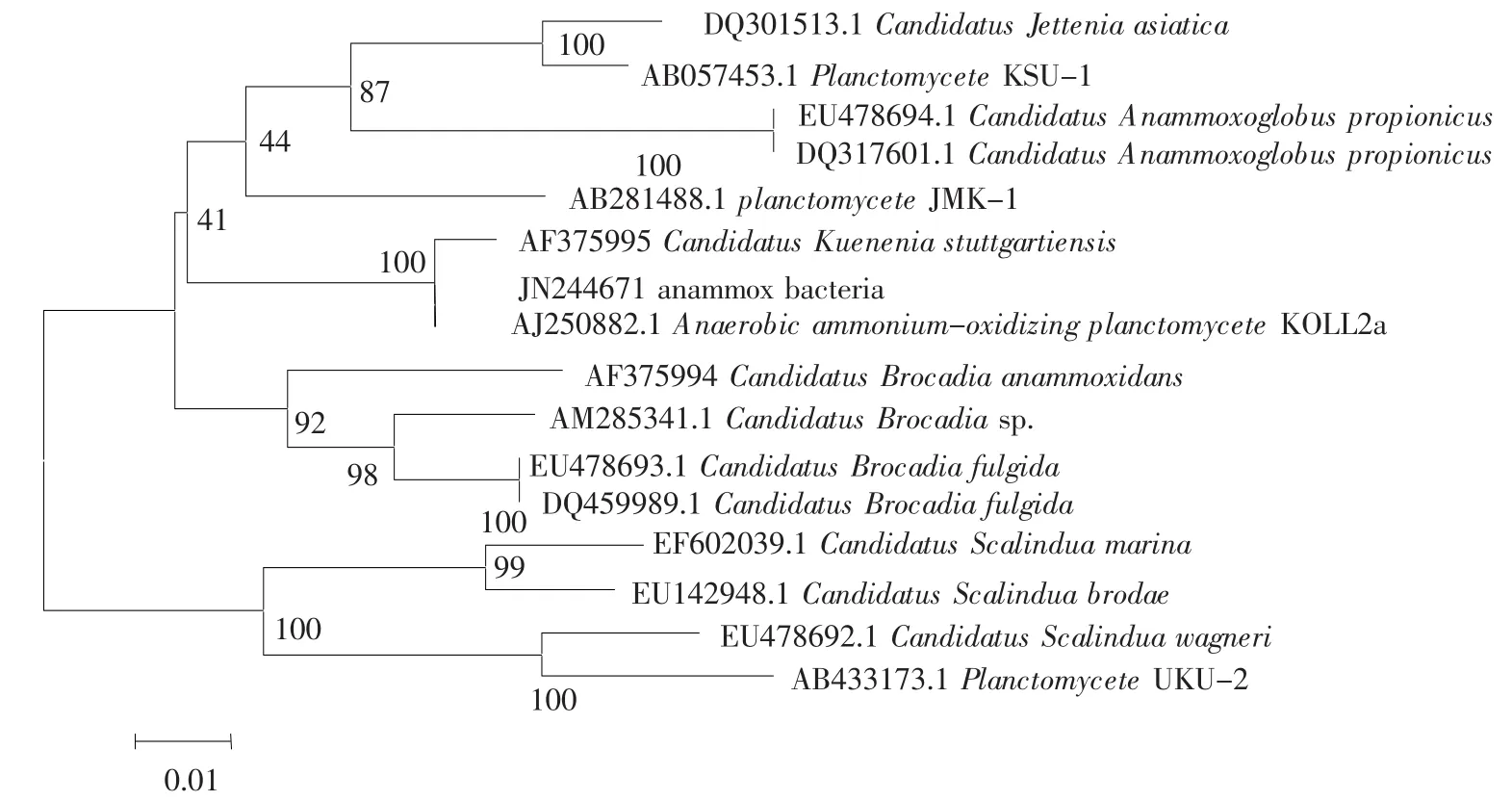
图3 厌氧氨氧化细菌的系统发育树分析
图3代表生物滤池中厌氧氨氧化细菌和已知的5类厌氧氨氧化细菌及相关浮霉状菌的系统发育关系.序列 JN244671与 Candidatus Kuenenia stuttgartiensis(AF375995)同源性最高,相似度达99%.同时,与 Anaerobic ammonium-oxidizing planctomycete KOLL2a(AJ250882.1)在同一个分支上,显示它们在进化关系上最为接近.Candidatus Kuenenia stuttgartiensis最早发现于生物滤池中[14],通过环境基因组方法,已经完成全基因组测序[17],这为研究厌氧氨氧化细菌提供了新的蓝图.Candidatus Kuenenia stuttgartiensis菌体呈球状,直径1 μm左右,化能自养型,具有典型的厌氧氨氧化菌细胞结构特征,本实验中观察到的厌氧氨氧化菌与这些特征十分吻合.
已报道的大多数厌氧氨氧化反应器操作温度保持在30~40℃[2],而一般废水的温度都处于常温(25℃)或更低温度,在实际污水处理中,对废水进行加热和保温成本太高也不实际,这成为厌氧氨氧化工艺应用的一个难题.本研究中,火山岩填料厌氧氨氧化生物滤池(B2)在较低温(15.0~16.5℃)条件下脱氮效果良好;另外,反应器长时间运行中,一直在室内自然光线环境中,并不需要严格黑暗避光,这对 CandidatusKuenenia stuttgartiensis的认识更深了一步.综合研究表明,可以采用火山岩填料填充生物滤池反应器,并富集Candidatus Kuenenia stuttgartiensis,有助于厌氧氨氧化工艺在较低温度(15.0~16.5℃)条件下稳定运行,从而节省废水加热与保温费用.
3 结论
1)SEM结果显示,陶粒填料反应器(B1)内丝状菌比较多,球形细菌分布密度较低,火山岩填料反应器(B2)没有发现丝状菌,而球形细菌分布密集.丝状菌的存在不利于anammox菌的生长,火山岩填料更有利于球形细菌的富集.
2)细菌DGGE结果表明,不同填料反应器内微生物种类有所差别,B1和B2细菌种群的相似性仅为68.1%,B2反应器内细菌丰度更高.虽然B1和B2填料不同,但经过较长时间运行后,能富集到同一种厌氧氨氧化细菌.
3)通过细菌及厌氧氨氧化细菌DGGE条带16S rRNA克隆测序,鉴定反应器内功能微生物为厌氧氨氧化菌Candidatus Kuenenia stuttgartiensis.可以采用火山岩填料生物滤池反应器形式,通过富集Candidatus Kuenenia stuttgartiensis,有助于厌氧氨氧化工艺在较低温度(15.0~16.5℃)下稳定运行,从而节省废水加热与保温费用.
[1]KARTAL B,KUENEN J G,LOOSDRECHT van M C.Engineering.Sewage treatment with anammox[J].Science,2010,328(5979):702-703.
[2]ZHANG L,YANG J C,HIRA D,et al.High-rate nitrogen removal from anaerobic digester liquor using an upflow anammox reactor with polyethylene sponge as a biomass carrier[J].Journal of Bioscience and Bioengineering,2011,111(3):306-311.
[3]杨洋,左剑恶,全哲学,等.UASB反应器中厌氧氨氧化污泥的种群分析[J].中国环境科学,2006(1):52-56.
[4]田智勇,李冬,杨宏,等.上向流厌氧氨氧化生物滤池的启动与脱氮性能[J].北京工业大学学报,2009(4):509-515.
[5]李冬,王俊安,陶晓晓,等.常温条件下厌氧氨氧化生物滤池影响因素[J].哈尔滨工业大学学报,2010(6):869-872.
[6]WINKLER M K H,KLEEREBEZEM R,KUENEN J G,et al.Segregation of biomass in cyclic anaerobic/aerobic granular sludge allows the enrichment of anaerobic ammonium oxidizing bacteria at low temperatures[J].Environmental Science& Technology,2011,45(17):7330-7337.
[7]马金明,李冬,吴迪,等.低温城市生活污水再生全流程中A/O高效除磷研究[J].水处理技术,2012,37(12):103-108.
[8]ZHOU J,BRUNS M A,TIEDJE J M.DNA recovery from soils of diverse composition[J].Appl Environ Microbiol,1996,62(2):316 -322.
[9]OVREAS L,FORNEY L,DAAE F L,et al.Distribution of bacterioplankton in meromictic Lake Saelenvannet,as determined by denaturing gradient gel electrophoresis of PCR-plified gene fragments coding for 16S rRNA[J].Appl Environ Microbiol,1997,63(9):3367 -3373.
[10]SCHMID M,WALSH K,WEBB R,et al.Candidatus"Scalindua brodae",sp.nov.,Candidatus"Scalindua wagneri",sp.nov.,two new species of anaerobic ammonium oxidizing bacteria[J].Syst Appl Microbiol,2003,26(4):529-538.
[11]BASSAM B J,CAETANO-ANOLLES G,GRESSHOFF P M.Fast and sensitive silver staining of DNA in polyacrylamide gels[J].Anal Biochem,1991,196(1):80 -83.
[12]SHANNON C E.The mathematical theory of communication.1963[J].MD Comput,1997,14(4):306 -317.
[13]杨宏,姚乾,黄春雷,等.A/O生物除磷工艺丝状菌膨胀的控制[J].北京工业大学学报,2009,35(12):1663-1669.
[14]郑平,张蕾.厌氧氨氧化菌的特性与分类[J].浙江大学学报:农业与生命科学版,2009,35(5):473-481.
[15]秦玉洁,周少奇,朱明石.厌氧氨氧化反应器微生态的研究[J].环境科学,2008(6):1638-1643.
[16]KARTAL B,RATTRAY J,NIFTRIK van L A,et al.Candidatus"Anammoxoglobus propionicus"a new propionate oxidizing species of anaerobic ammonium oxidizing bacteria[J].Syst Appl Microbiol,2007,30(1):39 -49.
[17]STROUS M,PELLETIER E,MANGENOT S,et al.Deciphering the evolution and metabolism of an anammox bacterium from a community genome[J].Nature,2006,440(7085):790-794.

