双翅目昆虫线粒体基因组研究进展
梅 琰,岳巧云,贾凤龙*
(1.中山大学生命科学学院,广州 510275;2.中山市出入境检验检疫局,广东中山 528403)
双翅目Diptera 是昆虫五大目之一。近年来,由于mtDNA 因母性遗传(徐庆刚等,2001)、拷贝多、易用通用引物扩增、相对核基因小等特点,越来越多地被运用于双翅目昆虫系统发育、群体遗传变异、近缘种的识别(Schroeder et al.,2003)等研究。线粒体基因组研究方法的成熟,特别是长片段PCR 扩增技术的应用,使得双翅目昆虫线粒体基因组的研究飞速发展。截止2011年12月,共有49种双翅目线粒体基因组序列提交到了NCBI的GenBank 中,另外还有12种果蝇Drosophila和尖音库蚊Culex pipiens pipiens 的线粒体DNA 序列除了A+T富含区外其他部分完成了测序提交到了NCBI 的GenBank 中。线粒体基因在双翅目系统学研究中已被广泛应用,尤其在环裂亚目中(Wiegmann et al.,2003,2011;Kutty et al.,2008,2010)。关于应用线粒体基因进行双翅目分子系统学、近缘物种的鉴定、COI 基因等方面的应用国内已经有人做了综述(葛振萍等,2007;池宇等,2010),此文不再涉及。本文目的是将双翅目线粒体基因组的研究进展进行如下介绍.
1 双翅目昆虫mtDNA 基因组的结构特点
双翅目昆虫mtDNA和其他的动物的一样也是一个双螺旋共价闭合环状分子,大小在14~20 kb之间。基因组的大小主要是由A+T富含区(又称控制区)的长短决定的(Boore et al.1999;Oliveira et al.,2007)。在已知双翅目mtDNA 基因组中,瘿蚊科Cecidomyiidae 的Rhopalomyia pomum的长度为14503 bp,是已知双翅目中基因组最小的,该物种的tRNA 基因发生了类似蜘蛛纲Arachnida中Habronattus oregonensis的缺失(Beckenbach et al.,2009)。最大的mtDNA 基因组是黑腹果蝇Drosophila melangaster,为19517 bp,A+T富含区长达4601 bp(Lewis et al.,1994)。
双翅目昆虫mtDNA 基因组相对保守,亚库巴果蝇Drosophila yakuba 为其中的代表,mtDNA 基因组由37个基因组成,其中13个蛋白质编码基因是与氧化磷酸化相关的功能基因,分别是:NADH-Q 还原酶7个亚基(ND1-6 及ND4L),细胞色素氧化酶3个亚基(COI-III),ATP 合成酶的2个亚基(ATP6和ATP8),细胞色素还原酶的1个亚基(Cytb)。22个氨基酸转运RNA 编码基因,其中有18个tRNA 基因分别对应18种不同的氨基酸,但有两种比较特殊,丝氨酸和亮氨酸tRNA 各有两种,分别是tRNA-leu(CUN)、tRNA-leu(UUR)、tRNA-ser(AGN)、tRNA-ser(UCN)。另2个为线粒体核糖体RNA 基因(12S rRNA和16S rRNA),此外还有一个影响mtDNA 基因组大小的A+T富含区,也是线粒体基因组中最大的非编码区,长度变化很大。据NCBI 中GenBank 的数据,双翅目中家蚕追寄蝇Exorista sorbillans 的A+T富含区长最短,仅104 bp,黑腹果蝇最长,为4601 bp。除了这个最大的非编码区外,线粒体基因组中在两个相邻的基因之间还存在着短的非编码区,又称基因间隔区。如在螺旋蝇Cochliomyia hominivorax 线粒体基因组中有14个基因间隔区,长度在1~18 bp 不等,总长120 bp(Lessinger et al.,2000)。
双翅目昆虫线粒体基因组的两条链都参与基因的编码,以Drosophila yakuba 为典型的代表,如图1 所示。其中J(majority)链中包含了9种蛋白质编码基因和14种tRNA 基因,9种蛋白质编码基因分 别 是COI-III,ND2,ND3,ND6,ATP6,ATP8,CYTB。14种tRNA 基因分别为tRNA-Ile,tRNA-Met,tRNA-Trp,tRNA-Leu,tRNA-Lys,tRNA-Asp,tRNA-Gly,tRNA-Ala,tRNA-Arg,tRNA-Asn,tRNA-Ser,tRNA-Glu,tRNA-Thr,tRNA-Ser。N 链编码剩余的4种蛋白质(ND1,ND4,ND4L,ND5)和2种线粒体核糖体RNA(12S rRNA,16S rRNA),以及8种tRNA 基因(tRNA-Gln,tRNA-Cys,tRNA-Tyr,tRNA-Phe,tRNA-Pro,tRNA-Leu,tRNA-His,tRNAVal)。但也有些种类存在着重排现象,其中比较普遍的现象是tRNA 基因的增加或者发生倒位,转移到另一条链上。据GenBank 中的数据,狂蝇科Oestridae 的Dermatobia hominis 线粒体基因组中在tRNA-lys(K)和tRNA-asp(D)之间有一个额外的 tRNA-val(V)。实蝇科 Tephritidae 的Bactrocera carambolae,B.philippinensis和B.papayae的线粒体基因组中位于Cytb和ND1 之间的tRNA-ser(S)由经典位置J 链上转移到了N 链上。丽蝇科Calliphoridae 金蝇属Chrysomya 的C.putoria、C.megacephala 的线粒体基因组中在A+T富含区靠近12S rRNA 基因的位置有一个额外的tRNA-ile 基因,这种现象被认为是金蝇属蝇类的的共性(Junqueira et al.,2004;Stevens et al.,2008)。低等的双翅目如毫蚊科Trichoceridae 的Paracladura trichoptera 线粒体基因组的排序与典型的序列相比发生了巨大的变化,不仅涉及到tRNA 基因的位置,蛋白质编码基因的位置也发生了很大的变化。线粒体基因组包含的37个基因在理论上存在很大的重排的潜力,但双翅目中特别是进化地位较高的种重排现象并不频繁,比较常见的现象是涉及个别tRNA 基因的重排。不同种共享重排序列可能预示着它们系统发育关系比较近,而低等的双翅目昆虫基因序列发生较大的改变可能预示着在进化的早期产生了独立的进化分支。
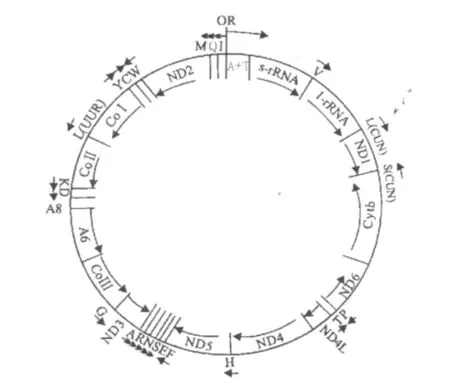
图1 典型的双翅目昆虫线粒体基因组排列方式Fig.1 Typical arrangement in diptera
与其他动物一样,双翅目昆虫线粒体基因组与核基因相比较,A+T含量很高。从整个线粒体基因组上来看,即使含量最低的Bactrocera oleae,A+T含量也达到了72.7%(Nardi et al.,2003),而含量最高的瘿蚊Rhopalomyia pomum 高达85.2%(Beckenbach et al.,2009)。在37个基因中,13个蛋白质编码基因总体上A+T含量略低于整个基因组。如埃及伊蚊Aedes aegypti 的线粒体蛋白质编码基因的A+T含量为77.1%,略低于78.9%的平均值(Behura et al.,2011)。2 条链上蛋白质编码区的碱基组成存在不对称现象,如在Bactrocera oleae 基因组中,J 链上A和T 碱基的百分比非常接近(33.1±2.5%和35.9±1.7%),但是在N链上T 碱基的百分比比A 大得多(48.6±1.5%和25.8±1.7%)。这种链间碱基含量的差异可能对其中编码基因的核苷酸替换产生一定的选择压力(Nardi et al.,2003)。在密码子方面,第三位密码子A+T的含量非常高,而且比第一、二位密码子高很多,如Liriomyza trifolii 第三位密码子A+T的含量高达89.4%,而第一、二密码子位上的A+T的含量分别是69.5%和67.4%(Wang et al.,2011),可能的原因之一是绝大多数的密码子本身就以A/T 结尾,如在黑腹果蝇的七个蛋白质编码基因中包含2097个密码子,其中有1987个(约94.3%)是以A/T 结尾(Garesse,1988),此外还有可能是第三位密码子上相对较低的选择和突变压力(Jermiin et al.,1994)。
2 mtDNA 基因组中蛋白质编码基因、tRNA、rRNA和非编码区的一般属性
2.1 蛋白质编码基因
双翅目昆虫mtDNA 中的蛋白质编码基因和其他动物的线粒体一样是13个。已知62种双翅目昆虫的mtDNA 中最大的基因是ND5,长度在1665~1746 bp 之间,瘿蚊R.pomum 的ND5 最短,致倦库蚊Culex quinquefasciatus 的最长(Behura et al.,2011)。最小的基因是ATP8,在153~180 bp 之间。13个蛋白质编码基因中变化最小的是COI,已知62种中有61种该基因在1531~1540 bp 之间,而该基因最长存在于食蚜蝇Simosyrphus grandicornis,为1558 bp(Cameron et al.,2007),Rhopalomyia pomum 中唯一没有缩短的蛋白质的基因就是COI(Beckenbach et al.,2009)。
线粒体编码的13个蛋白质基因都是参与氧化磷酸化作用的功能基因。这些功能基因并非绝对的保守,而是有非常多的突变位点。由于氨基酸密码子的简并性,许多在第三位和一些第一位置的碱基受到限制比较小,这些位置发生的转换常常是沉默的,它们的改变并不能影响所编码的氨基酸的种类。在6种按蚊的蛋白质编码基因(PCGs)中共有795个核苷酸突变位点,其中94%是沉默性的,不能改变所编码的氨基酸的种类,有674个突变发生在第三位的密码子上(Krzywinski et al.,2010)。在蛋白质编码基因中,单个核苷酸的改变是由转换或颠换引起的,亲缘关系近的种间的替换形式主要是转换,如Drosophila simulans和D.mauritiana,而亲缘关系较远的种间替换的形式以颠换居多,如黑腹果蝇和亚库巴果蝇之间,这可能是由于转换引起的核苷酸水平上的改变是同义性的,而颠换引起的改变是非同义性的(Satta et al.,1987)。在这13个蛋白质编码基因中不同的基因变异情况不同,总体上来说COI-III 是相对保守的,变化大的是NADH 的几个亚基。对12种果蝇的蛋白质编码基因研究显示它们都受到了强的进化选择的压力,氨基酸变异率分别是NADH(0.059)>ATP(0.042)>Cytb(0.037)>COI-III(0.020)(Clark et al.,2007)。
双翅目昆虫蛋白质编码基因的起始密码子一般是ATN,但COI 基因的起始密码子比较特殊,如 TCG(Sperling et al.,1994;Weigl et al.,2010)、CCG(Andrianov et al.,2010)、ATAA(de Bruijn,1983)、ATCA(Behura et al.,2011)、GTGA(Satta et al.,1987)、GTAA(Satta et al.,1987)、ATTTAA(Mitchell et al,1993;Beard et al,1994),然而精确地定义某物种的COI 起始密码子还有赖于翻译后产生的氨基酸序列与初始的核苷酸序列之间的比较分析(Spanos et al.,2000)。这些特殊的密码子在参与后来的蛋白质翻译的过程中,先转录为mRNA 后经RNA 的编辑转换成正常的起始密码子完成翻译过程(Clary &Wolstenholme,1985)。蛋白质的终止子有常见的有四种类型,分别为正常的TAA、TAG和两个不完整的终止子TA和T。如潜叶蝇Liriomyza sativae 的线粒体DNA 的13个蛋白质编码基因中有10个的终止子是正常的TAA、TAG,另外ND5 的终止子是TA,ND2和Cytb 的终止子是T(Yang et al.,2011)。Bactrocera oleae 的mtDNA 中,有9个蛋白质编码基因的终止子为TAA,ND3和Cytb 则为TAG,而ND1和COI 分别以T/TA 作为终止子(Nardi et al.,2003)。双翅目mtDNA 编码的几个基因如ND3-ND5,COII,Cytb 中,终止密码子常常是不完整的TA 或T(Lessinger and Azeredo-Espin,2000;Oliveira et al.,2008),这些不完整的终止子经过转录后的编辑,对不完整的终止子进行多聚腺苷酸化便能形成完整的终止子TAA(Yokobori et al.,1997)。mtDNA 由于其独特的碱基组成,在密码子的使用上有明显的偏向,如丽蝇Calliphora putoria 的13个线粒体编码的蛋白质基因没有用CTG、CGC、AGG。对NCBI 数据库中的数据分析表明,螺旋蝇Cochliomyia hominivorax和黑腹果蝇中没有使用CTG 等,这些GC 含量高的密码子没有被使用可能是mtDNA 基因组中GC碱基含量低的原因之一(Junqueira et al.,2004)。
2.2 tRNA 基因
双翅目昆虫的mtDNA 编码的tRNA 基因一般都能形成三叶草型的二级结构,但tRNA-ser 的二级结构常缺少DHU 臂而不能形成典型的三叶草型形状,如亚库巴果蝇(Clary et al.,1983)、丽蝇Calliphora putoria(Junqueira et al.,2004)。对已知62个物种的tRNA 统计分析显示,其大小绝大部分在60~72 bp 之间,但也有特殊情况,如瘿蚊科的Rhopalomyia pomum 的tRNA 基因缺少TψC stem-loop 结构和3'端的氨酰基接受臂,22个tRNA 中长度最小的只有41 bp,而缺失的氨酰基接受臂可能通过RNA 编辑,以5'端酰胺基接受臂为模板形成完整的氨基酸臂(Beckenbach et al.,2009)。双翅目昆虫的mtDNA 中tRNA 基因的个数一般是22个,也有特殊的情况,如Liriomyza trifolii,在A+T富含区靠近12S rRNA 的一侧出现了两个额外的tRNA 基因tRNA-thr和tRNA-leu(Wang et al.,2011)。tRNA 基因中最保守的地方是氨酰基接受臂,反密码子环和反密码子臂,其他的区域大小变化较大,如在果蝇Drosophila yakuba 中氨酰基接受臂、反密码子环和反密码子臂的长度都是固定的,分别是7 bp、7 bp、5 bp,而其他部位如二氢尿嘧啶环(3~8 bp)、TψC 环(3~8 bp)多变环(4~5 bp)等(Clary and Wolstenholme,1985)。tRNA 基因中还存在这一些非Watson-Crick(DNA双螺旋)的错配碱基存在,如在潜叶蝇Liriomyza sativae 的22个tRNA 基因中共含有27个错配的碱基,其中G-U21个,U-U4个,A-A1个,A-C 1个,分别是氨酰基接受臂8个,DHU 臂10个,AC 臂5个,DHU 臂4个(Yang et al.,2011)。不同的物种中tRNA 的进化速率是有一定的变化,如在亲缘关系较近的黑腹果蝇组(簇)7个种中,共有3种tRNA 的进化速率存在相当大的差异,这种速率的差异可能与紧邻蛋白编码基因的tRNA 在蛋白处理过程中的断续性规则(punctuational role)有关(Nigro et al.,1991)。
2.3 rRNA 基因
双翅目昆虫mtDNA 编码的rRNA 基因与其他昆虫一致也有2个rRNA。其中一个较大的16S rRNA和一个较小的12S rRNA。90%的16S rRNA大小在1310~1350 bp 之间,但也有特例,瘿蚊Rhopalomyia pomum 16S rRNA 基因只有1264 bp(Beckenbach et al.,2009),而达林按蚊Anopheles darlingi 中,长度达到1394 bp(Moreno et al.,2010)。两种rRNA 基因在线粒体基因组中都是以单拷贝形式存在的。rRNA 基因的进化速率与功能相关,核糖体蛋白结合位点、mRNA 结合位点和轴心螺旋等是其发挥功能的主要作用区域,属于高保守区。rRNA 分子茎中长茎比短茎保守,短茎又比环保守些。在12S rRNA 中,3'端比5'端要保守的多,对6种果蝇的12S rRNA 研究显示,12S亚基3'端的进化速率比5'端慢得多,即使经过了3000~6000万年的进化,其分类单元之间的平均遗传距离依然非常近。这也进一步证明了rRNA 基因的核苷酸序列的变异是由严格的结构和功能限制所导致的,而不是简单的突变率减少(Simon et al.,1994)。
2.4 A+T富含区
双翅目mtDNA 中位于12S rRNA和tRNA-ile之间的区域,A、T 两种碱基的含量异常丰富,如黑腹果蝇的A、T 两种碱基的含量高达96%,称做A+T富含区。A+T富含区的长度在不同的物种变化非常大,甚至在种内不同个体之间也存在着差异。如角蝇Haematobia irritans 中,出现了3种不同的基因型(Oliveira et al.,2006),这主要与重复单元的长度和拷贝数的变化有关(Solignac et al.,1986;Monforte et al.,1993)。A+T富含区是线粒体基因组中进化速率最快的区域,对果蝇属DNA 序列的比较分析表明,大部分物种A+T富含区序列的分歧非常显著(Clary et al.,1987),但A+T富含区内也有许多个保守片段。Lessinger等对引起蝇蛆病的五种双翅目昆虫的A+T富含区进行研究时发现了8个保守片段(CSBs)(Lessinger et al.,2000),而这8个保守性片段也存在丽蝇科中(Lessinger et al.,2004)。Oliveira 等在蝇科的几个种如西方角蝇Haematobia irritans,家蝇Musca domestica,厩蜇蝇Stomoxys calcitrans和东方芒蝇Atherigona orientalis 中发现了这8个片段中的7个,其中前3种缺少CSB3,而在东方芒蝇中缺少CSB7(Oliveira et al.,2007)。Zhang 等总结的多聚T片段相当于CSB1,[TA(A)]n片段相当于CSB6(Zhang et al.,1997)。Oliveira 等对蝇科的4个种、丽蝇科的5个种、果蝇科的7个种、狂蝇科的1种的A+T富含区综合分析时发现了短角亚目中4个保守区(BCRs)和Gisland(Oliveira et al.,2007)。A+T富含区作为mtDNA 中最大的非编码区与mtDNA 的复制与转录有关。Clary 等认为位于MQI 基因附近的多聚T片段有促进朝着tRNA基因簇方面转录的起始的识别作用(Clary et al.,1987)。Saito 等将果蝇线粒体DNA 两条链复制的起始精确定位于两个多聚T片段下游的第一个碱基(Saito et al.,2005)。Goddard 等通过电镜技术将果蝇线粒体DNA 复制的起始定位在A+T富含区内一个300 bp 的保守区(Goddard et al.,1978;1980)。
3 结语
随着技术的不断成熟,利用mtDNA 中所蕴含的分子数据来研究昆虫系统学已成为一大热点,多种分子数据联合分析对单基因的优越性已经被广泛接受,如线粒体不同的基因间,线粒体与和核基因间。在昆虫分子系统学研究中,更重要的是利用哪些基因解决哪些问题,及如何恰当地分析分子数据(陈学新等,2001),这势必会加速昆虫系统学的发展。然而在分子生物学迅猛发展的今天,我们也不能忘记传统意义上系统学的作用。仅仅利用分子数据分析物种间的系统发育关系有时会得出非常荒谬的结论,特别是在还没确定哪些基因对哪些问题起作用的今天。分子数据也可以解决形态学上难以区分的物种,是对传统分类学的一个有益的补充(葛振萍等,2007)。传统意义上的形态学和分子系统学相结合,会得出更科学的结论,势必成为双翅目昆虫系统学未来的发展方向。
References)
Chen XX,2001.Insect Molecular Science.Beijing:China Science Press.1-44.[陈学新,2001.昆虫分子科学.北京:科学出版社.1-44]
Chi Y,Wang SD,Zhang CT,2010.Advance of Diptera based on mitochondrial COI gene.Entomotaxonomia(Suppl.),8:71-77.[池宇,王诗迪,张春田,2010.基于线粒体COI 基因的双翅目昆虫研究进展.昆虫分类学报(增刊),8:71-77]
Ge ZP,Liu JY,Zhang CT,2007.Progress of studies on molecular systematics of flies(Diptera).Journal of Shenyang Normal University(Natural Science Edition),25(2):233-236.[葛振萍,刘家宇,张春田,2007.双翅目蝇类的分子系统学研究进展.沈阳师范大学学报(自然科学版),25(2):233-236]
Xu QG,Hua BZ,2001.Application of mtDNA in phylogenetic analysis of insects.Journal of Northwest A & F University(Natural Science Edition),29(1):79-83.[徐庆刚,花保祯,2001.线粒体DNA 在昆虫系统学研究中的应用.西北农林科技大学学报(自然科学版),29(1):79-83]
Andrianov BV,Goryacheva II,Mugue NS,Sorokina SY,Gorelova TV,Mitrofanov VG,2010.Comparative analysis of the mitochondrial genomes in Drosophila virilis species group(Diptera:Drosophilidae).Trends.Evol.Biol.,2(e4):22-31.
Beard CB,Mills Hamm D,Collins FH,1993.The mitochondrial genome of the mosquito Anopheles gambiae:DNA sequence,genome organization,and comparisons with mitochondrial sequences of other insects.Insect Mol.Biol.,2:103-124.
Beckenbach AT,Joy JB,2009.Evolution of the mitochondrial genomes of Gall Midges(Diptera:Cecidomyiidae):rearrangement and severe truncation of tRNA genes.Genome Biology Evolution,1:278-287.
Behura SK,Lobo NF,Haas B,deBruyn B,Lovin DD,Shumway MF,Puiu D,Romero-Severson J,Nene V,Severson DW,2011.Complete sequences of mitochondria genomes of Aedes aegypti and Culex quinquefasciatus and comparative analysis of mitochondrial DNA fragments inserted in the nuclear genomes.Insect Biochem.Mol.Biol.,41(10):770-777.
Boore JL,1999.Animal mitochondrial genomes.Nucleic Acids Res.,27(8):1767-1780.
Cameron SL,Lambkin CL,Barker SC,Whiting MF,2007.A mitochondrial genome phylogeny of Diptera:whole genome sequence data accurately resolve relationships over broad timescales with high precision.Syst.Entomol.,32(1):40-59.
Clark AG,Eisen MB,Smith DR,et al.,2007.Evolution of genes and genomes on the Drosophila phylogeny.Nature,450,203-218.
Clary DO,Wahleithner JA,Wolstenholme DR,1983.Transfer RNA genes in Drosophila mitochondrial DNA: related 5'flanking sequences and comparisons to mammalian mitochondrial tRNA genes.Nucleic Acids Res.,11(8):2411-2425.
Clary DO,Wolstenholme DR,1985.The mitochondrial DNA molecular of Drosophila yakuba:nucleotide sequence,gene organization,and genetic code.J.Mol.Evol.,22(3):252-271.
Clary DO,Wolstenholme DR,1987.Drosophila mitochondrial DNA:conserved sequences in the A+T-rich region and supporting evidence for a secondary structure model of the small ribosomal RNA.J.Mol.Evol.,25:116-125.
de Bruijn MH,1983.Drosophila melanogaster mitochondrial DNA,a novel organization and genetic code.Nature,304,234-241.
Garesse R,1988.Drosophila melanogaster mitochondrial DNA:gene organization and evolutionary considerations.Genetics,118(4):649-663.
Goddard JM,Wolstenholme DR,1978.Origin and direction of replication in mitochondrial DNA molecules from Drosophila melanogaster.Proc.Natl.Acad.Sci.,75:3886-3890.
Goddard JM,Wolstenholme DR,1980.Origin and direction of replication in the mitochondrial DNA molecules from the genus Drosophila.Nucleic Acids Res.,8:741-757.
Jermiin LS,Graur D,Lowe RM,Crozier RH,1994.Analysis of directional mutation pressure and nucleotide content in mitochondrial cytochrome b genes.J.Mol.Evol.,39:160-173.
Junqueira AC,Lessinger AC,Torres TT,Silva FR da,Vettore AL,Arruda P,Espin AMLA,2004.The mitochondrial genome of the blowfly Chrysomya chloropyga(Diptera:Calliphoridae).Gene,339:7-15.
Krzywinski J,Li C,Morris M,Conn JE,Lima JB,Povoa MM,Wilkerson RC,2010.Analysis of the evolutionary forces shaping mitochondrial genomes of a Neotropical malaria vector complex.Mol.Phylogenet.Evol.,58(3):469-477.
Kutty SN,Pape T,Pont A,Wiegmann BM,Meier R,2008.The Muscoidea(Diptera:Calyptratae)are paraphyletic:evidencefrom four mitochondrial and four nuclear genes.Mol.Phylogenet.Evol.,49(2),639-652.
Kutty SN,Pape T,Wiegmann BM,Meier R,2010.Molecular phylogeny of the Calyptratae(Diptera:Cyclorrhapha)with an emphasis on the superfamily Oestroidea and the position of Mystacinobiidae and McAlpine's fly.Systematic Entomology,35(4):614-635.
Lessinger AC,Azeredo-Espin AML,2000.Evolution and structural organization of mitochondrial DNA control region of myiasis-causing flies.Med.Vet.Entomol.,14:71-80.
Lessinger AC,Junqueira ACM,Conte FF,Azeredo-Espin AML,2004.Analysis of a conserved duplicated tRNA gene in the mitochondrial genome of blowflies.Gene,339:1-6.
Lessinger AC,Martins-Junqueira AC,Lemos TA,Kemper EL,da Silva FR,Vettore AL,Arruda P,Azeredo-Espin AM,2000.The mitochondrial genome of the primary screwworm fly Cochliomyia hominivorax(Diptera:Calliphoridae).Insect Mol.Biol.,9(5):521-529 .
Lewis DL,Farr CL,Farquhar AL,Kaguni LS,1994.Sequence,organization,and evolution of the A+Tregion of Drosophila melanogaster mitochondrial DNA.Mol.Biol.Evol.,11:523-538.
Mitchell SE,Cockburn AF,Seawright JA,1993.The mitochondrial genome of Anopheles quadrimaculatus species A: complete nucleotide sequence and gene organization.Genome,36:1058-1073.
Monforte A,Barrio E,Latorre A,1993.Characterization of the length polymorphism in the A+T-rich region of the Drosophila obscura group species.J.Mol.Evol.,36:214-223.
Moreno M,Marinotti O,Krzywinski J,Tadei WP,James AA,Achee NL,Conn JE,2010.Complete mtDNA genomes of Anopheles darlingi and an approach to anopheline divergence time.Malar.J.,9:127.
Nardi F,Carapelli A,Dallai R,Frati F,2003.The mitochondrial genome of the olive fly Bactrocera oleae:two haplotypes from distant geographical locations.Insect Mol.Biol.,12(6):605-611.
Nigro L,Solignac M,Sharp PM,1991.Mitochondrial DNA sequence divergence in the Melanogaster and oriental species subgroups of Drosophila.J.Mol.Evol.,33:156-162.
Oliveira MT,Azeredo-Espin AML,Lessinger AC,2007.The mitochondrial DNA control region of Muscidae flies:evolution and structural conservation in a Dipteran context.J.Mol.Evol.,64:519-527.
Oliveira MT,Barau JG,Junqueira AC,Feijao PC,Rosa AC,Abreu CF,Azeredo-Espin AM,Lessinger AC,2008.Structure and evolution of the mitochondrial genomes of Haematobia irritans and Stomoxys calcitrans: the Muscidae(Diptera: Calyptratae)perspective.Mol.Phylogenet.Evol.,48(3):850-857.
Oliveira MT,da Rosa AC,Azeredo-Espin AML,Lessinger AC,2006.Improving access to the control region and tRNA geneclusters of dipteran mitochondrial DNA.J.Med.Entomol.,43:636-639.
Saito S,Tamura K,Aotsuka T,2005.Replication origin of mitochondrial DNA in insects.Genetics,171:1695-1705.
Satta Y,Ishiwa H,Chigusa SI,1987.Analysis of nucleotide substitutions of mitochondrial DNAs in Drosophila melanogaster and its sibling species.Mol.Biol.Evol.,4(6):638-650.
Schroeder H,Klotzbach H,Elias S,Augustin C,Pueschel K,2003.Use of PCR-RFLP for differentiation of calliphorid larvae(Diptera,Calliphoridae)on human corpses.Forensic Sci.Int.,132:76-81.
Simon C,Farti F,Beckenbach AC,1994.Evolution,weihgting and phylogenetic utility of mitochondrial gene sequence and compilation of conserved polymerase chain reaction primers.Ann.Entomo.Soc.Am.,87:651-701.
Solignac M,Monnerot M,Mounolou JC,1986.Concerted evolution of sequence repeats in Drosophila mitochondrial DNA.J.Mol.Evol.,24:53-60.
Spanos L,Koutroumbas G,Kotsyfakis M,Louis C,2000.The mitochondrial genome of the mediterranean fruit fly,Ceratitis Capitata.Insect Mol.Biol.,9(2):139-144.
Sperling FA,Anderson GS,Hickey DA,1994.A DNA-based approach to the identiflcation of insect species used for postmortem interval estimation.Journal of Forensic Sciences,39:418-427.
Stevens JR,West H,Wall R,2008.Mitochondrial genomes of the sheep blowfly,Lucilia sericata,and the secondary blowfly,Chrysomya megacephala.Med.Vet.Entomol.,22:89-91.
Wang S,Lei Z,Wang H,Dong B,Ren B,2011.The complete mitochondrial genome of the leafminer Liriomyza trifolii(Diptera:Agromyzidae).Mol.Biol.Rep.,38(2):687-692.
Weigl S,Testini G,Parisi A,Dantas-Torres F,Traversa D,Colwell DD,Otranto D,2010.The mitochondrial genome of the common cattle grub,Hypoderma Lineatum.Med.Vet.Entomol.,24(3):329-335.
Wiegmann BM,Trautwein MD,Winkler IS,et al.,2011.Episodic radiations in the fly tree of life.PNAS,108(14):5690-5695.
Wiegmann BM,Yeates DK,Thorne JL,Kishino H,2003.Time flies,a new molecular time-scale for brachyceran fly evolution without a clock.Syst.Biol.,52(6):745-756.
Yang F,Du YZ,Wang LP,Cao JM,Yu WW,2011.The complete mitochondrial genome of the leafminer Liriomyza sativae(Diptera:Agromyzidae):Great difference in the A+T-rich region compared to Liriomyza trifolii.Gene,485(1):7-15.
Yokobori S,Pääbo S,1997.Polyadenylation creates the discriminator nucleotide of chicken mitochondrial tRNA(Tyr).J.Mol.Biol.,265:95-99.
Zhang DX,Hewitt GM,1997.Insect mitochondrial control region:a review of its structure,evolution and usefulness in evolutionary studies.Biochem.Syst.Ecol.,25:99-120.
- 环境昆虫学报的其它文章
- 半翅目沫蝉总科化石研究进展

