羟基脲抑制盐酸胍对于酵母朊病毒[PSI+]的治愈*
王莉洁,沈曼莉,李 辉,宋有涛,
2.辽宁大学环境学院,沈阳 110036
酵母朊病毒[PSI+],是由酵母细胞的翻译终止因子Sup35p(eRF3)在细胞质中发生淀粉样聚集引起的[1]。研究表明,酵母朊病毒[PSI+]在细胞内的聚集、繁殖和遗传受到分子伴侣Hsp104,Hsp70家族及其辅助因子的调控[2],其中分子伴侣 Hsp104的活性对酵母朊病毒[PSI+]的维持起着重要的作用,当其过表达或活性受到抑制时可以导致[PSI+]的丢失[3]。早期的研究发现,低浓度盐酸胍(3~5 mmol/L GuHCl)能够通过竞争性抑制 Hsp104的活性,在细胞持续分裂的情况下治愈[PSI+]为[psi-
]。后来的研究表明,盐酸胍治愈[PSI+]的机制可能与盐酸胍在细胞分裂时逐步降低[PSI+]遗传繁殖过程中的“种子”数目有关,而与盐酸胍在细胞外促进蛋白变性的功能无关[4-5]。那么细胞分裂在被抑制的情况下,盐酸胍能否治愈酵母朊病毒[PSI+],这个问题引起了研究者的广泛关注。2005年Wu等通过利用荧光漂白后恢复技术(FRAP)监测[PSI+]酵母活细胞中Sup35p-GFP聚集体变化情况时发现,α因子和法尼醇(抑制DNA连接酶和组蛋白转移酶活性)虽然抑制了酵母细胞分裂,但是不影响盐酸胍对[PSI+]的治愈[6]。2007年 Tuite等通过遗传表型实验发现,α因子和法尼醇(或两性霉素B)在抑制细胞分裂的同时也抑制了盐酸胍对酵母朊病毒[PSI+]的治愈[7]。受到实验手段的局限,迄今细胞分裂受抑制与盐酸胍治愈[PSI+]的关系仍未十分明确。
本研究采用羟基脲(阻止核苷酸还原为脱氧核苷酸,选择性地阻碍DNA合成,对RNA及蛋白质合成并无阻断作用)作为细胞分裂的抑制剂,利用半变性琼脂糖凝胶电泳(SDD-AGE)结合蛋白质印迹(Western blot)技术[8],比较羟基脲和盐酸胍共同作用下[PSI+]细胞裂解液中Sup35p的聚集体存在形式,从蛋白水平分析了羟基脲抑制细胞分裂情况下的盐酸胍对酵母细胞内朊病毒[PSI+]聚集体大小的影响及两者相关性。结果表明,当羟基脲抑制酵母细胞分裂时盐酸胍不能将[PSI+]细胞治愈为[psi-],其原因可能与羟基脲使得盐酸胍解聚酵母朊病毒聚集体的能力明显下降有关。这些数据在一定程度上支持了Tuite等提出的盐酸胍治愈需要细胞分裂的假说。
1 材料与方法
1.1 材料
1.1.1 主要试剂 酵母浸粉、蛋白胨、葡萄糖、琼脂粉均购自上海奥博星生物科技有限公司;尿嘧啶、色氨酸、亮氨酸均购自国药集团化学试剂有限公司;腺嘌呤购自美国Solarbio公司;盐酸胍(GuHCl)、羟基脲(Hydroxyurea)购自Sigma-Aldrich公司,ECL试剂购自碧云天生物有限公司,兔抗-GFP抗体及兔抗绵羊IgG二抗均购自金斯特生物科技有限公司。
1.1.2 菌株 野生型酵母(NMC)MATαkar1SUQ5ade2-1his3Δ202leu2Δ1trp1Δ63ura3-52sup35::KanMX/pJ501,[PSI+],美国国立卫生研究院(NIH)Masison D.C.博士惠赠。重组型酵母(NGMC):MATαkar1SUQ5ade2-1his3Δ202leu2Δ1trp1Δ63ura3-52sup35::KanMX/pJ510,[GPSI+],本实验室构建。将[PSI+]或[GPSI+]细胞划线于含有5mmol/L盐酸胍的1/2YPD固体平板上培养7d后,分离红色单菌落得到[psi-]或[Gpsi-
]细胞。
1.1.3 培养基1/2YPD培养基 酵母浸粉0.5%,蛋白胨2%,葡萄糖2%;SD-Ade培养基:酵母氮源0.7%,葡萄糖2%,尿嘧啶20mg/L,色氨酸20mg/L,亮氨酸60mg/L;1/2YPD+5mmol/L GuHCl培养基:同1/2YPD,灭菌后加入0.5%的1mol/L GuHCl储液;1/2YPD+ Hydro 培 养 基:同 1/2YPD,灭菌后加入固体羟基脲1.521g/50mL,使得终浓度为0.4mol/L。固体平板加入2%琼脂粉。
1.2 方法
1.2.1 羟基脲对于酵母朊病毒[PSI+]的致死率和生长曲线分析 从新鲜YPD平板挑取的酵母[PSI+]菌落经12h活化培养至OD600=1.0左右,接种到含有不同药物的培养基中(菌液终浓度为OD600=0.02),30℃,170r/min培养,每隔2h取药物作用后的菌悬液,采用光电比浊法测定其OD值,绘制生长曲线;同时,将菌悬液稀释涂板,30℃,培养3d,待菌落长出后分别统计不同培养时间下羟基脲作用的酵母[PSI+]细胞致死率(致死率%=100%-存活菌落数目×稀释倍数/活菌总数)。
1.2.2 羟基脲和盐酸胍共同作用下酵母朊病毒[PSI+]表型及治愈率统计 将不同培养时间下羟基脲和盐酸胍共同作用与单独盐酸胍作用下的[PSI+]菌落分别进行稀释涂布于1/2YPD,30℃培养5d~7d。待菌落颜色不再变化时,统计总菌落数以及颜色变红的菌落数,计算两种情况下酵母朊病毒[PSI+]菌落治愈率(治愈率=红色菌落总数/总菌落数×100%)。
1.2.3 羟基脲和盐酸胍共同作用下的酵母朊病毒[PSI+]表型分析 挑取不同时期盐酸胍单独作用、盐酸胍和羟基脲共同作用后的固体培养基上生长的红色和白色菌落,分别划线于1/2YPD固体平板上,30℃培养1d,随后分别影印到SD-Ade和含有5 mmol/L盐酸胍的1/2YPD培养基上,30℃培养3d~5d,观察颜色变化及菌落生长状态。
1.2.4 SDD-AGE/Western blot检测 将羟基脲和盐酸胍作用后的酵母朊病毒[PSI+]菌悬液,离心后收集菌体;采用无菌蒸馏水清洗去除残余药物后悬浮菌液,离心后弃上清;随后加1ml裂解液将菌体悬浮,采用珠磨破碎仪振荡破碎细胞,30s4次,随后,离心收集上清蛋白样品。取新鲜蛋白样品加入0.1%SDS,42℃温浴10min,采用1.5%琼脂糖(0.1%SDS)进行半变性琼脂糖凝胶电泳(SDDAGE),恒压100V,1h;随后转移到固相载体PVDF膜上,采用5%脱脂奶粉封闭1h,以兔抗-GFP抗体(1∶1 000)孵育4℃过夜,随后加入到HRP标记的羊抗兔IgG抗体(1∶5 000)孵育1h,采用ECL发光试剂盒对其进行放射自显影检测[9]。
2 结 果
2.1 羟基脲对于酵母朊病毒[PSI+]活细胞的毒性分析 由于羟基脲对于酵母细胞生长存在着较高的毒性,本研究分别对单独盐酸胍、单独羟基脲、羟基脲和盐酸胍共同作用3种条件下的酵母[PSI+]细胞生长情况进行了研究。从图1的生长曲线可以看出,单独盐酸胍作用的酵母[PSI+]细胞能够正常生长,前6h处于延滞期,在6h~24h处于对数期,24h后处于稳定期。而单独羟基脲、羟基脲和盐酸胍共同作用的酵母[PSI+]细胞生长受到抑制,延滞期后细胞分裂停止。根据稀释后涂板的菌落生长数目,单独羟基脲、羟基脲和盐酸胍共同作用6h以内的酵母[PSI+]细胞致死率为50%左右,与对照和单独盐酸胍作用的区别不大;6h后,对照和单独盐酸胍作用的酵母[PSI+]细胞致死率逐渐下降到20%左右,而羟基脲和盐酸胍共同作用的细胞致死率>70%,24h后>90%。
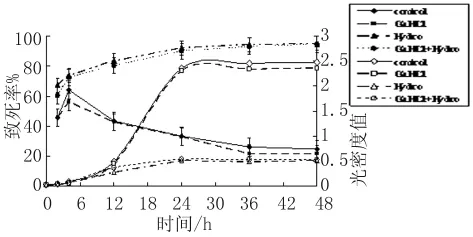
图1 羟基脲对于酵母朊病毒[PSI+]细胞致死率和生长曲线影响Fig.1 Effects of hydroxyurea on the cell mortality(left)and growth(right)of[PSI+]cells
2.2 羟基脲对盐酸胍治愈[PSI+]表型的影响 在[PSI+]细胞中,Sup35p主要以聚集体形式存在,导致无义突变抑制ade2-1突变株产生终止密码子通读现象,使[PSI+]细胞在腺嘌呤缺乏的培养基上生长为白色菌落;在 [psi-]细胞中,Sup35p主要以可溶形式存在,非无义抑制的ade2-1突变株需要在含有腺嘌呤的培养基上生长,细胞由于累积腺嘌呤合成途径中的红色中间产物而形成红色菌落,这个颜色指示系统可用来初步分析酵母朊病毒的存在状态[10]。
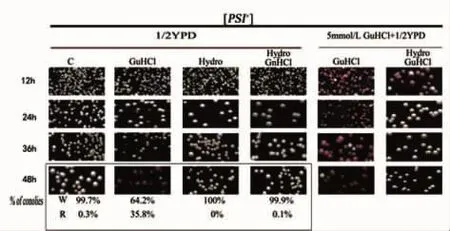
图2 羟基脲抑制细胞分裂下的盐酸胍治愈酵母朊病毒[PSI+]的情况Fig.2 Effects of hydroxyurea on guanidine hydrochloride curing yeast[PSI+]prion
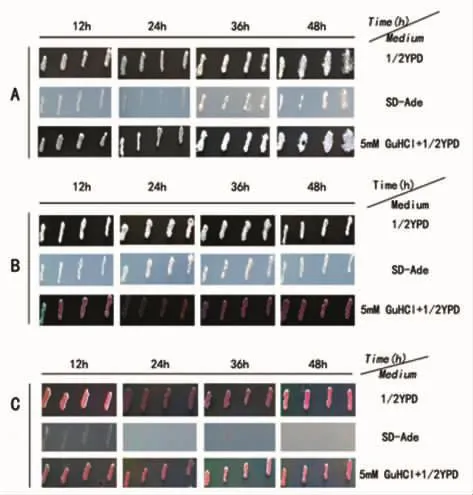
图3(A)羟基脲和盐酸胍共同作用出现的白色菌落表型分析;(B)、(C)单独盐酸胍治愈下出现的红色和白色菌落表型分析Fig.3(A):Phenotype analysis of the white colonies from combined action of hydroxyurea and guanidine hydrochloride;(B)and(C):Phenotype analysis of the white and red colonies from guanidine hydrochloride treatment,respectively
如图2所示,细胞正常分裂状态下[PSI+]细胞在1/2YPD液体培养48h之后涂板始终保持白色菌落形态,菌落数目呈现增加趋势;而加入盐酸胍的[PSI+]细胞,在培养24h之后逐渐出现红色菌落,且红色菌落比率随着培养时间的增加而上升。只加入羟基脲的[PSI+]细胞,在48h稀释后单菌落经过平板培养观察仅呈现白色菌落形态,菌落数目随培养时间的增加而逐渐减少,说明细胞分裂受到了抑制。值得注意的是,羟基脲和盐酸胍共同作用下的[PSI+]细胞虽然菌落数目随培养时间的增加而逐渐减少,但是在48h后几乎没有出现红色菌落。通过红、白表型颜色进行统计的结果显示,48h之后[PSI+]细胞的自发回复突变率为0.3%,盐酸胍对[PSI+]细胞的治愈率为35.8%,而羟基脲和盐酸胍共同作用的治愈率为0.1%。从表型来看,这暗示着羟基脲的加入使盐酸胍基本不能将[PSI+]治愈为[psi-]菌落。
2.3 羟基脲和盐酸胍共同作用[PSI+]酵母细胞的表型分析 正常的[PSI+]细胞能够在SD-Ade培养基上生长(图3B),同时能够在含有盐酸胍的1/2YPD固体培养基上被治愈为红色[psi-]菌落,且颜色表型能稳定遗传(图3C)[10]。
为了详细研究羟基脲对盐酸胍作用白色酵母朊病毒[PSI+]表型的影响,我们将图2中羟基脲和盐酸胍同时作用后形成的白色[PSI+]菌落随机挑取50个,并采用影印方法观察其菌落在不同培养基上的生长情况。如图3A所示,羟基脲和盐酸胍共同作用后的白色[PSI+]菌落,虽然在SD-Ade培养基上能够正常生长,但不能在含有盐酸胍的1/2YPD固体培养基上被治愈。因此,根据以上表型分析的结果,我们初步推测羟基脲和盐酸胍共同作用后产生的白色[PSI+]表型,可能是由于羟基脲的遗传毒性导致了酵母细胞某些代谢途径过程中的基因发生了改变,生成不能被盐酸胍治愈的[PSI+]菌落,即生成了不依赖于 Hsp104的[PSI+][11]。
2.4 羟基脲和盐酸胍共同作用[PSI+]酵母细胞的蛋白水平分析 在[PSI+]细胞中,Sup35p蛋白呈现具有一定SDS抗性的不可溶状态,形成分子量较大的聚集体;而在[psi-]细胞中,Sup35p为可溶性的单体。聚集体的分子量大小是反映酵母朊病毒[PSI+]聚集程度的重要指标,而利用SDD-AGE/Western Blot技术,可以在蛋白水平检测细胞内朊病毒聚集体分子量的大小[12]。
基于图1的实验结果,由于羟基脲对于细胞正常分裂和生长存在较强的抑制作用,因此我们仅选取羟基脲和盐酸胍共同作用4h的[PSI+]菌悬液,用珠磨仪破碎细胞提取蛋白样本,进行SDD-AGE/Western Blot检测。如图4A所示,对照的[PSI+]细胞中的Sup35p主要以混合的聚集体的形式存在,其分子量在600kDa以上;而[psi-]细胞中的Sup35p主要以单体形式存在,由于本蛋白实验体系使用了带有GFP标签的Sup35p(NGMC),其单体分子量为100kDa左右,在半变性的琼脂糖凝胶电泳中呈现拖尾的单带。单独加入羟基脲4h后对[PSI+]细胞中Sup35p蛋白的聚集形式并没有明显的影响。单独加入盐酸胍4h后,[PSI+]细胞中Sup35p蛋白的聚集体分子量逐渐变大,并伴随有少量的单体出现,说明盐酸胍抑制了Hsp104的“切割”作用,同时降低了朊病毒“种子”聚集Sup35p的单体能力,这与此前的研究结果是一致的[13]。值得注意的是,羟基脲和盐酸胍同时加入4h后,[PSI+]细胞中Sup35p蛋白聚集体的分子量与对照的[PSI+]细胞相比略微增大,但远小于盐酸胍单独作用的[PSI+]细胞,并且未出现单体形式的Sup35p蛋白。随后,我们利用Quantity One软件对SDD-AGE/Western Blot结果进行了定量分析(图4B)。以上蛋白水平的结果暗示着羟基脲能够抑制盐酸胍对酵母朊病毒[PSI+]的治愈,这与表型分析的结果是一致的。
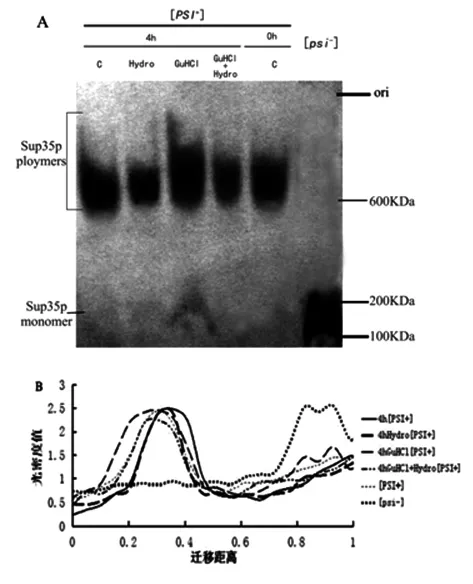
图4 羟基脲和盐酸胍共同作用下Sup35p蛋白聚集体大小的SDD-AGE/Western Blot分析Fig.4 Size of SDS-resistant Sup35p polymers in hydroxyurea and guanidine hydrochloride-treated cells by SDDAGE/Western blot analysis
3 结 论
以上研究结果表明,羟基脲抑制细胞分裂的情况下,从表型的实验数据来看,盐酸胍不能治愈酵母朊病毒[PSI+]。蛋白水平的实验数据证实了这一结论并发现,羟基脲的存在使得盐酸胍解聚酵母朊病毒聚集体的能力明显下降。这暗示着盐酸胍治愈朊病毒[PSI+]是需要细胞进行分裂的,与Tutie等提出的假说相一致。本研究弥补了单纯的基于表型研究与细胞水平分析盐酸胍治愈酵母朊病毒[PSI+]的一些不足,为进一步研究酵母朊病毒[PSI+]的聚集、解聚机制及传代、繁殖机制也提供了一个新的思路。
[1]Cox BS.Ψ,a cytoplasmic suppressor of super-suppressor in yeast[J].Heredity,1965,20(2):505-521.DOI:10.1038/hdy.1965.65
[2]Glover JR,Lindquist S.Hsp104,Hsp70,and Hsp40:A novel chaperone system that rescues previously aggregated proteins[J].Cell,1998,94(1):73-82.DOI:10.1016/S0092-8674(00)81223-4
[3]Chernoff YO,Lindquist SL,Ono B,et al.Role of the chaperone protein Hsp104in propagation of the yeast prion-like factor[PSI+][J].Science,1995,268(5212):880-884.DOI:10.1126/science.7754373
[4]Eaglestone SS,Ruddock LW,Cox BS,et al.Guanidine hydrochloride blocks a critical step in the propagation of the prion-like determinant[PSI+]ofSaccharomycescerevisiae[J].Proc Natl Acad Sci U S A,2000,97(1):240-244.DOI:10.1073/pnas.97.1.240
[5]Cox B,Ness F,Tuite M.Analysis of the generation and segregation of propagons:entities that propagate the[PSI+]prion in yeast[J].Genetics,2003,165(1):23-33.
[6]Wu YX,Greene LE,Masison DC,et al.Curing of yeast[PSI+]prion by guanidine inactivation of Hsp104does not require cell division[J].Proc Natl Acad Sci U S A,2005,102(36):12789-12794.DOI:10.1073/pnas.0506384102
[7]Byrne LJ,Cox BS,Cole DJ,et al.Cell division is essential for elimination of the yeast[PSI+]prion by guanidine hydrochloride[J].Proc Natl Acad Sci U S A,2007,104(28):11688-11693.DOI:10.1073/pnas.0701392104
[8]Halfmann R,Lindquist S.Screening for amyloid aggregation by semi-denaturing detergent-agarose gel electrophoresis[J].J Vis Exp,2008,17:e838.DOI:10.3791/838
[9]Song Y,Lan W,Wu X,et al.Quantitative effects of magnesium chloride stress on aggregation of Sup35p in[psi-]yeast cells[J].Protein Pept Lett,2010,17(12):1489-1494.
[10]Bach S,Talarek N,Andrieu T,et al.Isolation of drugs active against mammalian prions using ayeast-based screening assay[J].Nat Biotechnol,2003,21(9):1075-1081.DOI:10.1038/nbt855
[11]Jung G,Jones G,Wegrzyn RD,et al.A role for cytosolic Hsp70in yeast[PSI+]prion propagation and[PSI+]as a cellular stress[J].Genetics,2000,156(2):559-570.
[12]Song YT,Wu YX,Jung G,et al.Role for Hsp70chaperone inSaccharomycescerevisiaeprion seed replication[J].Eukaryot Cell,2005,4(2):289-297.DOI:10.1128/EC.4.2.289-297.2005
[13]Song YT,Song Y,Zhong ZW,et al.Illuminating precise quantification in yeast-based model for antiprion compounds screening[A].Proceedings of 2010first international confSerence on cellular,molecular biology,biophysics and bioengineering(Volume 3)[C],2010,1(3):296-299.

