基于EF1-a基因对常见禾本科作物的聚类分析
押辉远, 李卫涛, 王卫东, 焦 贞
(1.洛阳师范学院 生命科学系 河南 洛阳 471022; 2.郑州大学 河南省离子束生物工程重点实验室 河南 郑州 450052)
0 引言
禾本科(Poaceae)植物是单子叶植物(Orchidaceae)中的第2大科,有近700属,约11 000种.我国有190多属,1 200多种[1].不仅包括了水稻(OryzasativaL.)、小麦(TriticumaestivumL.)和玉米(ZeamaysL.)三大粮食作物,还包括高粱(SorghumMoench)、甘蔗(SaccharumL.)等其他重要经济作物,其中一些杂草是生态学的优势物种和先锋植物[2].然而禾本科的亚科和族的划分一直存在较大争议:Clayton等[3]将禾本科分成6个亚科;而Soderstrom等[4]则把稻族和菰族从亚科中分离出来,置于稻亚科中,形成7亚科;GPWG[5]通过对质体DNA和核DNA数据分析及结合形态学的方法将禾本科划分成12个亚科.
随着分子生物学的发展,基于基因序列的分子数据已被广泛应用于物种分类学、系统学和种群遗传学的研究中.植物延伸因子EF1-a是真核生物细胞内的第二大蛋白,并且在蛋白质合成延伸过程中起重要作用[6].许多禾本科植物如大麦、玉米、小麦、水稻等的EF1-a互补DNA (complement DNA, cDNA)序列已经测定,通过比较分析多种植物的EF1-a基因编码的氨基酸序列,发现种间的EF1-a基因编码的氨基酸序列高度保守,同源性高于95%[7],适合用于种系发生分析,可以作为研究系统发育的有力工具[8].目前,依据形态学、不同来源(核基因组、叶绿体基因组或线粒体基因组)的基因序列开展的禾本科植物系统分类研究获得的结果也都有所差异,而基于EF1-a基因对禾本科植物分类的研究少有所见.作者对水稻、小麦、大麦(Hordeumvulgare)、玉米、芒(Miscanthussinensis)、发草(Deschampsiaantarctica)等禾本科植物的EF1-a基因核苷酸和氨基酸序列进行比对分析,构建分子系统进化树.旨在进一步了解禾本科植物之间的进化亲缘关系,为禾本科植物种属间的分类提供确切的分子生物学依据.
1 材料和方法
1.1 材料
本文中所有EF1-a序列信息来自NCBI(http://www.ncbi.nlm.nih.gov/).
本研究所用植物材料如下:水稻(OryzasativaL.)、小麦(TriticumaestivumL.)、玉米(ZeamaysL.)、大麦(Hordeumvulgare)、芒(Miscanthussinensis)、发草(Deschampsiaantarctica)、大豆(Glycinemax).
1.2 方法
利用NCBI中的BLAST软件对6种禾本科植物的EF1-a基因序列进行同源性比对.运用Clustal X version 1.8分析软件[9],参数设为默认值, 对28个基因序列进行多重对比并人工校正.利用MEGA 5.0构建分子系统进化树[10].ME、NJ、UPGMA分子系统进化树均采用软件中的默认参数.
2 结果与分析
2.1 核苷酸和氨基酸序列的相似性
稻属中9个EF1-a核苷酸序列之间的相似值达到95%以上,而氨基酸序列之间的相似值达到99%以上;玉米属中15个EF1-a核苷酸序列之间的相似值多数在90%左右,但也出现了84%左右的低相似值,氨基酸序列之间的相似值相比于水稻也略显低些.研究表明,水稻EF1-a基因核苷酸序列相似性普遍较高,玉米相似性存在差异,但是氨基酸序列的相似性高于核苷酸序列的相似性,研究结果支持禾本科植物EF1-a氨基酸序列保守的观点[11].
2.2 多重序列比对分析
对6种禾本科植物28个EF1-a基因核苷酸序列和氨基酸序列进行多重序列比对分析,结果表明:核苷酸序列同源性高,氨基酸序列比核苷酸序列的同源性更高,所以氨基酸序列比较适合分子系统发育重建.
2.3 分子系统进化树分析
用gi|xxxxx代表NCBI中不同的EF1-a基因序列的编号,利用ME法构建核苷酸分子系统进化树,结果见图1.6种禾本科植物被分为4个组群,引自水稻属的9个核苷酸序列材料聚为一支,靴带支持率100%;引自小麦和大麦的2个序列材料聚在一起,靴带支持率100%,它们的亲缘关系更近;引自发草的材料聚为一类;引自玉米的15个材料在聚为一个组群的前提下又分为两个组群,其中引自芒的一个材料与玉米组群中的第二个组群聚为一类.
利用NJ法对核苷酸序列构建分子系统进化树,结果见图2.来自水稻的9个氨基酸序列聚为一支,靴带支持率100%;来自小麦和大麦的两个核苷酸序列聚为一支,节点支持率100%;来自水稻和麦属的材料聚在一起,亲缘关系较近;引自玉米的核苷酸序列在聚为一个组群的前提下又聚为两类,其中引自芒的材料聚在玉米的第二个类群中,表现出一定的亲缘关系;发草材料单独聚为一个类群.
利用ME法对氨基酸序列构建分子系统进化树,结果见图3.引用材料被聚类分为三大类群,来自水稻的9个氨基酸序列聚为一类,靴带支持率98%;来自小麦、大麦的两个氨基酸序列聚为一类,靴带支持率100%,然后与发草聚在一起,靴带支持率53%;引自玉米的材料被聚类分为两个大类,靴带支持率分别为90%、65%,其中在第二个大类中,芒以靴带支持率63%聚在其中.
利用NJ法对氨基酸序列构建分子系统进化树,结果见图4.来自水稻的氨基酸序列聚为一类,靴带节点支持率98%;来自小麦、大麦的氨基酸序列聚为一类,靴带支持率100%,然后与发草聚为一个类群;来自玉米的氨基酸序列聚为一支,靴带支持率54%,然后被聚为两个类群.来自芒的序列插在其中.
利用UPGMA法对氨基酸序列构建分子系统进化树,结果见图5.来自水稻的9个序列聚在一支,靴带支持率96%;来自小麦和大麦的序列也聚在一支,靴带支持率100%;而玉米的氨基酸序列分布在不同分支,分析价值不高,但是其他的物种聚类明显.因此,此种方法运用在禾本科植物氨基酸序列的聚类分析还有待进一步的研究.
利用UPGMA法对核苷酸序列构建分子系统进化树,结果见图6.核苷酸序列被聚类分析分为四大分支,引自发草的材料单独聚为一类;引自水稻和麦属的材料聚为一个大类,然后麦属和水稻各自聚在一起;引自玉米的材料被聚为了两个大类,而芒聚在了其中一个大类里面.
通过对核苷酸和氨基酸序列构建的分子系统进化树进行比较分析,得出以下结论:利用核苷酸序列构建的3种进化树都能准确反映禾本科植物的分类地位和亲缘关系,并且与传统的分类基本吻合.利用氨基酸序列构建的ME树和NJ树也能反映禾本科植物的分类地位和亲缘关系,但构建的UPGMA树对玉米的分类准确性不高,原因可能是与玉米亚种较多,基因具有多态性有关.因此核苷酸序列构建的系统进化树更适合于属间的聚类进化分析,而氨基酸序列保守性高,更适合于种间的系统进化分析,3种构建方法以UPGMA法对亚种间的细化分类最为合适.
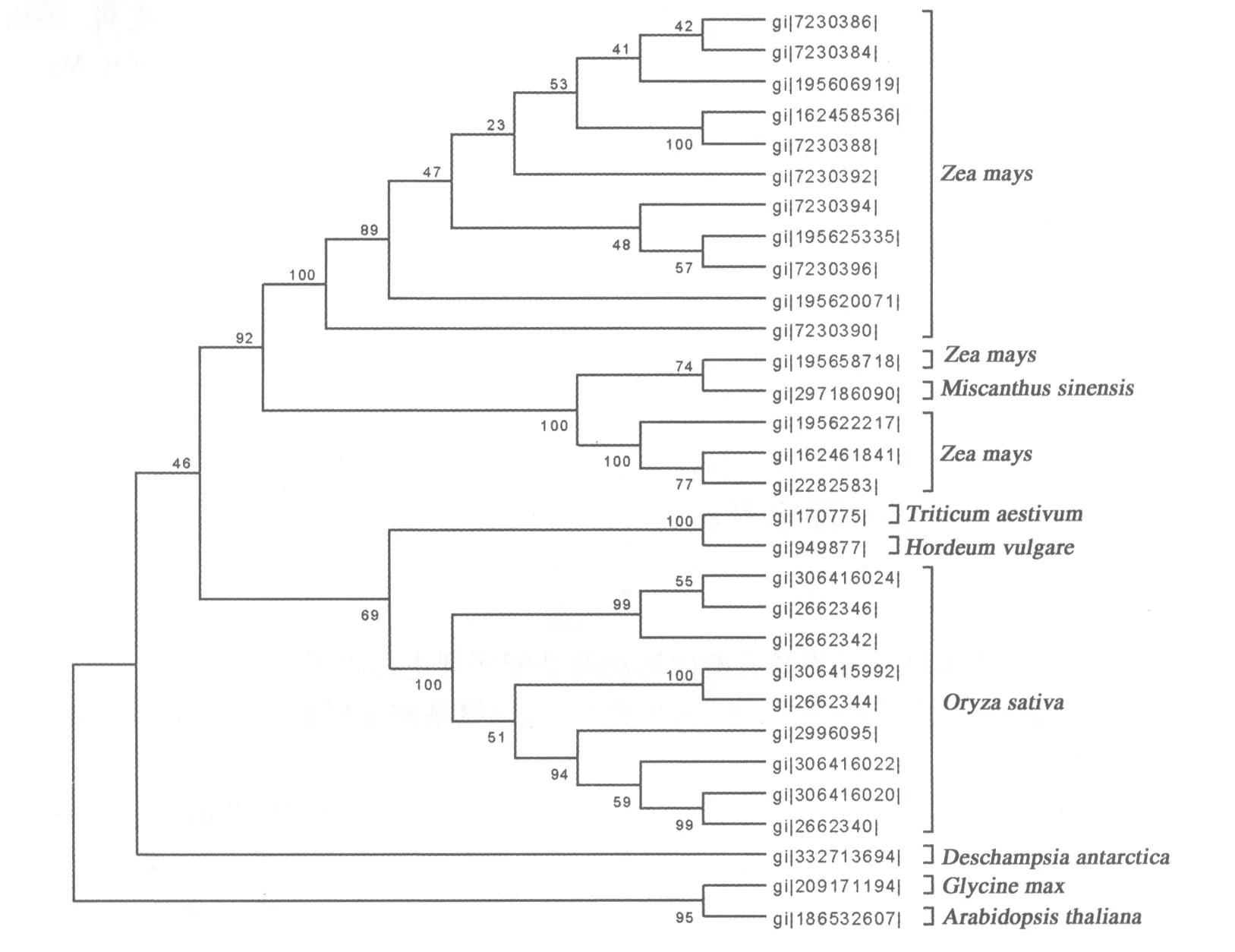
图1 基于EF1-a核苷酸序列构建的ME树Fig.1 ME tree established on the basis of EF1-a cDNA sequences
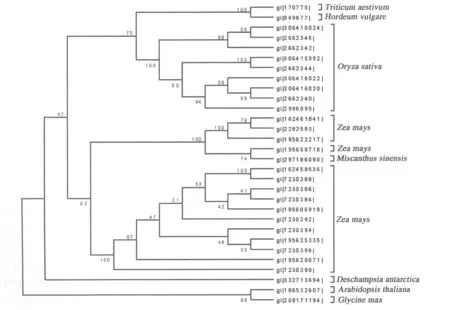
图2 基于EF1-a核苷酸序列构建的NJ树Fig.2 NJ tree established on the basis of EF1-a cDNA sequences
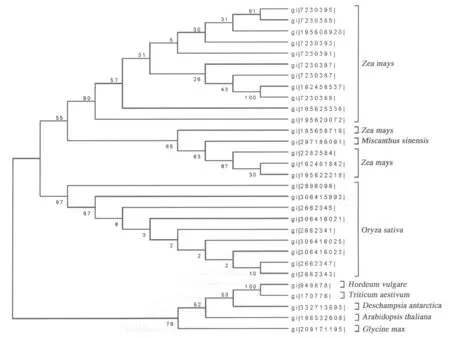
图3 基于EF1-a氨基酸序列构建的ME树Fig.3 ME tree established on the basis of EF1-a amino acid sequences
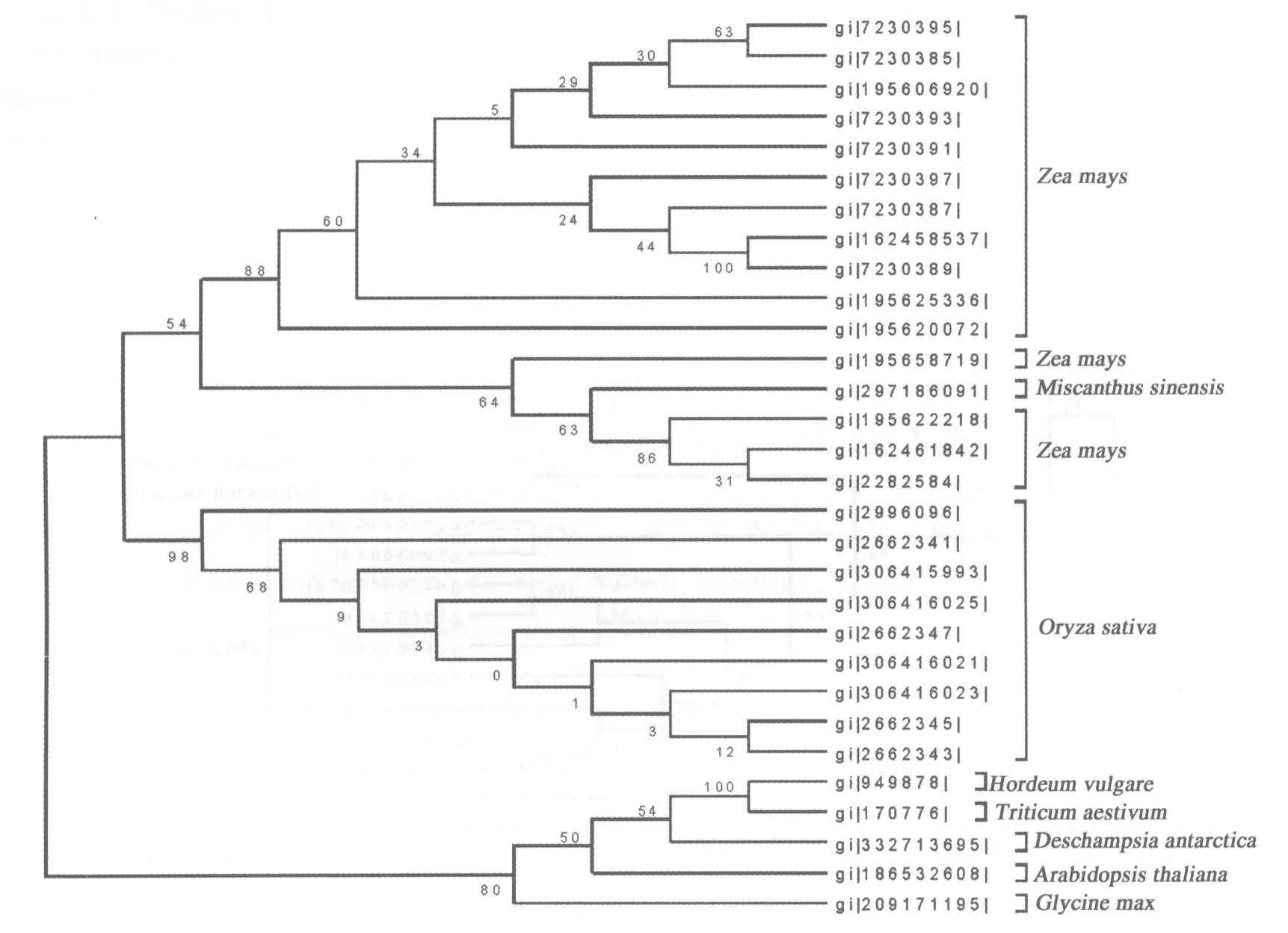
图4 基于EF1-a氨基酸序列构建的NJ树Fig.4 NJ tree established on the basis of EF1-a amino acid sequences
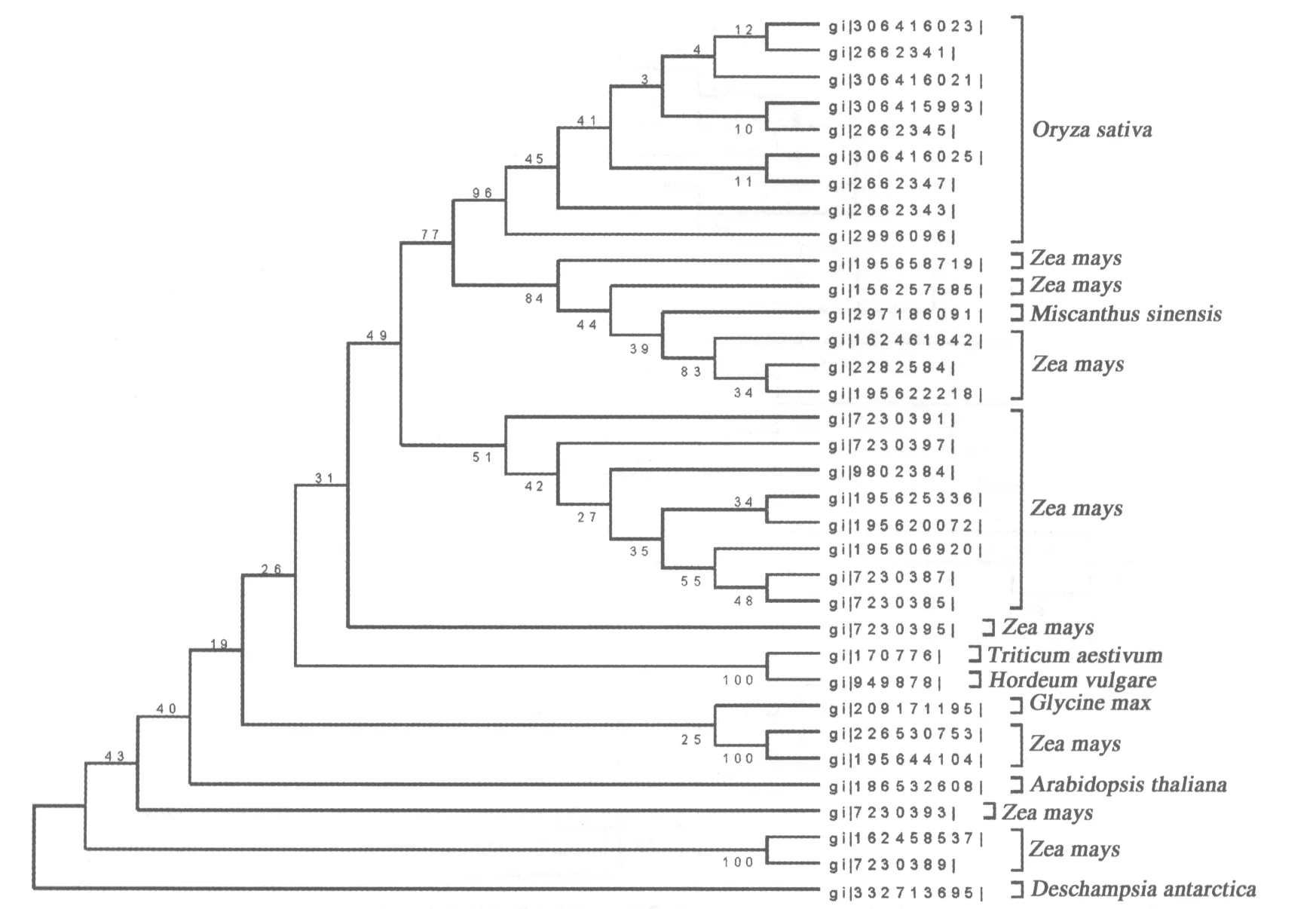
图5 基于EF1-a氨基酸序列构建的UPGMA树Fig.5 UPGMA tree established on the basis of EF1-a amino acid sequences
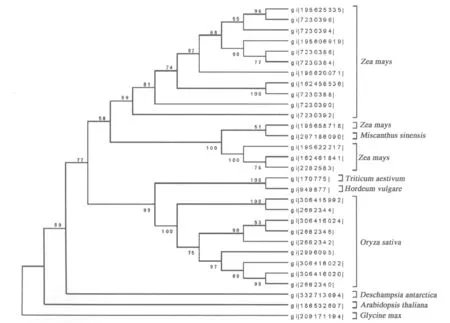
图6 基于EF1-a核苷酸序列构建的UPGMA树Fig.6 UPGMA tree established on the basis of EF1-a cDNA sequences
3 讨论
核苷酸序列是物种进化的忠实记录者,直接研究其序列变异可以再现物种进化历程,为物种系统演化提供有益的线索[12].因此,基于分子水平的系统发育研究,对于揭示植物系统进化的本质具有重大的意义.作者通过对主要禾本科植物EF1-a的基因序列和氨基酸序列比对分析及构建分子系统进化树,结果表明EF1-a基因对禾本科植物种属间分类具有一定的应用价值.植物延伸因子EF1-a广泛存在于真核细胞内,其表达调控十分保守,与植物细胞的多种生理生殖反应有关.植物种间的EF1-a氨基酸序列高度保守,在种间遗传进化分析研究中作用显著,所以其不失为遗传进化分析的另一种良好材料.可以根据该基因的保守序列克隆某一分类位置有争议的禾本科植物的EF1-acDNA序列,与已有的禾本科该基因的cDNA共同构建合适的系统进化树,作为该物种分类的重要参考依据.另外,由于对植物延伸因子的研究不是很完善,本研究的覆盖面还不是很广,要达到更完善的程度,需要对植物延伸因子进行更多的研究.
参考文献:
[1] 郭本兆.中国植物志:第九卷第三分册[M]. 北京:科学出版社,1987:116-118.
[2] Shantz H L. The place of grasslands in the earth’s cover of vegetation [J]. Ecology, 1954, 35(2): 143-145.
[3] Clayton W D, Renvoize S A. Genera Graminum [M]. London: Her Majesty’s Stationery Office, 1986:303-310.
[4] Soderstrom T R, Hilu K W, Campbell C S, et al. Grass Systematic and Evolution [M]. Washington D C: Smithsonian Institute Press, 1987:458-466.
[5] Grass Phylogeny Working Group. Phylogeny and subfamilial classification of the grasses (Poaceae) [J]. Annals of the Missouri Botanical Garden, 2001, 88(3): 373-457.
[6] Negrutskii B S, El’skaya A V. Eukaryotic translation elongation factor 1 alpha: structure, expression, functions and possible role in aminoacyl-tRNA channeling[J]. Prog Nucleic Acid Res Mol Biol, 1998, 60: 47-78.
[7] Browning K S. The plant translational apparatus[J]. Plant Molecular Biology, 1996, 32(1/2): 107-144.
[8] Gaut B S. Evolutionary dynamics of grass genomes [J]. New Phytologist, 2002, 154(1): 15-28.
[9] Thompson J D, Gibson T J, Plewniak F, et al. The Clustal X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools [J]. Nucleic Acids Research, 1997, 25(24):4876-4882.
[10] 张阳德.生物信息学[M].北京:科学出版社,2004:163-178.
[11] Gaut B S. Evolutionary dynamics of grass genomes [J]. New Phytologist, 2002, 154(1): 15-28.
[12] 赵建国,李辉,王启贵,等.鸡偶联蛋白(UCP)基因内含子的克隆与系统发生树的构建 [J].遗传,2004, 26(1):50-54.

