赤眼鳟线粒体D-loop和Cyt b基因序列的对比分析*
杨慧荣,赵会宏 ,蒙子宁 ,刘 丽 ,林权卓
(1.华南农业大学动物科学学院,广东 广州 510642;2.中山大学水生经济动物研究所暨广东省水生经济动物良种繁育重点实验室,广东 广州 510275)
赤眼鳟Squaliobarbuscurriculus属鲤科Cyprinidae,雅罗鱼亚科Leuciscinae,赤眼鳟属Squaliobarbus,俗称红眼棒、野草鱼、参鱼,是广布中国主要水系的野生经济鱼类[1]。赤眼鳟因其肉质细嫩,味道鲜美,具有抗病力强,食性杂,生长较快等特点,成为我国重要的经济鱼类之一。目前,有关赤眼鳟的研究主要集中于资源状况、基础生物学、养殖和人工繁殖方面[2-3],而关于其群体遗传学与基因多样性研究则鲜有报道,仅2008年杨太有等[4-6]用RAPD和ISSR技术对丹江口水库、青龙湖和宿鸭湖3地理群体进行了遗传多样性分析。
线粒体DNA(Mitochondrial DNA,mtDNA)具有母性遗传、进化速度快、核苷酸替代率高等特点,已成为鱼类进化生物学和群体遗传学研究的重要遗传标记[7-8]。mtDNA基因组内不同的区域进化速度存在差异,适合不同水平的进化研究。在mtDNA上,位于tRNAPro和tRNAPhe基因之间的D控制区(D-loop)是整个mtDNA上序列和长度变异最大的区域[9],也线粒体基因中进化最快的部分,适用于种群水平差异的检测,也可用于种间分析[7-10]。而Cytb基因是线粒体DNA上的蛋白质编码基因,其进化速度适中,适合种群水平差异的检测,是探讨种间和种内遗传分化程度的良好标记[11-13]。本研究开展了珠江水系和长江水系不同水域赤眼鳟mtDNA D-loop和Cytb基因序列的对比分析,旨在研究这两种遗传标记对赤眼鳟遗传多样性状况和群体遗传结构的差异,探讨mtDNA D-loop和Cytb基因对于群体遗传多样性分析的优缺点,进而为针对不同研究材料、不同研究目的,选择不同的分析方法提供技术上的指导,使得结论更客观真实。
1 材料与方法
1.1 材料
赤眼鳟样品于2005年6月分别采自长江水系的宜昌和武汉江段(编号为YC群体、WH群体),珠江水系梧州和新丰江段(编号为WZ群体、XF群体)。YC群体取自宜昌巴东县的官渡口,属于长江上游干流巫峡的起点;WH群体取自武汉市境内江段,属于长江中游水域;WZ群体和XF群体分布代表了珠江水系的西江和东江流域。共计24个个体,均为野生群体。活体运回实验室后取背部肌肉并置于超低温冰箱,取样后的赤眼鳟用福尔马林固定保存。
1.2 方法
基因组DNA的提取参考《分子克隆实验指南》[14]的方法略加改进。PCR反应在Biometra PCR仪上进行。D-loop和Cytb引物由Invitrogen生物技术有限公司合成,D-loop引物序列如下:DL1:5'-ACCCCTGGCTCCCAAAGC-3',DH2:5'-ATCTTAGCATCTTCAGTG-3'[15]。Cytb引物序列如下:L14724:5'-GACTTGAAAAACCACCGTTG-3',H15915:5'-CTCCGATCTCCGGATTACAAGAC-3'[12]。反应总体积50 μL,其中10×Taq Buffer 5 μL,Taq Polymerase 0.5 μL (5 U·μL-1),dNTPs(2.5 mmol·L-1)4 μL,Mg2+(25 mmol·L-1)2.5 μL,引物1和2(10 umol·L-1)各2 μL,模板DNA 50 ng,无离子超纯水35.5 μL。扩增条件为:94 ℃预变性2 min后,再进行35个循环,每一循环包括:94 ℃ 45 s,58 ℃ 45 s,72 ℃ 1 min;最后72 ℃延伸7 min。每次PCR反应均设不含模板DNA的空白对照。扩增产物经w=1.5%TBE琼脂糖凝胶电泳分离,EB染色,紫外灯光下检测、拍照。
1.3 测序与数据分析
产物纯化和序列测定由上海基康公司(Gene core Co.)完成,通过双向测序获得赤眼鳟D-loop和Cytb序列,经SeqManII (DNASTAR Inc)软件拼接并赋予人工校对后,用CLUSTAL-X程序对所获得的序列进行比对[16],采用软件DAMBE统计单倍型并作图,检测转换、颠换是否达饱和。群体的遗传多样性指标单倍型多样性(Haplotype diversity,H)和核苷酸多样性(Nucleotide diversity,π)由ARLEQUIN[17]统计软件计算获得,群体间的分化指数(F-statistics,FST)[18]的计算在ARLEQUIN中完成,群体间的基因流[19]由公式Nm=[(1/FST)-1]/2计算得出,群体遗传变异的分子方差分析(AMOVA)[20]在ARLEQUIN中完成。采用MEGA4.1软件[21]统计DNA序列的碱基组成,转换/颠换、插入/缺失位点数,单突变位点,简约信息位点和平均转换/颠换率;利用Kimura双参数模型计算个体间、群体内和群体间的遗传变异率;用NJ法进行聚类分析,构建聚类关系树。
2 结 果
2.1 D-loop和Cyt b基因序列信息
由软件DAMBE统计可知:在24个598 bp的D-loop基因序列中共检测出18个单倍型。由MEGA4.1软件对D-loop基因序列进行分析,检测到18个单突变位点,34个简约信息位点数;在67个突变位点中:转换位点40个,颠换位点15个(其中有2个位点即发生了转换,又发生了颠换),插入/缺失位点14个,转换明显高于颠换,平均的转换发生频率是颠换的3.192。24个个体D-loop基因片段中,T、C、A、G碱基的平均含量分别为35.1%,17.5%,33.7%,13.7%,其中,A+T的含量(68.8%)明显高于C+G的含量(31.2%)。同样,在24个752 bp的Cytb基因序列中共检测出18个单倍型,检测到4个单突变位点,68个简约信息位点数;在72个突变位点中:转换位点60个,颠换位点15个(其中有3个位点即发生了转换,又发生了颠换),无插入/缺失位点,转换明显高于颠换,平均的转换发生频率是颠换的4.636。T、C、A、G碱基平均含量分别为26.4%,29.8%,29.3%,14.5%,其中,A+T的含量(55.7%)略高于C+G的含量(44.3%)。对比分析可知:Cytb除简约信息位点百分比大于D-loop外,多态位点百分比和单突变位点百分比均小于D-loop(表1)。

表1 赤眼鳟线粒体D-loop 及Cyt b碱基组成及变异分析
2.2 D-loop和Cyt b基因序列的遗传变异分析
用软件MEGA4.1统计出两水系4个不同水域赤眼鳟各个体间的转换/颠换数及遗传变异率。D-loop序列中,各个体间的转换位点为0~24,颠换位点为0~8,均为转换大于颠换。两大水系4个不同群体个体间的遗传变异率在0~5.93%之间,赤眼鳟不同个体与青鱼的遗传变异率在19.09%~20.43%之间。4个不同群体赤眼鳟(WZ、XF、WH和YC)群体内的遗传距离分别是0.012 0、0.016 2、0.009 9和0.004 8,群体间的遗传距离值为0.007 0~0.049 1,总平均遗传距离为0.029 3(不含外群)。Cytb序列中,各个体间的转换位点为0~51,颠换位点为0~13,均为转换大于颠换。两大水系4个不同群体个体间的遗传变异率在0~8.78%之间,而赤眼鳟不同个体与青鱼的遗传变异率在11.61%~12.48%之间。4个不同群体赤眼鳟(WZ、XF、WH和YC)群体内的遗传距离分别是0.010 9、0.007 3、0.003 8和0.005 2,群体间的遗传距离值为0.004 3~0.082 7,总平均遗传距离为0.045 5(不含外群)(表2)。
4个群体的D-loop和Cytb序列的单倍型多样性(H)、平均核苷酸差异数(K±SD)、核苷酸多样性(π)见表3。4个群体间的FST及Nm值见表4。从表4可以看出:武汉群体与新丰群体及梧州群体的遗传差异显著,宜昌群体与新丰群体及梧州群体的遗传差异显著,而同一水系内的不同群体差异不显著。把4个群体作为一个组进行群体间的分子变异等级分析(AMOVA)(表4),结果表明:不管D-loop,还是Cytb,群体间都存在显著性遗传差异(FST=0.689 77或0.871 94,P=0.000 0<0.05)。

表2 两大水系4个不同水域赤眼鳟D-loop和Cyt b基因序列群体内(对角线上)、群体间(对角线下)以及总平均(最后行)遗传距离1)
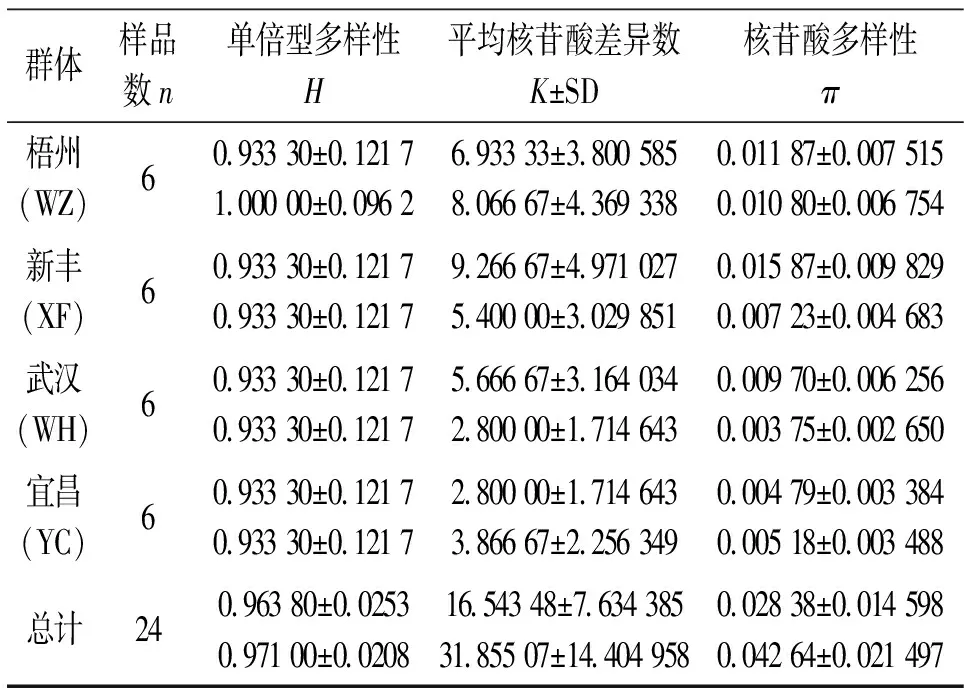
表3 4个赤眼鳟群体相关遗传多样性指标1)

表4 群体间的FST(对角线下方)和Nm(对角线上方)1)
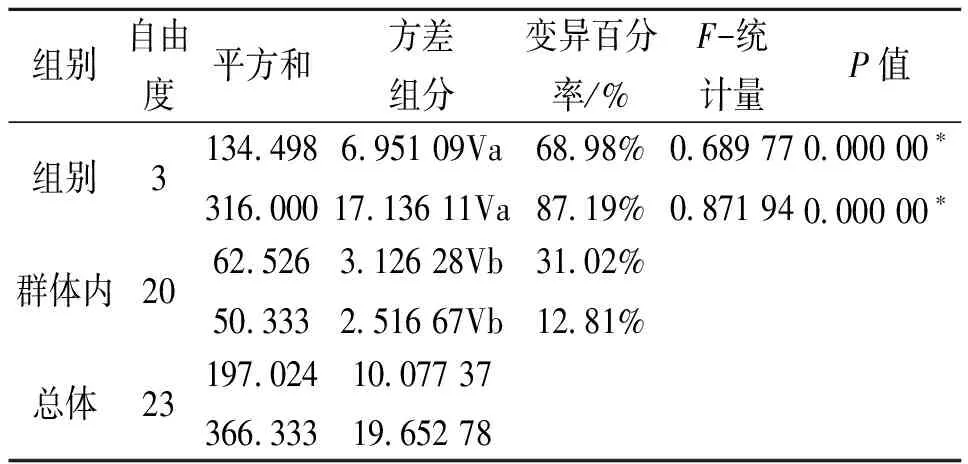
表5 群体间的遗传变异的分子变异等级分析(AMOVA)1)
2.3 分子系统树
以青鱼MylopharyngodonpiceusD-loop和Cytb基因序列为外群,用MEGA4.1中的“NJ”法构建分子系统树,其拓扑结构见图1。用Bootstrap 1000给出各支的置信度,最高为100,最低为17。两大水系4个不同水域赤眼鳟24个个体的系统树明显分为两支:珠江12个个体聚为一支,长江12个个体聚为另一支,且都有较高的置信度,各大水系内的不同水域个体却混杂在一起。
3 讨 论
3.1 D-loop和Cyt b对赤眼鳟遗产多样性的分析
单倍型多样性(H)、平均核苷酸差异数(K±SD)和核苷酸多样性(π)是反映物种遗传变异水平的3个重要参数,赤眼鳟D-loop和Cytb的H、K和π值分别为0.963 8、16.543 48、0.028 38和0.971 0、31.855 07、0.042 6,与相同技术对光唇裂腹鱼[22-25]的研究结果相比(表5),本研究中赤眼鳟各遗传多样性参数值较高。另外,根据碱基组成的基本信息,赤眼鳟具有较高的单倍型检出率(数)、多态位点百分比(数)以及简约信息位点百分比(数)[22-26]。所以,无论是D-loop,还是Cytb,两者都很相似地得出赤眼鳟总体较高遗传多样性的研究结果。由于物种特定的遗传结构、分类地位、生态习性及淡、海水鱼类在基因交流方面的不同,可能导致它们彼此之间在遗传分化水平上存在差异,但上述比较,在一定程度上能够间接反映出赤眼鳟的遗传多样性状况。

图1 两大水系4个不同水域赤眼鳟D-loop(A)和Cyt b(B)基因序列的NJ分子系统树(数值表示1 000次重复抽样的百分比)
一般来说,物种遗传多样性的高低与其适应能力、生存能力和进化潜力密切相关,遗传变异是有机体适应环境变化的必要条件[27-28]。虽然赤眼鳟具有较高的遗传多样性水平,但由表2、4可以看出,D-loop和Cytb很一致的表明:长江和珠江水系间存在明显的遗传变异,可同一水系内群体间的遗传变异和遗传多样性并不十分丰富。分子变异分析(AMOVA)结果也显示:4个群体总的遗传分化指数为0.689 77和0.871 94(P<0.001),其中68.98%、87.19%遗传变异存在于群体间,群体内的遗传变异率对总的遗传变异贡献较小。所以,在同一水系内部,赤眼鳟的遗传多样性状况并不十分乐观,加强对现有资源的科学管理和保护、恢复有效种群大小、丰富遗传多样性是保护赤眼鳟自然资源的有效举措。
3.2 D-loop和Cyt b基因的变异位点比较
在所研究的不同水域赤眼鳟个体D-loop序列中检测出了67个突变位点,多态位点的比例高达11.2%,再一次验证了D-loop序列的高进化速率。其中插入/缺失位点14个,在检测的Cytb中就没有发现插入/缺失位点,在其他的研究中也证实了该观点[29,30],这主要是由于Cyt b用于编码蛋白质,如有插入/缺失的发生很容易被淘汰;而D-loop片段为非编码基因,主要与线粒体基因的调控有关,不受蛋白功能上的需要和三联体密码的限制,所以插入/缺失突变后比较容易被保留。在D-loop和Cytb的变异位点中,转换和颠换比值分别为3.192和4.636,通过软件DAMBE分析所得的转换、颠换对遗传距离图可知:D-loop和Cytb的转换和颠换数没有达到平台效应,即没有达到饱和状态,说明供统计分析的数据准确可靠,序列中的碱基替代均具有系统发育意义[31],可以用来作为赤眼鳟种内的遗传分析。统计每个个体两两之间的转换和颠换数,每条序列的转换均大于颠换,Gerlach等[32]的研究成果也证实了该观点,其原因仍不清楚,需要进一步研究。在D-loop和Cytb基因碱基组成中,A+T含量均高于C+G含量,且D-loop基因中A+T含量要高出Cytb近11.1%,这与其他的研究结果相一致[33,34]。

表6 不同物种相关遗传多样性指标比较1)
3.3 D-loop和Cyt b基因的进化比较
线粒体基因组的特性使它已成为进化生物学和群体遗传学研究中重要的分子标记。目前,研究最多的是Cyt b、12S rRNA、16S rRNA、COⅠ基因和D-loop区[22-26,30,34-35]。本研究中,尽管D-loop和Cytb很一致的表明长江和珠江水系赤眼鳟存在明显的遗传分化,但同一水系内群体间的遗传变异并不丰富,且D-loop和Cytb基因之间也存在差异。由群体间的遗传距离值和FST值(表2和表4)可以看出,对于同一水系群体间(如WZ-XF,YC-WH)的遗传差异较小,Cytb则更小,对于不同水系群体间(如WZ-YC和WH,XF-YC和WH)的遗传差异较大,Cytb则更大,呈现出“小则越小,大则越大”的态势。分析其原因,可能与D-loop和Cytb进化速率密切相关。
有研究表明,线粒体不同基因的进化速率存在差异[37]。D-loop区由于不编码蛋白,受到的选择压力最小,所以进化速度最快[23,36]。Cytb基因由于编码线粒体细胞色素b蛋白,其进化速度适中,适合研究种内到种间甚至科间的系统发育关系,近年来广泛地应用于爬行类、鱼类等的系统发育、种类鉴别等的研究[38-40]。蒙子宁等[26]认为,石斑鱼线粒体细胞色素b基因序列在种内变异极小、变异位点数和平均核苷酸差异数等均非常低;但该基因序列在石斑鱼种间却存在较大的核苷酸差异,UPGMA系统树更是以100%的置信度将斜带石斑鱼和赤点石斑鱼区分开。由此可见,Cytb基因具有保守和变异的双重特性、保守性决定了该基因不适合做石斑鱼的种内遗传变异分析;但其变异性使得该基因适合于种以上水平的种类鉴定。李娜等[23]的研究中,将中国香鱼与日本香鱼的Cytb基因比对后,仅发现2个变异位点,说明Cytb基因在香鱼种内的变异很小,所以作者认为其不适合作为种内遗传分析的标记。而在D-loop区发现17个变异位点,说明香鱼与其他脊椎动物一样线粒体D-loop区的进化速度较快。
据此分析,是否可以推测:Ctyb在遗传变异显著(或遗传分化明显)的群体中变异较大,更适合种间或以上阶元系统进化分析,而D-loop在遗产差异不显著的群体中变异相对较大,适合同种不同地理居群的多样性分析?
由于各种遗传分析方法都有优缺点,因此在实践应用中,需根据研究材料、目的不同,选择不同的分析方法,且最好是几种方法同时运用,将结果相互印证,以便使得到的结论更客观,更真实。
参考文献:
[1]杨干荣,黄宏金.雅罗鱼亚科[M] // 伍献文.中国鲤科鱼类志.上海: 上海科学技术出版社,1982: 52-53.
[2]李桂峰,康裕财,孙际佳,等.酵母多糖对赤眼鳟非特异性免疫技能的影响[J].中山大学学报:自然科学版,2003,42(4): 55-58.
[3]何学福,阳清发.嘉陵江西河赤眼鳟的生长研究[J].西南师范大学学报:自然科学版,1997,22(6): 680-685.
[4]杨太有,关建义,陈宏喜,等.丹江口水库赤眼鳟(Squaliobarbuscurriculus)遗传多样性的RAPD和ISSR分析[J].海洋与湖沼,2008,39(2): 157-161.
[5]杨太有,关建义,陈宏喜.三个地理群体赤眼鳟遗传多样性的ISSR分析[J].水生生物学报,2008,32(4): 529-533.
[6]杨太有,关建义,陈宏喜,等.三个地理群体赤眼鳟遗传多样性的RAPD分析[J].四川动物,2008,27(5): 783-784.
[7]郭新红,刘少军,刘巧,等.鱼类线粒体DNA研究新进展[J].遗传学报,2004,31(9): 983-990.
[8]肖武汉,张亚平.鱼类线粒体DNA的遗传与进化[J].水生生物学报,2000,24(4): 384-391.
[9]LEE W J,KOCHER T D.Complete sequence of a sea Lamprey (Petromyzoamarinus) mitochondrial genome: Early establishment of the vertebrate genome organization [J].Genetics,1996,139: 873-887.
[10]葛陇利,赫崇波,高祥刚,等.利用mtDNA控制区序列分析斑点叉尾鮰的遗传多样性[J].水产科学,2007,26(10): 547-550.
[11]MOMMSEN H.Biochemistry and molecular biology of fishes[M].London: Elsevier Science Publishiers,1993: 1-38.
[12]XIAO Wuhan,ZHANG Yaping,LIU Huanzhang.Molecular systematics of Xenocyprinae(Telestei: Cyprinidae): taxonomy,biogeography,and coevolution of a special group restricted in east Asia[J].Mol Phylogenet Evol,2001,18: 163-173.
[13]韩虎峰,代应贵.珠江水系光唇裂腹鱼可渡河种群mtDNA D-loop序列多态性分析[J].中国水产科学,2010,17(1): 143-149.
[14]SAMBROOK J,RUSSELL D.分子克隆实验指南 [M].2版.金冬雁,黎孟枫,译.北京: 科学出版社,1996: 464-469.
[15]唐琼英,刘焕章,杨秀平,等.沙鳅亚科鱼类线粒体DNA控制区结构分析及系统发育关系的研究[J].水生生物学报,2005,29(6): 645-653.
[16]THOMPSON J D,GIBSON T J,PLEWNIAK F,et al.The Clustal_X windows interface: flexible strategies for multiple sequences alignment aided by quality analysis tools[J].Nucleic Acids Research,1997,25 (24): 4876-4882.
[17]SCHNEIDER S,ROESSLI D,EXCOFFIER L.ARLEQUIN,version2000: a software for population genetics data analysis[M].Geneva: University of Geneva,2000.
[18]WRIGHT S.The interpretation of population structure byF2statistics with special regard to systems of mating[J].Evolution,1965,19: 395-420.
[19]NEI M.Evolution of human races at the gene level[M] // BONNE T B,COHEN T,GOODMAN R M,et al.Human Genetics,Part A: The unfolding genome.New York: Alan R Liss,1982: 167-181.
[20]EXCOFFIER L,SMOUSE P E,QUATTRO J M.Analysis of molecular variance inferred from metric distances among DNA heliotypes: application to human mitochondrial DNA restriction data[J].Genetics,1992,131: 479-497.
[21]KUMAR S,TAMURA K,JAKOBSEN I B,et al.MEGA2.0: molecular evolutionary genetics analysis software[M].Tempe: Arizona State University,2001.
[22]韩虎峰,代应贵.珠江水系光唇裂腹鱼可渡河种群mtDNA D-loop序列多态性分析[J].中国水产科学,2010,17(1): 143-149.
[23]李娜,陈少波,谢起浪,等.闽浙地区香鱼线粒体Cytb基因和D-loop区序列多态性分析[J].遗传,2008,30(7): 919-925.
[24]彭士明,施兆鸿,陈超,等.根据mtDNA D-loop序列分析东海银鲳群体遗传多样性[J].海洋科学,2010,34(2): 28-32.
[25]孙鹏,尹飞,彭士明,等.条石鲷线粒体 COⅠ和 Cytb序列的遗传变异分析[J],水产学报,2011,35(5): 327-333.
[26]蒙子宁,杨丽萍,吴丰,等.斜带石斑鱼、赤点石斑鱼RAPD和线粒体Cytb基因序列变异分析[J].中山大学学报:自然科学版,2007,46(1): 75-80.
[27]CONRAD M.Adaptability:The significance of variability from molecular to ecosystem[M].New York: Plenum Press,1983.
[28]FERAL J P.How useful are the genetic markers in attempts to understand and manage marine biodiversity[J].Journal of Experimental Marine Biology and Ecology,2002,268: 121-145.
[29]唐琼英,杨秀平,刘焕章.刺鲃基于线粒体细胞色素b基因的生物地理学过程[J].水生生物学报,2003,27(4): 352-356.
[30]BILLINGTON N,HEBERT P D N.Mitochondrial DNA diversity in fishes and its implications for introductions[J].Canadian Journal of Fisheries and Aquatical Sciences,1991,48(suppl.1): 80-94.
[31]KOCHER T D,WILSON A C.Sequence evolution of mitochondrial DNA in humans and chimpanzees: Control region and a protein-coding region[M] // OSAWA S ,HONJO T.Evolution of life: Fossils,molecules and culture.Tokyo: Springer-Verlag,1991: 391-413.
[32]GERLACH G,MUSOLF K F.Fragmentation of landscape as a cause for genetic subdivision in bank voles[J].Conservation Biology,2000,14: 1066-1074.
[33]TANG Qiongying,LIU Huanzhang,MAYDEN R,et al.Comparison of evolutionary rates in the mitochondrial DNA cytochromebgene and control region and their implications for phylogeny of the Cobitoidea (Teleostei: Cypriniformes)[J].Molecular Phylogenetics and Evolution,2006,39: 347-357.
[34]孙希福,高天翔,窦存银,等.江豚线粒体Cytb和D-loop片段序列分析及其分子系统学研究[J].南方水产,2009,5(5): 24-31.
[35]梁华芳,徐晓鹏,黄志坚,等.中国沿海龙虾属8种龙虾COI基因序列的分子系统学研究[J].中山大学学报:自然科学版,2011,50(6): 94-98.
[36]黄志坚,徐晓鹏,唐晶晶,等.淡水鱼类线粒体DNA D-loop基因的引物设计和应用[J].中山大学学报:自然科学版,2009,48(4): 84-88.
[37]MEYER A.Evolution of mitochondria DNA in fishes[M]//MOMMSEN T P,HOCHACHKA P W.Biochemistry and Molecular Biology of Fishes,Vol.2.New York: Elsevier,1993: 1-38.
[38]周继亮,张亚平,黄美华,等.蝮亚科蛇线粒体细胞色素b基因序列分析及其系统发育[J].动物学报,2001,47(4): 361-366.
[39]FARIAS I P,ORTI G,SAMPAIO I,et al.The cytochromebgene as a phylogenetic marker: the limits of resolution for analyzing relationships among cichlid fishes[J].J Mol Evol,2001,53(2):89-103
[40]项方,邹记兴,邓凤姣,等.用细胞色素b部分序列研究斑马鱼的分子分类与系统发育[J].动物学杂志,2004,39(5): 13-18.

