发酵法生产硫酸软骨素的研究进展
吴秋林,刘立明,陈坚
1 江南大学食品科学与技术国家重点实验室,江苏 无锡 214122
2 江南大学工业生物技术教育部重点实验室,江苏 无锡 214122
3 江南大学食品微生物制造工程实验室,江苏 无锡 214122
硫酸软骨素 (Chondroitin sulfate,CS) 作为糖胺聚糖类大分子酸性多糖,广泛存在于人体软骨、肌腱、椎间盘等结缔组织中,与透明质酸、硫酸角质素及核心蛋白聚合为蛋白聚糖 (主要成分为硫酸软骨素),赋予软骨凝胶样特性和抗变形能力,被称为人体“软黄金”[1]。鉴于其潜在的医药价值,2010年《中华人民共和国药典》将硫酸软骨素列为防治心血管疾病和关节病的最佳药物。同时硫酸软骨素作为膳食补充剂和保湿剂,广泛应用于食品和化妆品领域[2-3]。硫酸软骨素二糖单体结构为 4-GlcA-β-1,3-GalNAc-β-1 (GlcA:葡糖醛酸,GalNAc:乙酰半乳糖胺),由于硫酸化发生位点的不同,而将硫酸软骨素主要分为CS-O, A, B, C, D, E, F等类型 (图1),其中,动物来源的硫酸软骨素单体多为CS-A, C[4]。
硫酸软骨素的工业化生产是采用酶法、碱法等提取工艺,从气管、鼻中隔、鸡龙骨及鲨鱼软骨等动物软组织中提取硫酸软骨素[5]。全世界硫酸软骨素产业主要集中于我国,约占据全球产量的80%,是硫酸软骨素最大生产国和出口国。尽管我国硫酸软骨素产业发展强劲,但仍然面临原料生产周期长 (动物软骨的生产周期较长)、产业水平不高 (工艺繁琐、收率不高、质量不稳)、产品质量缺陷 (采用γ射线杀菌导致辐射残留)、污染严重 (产生大量的有机物和蛋白废水) 等制约产业发展的关键瓶颈。因此,如何发展原料丰富、技术先进、质量优异、清洁环保的生产工艺是促进硫酸软骨素产业发展的关键。除动物软骨组织外,硫酸软骨素还以荚膜多糖的形式存在于某些微生物细胞壁上,因此,通过培养微生物细胞而获得大量的硫酸软骨素是最具经济效益和开发潜力的生产工艺。发酵法生产硫酸软骨素的优点体现在:1) 原料来源广泛,成本低;2) 工艺条件温和;3) 产品质量稳定安全;4) 环境友好、无污染等。
因此,近年来采用微生物发酵法生产硫酸软骨素成为国内外研究的热点。本文就硫酸软骨素的生产菌株、发酵工艺及其合成途径与调控等方面的研究进展进行总结,以促进发酵法生产硫酸软骨素产业的发展。
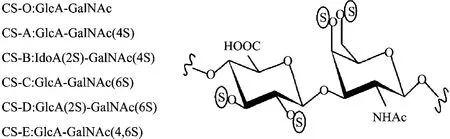
图1 硫酸软骨素的结构[4]Fig. 1 Structure of chondroitin sulfate[4].
1 发酵法生产硫酸软骨素及其类似物
1.1 硫酸软骨素的生产菌株及其改造
自然界中许多真菌和细菌等微生物均能合成低聚合度的硫酸软骨素或其类似物[6]。但作为工业生产菌株需具备:1) 人畜安全;2) 能利用廉价的糖质原料;3) 生长迅速,发酵周期短;4) 遗传背景清楚;5) 能大量合成硫酸软骨素或其类似物等条件。目前研究主要集中在巴斯德杆菌 Pasteurella multocida、大肠杆菌 Escherichia coli和枯草芽胞杆菌Bacillus subtilis等3种微生物 (表1)。
首先,大肠杆菌作为原核模式微生物,其遗传背景清楚,同时对其荚膜多糖合成与转运机制研究较多,是目前硫酸软骨素发酵研究的主要菌株。但E. coli K4发酵产物为K4CPS,即果糖软骨素 (4-β-GlcA(Fru)-1,3-β-D-N-GalNAc-1)[7],需经过脱果糖和硫酸化修饰等步骤才能获得硫酸软骨素[8]。这些修饰步骤不仅增加工艺复杂程度、增加生产成本,还会降低目标产品的最终得率。虽然P. multocida能合成软骨素类荚膜多糖,但其为家禽霍乱致病菌[9-11]。因此近年来主要是利用来源于 P. multocida type F的软骨素合成酶pmCS进行体外酶转化法合成软骨素多糖链,但单糖供体成本太高,尚无产量报道。在2010年,美国Amano Enzyme公司利用纳豆芽胞杆菌Bacillus natto直接发酵生产硫酸软骨素,但产量较低仅为0.24 g/L[6]。作者在筛选获得一株高产硫酸软骨素的枯草芽胞杆菌的基础上[12],通过发酵过程优化使硫酸软骨素产量达到4.2 g/L (32 h),这是目前公开文献报道的硫酸软骨素最高产量。枯草芽胞杆菌作为硫酸软骨素生产菌的优势在于:1) 由于是食品级微生物,安全性很高;2) 直接获得硫酸软骨素,无需硫酸化修饰。因此枯草芽胞杆菌将是发酵法生产硫酸软骨素的最佳菌株。
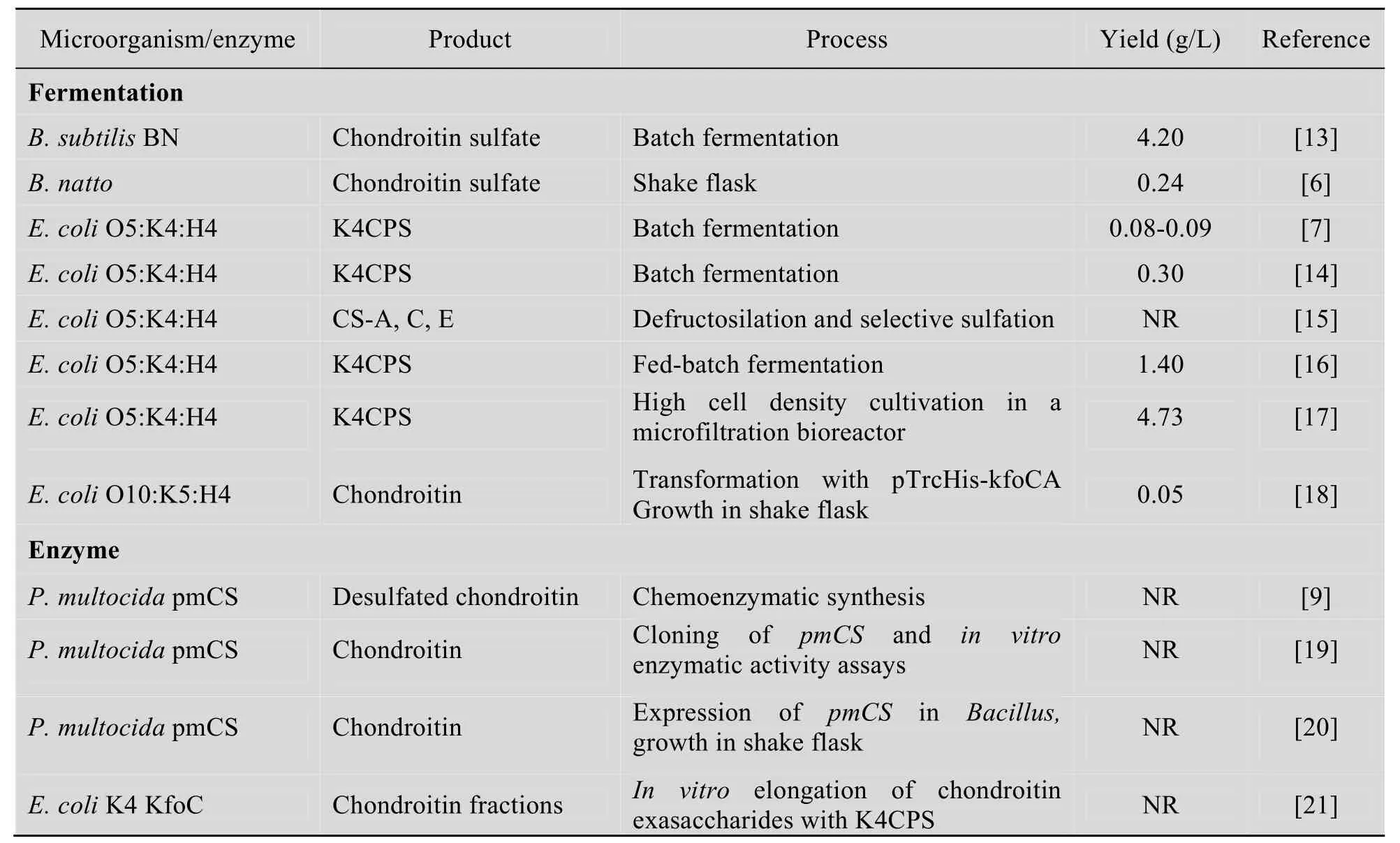
表1 生物技术法生产硫酸软骨素或类似物Table 1 Progress in biotechnological production of chondroitin and chondroitin sulfate
为了进一步提高硫酸软骨素的产量,Zanfardino 以E. coli O5:K4:H4为出发菌株,经过N-甲基-N-硝基-N-亚硝基胍 (NNNG) 随机诱变,以荚膜圈为筛选条件,筛选到1株产量提高80%的突变株 VZ15。进一步解析其高产的生理机制,发现其软骨素合成酶KfoC的第313个密码子发生错义突变 (Arg→Glu),使得 KfoC对UDP-GalNAc的亲和力更强[22]。同时,Cimini在E. coli K4中过量表达kfoC,将K4CPS产量提高了2倍[23]。上述结果表明,软骨素合成酶是影响硫酸软骨素高效合成的关键酶。通过分析荚膜多糖合成途径,发现单糖前体作为多糖聚合酶底物,其含量丰富程度也是影响硫酸软骨素多糖合成效率的关键因素。为此,Roman在大肠杆菌中过量表达UDP-葡萄糖脱氢酶 (UDPGDH),将UDP-GlcA含量提高3倍,不仅没有提高荚膜多糖的产量,反而降低了3倍左右[24]。这一研究结果表明,仅仅增加单个前体物质的含量对多糖合成酶具有竞争性抑制作用[24]。由于硫酸软骨素等荚膜多糖的合成涉及到单糖合成、核糖供应、酶动力学反应以及膜转运蛋白等步骤。如要提高硫酸软骨素的产量,可从转录水平上调节荚膜多糖合成基因簇的表达,如过量表达region 2转录表达的必需因子rfaH基因,显著提高了region 2基因表达水平,使硫酸软骨素的产量提高了58%[25]。此外,由于E. coli K4的野生质粒pK4EC05排斥外源质粒,导致重组质粒容易丢失,研究发现在发酵罐培养24 h后重组质粒全部丢失,导致重组菌难以扩大培养[23]。因此,如何增加目的基因在K4菌株中的稳定性成为分子改造的首要目标,采用的方法包括:1) 消除内源质粒,解除排斥性;2) 与内源质粒兼容的外源质粒作为载体;3) 以内源质粒为载体。但上述策略的重要基础在于解析质粒pK4EC05特性,为此,Cimini等在完成pK4EC05测序的基础上发现其能够插入外源基因。通过以 pK4EC05为载体在胞内稳定表达外源基因,并对重组菌成功进行了放大培养,最终K4CPS的产量达到9.2 g/L,为目前报道的最高水平[25]。
1.2 发酵条件优化与提取纯化工艺的研究进展
尽管枯草芽胞杆菌是工业生产硫酸软骨素的潜在生产菌株,但由于研究较晚,尚处于初级阶段。而早在1988年就发现E. coli O10:K4:H4荚膜多糖结构为果糖软骨素,1996年利用K4菌株进行发酵研究,采用Rodriguez的发酵培养基,在14 L发酵罐上培养24 h后,硫酸软骨素的产量为 300 mg/L[14]。这一研究引发了发酵法生产硫酸软骨素的研究热潮。相关研究主要集中在通过优化发酵条件和改造菌株以提高菌体浓度、硫酸软骨素产量和生产强度。
1.2.1 发酵条件
1) 碳源:荚膜多糖合成与细胞生长在营养物质和能量方面存在着竞争关系,因此碳源作为调节细胞生长速率的关键营养元素,其选择至关重要。Cimini等比较了葡萄糖和甘油对E. coli K4产荚膜多糖和生长的影响,两者浓度的增加均能提高荚膜多糖的产量,但甘油更佳[16]。分析发现当氮源不足时,葡萄糖浓度的增加会促进发酵液中乙酸等副产物的产生,抑制荚膜多糖的合成。而吸收率较低的甘油能降低糖酵解途径中的代谢流,减少乙酸等副产物的产生,从而其浓度的增加更能有效促进荚膜多糖的合成。
2) 氧气:荚膜多糖是组成细胞壁的重要成分,其高效合成依赖于胞内能量状态。高溶氧将显著增加胞内能量的供应,促进多糖的合成。研究发现,当DO值低于10%时,硫酸软骨素比合成速率急剧下降,而甘油消耗速率加快,同时在发酵液中积聚大量有机酸等副产物[16]。
3) 发酵工艺:荚膜多糖作为细胞表面成分,当微生物进入指数生长期时开始合成硫酸软骨素,在稳定期后产量达到最大值。Cimini在高溶氧条件下,通过补充碳氮源使细胞浓度达到56 g/L,从而使荚膜多糖产量达到1.4 g/L (比分批发酵提高5倍)[16]。但由于副产物的积累,导致细胞得率和转化率较低。为此,Schiraldi采用新型的微膜生物反应器截留微生物细胞而排除低分子量的副产物,以解除细胞在高密度培养过程中的副产物抑制作用,将K4CPS的产量提高到了4.73 g/L,细胞得率达到0.13 gK4CPS/gcdw。采用这一策略,使硫酸软骨素的产量比分批发酵(295 mg/L) 和补料分批发酵 (1.4 g/L) 分别提高了16倍和3.3倍[17]。
1.2.2 硫酸软骨素的提取工艺
从发酵液中提取K4CPS首先经过初提,即除去发酵液中菌体并回收得到初级K4CPS,再经膜超滤去除小分子杂质,乙醇沉淀得到粗多糖;加入蛋白酶降解蛋白质,最后经过透析或膜过滤等可得精制K4CPS[14,25]。纯化所得K4CPS还需经过脱果糖和硫酸化修饰等步骤制成硫酸软骨素。第一步,用乙酸调节pH值至2.8~3.0,100 ℃保温至少1 h,即可脱去果糖[25]。脱果糖后的软骨素可以酸形式或甲酯形式经过约6步的化学处理,最终得到CS-A, C, E[15]。但此法的缺点在于只能得到均一的硫酸软骨素单体,不能在同一多糖链的不同位点硫酸化。为解决这一问题,Bedini等提出了循环保护策略,首先将GlcA的2,3位醇基乙酰化后用循环基团选择性保护GalUAc的4,6位醇基,再在不同条件下循环打开,使每个二糖单体的单个羟基随机解除保护后被硫酸化,即可得到4位和6位随机硫酸化的非均一硫酸软骨素[8]。此法不但产率高,成本低,而且与医药级CS-A, C非常相似,具有重要的应用价值。
2 代谢途径与基因调控
2.1 合成基因簇
根据大肠杆菌荚膜多糖的遗传学特征、生物合成方式及其分子结构等特性,可将其分为4大类,即group 1~4[26]。E. coli K4属于group 2,如图2所示,其荚膜多糖合成基因簇包括3个功能区:region 1、region 2和region 3。
其中region 1和region 3主要负责新合成多糖链的修饰、转运与定位 (功能见表2)[28]。在多糖链合成后,region 1中的KpsS和KpsC对其还原性末端进行Kdo修饰,其中Kdo由CMP-Kdo供给,而CMP-Kdo则由region 1中的KpsF和KpsU参与合成。同时,KdsD和KdsB也具有合成 CMP-Kdo的功能[29-30]。在完成修饰后,由region 3中KpsM和KpsT组成的ABC-A2型转运系统负责对多糖进行转运。其中KpsM为膜整合蛋白,包括6个跨膜结构区;而KpsT则含有一个ATP结合区域并具有ATP水解酶活性[31-32],在转运过程中,KpsT通过识别多糖链还原性末端的phosphatidyl-Kdo,与多糖链相互作用。
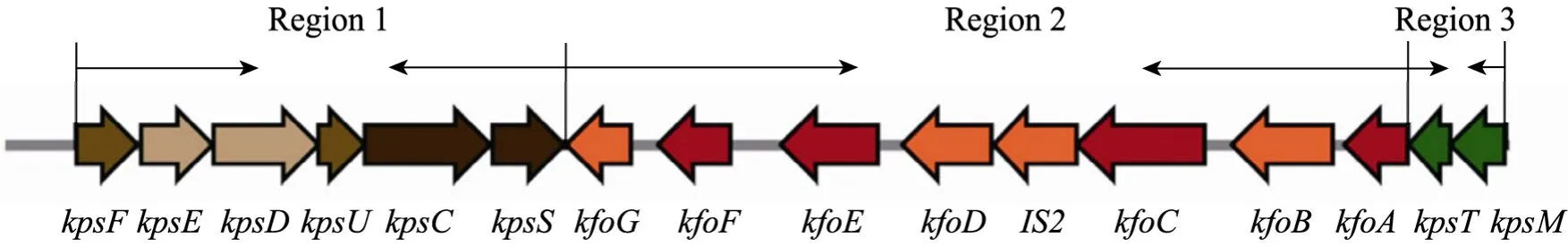
图2 K4荚膜多糖合成基因簇[27]Fig. 2 The gene cluster for K4CPS biosynthesis [27].

表2 与K4CPS合成相关的蛋白[26,34]Table 2 Proteins involved in the biosynthesis of K4CPS[26,34]
Region 2主要负责编码多糖及其前体合成相关的酶,多糖结构与基因型有关。E. coli K4 region 2长达14 kb (图2),包括kfoA~G等7个基因和1个插入序列IS2[27]。其中KfoA和KfoF分别参与了前体物质UDP-GalNAc与UDP-GlcA的合成。kfoC是编码具有双功能糖基转移酶活性的软骨素合成酶的基因,该酶N端和C端的糖基转移酶活性位点分别与前体 UDP-GlcA和UDP-GalNAc相结合[33],并交替转移到软骨素多糖链的非还原末端,从而延伸软骨素多糖链[34]。与软骨素比较,K4CPS带有1个果糖残基,其添加机制引起了研究者的兴趣。早期预测可能由含有糖基转移酶活性位点的 KfoG完成,但Krahulec通过同源重组技术将kfoG基因敲除后,K4CPS结构并没有发生预期的改变,从而否定了这一推测[35]。直到2012年,Trilli才发现KfoE蛋白为果糖转移酶,并敲除kfoE基因得到生产软骨素的E. coli (△ kfoE)菌株[36]。另外,在kfoC和kfoD两基因之间有1个长度为1 331 bp的插入序列IS2,是一个转运DNA元件,与转座酶同源性 96%,表明部分基因由其他地方转移过来[27]。
2.2 生物合成途径
K4CPS在细胞质中合成,并由一系列的转运蛋白将其转送至细胞膜表面,最终在细胞周围形成一圈黏液层。其合成过程可分为:1) 单糖前体的合成;2) 多糖的聚合与输出等两个阶段。
2.2.1 单糖前体的合成
以葡萄糖为碳源时,前体物质UDP-GlcA和UDP-GalNAc的合成途径如图3所示。将葡萄糖从胞外转运到细胞内是两前体合成的第一步,通过PEP-PST转运系统、ABC转运子和质子泵等方式完成。PEP-PST系统作为E. coli吸收葡萄糖的主要方式,在完成葡萄糖转运的同时实现了葡萄糖的磷酸化。随后以葡萄糖-6-磷酸 (Glc-6-P)为起点,通过两条代谢途径而合成UDP-GlcA和UDP-GalNAc。
1) UDP-GlcA的合成:Glc-6-P通过变位酶(Phosphoglucomutase) 转化为葡萄糖-1-磷酸(Glc-1-P),再由尿苷酰二磷酸葡萄糖焦磷酸化酶(UTP-glucose-1-phophate uridylyltransferase) 催化Glc-1-P与UTP合成UDP-Glc。UDP-Glc在UDP-葡萄糖脱氢酶 (UDP-glucose dehydrogenase) 催化下氧化脱氢形成UDP-GlcA。
2) UDP-GalNAc的合成:首先,Glc-6-P由葡糖磷酸异构酶 (Phosphogluco isomerase) 异构为 Fru-6-P,再通过转氨酶 (Glutamine-fructose-6-phophate transaminase) 从谷氨酰胺得到氨基,合成氨基糖GlcN-6-P,磷酸基团同样通过变位酶生成 GlcN-1-P。然后通过乙酰基转移酶(Glucosamine-1-phosphate N-acetyltransferase) 从乙酰辅酶 A (Acetyl-CoA) 转移乙酰基到GlcN-1-P合成GlcNAc-1-P。GlcNAc-1-P在UDP-乙酰葡糖氨二磷酸酶 (UDP-N-acetylglucosamine diphosphorylase) 催化下合成核苷糖UDP-GlcNAc。最后由差向异构酶 (UDP-glucose-4-epimerase)异构为前体UDP-GalNAc。通过上述生化途径解析发现,在单糖前体合成过程中,变位酶和转氨酶是荚膜多糖合成途径与糖酵解途径分流的第一个酶,其酶活直接决定荚膜多糖合成途径代谢通量的大小,是调节细胞生长与多糖合成的关键酶。此外脱氢酶 (KfoF) 和异构酶 (KfoA)是合成两前体的最后一个酶,也是控制前体合成的关键步骤,其过量表达能有效增加前体的合成量[24]。
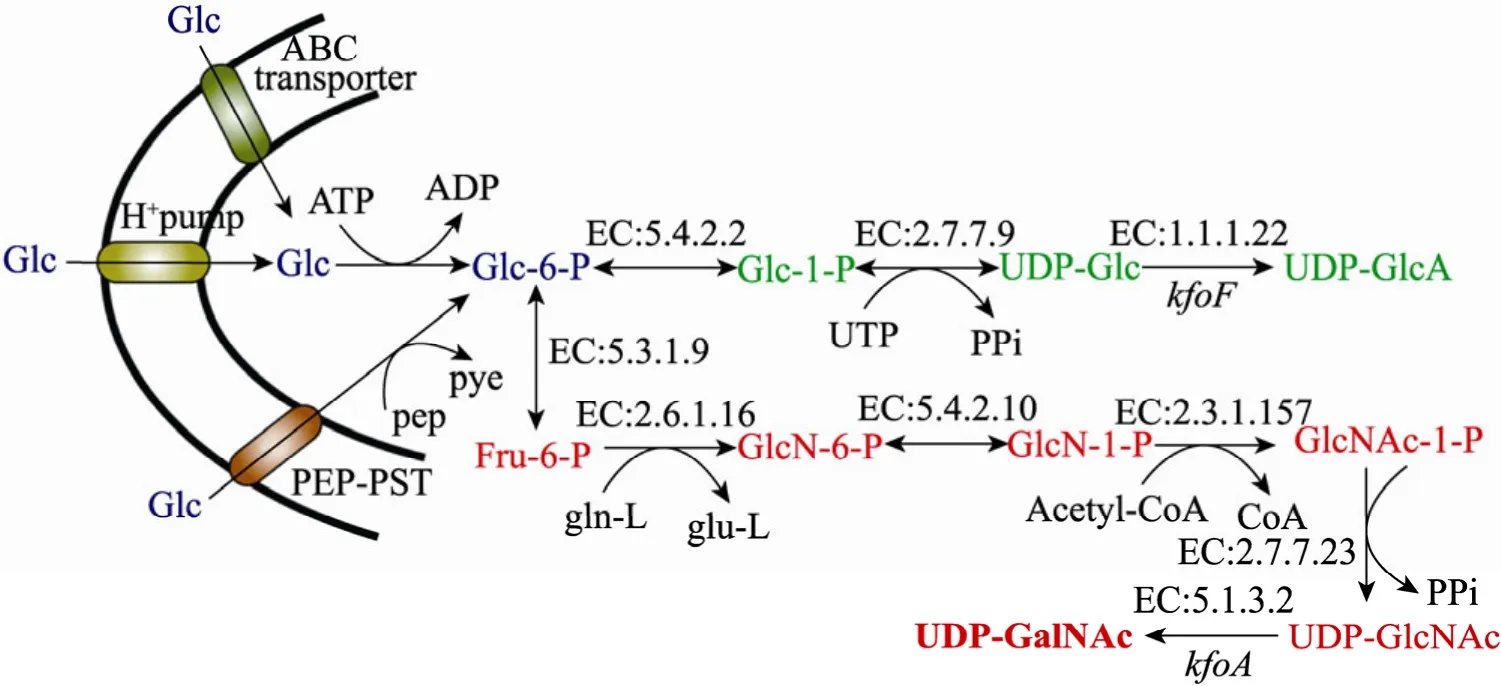
图3 K4荚膜多糖前体合成途径Fig. 3 Biosynthesis pathway for precursor of K4CPS in E. coli K4.
2.2.2 多糖的聚合与输出
多糖的聚合与输出紧密相连,由位于细胞两极的大型复合蛋白完成,这种复合结构有利于聚合形成300 kDa以上的多糖链并迅速穿过细胞内外膜运输至胞外[26,37]。这一过程包括 A~D 4步(图 4)。A:胞质中的软骨素合成酶 (KfoC) 将UDP-GlcA和UDP-GalNAc交替转移至软骨素多糖链的非还原性末端,延伸多糖并释放出UDP[27]。同时UDP亦能与软骨素合成酶结合,推测UDP的结合能影响合成酶的活性,参与多糖合成的调节[38]。多糖链形成后再由果糖转移酶(KfoE) 在GlcA的C3位添加果糖残基,形成完整的K4CPS多糖链[36]。B:在多糖链聚合的同时,KpsF催化5-磷酸核糖 (Ru5P) 异构为5-磷酸阿拉伯糖 (A5P)[39],再由KpsU蛋白合成Kdo的前体 CMP-Kdo[40]。Kdo为多糖链输出细胞膜的介质,由KpsS和KpsC负责在新合成多糖链的还原性末端连接上 phosphatidyl-Kdo,起始多糖的输出[37]。C:新合成的多糖链在转运过程中并非其特异性糖链结构被转运蛋白所识别,而是通过多糖链还原性末端的磷脂介质 Kdo与转运蛋白KpsT相互作用。而KpsT在ATP结合下发生构象变化并插入细胞内膜,同时KpsM在其周围形成孔通道。随后,ATP水解,释放出能量驱动KpsMT转运子将多糖链转移到周质空间。D:存在于周质空间的KpsE,N端与细胞膜外膜连接,C端与细胞膜内膜相连,协助多糖链进入 KpsD孔通道穿过细胞膜外膜,并帮助多糖定位到细胞膜表面[30,41]。荚膜多糖的合成是在一系列合成蛋白与转运蛋白的协同作用下完成的。过量表达单个蛋白难以提高荚膜多糖的产量,如 Bliss和Silver过量表达KpsD蛋白,在细胞外膜形成大量的孔道,打乱了细胞的渗透平衡,最终导致细胞死亡[42]。因此荚膜多糖合成基因簇的协调表达对荚膜多糖的合成至关重要。
2.3 合成基因簇的转录调控
大量研究表明,Group 2荚膜多糖的合成取决于微生物细胞生长的环境温度,当环境温度低于20 ℃时难以合成荚膜多糖,产生这一结果的机理在于荚膜多糖合成基因簇受到两个温度调控型启动子 (σ70启动子) 的调控 (图5)。Region 1启动子 (PR1) 和Region 3启动子 (PR3),分别位于Region 1上游225 bp和Region 3上游741 bp。PR1转录产生一条约为8 kb的多顺反子,随后相继转录出一条约1.3 kb的kpsS转录子[43]。在PR3转录从 region 3延伸至 region 2过程中,需要RfaH蛋白参与。RfaH与region 3上游30 bp处的ops序列结合,在转录过程中与RNA聚合酶复合物相互作用,调节region 2转录的正常进行。ops序列缺失或 rfaH基因突变均不能产生荚膜多糖[44]。
荚膜多糖合成基因簇的转录受到多种蛋白的调节,主要有H-NS与SlyA。其中,H-NS作为一种整体调节蛋白,具有负调控作用。其含量非常丰富,可达105个分子/基因组,能影响E. coli胞内约5%的基因表达[45]。而SlyA作为一种反抑制子,可与 H-NS同时结合于 DNA并抑制H-NS的结合,但其表达受到温度的控制[45]。因此,可通过温度调节H-NS/SlyA的比例对启动子进行低温抑制或高温激活。如图5所示,在低温条件下,胞内H-NS/SlyA比值较高,浓度很低的SlyA无法抑制H-NS与PR1、PR3结合,从而阻碍基因簇的转录。反之,在高温条件下,随着SlyA表达增多,与PR1、PR3的结合逐步增强,能解除H-NS与DNA的结合并激活基因簇的转录。此外,过量的SlyA蛋白将与H-NS形成核蛋白复合物结合于基因簇上,进一步促进其转录。因此,PR1、PR3的转录需要SlyA蛋白的高水平表达,同时其最大化转录需要 H-NS的参与[46]。

图4 K4荚膜多糖的合成与转运[41]Fig. 4 Biosynthesis and transportation of the K4 capsular polysaccharide[41]. (A) Synthesis of K4CPS occurs in the cytoplasmic. (B) KpsF and KpsU convert ribulose 5-phosphate (Ru5p) to CMP-Kdo, which is transferred to the reducing terminus of the finished polysaccharide by KpsS and KpsC. (C) Transport across the cytoplasmic membrane is achieved by KpsM and KpsT. (D) Transport to the cell surface requires KpsE and KpsD.
除上述主要调控蛋白外,还包括一些辅助调控因子,如整合宿主因子 (Integration host factor,IHF) 可发挥结构辅助作用,帮助DNA折叠以使核蛋白复合物结合到DNA;BipA蛋白通过与核糖体相互作用调节某种未知蛋白X的活性,X蛋白再调节PR1和PR3的转录[47]。同时在基因簇中还存在调节序列。首先,PR1中含有两个顺式作用元件 (Cis-acting elements) ——conA和conB,其缺失导致37 ℃时的转录活性降低,而在 20 ℃的条件下无影响。说明还有其他蛋白与元件结合参与37 ℃时的转录激活。其次,kpsM 基因起始密码子 5¢端非转录区 (741 bp) (Untranslated region,UTR) 会减弱下游基因的转录反应,但UTR并不含有任何顺式作用元件。141~219 bp是介导PR3温度调节的主要区域,缺失可解除20 ℃的抑制作用,而整个741 bp片段的缺失将极大提高PR3的转录活性[48]。

图5 E. coli K4荚膜多糖基因簇的表达与转录调控Fig. 5 Expression and regulation of capsular gene cluster in E. coli K4.
3 结论与展望
围绕发酵法生产硫酸软骨素这一研究主题,国内外研究人员已在高产微生物菌株筛选与改良、发酵条件优化与控制、硫酸软骨素合成机制与调控机理等方面开展了卓有成效的研究工作,为发酵法生产硫酸软骨素的工业化奠定了坚实的基础。然而,发酵法生产硫酸软骨素工业化进程的关键瓶颈在于较低硫酸软骨素的产量、产率和生产强度,其原因在于微生物自身代谢的经济学本能和工业生产环境与自然环境的巨大差异。因此,如何利用基因工程手段获得高产率的生产菌株和通过发酵优化实现硫酸软骨素的高效生产是未来的研究热点和重点,包括:1) 利用代谢工程手段强化硫酸软骨素的合成与转运途径;2) 从能量代谢途径和非牛顿假塑性流体氧气传递效率提高胞内能荷水平满足硫酸软骨素合成的高能量需求;3) 研究高密度培养细胞与高强度产物合成的两阶段发酵策略,实现硫酸软骨素发酵法生产的高产量、高产率和高生产强度的统一,为工业化生产奠定基础。
REFERENCES
[1] Qu ZH Wang XZ, Ding MX. Cell Biology. 3rd ed. Beijing: Higher Education Press, 2008: 528−529.翟中和, 王喜忠, 丁明孝. 细胞生物学. 3版. 北京: 高等教育出版社,2008: 528−529.
[2] Zhou S. Health food useful for improving water content of skin, obtained by mixing components including hyaluronic acid, chondroitin sulfate, fish collagen, vitamin C powder, glycine, proline, extract of grape seeds and starch: CN, 102210425A, 2011-10-12.
[3] Wada T, Mano T, Tanouchi M. Composition useful in pharmaceuticals and food-drinks for treating asthenopia, comprises mixture of chondroitin sulfate: WO, 2011136159A1, 2011-03-11.
[4] Schiraldi C, Cimini D, DeRosa M. Production of chondroitin sulfate and chondroitin. Appl Microbiol Biotechnol, 2010, 87(4): 1209−1220.
[5] Lauder R. Chondroitin sulphate: a complex molecule with potential impacts on a wide range of biological systems. Complement Ther Med, 2009, 17(1): 56−62.
[6] Jolly JF, Klimaszewski K, Nakanishi Y, et al. Microbial-derived chondroitin sulfate: US, 20100063001A1, 2009-06-03.
[7] Rrodriguez ML, Jann B, Jann K. Structure and serological characteristics of the capsular K4 antigen of Escherichia coli O5: K4: H4, a fructose-containing polysaccharide with a chondroitin backbone. Eur J Biochem, 1988, 177(1): 117−124.
[8] Bedini E, DeCastro C, DeRosa M, et al. A microbiological-chemical strategy to produce chondroitin Sulfate A,C. Angew Chem Int Ed Engl, 2011, 50(27): 6160−6163.
[9] DeAngelis PL, Gunay NS, Toida T, et al. Identification of the capsular polysaccharides of Type D and F Pasteurella multocida as unmodified heparin and chondroitin, respectively. Carbohydr Res, 2002, 337(17): 1547−1552.
[10] Chong BF, Blank LM, Mclaughlin R, et al. Microbial hyaluronic acid production. Appl Microbiol Biotechnol, 2005, 66(4): 341−351.
[11] Vann WF, Schmidt MA, Jann B, et al. The structure of the capsular polysaccharide (K5 antigen) of urinary-tract-infective Escherichia coli 010:K5:H4. A polymer similar to desulfo-heparin. Eur J Biochem, 1981, 116(2): 359−364.
[12] Liu LM, Wu QL, Liu J, et al. A screening method of chondroitin sulfate production strain and fermentation chondroitin sulfate: CN, 201110127831.1, 2011-05-18.刘立明, 吴秋林, 刘佳, 等. 一种产硫酸软骨素菌株的筛选方法及用该菌株发酵生产硫酸软骨素: CN, 201110127831.1, 2011-05-18.
[13] Liu LM Liu J, Wu QL. A method to improve the productivity of chondroitin sulfate by fermentation: CN, 201110317393.5, 2011-10-21.刘立明, 刘佳, 吴秋林. 一种提高硫酸软骨素发酵强度的生产方法: CN, 201110317393.5, 2011-10-21.
[14] Manzoni M, Bergomi S, Molinari F, et al. Production and purification of an extracellularly produced K4 polysaccharide from Escherichia coli. Biotechnol Lett, 1996, 18(4): 383−386.
[15] Zoppetti G, Oreste. Process for the preparation of chondroitin sulfates from K4 polysaccharede and obtained products: US, 6777398B2, 2002-10-21.
[16] Cimini D, Restaino OF, Catapano A, et al. Production of capsular polysaccharide from Escherichia coli K4 for biotechnological applications. Appl Microbiol Biotechnol, 2010, 85(6): 1779−1788.
[17] Schiraldi C, Restaino OF, Cimini D, et al. High cell density cultivation of Escherichia coli K4 in a microfiltration bioreactor: a step towards improvement of chondroitin precursor production. Microb Cell Fact, 2011, 10(1): 10−19.
[18] Suzuki K, Miyamoto K, Kaseyama H. Chondroitin-producing bacterium and method of producing chondroitin: US, 20100151532A1, 2008-06-24.
[19] DeAngelis PL. Chondroitin synthase gene and methods of making and using same: US, 7569386B2, 2005-06-24.
[20] DeAngelis PL, Padgett-McCue AJ. Methods of expressing Gram-negative glycosaminoglycan synthase genes in Gram-positive hosts: US, 7642071B2, 2007-02-01.
[21] Sugiugura N, Koji J. Novel process for preparation of chondroitin fraction: US, 20090155851, 2006-08-23.
[22] Zanfardino A, Restaino OF, Notomista E, et al. Isolation of an Escherichia coli K4 kfoC mutant over-producing capsular chondroitin. Microb Cell Fact, 2010, 9(1): 34−41.
[23] Cimini D, DeRosa M, Viggiani A, et al. Improved fructosylated chondroitin production by kfoC overexpression in E. coli K4. J Biotechnol, 2010, 150(3): 324−331.
[24] Roman E, Roberts IS, Lidholt K, et al. Overexpression of UDP-glucose dehydrogenase in Escherichia coli results in decreased biosynthesis of K5 polysaccharide. Biochem J, 2003, 374(3): 767−772.
[25] DeRosa M, Schiraldi C, Cimini D. Biotechnological production of chondroitin: WO, 2010136435A1, 2009-05-25.
[26] Whitfield C, Roberts IS. Structure, assembly and regulation of expression of capsules in Escherichia coli. Mol Microbiol, 1999, 31(5): 1307−1319.
[27] Ninomiya T, Sugiura N, Tawada A, et al. Molecular cloning and characterization of chondroitin polymerase from Escherichia coli strain K4. J Biol Chem, 2002, 277(24): 21567−21575.
[28] Roberts IS, Mountford R, Hodge R, et al. Common organization of gene clusters for production of different capsular polysaccharides (K antigens) in Escherichia coli. J Bacteriol, 1988, 170(3): 1305−1310.
[29] Bronner D, Sieberth V, Pazzani C, et al. Expression of the capsular K5 polysaccharide of Escherichia coli: biochemical and electron microscopic analyses of mutants with defects in region 1 of the K5 gene cluster. J Bacteriol, 1993, 175(18): 5984−5992.
[30] Meredith TC, Woodard RW. Characterization of Escherichia coli D-arabinose 5-phosphate isomerase encoded by kpsF: implications for group 2 capsule biosynthesis. Biochem J, 2006, 395(2): 427−432.
[31] Pavelka MS, Wright LF, Silver RP. Identification of two genes, kpsM and kpsT, in region 3 of the polysialic acid gene cluster of Escherichia coli K1. J Bacteriol, 1991, 173(15): 4603−4610.
[32] Nsahlai CJ, Silver RP. Purification and characterization of KpsT, the ATP-binding component of the ABC-capsule exporter of Escherichia coli K1. FEMS Microbiol Lett, 2003, 224(1): 113−118.
[33] Sobhany M, Kakuta Y, Sugiura N, et al. The chondroitin polymerase K4CP and the molecular mechanism of selective bindings of donor substrates to two active sites. J Biol Chem, 2008, 283(47): 32328−32333.
[34] Lidholt K, Fjelstad M. Biosynthesis of the Escherichia coli K4 capsule polysaccharide. A parallel system for studies of glycosyltransferases in chondroitin formation. J Biol Chem, 1997, 272(5): 2682−2687.
[35] Krahulec J, Krahulcová J, Medová M, et al. Influence of KfoG on capsular polysaccharide structure in Escherichia coli K4 strain. Mol Biotechnol, 2005, 30(2): 129−134.
[36] Trilli A, Busiello I, Daly S, et al. Biotechnological production of chondroitin: US, 20120010399A1, 2011-06-24.
[37] Rigg GP, Barrett B, Roberts IS. The localization of KpsC, S and T, and KfiA, C and D proteins involved in the biosynthesis of the Escherichia coli K5 capsular polysaccharide: evidence for a membrane-bound complex. Microbiology, 1998, 144(10): 2905−2914.
[38] Osawa T, Sugiura N, Shimada H, et al. Crystal structure of chondroitin polymerase from Escherichia coli K4. Biochem Biophys Res Commun, 2009, 378(1): 10−14.
[39] Tzeng YL, Datta A, Strole C, et al. KpsF is the arabinose-5-phosphate isomerase required for 3-deoxy-D-manno-octulosonic acid biosynthesis and for both lipooligosaccharide assembly and capsular polysaccharide expression in Neisseria meningitidis. J Biol Chem, 2002, 277(27): 24103−24113.
[40] Rosenow C, Esumeh F, Roberts IS, et al. Characterization and localization of the KpsE protein of Escherichia coli K5, which is involved in polysaccharide export. J Bacteriol, 1995, 177(5): 1137−1143.
[41] Corbett D, Roberts IS. Capsular polysaccharides in Escherichia coli. Adv Appl Microbiol, 2008, 65:1−26.
[42] Bliss JM, Garon CF, Silver RP. Polysialic acid export in Escherichia coli K1: the role of KpsT, the ATP-binding component of an ABC transporter, in chain translocation. Glycobiology, 1996, 6(4): 445−452.
[43] Simpson DA, Hammarton TC, Roberts IS. Transcriptional organization and regulation of expression of region 1 of the Escherichia coli K5 capsule gene cluster. J Bacteriol, 1996, 178(22): 6466−6474.
[44] Stevens MP, Clarke BR, Roberts IS. Regulation of the Escherichia coli K5 capsule gene cluster by transcription antitermination. Mol Microbiol, 1997, 24(5): 1001−1012.
[45] McVicker G, Sun L, Sohanpal BK, et al. SlyA protein activates fimB gene expression and type 1 fimbriation in Escherichia coli K-12. J Biol Chem, 2011, 286(37): 32026−32035.
[46] Corbett D, Bennett HJ, Askar H, et al. SlyA and H-NS regulate transcription of the Escherichia coli K5 capsule gene cluster, and expression of slyA in Escherichia coli is temperature-dependent, positively autoregulated, and independent of H-NS. J Biol Chem, 2007, 282(46): 33326−33335.
[47] Rowe S, Hodson N, Griffiths G, et al. Regulation of the Escherichia coli K5 capsule gene cluster: evidence for the roles of H-NS, BipA, and integration host factor in regulation of group 2 capsule gene clusters in pathogenic E. coli. J Bacteriol, 2000, 182(10): 2741−2745.
[48] Xue P, Corbett D, Goldrick M, et al. Regulation of expression of the region 3 promoter of the Escherichia coli K5 capsule gene cluster involves H-NS, SlyA, and a large 5¢ untranslated region. J Bacteriol, 2009, 191(6): 1838−1846.

