多基因表达计数技术及其生物医学应用
陈阳,何琪杨
许多慢性病如心脑血管疾病、糖尿病、肿瘤或是慢性传染病如艾滋病、病毒性肝炎均涉及多种基因的相互作用,检测疾病和药物治疗过程中大量基因的表达变化,将为疾病的诊断和疗效评价提供可靠的分子标志物。除了共性变化之外,疾病的发生和治疗还具有明显的个体差异,发展整合个体组学鉴定可能是揭示个体差异的重要途径,斯坦福大学医学院的 Michael Snyder 教授课题组首次报道了此方面的研究成果。使用基因芯片、蛋白质组学和代谢组学的方法,历时两年半跟踪监测了一名志愿者体内 4 万余种基因以及蛋白质水平的分子变化,比临床其他指标更早地发现了该志愿者已罹患糖尿病[1]。虽然基因表达的数据在疾病诊断和治疗中起重要的作用,但由于不能对细胞内基因表达的数量进行准确定量,根据对照组样本得到的相对量,不同实验或不同实验室均存在巨大的差异。可比性差是现在基因表达研究中存在的最大障碍,限制了基因表达评价指标的临床应用。近年来,美国 NanoString 公司研发的多基因表达计数系统(nCounter assay system,NAS)有可能克服上述难题。
NAS 技术诞生于 Leroy Hood 博士创立的系统生物学研究所。管理该研究所基因芯片实验室的 Krassen Dimitrov首先提出了分子条形码的概念,并进行了原理验证。2008年,Nature Biotechnology 杂志上以封面故事的形式报道了他们的研究成果[2]。该技术可以直接定量同一个样本中的800 种基因的表达量,无需 RNA 提取、逆转录以及 PCR扩增步骤,显著提高了表达谱分析的准确度与灵敏度。此项革命性技术自 2008 年出现以来,已经被越来越多的研究者所使用。
1 NAS 技术的原理及优点
NAS 技术利用分子条形码及双探针标志,对转录本进行特异性标定;通过影像化的数字计数准确量化样本的表达量,实现高通量、自动化地直接测定多种基因的表达,在确保实验准确性的同时,最大程度上减少了实验操作可能产生的误差。
1.1 NAS 技术的基本原理
NAS 技术的基本原理是使用了两种探针——捕获探针(capture probe)和报告探针(reporter probe)进行标定,它们各包含了一段长为 35 ~ 50 bp,能特异性地结合到同一种 mRNA 的序列,共同标记目标基因(图 1)。捕获探针、报告探针以及目标基因产物序列特异性结合后,形成探针/目标分子荧光复合物,一个复合物即代表一条 mRNA 分子,不同的基因序列以不同的荧光分子排列组合代表[2]。
报告探针是 NAS 的核心技术,其作用是显示特定的基因。它由一段特异性结合序列、一段线性化的 M13 噬菌体DNA 和 4 组 15 bp 长的重复序列(5'-repeat)组成。线性化的 M13 噬菌体 DNA 上标记了 4 色 7 位的荧光编码(分子条形码),所选择的荧光染料分别为 Alexa488、Alexa594、Alexa647 和 Cy3,每一个荧光编码代表一种特定的基因。理论上,7 位的 4 色荧光最多可以编码 16384(47)个基因。捕获探针的作用是固定杂交后的复合物,除包含特异结合序列外,还有 2 组 15 bp 长的重复序列(3'-repeat),该序列的末端连接有生物素(biotin),可以与包被链亲和素的样品处理板(sample cartridge)结合。
在设计探针时,首先按照引物设计的一般原则为每一个目标基因选择一段 100 bp 左右长度的互补序列,再将其分为两个 50 bp 的序列,两个序列都要满足与非特异扩增序列的同源性应小于 85%,或少于连续 15 个的互补碱基时,才能够作为捕获探针和报告探针的特异性结合序列。科研人员已经建立了人和小鼠的基于目标基因、互补序列、分子条形码的数据库,并根据不同研究的需要,提供相关基因的检测试剂盒。
1.2 NAS 的实验流程
NAS 的系统组成,包括样品制备工作站(Prep Station)与数据分析仪(Digital Analyzer)两个部分。其实验流程包括杂交、纯化固定、计数 3 个步骤[3]。
在实验过程中,将提供的缓冲液、探针以及样品混合,杂交过夜后,将样品送进样品制备工作站,工作站自动完成后续的工作。游离未杂交的报告探针和捕获探针经过两次磁珠筛选后去除,探针/目标分子复合物通过捕获探针的3'-repeat 携带的生物素与包被有链亲和素的样品处理板相连后通电,电流会使探针/目标分子复合物朝同一方向延展。此时,报告探针的 5'-repeat 呈游离散乱状态,会干扰对分子条形码的读数。加入与 5'-repeat 互补的带有生物素标记的寡核苷酸片段溶液,生物素与样品板相连,5'-repeat 端也被固定在样品板上。处理完成的样品板比较稳定,可以储存。将处理好的样品板放入数据分析仪进行信号数据采集,仪器会自动统计在样品板表面的荧光编码探针,并将其列表表示出来。
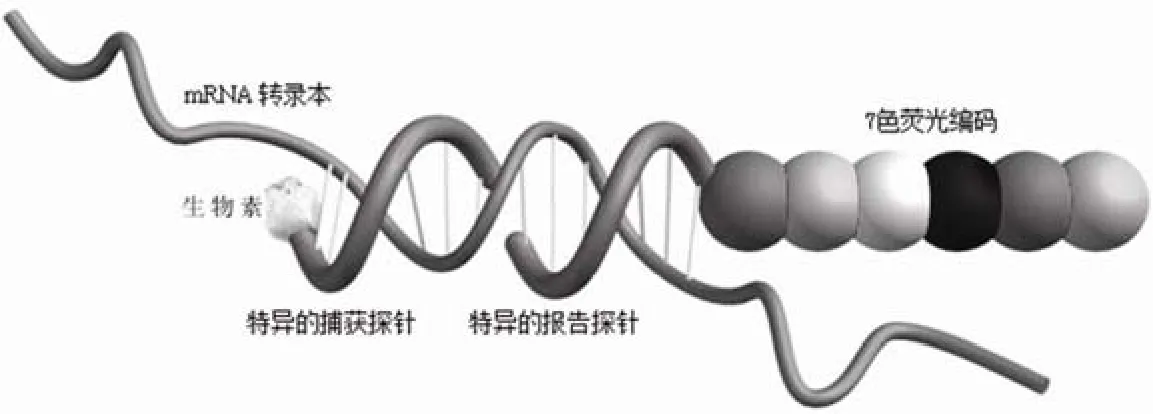
图 1 NAS 检测原理示意图
1.3 NAS 技术的特点
在基因表达检测技术发展过程中,Northern 杂交、RT-PCR 是第一代的技术,主要是检测单基因表达的变化。第二代技术包括基因芯片和检测少量基因表达的实时定量PCR 技术等。这些技术的特点是对基因表达的相对定量,数据必须与相应的对照组比较才能得到,这样导致不同实验之间,不同实验室之间得到的数据差异大。虽然建立了一些统计规则和转换方法,如以管家基因作为数据标准,比较和评价不同实验室得到的基因芯片数据[4],但目前仍缺乏被所有研究者和临床医生所接受的方法和规则。
NAS 属于第三代基因表达检测技术,其检测原理与基因芯片完全不同,以数字方式准确显示样品中的基因表达真实数量,高通量准确检测。在控制仪器检测精度和统一提供试剂盒的情况下,不用数据转换,就能实现对某一疾病关键表达基因的定量比较。基于该技术,将来有可能像目前临床血液的生化数据一样,建立人类相关疾病基因表达量的参考值范围。由于该技术没有 RNA 提取或产物扩增步骤,极大地降低了检测过程中发生差错的几率,减少数据误差。NAS通用性强,能够高度灵敏地检测多种来源的样品:如总RNA、细胞和组织裂解液、从石蜡包埋样品提取的 RNA 以及血液样品等。不过,该技术只能检测已知的基因,配套试剂价格高昂,也有可能限制其推广使用。
目前检测基因表达或全基因组分析的技术层出不穷,如对新 RNA 的 RNA 测序技术,目前就有罗氏公司的 454技术、Illumina 公司 Solexa 技术以及 ABI 公司的 SOLiD技术[5]。除了进行个人基因组检测外,还有寡核苷酸多态性(SNP)检测以及从 10 ~ 20 个细胞中,低成本进行单体型分析的长片段读数技术(long fragment read,LFR)[6]。由于疾病发生的复杂性和检测成本等原因,从临床实用角度看,NAS 技术最有可能在临床推广。
1.4 NAS 技术的可靠性评价
在 2008 年 NAS 技术的首次报道中,检测了人肺癌A549 细胞野生型和骨髓灰质炎(poliovirus,PV)感染型的509 种基因,结果表明,NAS 检测结果重复性高,与样品浓度的相关性 r2达到 0.9988 以上,与 Taqman PCR 技术检测结果的相关性 r2达到 0.945,准确性和检出率均优于基因芯片;检测海胆胚胎相关基因表明 NAS 的灵敏度和准确度与 SYBR Green 实时 PCR 技术相当[2]。此后,不同实验室的研究人员进一步验证了 NAS 技术的可靠性。Malkov 等[7]检测了新鲜冻存(fresh frozen,FF)、石蜡包埋(formalin fixed paraffin embedded,FFPE)以及组织粗提液(crude tissue lysates,CTL)样品中 48 种基因的表达,并与 Taqman PCR 检测结果相比较,证实 NAS 在多基因检测中确实能获得准确、高质量的数据。Reis 等[8]用 19 对口腔上皮癌组织和正常组织为实验材料,分别用 NAS、实时定量 PCR 技术检测了 FF 和 FFPE 样品中 20 种mRNA 的表达,进一步证实在 FFPE 样品 mRNA 质量不佳的情况下,NAS 甚至表现出比实时定量 PCR 更好的准确性。Sun 等[9]也在对乳腺癌细胞相关研究中证实,NAS 技术与 mRNA-seq 技术得到的结果具有很高的相关性。
2 NAS 技术的检测应用
NAS 除了能够检测 mRNA 外,又陆续实现了对miRNA 及 DNA 拷贝数变异的检测分析。miRNA 是一类22 碱基左右的非编码 RNA,能够被整合到沉默复合体(RISC)中,使碱基互补的目标 mRNA 沉默或降解,以实现其调控作用,与生物正常的生长发育以及各种疾病的发生都有着密切的关系[10]。目前的检测手段包括 Northern Blot 技术、芯片技术、基于 PCR 或滚环扩增(rolling circle amplification,RCA)的检测等,这些技术很难同时满足高灵敏度和高通量的需求。NAS 技术能够高通量的检测700 余种人和人-病毒 miRNA、600 余种小鼠 miRNA 和400 余种大鼠 miRNA 并进行图谱分析,而其灵敏度和特异性更与 qPCR 相当。其原理如图 2 所示,一段特殊的桥接寡核苷酸(bridge oligo)分别特异的结合目标 miRNA 和其对应的 miRtag,并使两者共价连接,经过变性,miRNA:miRtag 复合物成为一条单链,这个复合物能够和特定的捕获探针、报告探针结合,并像普通的 mRNA 一样被定量检测[3]。
DNA 拷贝数变异(copy number variations,CNVs)指DNA 片段范围从千碱基到兆碱基规模的缺失、插入、重复和多位点变异等亚微观突变,现有的检测技术包括 Real Time PCR、多重可扩增探针杂交技术(multiplex amplifiable probe hybridization,MAPH)、动态等位基因特异杂交(dynamic allele-specific hybridization,DASH)等,这些技术都不同程度上受到通量和准确性的限制[11]。基于 NAS 技术的 CNVs 分析可以在单个反应中检测最多 800 段 DNA区域,而准确度同样与 qPCR 相当。基因组 DNA 用 AluI限制性内切酶酶切、变性,使之形成单链 DNA,进而通过捕获探针和报告探针对特定 DNA 片段进行计数。利用此技术,研究人员进行了 20 个 CNV 区域的分析,并将其结果与人类基因组单体型图(HapMap)进行对比,准确率达到 94.2%。
3 NAS 技术在生物医学中的应用
由于该技术具有明显的优势,可以预计在生物和医学各领域中将得到广泛的应用。美国许多著名的生物医学研究机构均购置了该仪器,目前利用该仪器得到结果发表的研究论文大量增加。为了启发研究和应用思路,我们在此介绍一些应用的实例。
3.1 在信号通路研究中的应用
在基因表达的研究领域中,研究人员通常采取的策略是使用基因芯片获得高通量的基因表达谱后,再锁定几个基因用准确性更高的实时逆转录 PCR 进一步验证。NAS 技术简化了传统基因表达的研究方法,使基因表达研究更加简单。蛋白合成体(peroxisome)对天然免疫抗病毒机制过程中,线粒体蛋白 MAVS 起重要作用。使用 NAS 比较相关基因表达,同时用 9 个稳定表达基因作为内部对照,更深入地阐明了其作用机制[12]。混合系白血病基因(mixed lineage leukemia,MLL)重排常见于急性淋巴细胞白血病,Orlovsky 等[13]发现通过下调与 MLL 基因关系密切的MEIS1 和 3 个 HOXA 家族相关基因,可以修复 MLL 重排并抑制肿瘤细胞的生长,通过 NAS 检测到这 4 种基因并不会相互影响各自的表达,独立地实现修复重排和抑瘤作用。
利用 NAS 技术和定量质谱测定法,分别对 5000 余种基因的 mRNA 和蛋白质进行量化的检测,通过对 mRNA和蛋白质的丰度及周期变化的分析得出结论:与转录水平相比,翻译水平在调控蛋白质含量上起更加主要的作用[14]。Delmore 等[15]发现通过干扰 BET 蛋白功能,可降低 Myc等重要癌基因的转录,运用 NAS 技术检测 Myc 蛋白下游200 多个靶基因也呈现下调趋势,并最终使细胞周期循环停滞并导致细胞凋亡。
3.2 在表观遗传调节研究中的应用
表观遗传学主要研究与基因序列无关的遗传变化,涉及组蛋白修饰和染色质重塑(chromatin remodeling),其调节机制异常与疾病的发生和治疗密切相关[16]。Ram 等[17]将染色质免疫沉淀技术(chromatin immunoprecipitation,ChIP)和 NAS 技术相结合,称之为“ChIP-string”,监测染色质调控蛋白(chromatin regulators,CRs)与特定染色质区域相互结合的情况。研究人员通过该项技术对 30 种 CRs在染色质上的定位进行了检测,分析表明这些调控因子以 6 种特异性组合的形式与染色质相结合,每个组合内都包括特定激活物和抑制物,共同实现对染色质活性的调控。Guttman等[18]利用 NAS 技术检测分析了与蛋白质结合的 lincRNA,证实 lincRNA 能与特定蛋白质结合形成复合物,调控胚胎肝细胞的基因表达,进而影响肝细胞的多能性和发育。Lebedeva 等[19]在 RNA 结合蛋白 HuR 的研究中,利用NAS 技术及其他技术定位了 HuR 的结合位点在 3' 端非编码区,与 miRNA 结合位点临近,并检测了干扰 HuR 表达会影响许多蛋白在 mRNA 水平表达的变化。
3.3 在临床检测中的应用
多基因表达的检测是临床研究病因、诊断病情、评价治疗效果以及实现个体化用药的必要手段。临床样品量大、目标基因繁多,传统的检测技术会造成巨大的工作量。NAS 技术不仅能够实现多基因检测,且自动化程度高、操作简单,已经逐步被研究人员用于临床研究。Guiducci 等[20]通过NAS 技术分析临床样本相关免疫因子的表达,证实自身核苷酸对 Toll 样受体的识别影响了糖皮质激素治疗系统性红斑狼疮的效果。Lin 等[21]应用 NAS 技术分析临床血液样本,证实 TLR 在抗肿瘤免疫中起重要的作用。McNab 等[22]用 NAS 检测结核患者血液样本中不同细胞程序性死亡配体基因的表达,发现了与正常人相比中性粒细胞过表达程序性死亡配体基因。
3.4 在药物研发和评价中的应用
药物的有效性和安全性问题是临床失败的 2 大主要原因。2011 年 Nature 杂志公布的数据显示,2008 – 2010 年期间美国申请 II 期临床但失败的药物中,51% 的药物是无效的,另外有 19% 的药物存在安全性问题[23]。而在 2007 –2010 年 III 期临床的药物中,以上两种原因导致失败的比率分别达到了 66% 和 21%[24]。由此可见,加强药物的早期评价,在临床实验之前尽可能地排除存在安全性问题和无效的候选药物,不但有利于加速药物研发过程,同时也降低了药物研发的成本。计算机模型可以预测药物可能引起的副作用,研究发现一个新的计算机模型识别出 656 种药物的1241 个可能的副作用位点,其中 348 种在专有数据库中获得确认,而另外 151 种的副作用之前未被发现,并通过进一步的实验被证实[25]。而在药物早期评价的实验中,生物标志物的发现和评价显得尤为重要[26]。在肿瘤研究中,通过对 NCI 60 种肿瘤标准细胞株和 639 种其他细胞系的综合研究,揭示了基因组标志物在药物敏感性中的指标意义[27]。NAS 技术为药物实际研发中多位点的检测提供了便捷的研究手段。去甲基化制剂 zebularine 对肝癌的治疗有着显著差别,Andersen 等[28]定位了 70 个基因以判断是否适合用该药物进行治疗,并利用 NAS 技术检测了 57 个临床患者相关基因的表达,预测临床愈后的准确率达到了84% ~ 96%。
3.5 在干细胞质量评估中的应用
由于 NAS 多基因表达计数技术能准确定量关键基因的表达程度,通过检测干细胞分化基因的表达,可用于确定干细胞的质量。Bock 等[29]对 20 株胚胎干细胞、12 株诱导多能干细胞进行研究,证实细胞系基因组 DNA 甲基化和基因表达存在的微小差异决定了细胞株的分化倾向和分化能力。研究人员依此建立了一个评估多能干细胞有效性的评分系统,使用该技术测定经过非定向分化培养的细胞系中500 余种 mRNA 表达量,从而获知该多能干细胞分化成不同细胞系的倾向。
4 结语
NAS 技术的出现使系统生物学的研究结果更接近于临床应用,为疾病的诊断和治疗提供可靠、实用的分子标记物。但目前 NAS 技术所使用的配套试剂十分昂贵,且必须向NanoString 公司订购,如果在临床检验中使用费用昂贵。根据 NAS 技术的原理,发展类似的检测仪器和整套检测试剂值得国家和相关研究单位重视。
[1] Chen R, Mias GI, Li-Pook-Than J, et al. Personal omics profiling reveals dynamic molecular and medical phenotypes. Cell, 2012,148(6):1293-1307.
[2] Geiss GK, Bumgarner RE, Birditt B, et al. Direct multiplexed measurement of gene expression with color-coded probe pairs. Nat Biotechnol, 2008, 26(3):317-325.
[3] Kulkarni MM. Digital multiplexed gene expression analysis using the NanoString nCounter system. Curr Protoc Mol Biol, 2011, Chapter 25:Unit25B.10.
[4] Lee PD, Sladek R, Greenwood CM, et al. Control genes and variability: absence of ubiquitous reference transcripts in diverse mammalian expression studies. Genome Res, 2002, 12(2):292-297.
[5] Chen G, Wang C, Shi T. Overview of available methods for diverse RNA-Seq data analyses. Sci China Life Sci, 2011, 54(12):1121-1128.[6] Peters BA, Kermani BG, Sparks AB, et al. Accurate whole-genome sequencing and haplotyping from 10 to 20 human cells. Nature, 2012,487(7406):190-195.
[7] Malkov VA, Serikawa KA, Balantac N, et al. Multiplexed measurements of gene signatures in different analytes using the Nanostring nCounter Assay System. BMC Res Notes, 2009, 2:80.
[8] Reis PP, Waldron L, Goswami RS, et al. mRNA transcript quantification in archival samples using multiplexed, color-coded probes. BMC Biotechnol, 2011, 11:46.
[9] Sun Z, Asmann YW, Kalari KR, et al. Integrated analysis of gene expression, CpG island methylation, and gene copy number in breast cancer cells by deep sequencing. PLoS One, 2011, 6(2):e17490.
[10] Bhayani MK, Calin GA, Lai SY. Functional relevance of miRNA sequences in human disease. Mutat Res, 2012, 731(1-2):14-19.
[11] Freeman JL, Perry GH, Feuk L, et al. Copy number variation: new insights in genome diversity. Genome Res, 2006, 16(8):949-961.
[12] Dixit E, Boulant S, Zhang Y, et al. Peroxisomes are signaling platforms for antiviral innate immunity. Cell, 2010, 141(4):668-681.
[13] Orlovsky K, Kalinkovich A, Rozovskaia T, et al. Down-regulation of homeobox genes MEIS1 and HOXA in MLL-rearranged acute leukemia impairs engraftment and reduces proliferation. Proc Natl Acad Sci U S A, 2011, 108(19):7956-7961.
[14] Schwanhäusser B, Busse D, Li N, et al. Global quantification of mammalian gene expression control. Nature, 2011, 473(7347):337-342.
[15] Delmore JE, Issa GC, Lemieux ME, et al. BET bromodomain inhibition as a therapeutic strategy to target c-Myc. Cell, 2011, 146(6):904-917.
[16] Dawson MA, Kouzarides T. Cancer epigenetics: from mechanism to therapy. Cell, 2012, 150(1):12-27.
[17] Ram O, Goren A, Amit I, et al. Combinatorial patterning of chromatin regulators uncovered by genome-wide location analysis in human cells. Cell, 2011, 147(7):1628-1639.
[18] Guttman M, Donaghey J, Carey BW, et al. lincRNAs act in the circuitry controlling pluripotency and differentiation. Nature, 2011,477(7364):295-300.
[19] Lebedeva S, Jens M, Theil K, et al. Transcriptome-wide analysis of regulatory interactions of the RNA-binding protein HuR. Mol Cell,2011, 43(3):340-352.
[20] Guiducci C, Gong M, Xu Z, et al. TLR recognition of self nucleic acids hampers glucocorticoid activity in lupus. Nature, 2010,465(7300):937-941.
[21] Lin Y, Zhang L, Cai AX, et al. Effective posttransplant antitumor immunity is associated with TLR-stimulating nucleic acid-immunoglobulin complexes in humans. J Clin Invest, 2011, 121(4):1574-1584.
[22] McNab FW, Berry MP, Graham CM, et al. Programmed death ligand 1 is over-expressed by neutrophils in the blood of patients with active tuberculosis. Eur J Immunol, 2011, 41(7):1941-1947.
[23] Arrowsmith J. Trial watch: phase II failures: 2008-2010. Nature Rev Drug Discov, 2011, 10 (5):328-329.
[24] Arrowsmith J. Trial watch: phase III and submission failures:2007-2010. Nat Rev Drug Discov, 2011, 10(2):87.
[25] Lounkine E, Keiser MJ, Whitebread S, et al. Large-scale prediction and testing of drug activity on side-effect targets. Nature, 2012,486(7403):361-367.
[26] Littman BH. An NIH National Center for Advancing Translational Sciences: is a focus on drug discovery the best option? Nat Rev Drug Discov, 2011, 10(6):471.
[27] Garnett MJ, Edelman EJ, Heidorn SJ, et al. Systematic identification of genomic markers of drug sensitivity in cancer cells. Nature, 2012,483(7391):570-575.
[28] Andersen JB, Factor VM, Marquardt JU, et al. An integrated genomic and epigenomic approach predicts therapeutic response to zebularine in human liver cancer. Sci Transl Med, 2010, 2(54):54-77.
[29] Bock C, Kiskinis E, Verstappen G, et al. Reference maps of human ES and iPS cell variation enable high-throughput characterization of pluripotent cell lines. Cell, 2011, 144(3):439-452.

