柄海鞘共附生细菌的分离培养与系统发育多样性研究*
苗婷婷,邢 翔,杜宗军,陈冠军
(山东大学 威海分校 海洋学院,山东 威海264209)
柄海鞘共附生细菌的分离培养与系统发育多样性研究*
苗婷婷,邢 翔,杜宗军,陈冠军*
(山东大学 威海分校 海洋学院,山东 威海264209)
通过纯培养和16SrRNA基因序列的相似性比对,并构建系统发育树对威海海域柄海鞘共附生细菌的多样性进行了研究。用2216E培养基从柄海鞘样品中分离到78株细菌,通过形态学特征和TCBS培养基选择性筛选后,选取30株具有代表性的菌株进行了16SrRNA基因测序和基于16SrRNA基因序列的系统发育多样性分析。结果表明,30株菌株分别属于细菌域的4个系统发育类群(Actinobacteria,Bacteroidetes,Firmicutes,Groteobacteria)的11个科,11个属。根据16SrRNA基因序列,大多数菌株与其系统发育关系最密切的模式菌株之间存在一定的遗传差异(16SrRNA基因序列相似性为91.82% ~98.93%)。其中菌株HQW7,HQYD1,HQWD4,HQE1和HQE2与相关模式菌株的16SrRNA基因最高相似度仅分别为91.82%,92.51%,92.99%,93.52%,93.87%,这5株菌可能代表新的分类单元。威海海域柄海鞘共附生细菌存在着较为丰富的物种多样性和系统发育多样性,并可能存在新的物种。
柄海鞘;共附生细菌;16SrRNA基因;系统发育分析;细菌多样性
海洋中的微生物分布非常广泛,有的自由生活在海水中,有的存在于海底沉淀物或海泥的表面,还有一部分与海洋动植物处于共生、共栖、寄生或附生的关系。共附生细菌可能在清除宿主代谢废物[1]和为宿主提供生物活性物质[2-4]等方面扮演着重要角色。由于共附生菌长期与宿主共进化,往往拥有特殊的代谢途径,故很有可能产生结构全新的生理活性物质,为研究与开发微生物制品和微生物药物提供了宝贵的源头线索,因此许多研究者对于利用共附生海洋微生物作为天然生物制品的来源产生相当大的兴趣[5]。从海洋生物分离出的具有重要生物功能的次级代谢产物,也往往是由附生于其上的微生物产生的。在许多情况下,若要得到大量宿主用于生产药物是非常困难的,这也是其商业化生产的障碍。而细菌则不同,大规模的培养就可以保证共生菌稳定的生产量[6]。而且微生物具有生长周期短、代谢易于控制、菌种易于选育等优势,因此共生微生物资源的利用和开发应无来源之忧。因此,从共附生细菌中分离生物活性物质就能够越过这个障碍,免除了从自然环境中收获大量宿主的需要。这样,既保护了环境也降低了商业化生产的成本。
海洋无脊椎动物的体表和体内存在着大量的微生物,它们与海洋无脊椎动物之间存在着高度特异性的共附生关系。目前,海洋无脊椎动物的研究对象主要集中在海绵、海葵、珊瑚和一些肠腔动物。Kennedy等[7]对海葵共附生细菌区系以及具有抗菌活性的细菌进行了研究。Xie等[8]指出从海葵、贻贝等分离到的活性菌株数高于从其他动物中分离的活性菌数。郑立等[9]的研究证明了无脊椎动物体表的共附生细菌对于赤潮藻具有抑制作用。近几年,在海鞘中也发现了许多结构新颖、活性独特的化合物[10-12],引起了许多研究人员的高度重视,已经逐渐成了海洋天然产物化学研究的热点,海鞘也成了除海绵之外人类获取有显著生理活性物质的重要海洋生物资源。由于海洋生物中的许多活性化合物是由其共附生微生物代谢产生的,因此采集海鞘共附生微生物资源,研究其系统发育多样性,将可以为海洋活性化合物开发提供新的源头资料。
本研究报道了采用纯培养法和基于16SrRNA基因序列的系统发育分析对威海近海海域柄海鞘中可培养共附生细菌的系统发育多样性的研究结果,以期为相关微生物资源的研究、保护、开发和利用打下一定的基础。
1 材料与方法
1.1 材 料
1.1.1 主要试剂
PCR引物由生工生物工程(上海)有限公司合成。细菌基因组DNA抽提试剂盒,PCR常规操作用试剂和酶均购自生工生物工程(上海)有限公司。DNA Marker购于宝生物工程(大连)有限公司。
1.1.2 培养基
2216E海洋细菌培养基:蛋白胨5g,酵母粉1g,柠檬酸铁0.01g,琼脂15~18g,陈海水1L,0.1g/L NaOH 调pH 值至7.2~7.6,121℃,20min灭菌。
TCBS培养基:成品TCBS(北京 陆桥)培养基加入0.02g/L的NaCl,煮沸后倒平板。
1.2 样品的采集与菌株的分离
1.2.1 样品采集
柄海鞘(Styela clava)样品于2009-09采自山东威海近海(37°37′14″N,122°01′05″E)。采到的柄海鞘立即保存到装有海水的标本袋里,密封(防止柄海鞘与空气直接接触),迅速带回实验室。在实验室超净工作台中及时处理。
1.2.2 样品处理
将采集的柄海鞘用无菌海水反复冲洗表面后,在超净工作台中解剖,分别取其胃肠消化系统、咽部和生殖腺称重,用无菌海水冲洗后研磨成浆,用无菌海水按照l0-1到10-6的比例进行梯度稀释。取柄海鞘体腔液500μL,用无菌海水按照l0-1到10-6的比例进行梯度稀释。在2216E[12]固体培养基上涂布接种,每个板接种0.1mL,在25℃下培养3~5d。用相同的培养条件分离纯化所得的菌株。
1.2.3 菌株的分离纯化与筛选
挑取平板上的单菌落,在2216E平板上进行划线分离,菌种达到纯培养以后,在-70℃冰箱进行菌种的长期保藏。将纯培养物接种到TCBS培养基上进一步观察其生长状况。
1.3 细菌16SrRNA基因序列测定与系统发育分析
1.3.1 细菌DNA的抽提
按照DNA柱式抽提试剂盒的相关说明操作进行抽提,0.8%的琼脂糖凝胶进行电泳检测。
1.3.2 细菌16SrRNA基因的扩增
利用50ng/μL的DNA作为模板对16SrRNA基因进行扩增,PCR引物为通用引物:正向引物27F:5′-AGAGTTTGATC(C/A)TGGCTCAG-3′;反向引物1492R:5′-TACGG(C/T)TACCTTGTTACGAC-3′。PCR反应体系:模板,1μL;正,反引物各1μL;2×PCR Mix Kit,25μL;Taq酶,1μL;ddH2O,21μL。PCR反应条件:94℃预变性6min;94℃变性45s,55℃复性45s,72℃延伸90s,35个循环;最后72℃温育8 min。PCR产物经电泳检测后直接送至测序公司进行测序或进行克隆后测序。
根据测序结果,运用Blast程序与数据库中已存在的细菌16SrRNA基因序列进行相似性比较分析;序列的比对及系统发育分析采用MEGA(4.0)软件。
1.4 Shannon-winner多样性指数
本研究采用16SrRNA基因序列相似性小于97%的菌株属于不同物种的归类原则[13],采用Shannonwinner指数(H)和均匀度指数(E)估算多样性。

式中,S为菌种数;Pi为第i种的多度比例,Pi=ni/N;n为第i种的菌株数;N为所有菌株数的总和。
2 结果与分析
2.1 菌株的分离
根据菌落大小、形态、颜色等特征,挑取分离平板上的单菌落进行划线分离纯化,用显微观察法检查纯化情况,最终从本次采集的样品中分离到78株纯培养物,其中55株细菌能够在TCBS培养基上生长。TCBS培养基,即硫代硫酸盐柠檬酸盐胆盐蔗糖琼脂培养基,主要用于弧菌科细菌的分离培养。能够在TCBS培养基上生长说明本次试验中的55株细菌属于弧菌科。
2.2 柄海鞘相关微生物类群的多样性
通过菌落形态、细胞显微形态观察以及TCBS培养基培养实验结果去冗余,最终从78株纯培养物中选择30株(其中有7株为能够在TCBS培养基上生长的菌株)并进行测序和基于16SrRNA基因序列的系统发育多样性分析。16SrRNA基因PCR扩增后电泳结果见图1,基因片段大小约为1 500bp。30株分离菌株的16SrRNA基因序列提交GeneBank数据库进行注册,获得注册号为JF721968~JF721997。
一般认为,根据16SrRNA基因序列,菌株间同源性在93%~95%,可以认为属于不同的属[14];同源性大于95%,可以认为属于同一属;16SrRNA序列同源性大于97%,可以认为属于同一个种。基于16SrRNA基因的系统发育分析见图2(系统发育树)。

图1 16SrRNA 基因PCR产物的琼脂糖凝胶电泳Fig.1 Agarosegel electrophoresis of PCR products of 16SrRNA gene
16SrRNA基因序列分析表明,30个分离菌株属于细菌域的4个系统发育类群(表1):放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和变形菌纲(Proteobacteria),其中包括11个科(Alteromonadaceae,Bacillaceae,Flammeovirgaceae,Flavobacteriaceae,Halomonadacea,Micrococcacea,Pseudomonadaceae,Psychromonadaceae,Shewanellaceae,Staphylococcaceae,Vibrionaceae)、11个属(Agarivorans,Arthrobacter,Bacillus,Flammeovirga,Halomonas,Shewanella,Staphylococcus,Tenacibaculum,Pseudomonas,Psychromonas,Vibrio)。其中,变形菌纲菌株都属于γ-变形菌纲,占分离菌株的绝大部分比例(22株,73.3%),其次是Bacteroidet菌门(4株,13.3%),Firmicutes菌门(3株,10%),Actinobacteria菌门(1株,3.3%)。
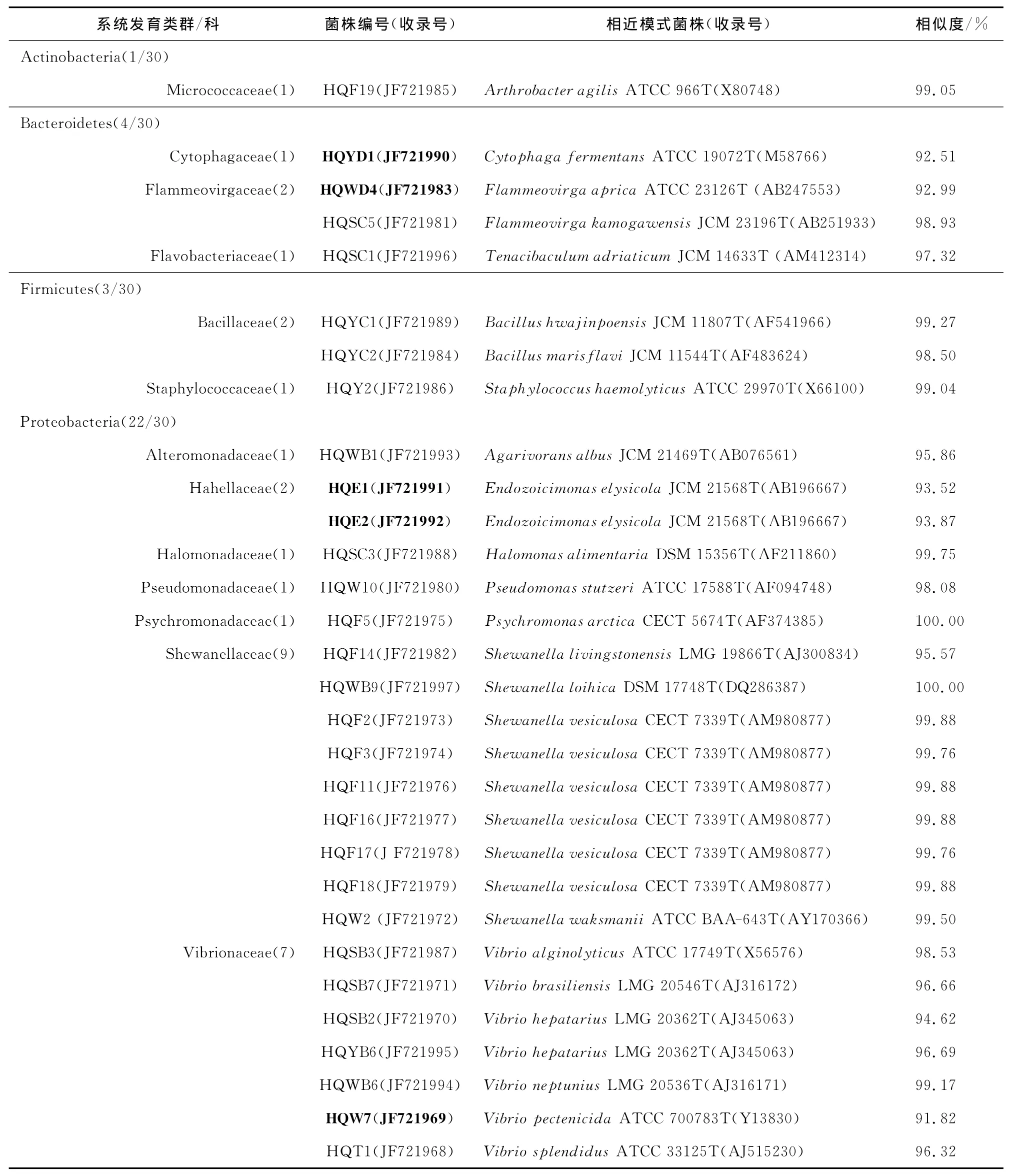
表1 柄海鞘可培养细菌与其系统发育关系最密切的典型菌株间的系统发育关系Table 1 Closest phylogenetic neighbors of strains isolated from Styela clava based on 16SrRNA sequence analysis
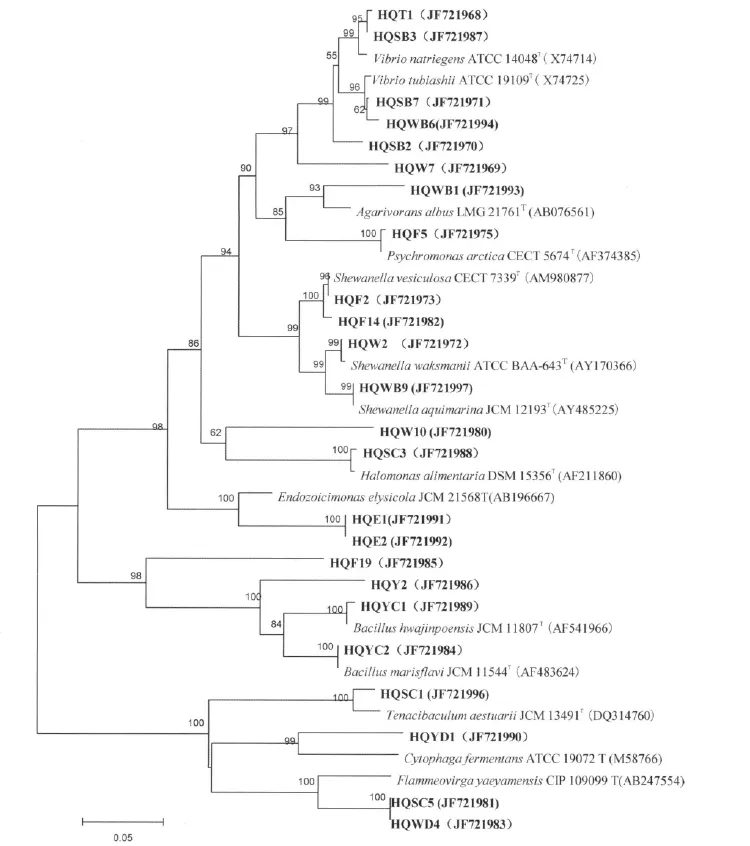
图2 基于16SrRNA基因序列构建的柄海鞘共附生可培养细菌的系统发育树Fig.2 Neighbor-Joining tree constructed based on 16SrRNA gene sequence analysis showing the phylogenetic relationships among strains isolated from Styela clava and related taxa
2.3 物种与遗传多样性
按16SrRNA基因序列相似性小于97%的菌株是属于不同物种的归类原则,30个分离菌株可以归为23个物种。柄海鞘相关可培养细菌的Shannon-winner多样性指数(H)为4.26,均匀度指数(E)为0.97,表明其具有较高的物种多样性和均匀度。除了2株(HQF5和HQWB9)分别与其相关的已知物种的典型菌株的16SrRNA基因序列相似性为100%外(表1),部分分离菌株与其相关的已知物种的典型菌株的16S rRNA基因序列相似性在91.82%~98.93%之间,说明部分菌株与其系统发育关系密切的相关菌株之间存在一定的遗传差异。
值得指出的是,菌株HQW7、HQYD1、HQWD4、HQE1和HQE2与模式菌株的16SrRNA基因系列存在较大差异,最高相似度仅分别为91.82%,92.51%,92.99%,93.52%,93.87%,这5株菌可能代表新的分类单元。目前这些细菌的新种鉴定工作正在进行中。
3 讨 论
本研究采用细菌纯培养以及基于16SrRNA基因序列的系统发育分析对威海海域柄海鞘的共附生细菌的多样性进行了研究。本次试验共获得78株纯培养物,通过TCBS培养基的选择性筛选可知,柄海鞘共附生细菌中有很大比例的菌株属于弧菌属(55株,70.5%)。作为近海河口环境中的微生物区系的主要成员,弧菌广泛分布于半咸水、河口、海洋的水体和沉积物中。弧菌在海洋环境中占据优势地位,除在营养物的循环中起着非常重要的作用外,弧菌还是鱼、虾、软体动物肠道内可培养细菌种的优势菌群,在消化和营养方面可能起着非常重要的作用[15-16]。于德华等[17]在调查研究青岛近岸不同环境中的海洋弧菌时发现,在海水、沉积物、鱼类和藻类等海洋生物表面,弧菌科都是优势种群,在鱼体表面甚至达到83.1%,在工业污染区沉积物中也占39.5%,其它环境中都达到50%左右甚至更多。在无污染海域,弧菌属在细菌区系中的比例为10%~14%,在海水养殖密集区的比例在6%~7.5%之间,在鱼体表面达47.7%,在工业污染区的比例为2.6%~5%。本次试验结果与此相符。
Webster等[18]等研究了海绵(Rhopaloeides odorabile)共附生细菌的系统发育多样性,在他们分离并测序的34株细菌中,放线菌门菌株占30%,γ-变形菌纲菌株占41%。Huang等[19]在调查硇洲海域马粪海胆可培养细菌多样性时发现,厚壁菌门菌(58.8%)是其中占优势的微生物类群,其次是γ-变形菌纲(26.5%)。Xiao等[20]发现海葵中可培养细菌中厚壁菌门也是优势菌群,占所分离细菌总数的40.5%,γ-变形菌纲菌株占33.3%。本研究结果也表明γ-变形菌纲是柄海鞘共附生细菌的优势菌群,这与其它海洋无脊椎动物具有相似之处。
用于系统发育分析的30株菌归属4个系统发育群11个科11个属,可以分为23个物种,大多数菌株与其系统发育关系最密切的已知物种的典型菌株之间存在一定的遗传差异(16SrRNA基因序列相似性为91.82%~98.93%),且发现5株菌 HQW7、HQYD1、HQWD4、HQE1和 HQE2与模式菌株的16SrRNA基因相似度较低,可能代表新的分类单元。上述结果揭示了柄海鞘可培养细菌的多样性。
(References):
[1]WILKINSON C R.Microbial associations in sponges.I.Ecology,physiology and microbial populations of coral reef sponges[J].Marine Biology,1978,49:161-167.
[2]UNSON M D,HOLLAND N D,FAULKNER D J.A brominated secondary metabolite synthesized by the cyanobacterial symbiont of a marine sponge and accumulation of the crystalline metabolite in the sponge tissue[J].Marine Biology,1994,119:1-11.
[3]SCHMIDT E W,OBRAZTSOVA A Y,DAVIDSOND S K,et al.Identification of the antifungal peptide-containing symbiont of the marine spongeTheonella swinhoeias a novelδ-teobacterium,“CandidatusEntotheonella palauensis”[J].Marine Biology,2000,136:969-977.
[4]HENTSCHEL U,SCHMID M,WAGNER M,et al.Isolation and phylogenetic analysis of bacteria with antimicrobial activities from the Mediterranean spongesAplysina aerophobaandAplysina cavernicola[J].FEMS Microbiology Ecology,2001,35(3):305-312.
[5]JOERN P.Metabolites from symbiotic bacteria[J].Nat.Prod.Rep.,2004,21:519-538.
[6]HILL R T.Microbes from marine sponges:a treasure trove of biodiversity for natural products discovery[M]∥Bull A T.Microbial diversity and bioprospecting.Washington,D.C.:American Society for Microbiology,2004:177-190.
[7]KENNEDY J,BAKER P,PIPER C,et al.Isolation and analysis of bacteria with antimicrobial activities from the marine sponge Haliclona simulans collected from Irish waters[J].Mar.Biotechnol.,2009,11:384-396.
[8]XIE X Q,LIN H P,YAN B,et al.Screening of cytotoxic activity of marine animal symbiotic and epiphyte microorganisms against B16 tumor cell[J].Chinese Journal of Marine Drugs,2006,25(6):26-30.
[9]ZHENG L,HAN X T,YAN X J,et al.A preliminary study on algicidal activity of marine bacteria associated with marine organism[J].Advances in Marine Science,2006,25(4):511-519.郑立,韩笑天,严小军等,海洋生物共栖细菌抑藻活性的初步研究[J].海洋科学进展,2006,25(4):511-519.
[10]RYUICHI S,STROH J G,SULLIN D W,et al.Seven new didemnins from the marine tunicateTrididemnum solidum[J].Journal of the American Chemical Society,1995,117(3):3734-3736.
[11]MCDONALD L A,SWERSEY J C,IRELAND C M,et al.Botryllamide A-D,new brominated tyrosine derivatives from styelid ascidians of the genusBotryllus[J].Tetrahedron,1995,51(18):5237-5240.
[12]WILLIAMS A B,JACOBS R S.A marine natural product,patellamide D,reverses multidrug resistance in a human leukemic cell line[J].Cancer Letters,1993,71(1-3):97-102.
[13]STACKEBRANDT E,COEBEL B M.Taxonomic note:aplace for DNA-DNA reassociation and 16SrRNA sequence analysis in the present species definition in bacteriology[J].International Journal of Systematic and Evolutionary Microbiology,1994,44:846-849.
[14]LIU Y,HUANG X H,HE P Q,et al.Molecular identification and phylogenetic analysis of cultivable bacteria from India Ocean hydrothermal vents[J].Advances in Marine Science,2009,27(4):193-200.刘艳,黄晓航,何培青,等.印度洋深海热液区可培养细菌的分子鉴定与系统发育分析[J].海洋科学进展,2009,27(4):193-200.
[15]MOSS S,LEAMASTER B R,SWEENEY J N.Relative abundance and species composition of gram-negative,aerobic bacteria associated with the gut of juvenile white shrimpLitopenaeus vannameiReared in oligotrophic well water and eutrophic pond water[J].World Aquac.Soc.,2000,31:255-263.
[16]SAWABE T,SUGIMURA I,OHTSUKA M,et al.Vibrio halioticolisp.nov.,a non-motile alginolytic marine bacterium isolated from the gut of the abaloneHaliotis discushannai[J].Int.J.Syst.Bacteriol.,1998,48:573-580.
[17]YU D H.The Investigation of marine vibrios in different environment around Qingdao and the rapid detection of pathogenicVbrio anguillarumby PCR amplification[D]于德华.青岛近岸不同环境中海洋弧菌的检测及用PCR方法快速检测病原鳗弧菌[D].青岛:中国海洋大学,2003.
[18]WEBSTER N S,WILSON K J,et al,Phylogenetic diversity of bacteria associated with the marine spongeRhopaloeides odorabile[J].Applied and Environental Microbiology,2001,67(1):434-444.
[19]HUANG K,ZHANG L,ZIU Z X,et al.Diversity of culturable bacteria associated with the sea urchinHemicentrotus pulcherrimusfrom Naozhou Island[J].Applied and Environmental Microbiology,2009,49(11):1424-1429.
[20]XIAO H,CHEN Y,LIU Z ,et al,Phylogenetic diversity of cultivable bacteria associated with a sea anemone from coast of the Naozhou island in Zhanjiang,China[J].Wei Sheng Wu Xue Bao,2009,49(2):246-250.
Isolation and Phylogenetic Analysis of Associated Bacteria From theStyela clava
MIAO Ting-ting,XING Xiang,DU Zong-jun,CHEN Guan-jun
(Marine college of Shandong University at Weihai,Weihai 264209,China)
Associated bacteria were isolated from Styela clava collected from the coast of Weihai,China.They were cultured by using conventional culture-dependent method and investigated by using phylogenetic analysis based on 16SrRNA gene sequence comparisons.The 78isolates were selected from samples on marine 2216medium (Difco).Based on morphological characters and weather growing on TCBS selective medium,we selected 30strains for molecular phylogenetic study using 16SrRNA gene sequences.Our results showed that 30isolates were members of 11genera of 11families in 4major phylogenetic groups(Actinobacteria,Bacteroidetes,Firmicutes,Proteobacteria).Most of the strains had certain genetic differences between the closely known type strains(16SrRNA gene sequence similarity were 95.57%~99.88%).The lower similarity suggested that at least 5strains represented new species.The results presented above showed that there were abundant species diversity and phylogenetic diversity of bacteria isolated fromStyela clava.
Styela clava;associated bacteria;16SrRNA gene;phylogenetic analysis;bacterial diversity
April 15,2011
Q938.1
A
1671-6647(2012)01-0111-08
2011-04-15
国家自然科学基金重点项目——渤海中南部底栖生物生产过程与生物多样性集成研究(40730847);山东大学自主创新基金项目——海洋微生物的资源采集及其次生代谢产物研究(IIFSDU)
苗婷婷 (1988-),女,山东济宁人,研究生,主要从事海洋生物学方面研究.E-mail:miaotingting456@163.com
*通讯作者,教授,博士生导师,主要从事纤维素的微生物降解、微生物生理生化及酶学方面研究.E-mail:guanjun@sdu.edu.cn
(王佳实 编辑)

