广东沿海杂色鲍养殖群体遗传多样性的微卫星分析*
许 新,王江勇,姜敬哲,区又君
(1.中国水产科学研究院 南海水产研究所,广东 广州510300;2.从化市畜牧兽医渔业局,广东 从化510900)
广东沿海杂色鲍养殖群体遗传多样性的微卫星分析*
许 新1,2,王江勇1*,姜敬哲1,区又君1
(1.中国水产科学研究院 南海水产研究所,广东 广州510300;2.从化市畜牧兽医渔业局,广东 从化510900)
实验选取的7个杂色鲍(Haliotisdiversicolor)养殖群体分别来自广东汕头、汕尾、惠东、湛江和徐闻,深圳野生驯养,利用7对微卫星引物对上述群体进行了遗传多样性和群体遗传结构的分析。结果显示:7个位点产生的等位基因数从7~13个不等,共检测到68个等位基因,平均每个位点的等位基因数为9.71个。有效等位基因数的范围为3.8~8.1,平均观测杂合度范围为0.422~0.906,平均期望杂合度范围为0.748~0.917。7个群体的多态信息含量变化范围为0.657~0.883,PIC值均在0.500以上,表现为高度多态性。与两个野生驯养的养殖群体相比,其他5个养殖群体均存在不同程度的遗传多样性降低。AMOVA分析显示,4.79%的遗传变异来自于群体之间,95.21%的变异来自于群体内的个体之间。研究表明,广东沿岸的杂色鲍具有较高的遗传多样性水平,7个群体之间呈现一定的遗传分化。
杂色鲍;微卫星标记;遗传多样性;养殖群体
(王佳实 编辑)
杂色鲍是分布在中国南方沿海一种具有重要经济价值的贝类,在我国海水养殖业中占有重要地位。然而,多年的养殖也引发了杂色鲍的苗种品质下降、病害多发、养殖生态失衡等诸多不良现象的出现,例如种质资源严重衰退和对环境敏感[1-2]。因此,对该物种的种质保护和改良是一项迫不及待的任务。目前,对杂色鲍研究多集中于基础生物学、养殖技术及病理学探讨等[3-6],在群体遗传多样性研究方面只有养殖群体和野生群体的同工酶比较和随机扩增多态DNA(RAPD)分析[7-9]。而有关杂色鲍的群体遗传多样性和遗传结构的微卫星(SSR)分析尚未有研究报导。本研究运用SSR技术对中国广东沿岸6个地区的杂色鲍养殖群体遗传多样性和遗传分化进行分析研究,以期为杂色鲍的遗传育种和种质资源保护利用等提供理论依据。
1 材料与方法
1.1 实验材料
本实验研究的7个养殖群体采样地点见图1,具体情况见表1。
1.2 DNA提取与SSR扩增
1.2.1 DNA提取
供试样品的基因组DNA利用海洋动物组织基因组DNA快速提取试剂盒(天根生化科技有限公司)提取。
1.2.2 PCR反应条件
本研究所用7对微卫星位点来自本实验室从文库中筛选得到的19对引物,由上海生工生物技术公司合成。核心重复序列、引物序列和最适PCR扩增条件见表1。PCR反应在C1000TM型热循环PCR仪上进行,反应体系为25μL(组份见表3)。PCR反应条件为:94℃预变性6min;然后94℃30s,退火45s(退火温度见表2),72℃1.5min,共35个循环;最后72℃延伸7min。PCR产物经6%变性聚丙烯酰胺凝胶电泳分离(60W跑1~2h),硝酸银染色后检测其多态性。使用100bp Ladder DNA(Takara.Co)来确定等位基因大小,电泳条带数目由人工统计。

表1 7个养殖群体的情况Table 1 The situation of seven cultured population
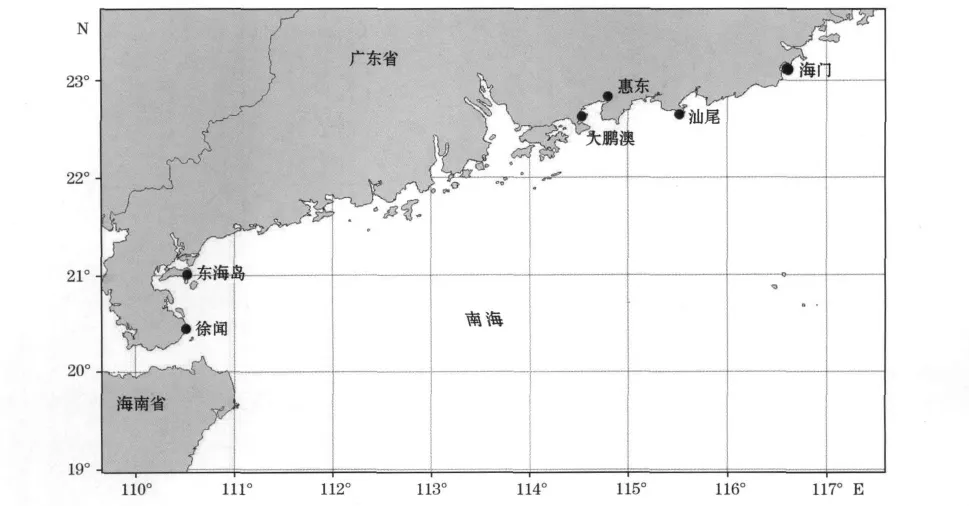
图1 7个杂色鲍采集地点(●代表采样点)Fig.1 Locations for seven Haliotisdiversicolor populations samples(●site of sampling)

表2 选取7个位点的引物序列和退火温度Table 2 Primer sequence and annealing temperature of seven microsatellites loci

表3 25μL PCR反应体系具体组份Table 3 Components of 25μL PCR reaction system
1.3 数据统计处理
统计每个位点所含的等位基因数量(A),用Popgene V1.32[10]软件计算群体的有效等位基因数(Ae)、观测杂合度(Ho)、期望杂合度(He)、群体间的Nei氏遗传距离微卫星卫位点的等位基因频率,并进行哈迪-温伯格平衡检测。
用Arlequin V2.0[11]软件计算遗传距离,估计群体间的遗传分化;对群体内和群体间的变异进行分子方差分析(hierarchical analysis of molecular variance,AMOVA)。根据Botstein等[15]计算每个微卫星位点的多态信息含量(polymorphism information content,PIC),计算公式如下:
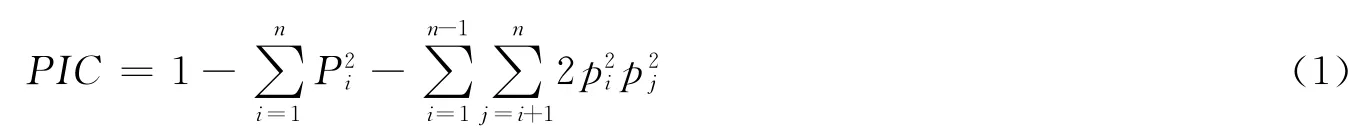
式中,pi和pj分别表示第i和第j个等位基因在群体中的频率;n为等位基因数。
根据群体间的遗传距离,利用MEGA 4.1[12]构建聚类关系树,各分支的置信度由自展(bootstrap)检验1 000次获得。
2 结果与分析
2.1 PCR扩增结果及多态性
采用6%的变性聚丙烯酰胺凝胶电泳对7个群体7个多态性位点进行检测,得到了清晰的谱带。7个多态位点在7个群体共检测出68个等位基因,每个位点平均产生9.71个等位基因。7个微卫星位点在7个杂色鲍群体中均获得了良好的多态性,可在杂色鲍遗传多样性分析中使用。部分聚丙烯凝胶电泳结果见图2。
图2 位点ZSB6(a)和位点ZSB13(b)聚丙烯凝胶电泳结果Fig.2 Results of some polymorphic loci by polyacrylamide electrophoresis
2.2 群体内遗传多样性
7个微卫星位点在7个杂色鲍养殖群体中检测到较高的多态性(表4)。不同的位点显示出不同的多态性水平。7个位点产生的等位基因数从5~13个不等,其中ZSB8位点在SW中所得到的等位基因数目最少,为5个,ZSB13位点在群体RB上所获得的等位基因数目最多,为13个(表2)。有效等位基因数反映了基因间的相互影响,可作为遗传变异的一个度量参数。它反映的群体遗传变异性大小,其数值越接近所检测到的等位基因的绝对数,表明等位基因在群体中分布越均匀。7个多态位点中,有效等位基因数目范围为3.8~8.1,其中ZSB13最大,ZSB8最小。平均有效等位基因数为5.46;说明这些位点上存在稀有等位基因。PIC值均在0.500以上变化范围0.657~0.883,ZSB13最大,ZSB4最小。全部表现为高杂合度(PIC>0.5),表明这个群体的遗传多样性比较丰富。平均观察杂合度范围0.422~0.906,平均期待杂合度范围为:0.748~0.917。所有位点的平均观察杂合度为0.625,平均期待杂合度为0.874。其中SY和RB的各项指标都比其他群体的高。
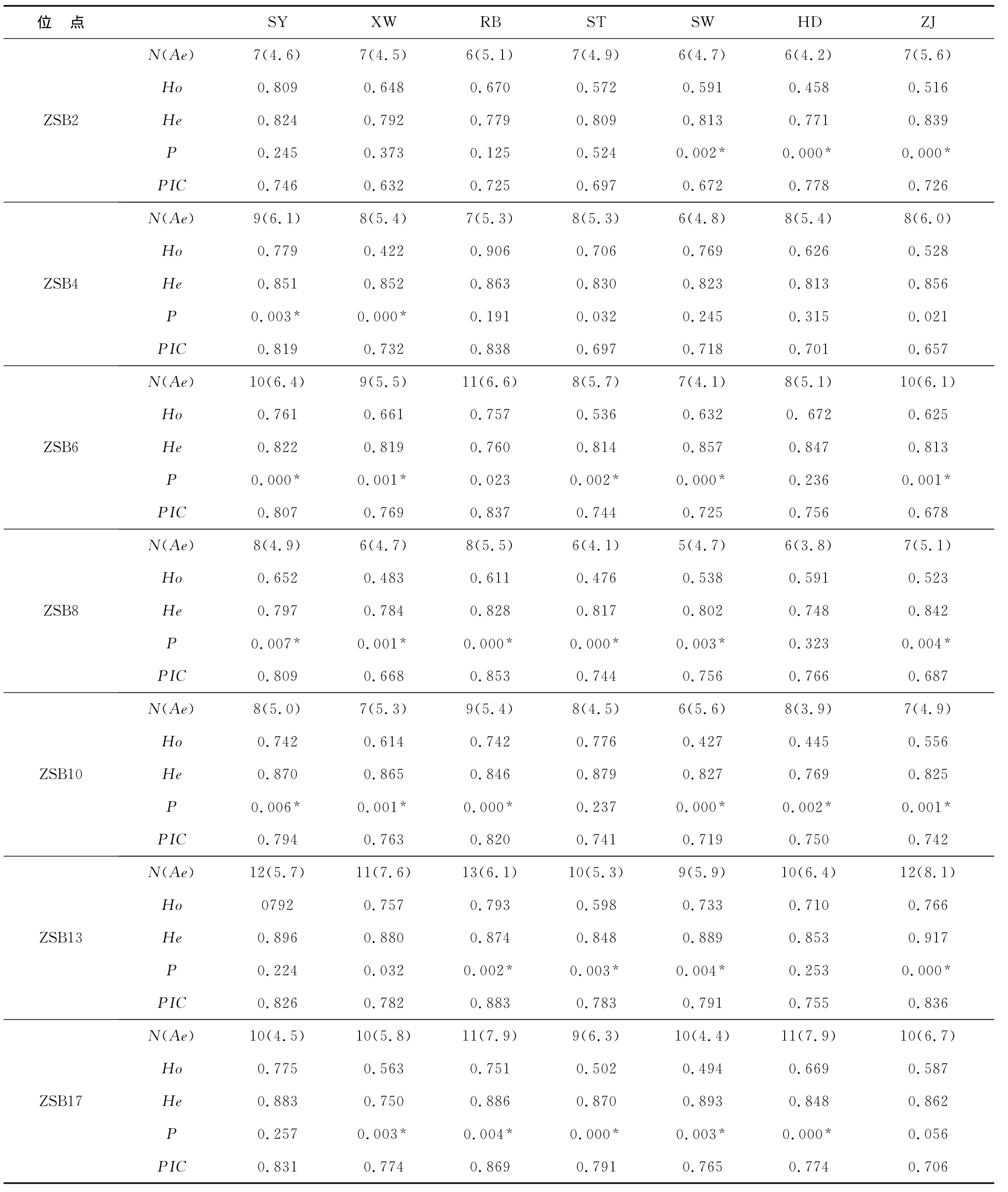
表4 不同位点在7个杂色鲍群体的遗传多样指数Table 4 Genetic diversity index of seven microsatellite loci in seven Haliotisdiversicolor populations
2.3 哈迪·温伯格(Hardy-Weinberg)平衡检验
经哈迪·温伯格平衡的卡方检测发现,所有位点在7个群体均存在不同程度的偏离平衡状态。所检测的49个位点(7个群体 ×7个位点)经过Bonferroni纠正后,有31个位点发生不同程度的偏离(表4)。可能是由于杂合子缺失造成,或者存在无效等位基因。遗传偏离指数从直观上表明了杂合子缺失或过剩,根据统计结果,表明所有群体存在不同程度杂合子缺失。
2.4 群体遗传结构
近交系数(Fis)是指个体的等位基因来自共同的祖先基因概率,表示近亲交配个体基因纯和程度的数量指标。7个位点在不同群体内的Fis值如表5所示。所有群体的平均近交系数0.204 7,表明群体之间的近交程度较小。分子方差分析(AMOVA)结果表明,群体内的方差占总方差的95.21%,群体间的遗传变异仅为总变异的4.79%(表6)。群体内的遗传变异远大于群体间的遗传变异,表明群体内的遗传变异绝大部分来源于群体内个体间的遗传变异。
2.5 群体间的遗传差异
遗传分化系数(Fst)是衡量所有群体之间遗传分化的重要指标。Wright[13]认为,Fst在0~0.05之间表示群体间遗传分化微弱;0.05~0.5之间表示群体遗传分化中等;0.15~0.25之间表示群体遗传分化较大;当Fst大于0.25时,表示遗传分化极大。本研究中两两群体间比较Fst值范围在0.015~0.162(表7)。最大的Fst值出现在日本与湛江群体之间,最小的Fst值出现在ZJ与XW之间。其中RB与其他群体有显著水平的差异,说明群体间可能存在显著遗传结构差异。
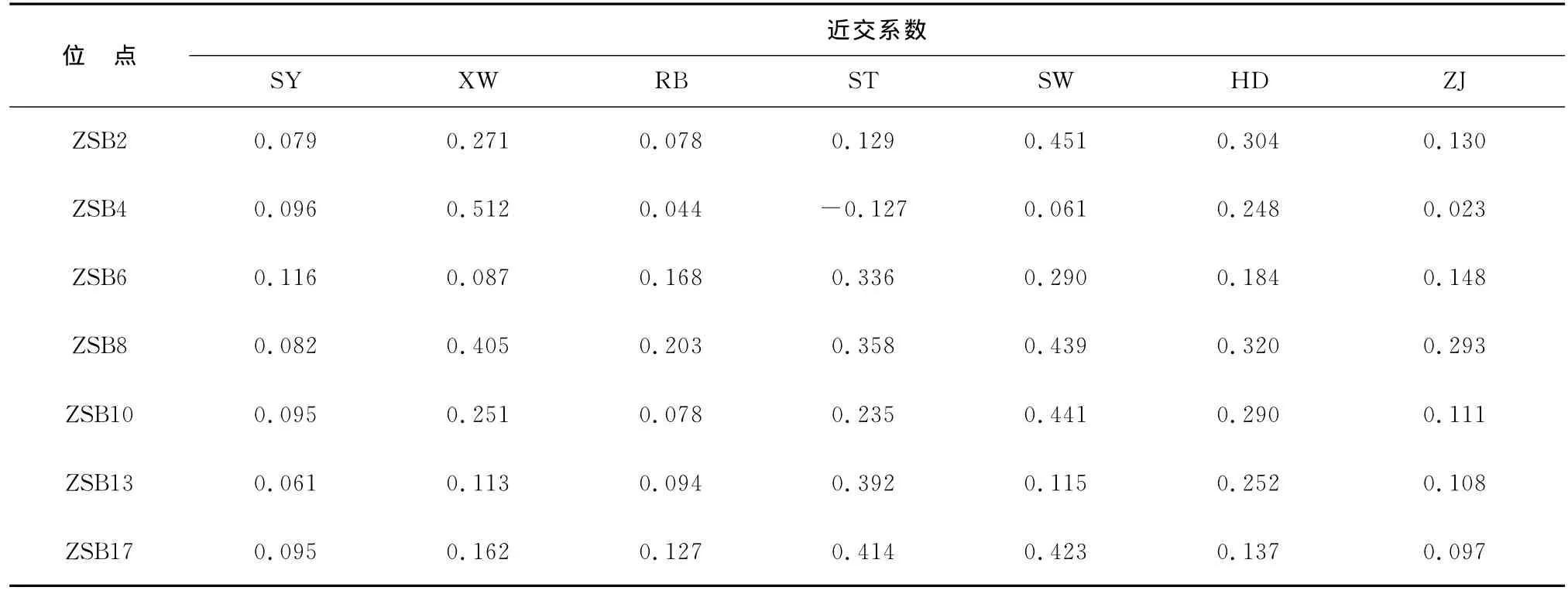
表5 7个位点在杂色鲍不同群体内的近交系数(Fis)Table 5 Inbreeeding coefficient(Fis)of seven loci in populations of Haliotisdiversicolor
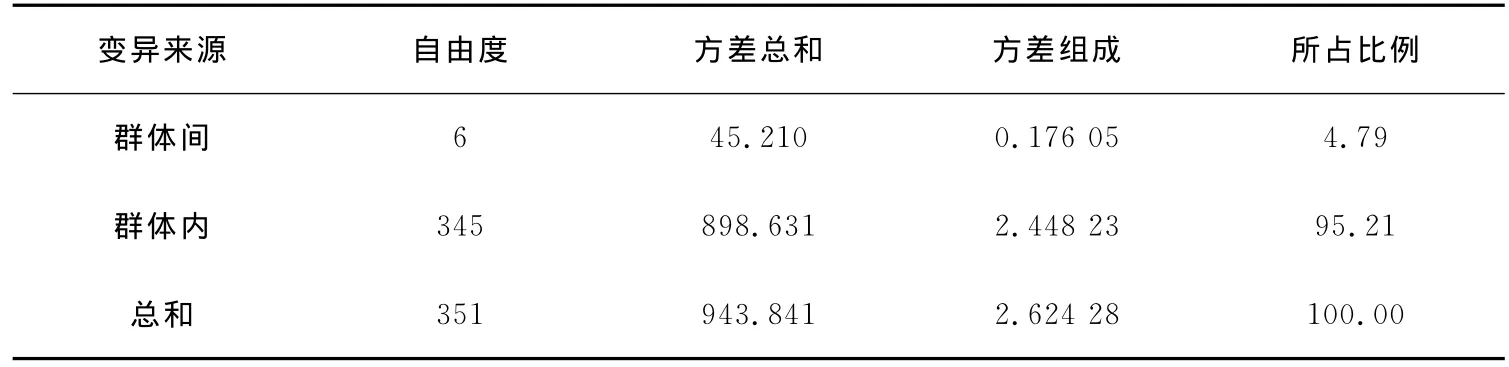
表6 杂色鲍7个群体的分子方差分析Table 6 AMOVA of analysis of seven populations of HaliotisDiversicolor
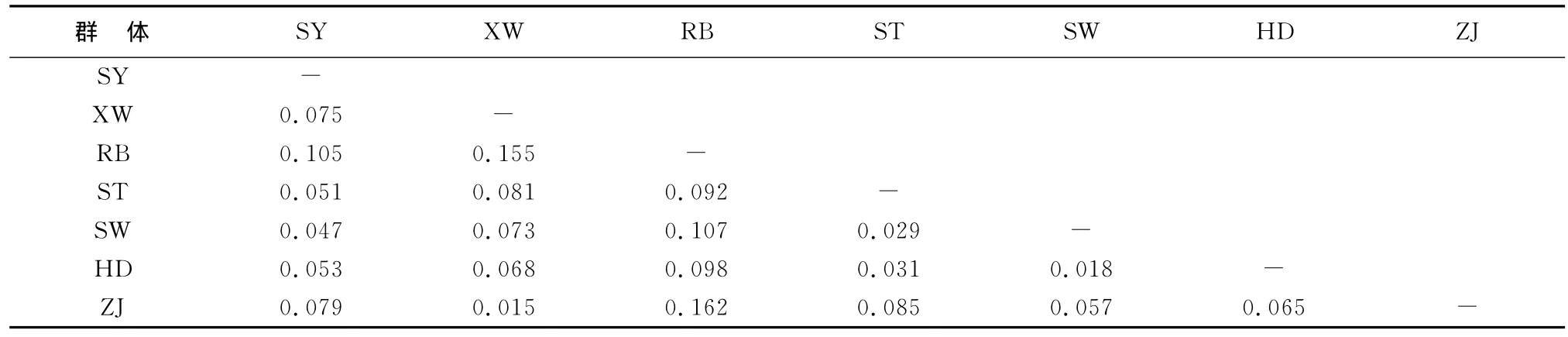
表7 基于群体间Fst值的遗传分化分析Table 7 Analysis of genetic differentiation between pairs of samples based on estimates of Fst
2.6 遗传距离与聚类分析
各个群体的AMOVA分析表明,群体间的遗传分化是显著的,群体之间的相似系数和遗传距离如表8所示,根据Nei[14]的方法计算群的遗传距离与相似系数,不同群体间偏差矫正后的相似系数为0.892 7~0.980 4,而杂色鲍各群体之间的遗传距离为0.016 8~0.118 7,其中ZJ与XW和SW与HD之间的遗传距离为0.016 8和0.022 7,明显小于其他群体之间的遗传距离,RB与ZJ之间的遗传距离最大(表8)。根据群体之间的遗传距离,采用软件MEGA 4.1的NJ程序进行群体间的亲缘关系聚类分析,其中ZJ、XW之间的亲缘关系最近,聚为一类;SY自成一类;而RB与ST,SW,HD聚为一类(图3)。
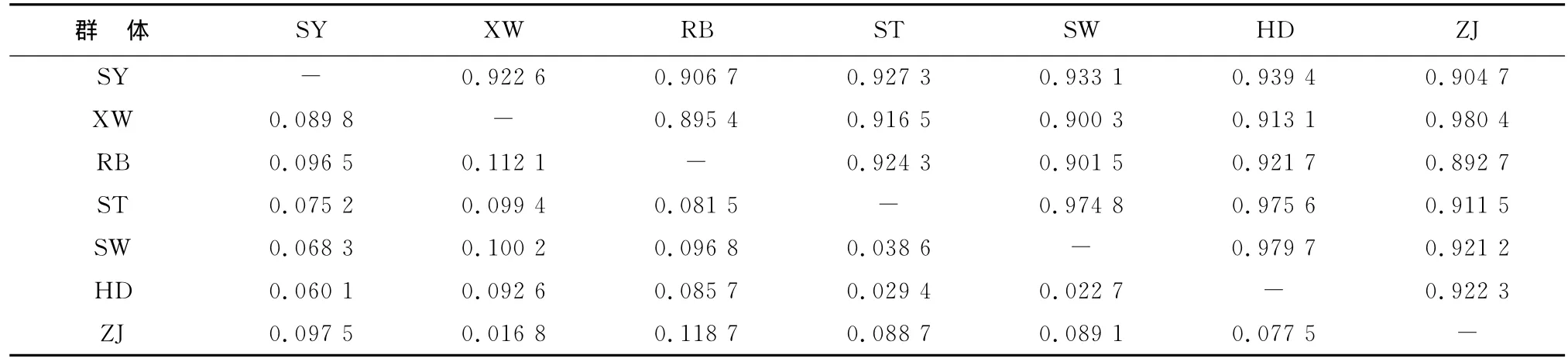
表8 杂色鲍7个群体的Nei相似性指数及遗传距离Table 8 Nei’s genetic identity and genetic distance of seven populations of Haliotisdiversicolor
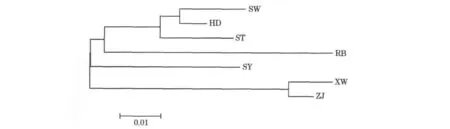
图3 7个杂色鲍群体间的NJ系统树Fig.3 NJ tree of seven Haliotisdiversicolor populations
3 讨 论
3.1 群体内的遗传多样性
在7个微卫星位点中我们均检测到丰富的遗传多样性,平均等位基因数为9.71(范围7~13),平均期待杂合度为0.874(范围0.748~0.917)。王鹭骁等[7]对采自海南三亚、香港和福建平潭的3个野生杂色鲍种群以及来自台湾的九孔鲍的1个养殖群体共4个群体的6种同工酶系统的遗传变异进行研究,结果表明,同工酶位点的平均等位基因数为1.2,预期杂合度和实际杂合度都很小,只有0.058~0.075。黎中宝等[8]对九孔鲍的养殖群体和杂色鲍的自然群体进行了10种等位的电泳检测和谱带遗传分析表明,只有少部分位点具有多态性,且平均每个位点有2~4个等位基因。与同工酶标记相比,本研究所采用7个微卫星位点具有更丰富的多态,更适合对杂色鲍群体进行遗传分析。
PIC是等位基因频率和数目变化的函数,反映了基因变异程度的高低和群体的遗传多样性水平。根据Bostein等[15]提出的衡量基因变异程度的多态信息含量指标,在一个群体中,当PIC>0.5时,该位点为高度多态位点;当0.25<PIC<0.5时,该位点为中度多态位点;当PIC<0.25时,该位点为低度多态位点。本研究所选择的7个位点在杂色鲍7个群体中的PIC值均大于0.5,均属于高度多态位点。说明广东沿岸养殖的各群体的遗传多样丰富,群体内的遗传变异大,多态信息含量较高。这与杜博对南方杂色鲍的RAPD的分析一致[9]。
杂合度是衡量群体遗传多样性的一个重要参数。杂合度越高,表明群体遗传多样性越高,反之则较低。分析7个群体的等位基因与杂合度,结果显示各群体的遗传多样性还处于较高水平。分析其原因可能为2个:其一,之前数量巨大且未受到干扰的群体是在近期遭到人为因素干扰才引起的群体数量急剧减少,但仍然保持较高水平的遗传多样性的现象;其二,ST、SW和HD等群体都是与RB杂交产生的杂交群体,通过杂交而获得了较多的基因,从而表现出较高的遗传多样性。与杂合度相比较,等位基因对“瓶颈效应”的敏感程度更高,本研究中野生驯养的养殖群体在等位基因数目总体表现出比其他养殖群体高,表明在养殖过程杂色鲍养殖群体的等位基因有所丧失。然而,其他养殖群体的群体内遗传丰度也都较高,可能是由于不同地区杂色鲍杂交所致。统计结果表明两个野生驯养的养殖群体遗传潜力比其他养殖群体的要高。
本研究中有两个群体是野生驯养的,即RB和SY,两个群体无论是从观察杂合度和多态信息含量方面来讲,都比其他养殖群体要高。这种现象与对皱纹盘鲍(Haliotisdiscushannai)的研究结论相一致[16]。养殖群体的遗传多样性较野生群体遗传多样性有所下降,主要原因如下:我国南方的杂色鲍在养殖过程中,育苗厂选取的亲鲍数量相对较少,且雄性个体数目远远少于雌性个体数[17],经过多代的繁殖后,由于遗传漂变和瓶颈效应,研究认为亲鲍数量少且性别比例不均衡现象势必会造成遗传多样性的下降。Vespoor等[18]和Winans等[19]在对鲑鱼(salmon)的遗传调控研究中认为,当有效亲本数量减少到小于100时,可以减低该群体的群体遗传多样性。在养殖多带后,养殖群体的群体遗传多样性与野生群体比较会存在不同程度的下降,这种现象在鲍(abalone)[20],对虾(Penaeusorientaliskishinouye)[21],牡蛎(ConchaOstreae)[28]和各种鱼类中[22-24]均有报道。与野生群体相比,遗传多样性降低已经成为养殖生物群体的常见现象。在养殖环境中,人工和自然选择会通过改变养殖群体中等位基因构成,进而影响遗传结构[25]。
3.2 哈迪-温伯格平衡
在49组群体位点组合数据中(7群体×7位点),31个群体位点组合检测为显著偏离哈迪-温伯格平衡(P<0.05/7)。导致哈迪-温伯格平衡偏离主要有两个原因:一是群体内杂合子缺失;二是无效等位基因的存在。经过检测,主要是由于无效等位基因的存在而导致哈迪-温伯格平衡的偏离。由于微卫星序列的点突变率(10-2~10-5)和复制突变率(10-3~10-4)都很高[26],很容易导致无效等位基因的产生。在哺乳动物、鱼类、甲壳类动物中都曾有报道[27-28]。在海洋贝类中,无效等位基因的存在也是一个比较常见的现象。Li等[27]在太平洋牡蛎(Crassostreagigas)和皱纹盘鲍微卫星的遗传分析的研究中,发现分别有51.9%和10.7%的位点存在无效等位基因。而在微卫星群体遗传分析中,如果无效等位基因的存在而不被考虑,就会出现把杂合子计数为纯合子的错误,从而影响哈迪-温伯格平衡。
3.3 遗传变异和遗传分化
Fst值是衡量群体间遗传分化程度的重要参数。本研究中Fst值的范围为0.015~0.162。其中RB与其他群体之间Fst值都大于0.08,表明RB与其他养殖群体之间都存在明显的遗传分化。通过AMOVA分子方差分析对变异来源的剖分表明,群体间变异仅占总变异的4.79%,表明选育过程中近交及瓶颈效应发生的可能性不大,群体遗传结构的改变主要是由人工选择压力造成的。梁俊等[29]对鳗鲡(Anguillajaponica)进行研究发现欧洲鳗与日本鳗间的分化系数(Fst)为0.310,推算其分化世代数为224 320,约为200多万年,该研究表明,在自然条件下,长期的地理隔离会造成不同群体间产生显著的遗传分化。
Lehoczky等[30]采用13个微卫星位点对养殖鲤与野生鲤各3个群体研究发现,通过Fst值比较,表明各群体间出现了明显的遗传分化。不过,Lehoczky等未能交代其研究的养殖群体经历了多久的驯养。该研究启示,在人工控制条件下,通过选择、人工诱变、杂交等手段可改变群体内的遗传平衡,也可能促使群体内的遗传结构发生改变,在较短时间内造成与长期地理隔离效应相当的群体遗传分化。刘必谦等[31]在对4个不同地区的大连湾牡蝠进行研究时发现,相邻群体的遗传差异不明显,且遗传差异与地理位置有关,地理位置相距越远,遗传差异越明显。
本研究的Fst值显示出各群体存在遗传分化,采用Dc遗传距离构建的NJ系统树未能表明一个严格按地区划分的结构,图3中把RB和ST,SW和HD归为一支,SY另一支,而ZJ,XW归为第三支。地理距离和遗传距离的不相关可能是因为在长期的养殖过程中各养殖厂之间亲鲍和子代频繁交流交换造的,或者是因为ST,SW和HD均为和RB杂交的杂交种,具有一定相似的基因。但总体上与地理划分还有一定的关系,比如SW和HD,ZJ和XW之间。这可能是与杂色鲍的生活习性有关,由于附着生活,不会长距离地移动势必会造成不同地理位置的遗传分化。而养殖群体之间的遗传分化,我们推测是养殖群体中亲鲍数目的降低加剧了遗传漂变的影响。这种现象在太平洋牡蛎[32]、鲑鱼[33]也有报道。广东沿岸杂色鲍群体的遗传多样性较为丰富,这与RAPD方法取得的研究结果相似[34]。但由于各养殖厂为获得优良性状,不同杂色鲍的地理群体之间均存在杂交现象,遗传背景比较复杂和混乱,而系统的遗传选育需要建立在对养殖对象遗传背景了解的基础上。因此,中国各个养殖区域应该建立良好的亲鲍和苗种引进等级制度,用以保证鲍产业的健康发展。
(References):
[1]DENG Y W,LIU X,ZHANG G F,et al.Inbreeding depression and maternal effects on the early performance of Pacific abalone[J].North American Journal of Aquaculture,2005,67:231-236.
[2]MARCHANT S,HAYE P A,MARIN S A,et al.Genetic variability revealed with microsatellite markers in an introduced population of the abaloneHaliotisdiscushannaiIno[J].Aquaculture Research,2009,40(3):298-304.
[3]CHEN C S,ZHONG Y P,WU Y P,et al.The effect of salinity on food intake,growth and survival ofHaliotisdiversicolorsupertexta[J].Journal of Fisheries of China,2000,24(l):41-45.陈昌生,钟幼平,吴永沛,等.盐度对九孔鲍摄食、生长及存活的影响[J].水产学报,2000,24(l):41-45.
[4]WANG J Y,CHEN B S,FENG J,et al.Primary observation on spherical virus in versicolor abalone(Haliotisdiversicolor)with crack shell disease[J].Tropic Oceanology,2000,19(4):82-85.王江勇,陈毕生,冯娟,等.杂色鲍裂壳病球状病毒的初步观察[J].热带海洋,2000,19(4):82-85.
[5]LüJ Y,CHEN Z S,WU J Y,et al.Embryonic development of abalone(Haliotisdiversicolorreeve)[J].Acta Zoologica Sinica,2001,47(3):317-323.吕军义,陈志胜,吴金英,等.杂色鲍的胚胎发育[J].动物学报,2001,47(3):317-323.
[6]ZHANG Z H,CONG J R,ZHU M Y.The effect of culture density on the growth of abalone,Haliotisdiversicolorsupertexta[J].Marine Sciences,2003,27(10):10-14.张朝晖,丛娇日,朱明远.养殖密度对九孔鲍生长的影响[J].海洋科学,2003,27(10):10-14.
[7]WANG L X,WANG Z Y,HE C H,et al.Genetic Relationship inHalitisdiversicolor[J].Journal of Xiamen University(Natural Science),2005,44(l):98-102.王鹭骁,王志勇,柯才焕,等.不同地理群体杂色鲍的同工酶分析[J].厦门大学学报(自然科学版),2005,44(l):98-102.
[8]LI Z B,TIAN Z,ZHU D R.Biochemical genetic analysis of allozymes ofHaliotisdiversicolorsupertextaandHaliotisdiversicolordiversicolor[J].Marine Sciences,2004a,28(2):27-31.黎中宝,田柱,朱冬蕊.九孔鲍和杂色鲍等位酶的生化遗传分析[J].海洋科学,2004a,28(2):27-31.
[9]DU B.Genetic diversity of hatchery-produced stocks of three economically important abalone species in southern China[D].Wuhan:Huazhong Agricultural University,2007,1-50.杜博.中国南方沿海3种经济鲍养殖群体遗传多样性分析[D].武汉:华中农业大学,2007,1-50.
[10]YEH F C,BOYLE T J B.Population genetic analysis of co-dominant and dominant marker and quantitative traits[J].Belgian Journal of Botany,1997,129:157.
[11]SCHNEIDER S,ROESSLI D,EXCOFFIER L.Arlequin(version 2.0):a software for population genetics data analysis,version 2000[CP/DK].Genera:University of Geneva,2000.
[12]KUMAR S,TAMURA K,NEI M.MEGA3:integrated software for molecular evolutionary genetics analysis and sequence alignment[J].Brief Bioinformatics,2004,5(2):150-163.
[13]WRIGHT S.Evolution and the genetics of populations[M].Chicago:The University of Chicago Press,1978.
[14]NEI M.Estimation of average heterozygosity and genetic distance from a small number of individual[J].Genetics,1978,89(3):583-590.
[15]BOTSTEIN D,WHITE R L,SKOLNICK M,et al.Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J].American Journal of Human Genetics,1980,32(3):314-331.
[16]SHU J.Molecular population genetics of the pacific abalone,haliotis discus hannai[D].Qingdao:Ocean University of China,2008,1-118.束靖.皱纹盘鲍分子群体遗传学研究[D].青岛:中国海洋大学,2008,1-118.
[17]ZHAN A B.Development,characterization and application of microsatellite markers in zhikong scallop(chlamys farreri)[D].Qingdao:Ocean University of China,2007,1-218.战爱斌.栉孔扇贝微卫星标记的筛选及应用[D].青岛:中国海洋大学,2007,1-218.
[18]VESPOOR E.Reduced genetic variability in first generation hatchery populations of Atlantic salmon(Salmo salar)[J].Canadian Journal of Fisheries and Aquatic Sciences,1988,45:1686-1690.
[19]WINANS G A.Genetic.variability in chinook salmon stocks from the Columbia River basin[J].North American Journal of Fisheries Management,1989,9(1):47-52.
[20]EVANS B,BARTLETT J,SWEIJD N,et al.Loss of genetic variation at microsatellite loci in hatchery produced abalone in Australia(Haliotisrubra)and South Africa(Haliotismidae)[J].Aquaculture,2004,233(1-4):109-127.
[21]XU Z,PRIMAVERA J H,PENA L D,et al.Genetic diversity of wild and cultured Black Tiger Shrimp(Penaeusmonodon)in the Philippines using microsatellites[J].Aquaculture,2001,199:13-40.
[22]NORRIS A T,BRADLEY D G,CUNNINGHAM E P.Microsatellite genetic variation between and within farmed and wild Atlantic salmon(Salmosalar)populations[J].Aquaculture,1999,180(3-4):247-264.
[23]PORTA J,PORTA J M,MART NEZ-RODR GUEZ G,et al.Genetic structure and genetic relatedness of a hatchery stock of Senegal sole(Soleasenegalensis)inferred by microsatellites[J].Aquaculture,2006,25(1):46-55.
[24]WAS A,WENNE R.Genetic differentiation in hatchery and wild sea trout(Salmotrutta)in the Southern Baltic at microsatellite loci[J].Aquaculture,2002,204(3-4):493-506.
[25]MJOLNEROD I B,REFSETH U H,KARLSEN E,et al.Genetic differences between two wild and one farmed population of Atlantic salmon(Salmo salar)revealed by three classes of genetic markers[J].Hereditas,1997,127:239-248.
[26]VIGOUROUX Y,JAQUETH S J,MATSUOKA Y,et al.Rate and pattern of mutation at microsatellite loci in maize[J].Molecular Biology and Evolution,2002,19(8):1251-1260
[27]LI Q,PARK C,KIJIMA A.Allelic transmission of microsatellites and application to kinship analysis in newly hatched Pacific abalone larvae[J].Fisheries Science,2003,69(5):883-889.
[28]REECE K S,RIBEIRO W L,GAFFNEY P M,et al.Mierosatellite marker development and analysis in the eastern oyster(Crassostrea virginiea):Confirmation of null alleles and non-Mendelian segregation ratios[J].Journal of Heredity,2004,95(4):346-352.
[29]LIANG J,LI D J,LU L Q.Microsatellite variation of Japanese eel(Anguillajaponica)and European eel(A.anguilla)[J].Oceanologia et Limnologia Sinica,2003,34(4):414-421.梁俊,李道季,卢莉琼.日本鳗鲡(Anguillajaponica)和欧洲鳗鲡 (A.anguilla)的微卫星差异[J].海洋与湖沼,2003,34(4):414-421.
[30]LEHOCZKY I,MAGYARY I,HANCZ C,et al.Preliminary studies on the genetic variability of six Hungarian common carp strains using microsatellite DNA markers[J].Hydrobiologia,2005,533:223-228.
[31]LIU B Q,DAI J X,YU Z N.The investigation of populations by RAPD markers on oyster,crassostreatalienwhanensis[J].Journal of Ocean University of Qingdao,1998,28(l):82-88.刘必谦,戴继勋,喻子牛.RAPD标一记在大连湾牡蛎群体研究中的应用[J].青岛海洋大学学报,1998,28(l):82-88.
[32]HEDGECOCK D,LIN J Z,DECOLA S,et al.Transcriptomic analysis of growth heterosis in larval Pacific oysters(Crassostreagigas)[J].Proceedings of the National Academy of Sciences of the United States of America,2007,104(7):2313-2318.
[33]ALLENDORF F W,PHELPS S R.Loss of genetic variation in a hatchery stock of cutthroat trout[J].Transactions of the American Fisheries Society,1980,109:537-543.
[34]DU B,TONG X,HUANG G J,et al.Genetic variation in hatchery produced abalone stocks in South China using RAPD[J].Journal of Tropical Oceanography,2007,26(6):52-57.杜博,童馨,黄桂菊,等.九孔鲍养殖群体遗传变异的RAPD分析[J].热带海洋学报,2007,26(6):52-57.
Studies on the Genetic Structure ofHaliotisdiversicolorReeve in Coastal Area of Guangdong Province by Microsatellite Markers
XU Xin1,2,WANG Jiang-yong1,JIANG Jing-zhe1,OU You-jun1
(1.SouthChinaSeaFisheriesInstitute,ChineseAcademyofFisherySciences,Guangzhou 510300,China;2.ConghuaAnimalHusbandry,Veterinary&FisheryBureau,Conghua 510900,China)
In the present study,Genetic diversity and genetic structure of 7cultured populations of theHaliotisdiversicolorReeve from Shantou,Shanwei,Huidong,Shenzhen,Zhanjiang,Xuwen(all are in Guangdong Province,China)was analyzed using 7microsatellite markers.Sixty eight alleles of these seven microsatellite loci were found in these seven populations.The observed number of alleles of these microsatellite loci was 7~13in each population.The average number of alleles was 9.71,and the average effective number of alleles was 3.8~8.1.The observed population heterozygosity was 0.422~0.906and the observed expected heterozygosity was 0.748~0.917.The polymorphism of the seven populations were all high,and the PIC of the pupations were 0.657~0.883(PIC>0.5).Comparing with two populations of wild domesticated breeding,the five breeding groups showed different degrees of overall reduction in genetic diversity.AMOVA analysis demonstrated that 95.21%of genentic divergence came from population and 4.79%came from individuals.These results reavealed that the abalones had a higher level of genetic diversity along the coast of Guangdong province and 7groups showed a certain some genetic differentiation.
Haliotisdiversicolor;microsatellite DNA;genetic diversity;cultured population
March 8,2011
Q953
A
1671-6647(2012)02-0284-11
2011-03-08
现代农业产业技术体系建设专项资金项目——贝类产业技术体系科学家岗位专项经费(nycytx-47);广东省海洋渔业科技推广专项项目——鲍抗病良种选育及产业化(A2008899E01);广东省农业攻关项目——贝类产业推进关键技术研究与示范(2010B20201014)
许新(1983-),男,山东临沂人,硕士研究生,主要从事水产经济动物种苗工程与繁殖生物学方面研究.E-mail:xuxin_0169@126.com
*通讯作者,男,研究员,主要从事贝类病害学方面研究.E-mail:wjy104@163.com

