抗生素抗性组学研究进展
徐春燕,苏建强,鄢庆枇,马 英
(1.集美大学水产学院,福建省高校水产科学技术与食品安全重点实验室,福建厦门 361021;2.中国科学院城市环境研究所,城市环境与健康重点实验室,福建厦门 361021)
抗生素抗性组学研究进展
徐春燕1,2,苏建强2,鄢庆枇1,马 英1
(1.集美大学水产学院,福建省高校水产科学技术与食品安全重点实验室,福建厦门 361021;2.中国科学院城市环境研究所,城市环境与健康重点实验室,福建厦门 361021)
抗生素抗性问题已成为当前国际上的研究热点之一.对抗生素抗性组学的内涵、研究意义及国内外研究进展进行了综述,并论述了宏基因组学技术在抗性组学研究方面的应用,这有助于促进国内开展相关方面的研究.
抗生素抗性;抗生素抗性基因;抗生素抗性组学;宏基因组学
抗生素是 20世纪最重要的医学发现之一,自其被发现以来,它在医疗领域得到了广泛的应用,对控制人类感染性疾病发挥了巨大的作用.与此同时,由于兽药抗生素具有防治动物疫病和促进生长的双重功效,因此,在养殖业和畜牧业中,常以亚治疗剂量长期添加于动物饲料中.目前,在全球范围内几乎所有地区都采用抗生素来实现追求产量、提高经济效益的目的[1].
然而,由于抗生素的大量使用使许多细菌获得了耐药性,导致抗生素的治疗效果日益下降,许多疾病的救治显得越来越棘手.近年来,临床医学领域不断发生由抗生素抗药性造成对一些致病菌无法有效控制的恶性事件.其实,早在 20世纪 60年代抗生素第一次进入临床应用之前,就分离到抗性细菌.随着抗生素的使用,抗性细菌在数量、多样性以及抗性强度上都显著增大[2].目前,每种抗生素在临床的使用上都有其抗性细菌,多重耐药菌株的检出也相当普遍,甚至出现了能抵抗绝大多数抗生素的“超级细菌”,为滥用抗生素敲响了警钟.抗生素抗性已成为一个医学难题.研究发现,许多细菌是通过抗生素抗性基因 (Antibiotic resistance genes,ARGs)来实现其抗药性机理的[3-4],但对于细菌,尤其是环境细菌抗性基因的起源、分布和多样性,尚不清楚[5].
抗生素抗性组 (Antibiotic resistome)是 2006年W right等人提出的新名词,是指微生物中所有抗生素抗性基因的集合[6].此后,抗性组学的方法便广泛用来研究土壤、猪粪、活性污泥等各种环境中的抗性基因[5,7-8].本文综述了抗性基因污染持续残留和广泛扩散的特点,并详述了抗性基因研究的新方法—抗性组学方法的内涵和研究程序,使人们对抗性以及抗性基因有了更进一步的认识,为促进国内开展相关方面的研究,进而研制新的抗菌药物,减少抗性带来的负面影响提供了参考.
1 抗生素抗性基因 (ARGs)——一种新型环境污染物
研究表明,用于预防与治疗人类和动物感染的抗生素,包括促进养殖动物快速增长的促进剂,只有15%可被吸收利用,大约 85%未被代谢而随排泄物直接排放至环境中[9].因此,养殖废水、医院废水的排放可导致环境中的抗生素浓度升高,从而诱导抗性基因;另外,长期使用抗生素的人类和动物,其体内编码的抗性基因的抗性菌株也随肠道细菌被排出体外,抗性基因也随之进入环境.兽药抗生素和医用抗生素诱导出的抗性基因是环境中抗性基因污染的主要来源,随着抗菌药物的开发和使用,抗性基因在抗生素的选择压力下得到进化,其编码的产物对新药的耐药性逐渐增加.这些抗性基因具有很高的迁移性和活性,可通过可移动遗传元件,如质粒、转座子、整合子及基因组岛等,在同种甚至于不同种菌株间水平转移,使得抗性基因在环境中广为传播,造成二次污染[1,10].这种方式获得的抗药性机率高,形成后较稳定,是引起抗药性传播的主要原因[11].研究表明,一些未受人类影响的偏远地区以及未施用抗生素的人或动物体内,存在与致病菌相同的抗性基因[5,7,12].这说明,ARGs在环境中普遍存在,而日益增长的抗生素选择压力加速了 ARGs的水平转移,甚至在世界范围内广泛传播[13].环境中的抗性菌可能会将抗性基因转移到人体的病原菌,增加人致病菌的抗生素耐药性,使细菌感染性疾病的治疗更加棘手.另外,携带 ARGs的微生物菌株死亡后,携带 ARGs的裸露 DNA可释放到环境,在特定成分的土壤或粘土中长期存在,而不被环境中脱氧核糖核酸酶 (DNAase)降解,环境中这些裸露的 DNA分子又可转入到其他生物体细胞内[14-16],造成抗性基因的传播和扩散.ARGs的存在和快速传播对人类健康及养殖业造成了很大的威胁.
研究表明[17],ARGs存在于各种环境中,包括河流沉积物、灌溉渠、牛奶加工厂的出水、污水处理厂污水回用的排污渠以及饮用水处理厂,并且人为活动干扰的环境介质中抗性基因的浓度明显高于未受到人类干扰地区.基于上述研究结果,Pruden等人首次提出将 ARGs作为一种环境污染物.ARGs具有持续残留和广泛扩散的特点,这些特点使其具有重大的生态风险,理应将 ARGs作为一类新型环境污染物,并给予足够重视.
2 抗生素抗性组的内涵及研究意义
以前,抗生素抗性研究的重点在于临床病原菌的抗性机制,但是,这些研究无法分析抗性基因的来源和进化.了解抗性基因的起源和进化是一项非常艰巨的任务,目的是为了将来制造出让细菌无法产生抗性的药物.更广阔的视角应该是对所有抗性基因——包括病原菌和非病原菌抗性基因,甚至是潜在的抗性基因进行研究[2].2006年,W right等人提出将微生物中所有ARGs的集合叫做抗生素抗性组 (Antibiotic resistome)(图 1)[6].致病菌携带的抗性基因只占抗性组很小的一部分,抗性组还包括抗生素生产者携带的抗性基因、隐秘的抗性基因 (Cryptic embedded genes)以及前体基因 (Precursor genes).隐秘的抗性基因是指嵌入到细菌染色体上,但通常不表达或表达水平很低的基因.前体基因可以编码有适度抗生素抗性活性或与抗生素密切相关的蛋白质,在一定的选择压力下,这种基因可以进化为有效的抗性基因,它们是抗生素抗性的最终外部来源.
抗性组概念的提出对于现代医学的发展具有重要影响:首先,虽然体内共生微生物是引起感染的主要原因,但环境细菌潜在的抗药性要比共生菌强;另外,临床上发现的抗性很多都源于环境抗性基因.过去 60年里,抗菌药物的广泛使用已经为环境中高抗性基因转移到病原菌提供了必要条件.所以有必要拓宽对于抗性基因的研究范围,将抗性基因的研究扩大到广阔的环境中,更多地了解全球抗性的流行和多样性[18],为发现新的抗性基因,了解新的抗性机制,研发新的抗生素提供理论依据.同时,抗性组的研究能够为抗生素抗性组提供预警机制,当某种抗性出现在临床上时,能够指导采取正确的诊断技术和治疗策略.
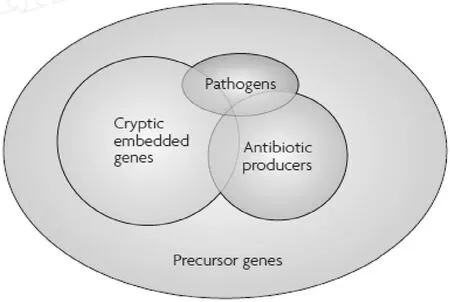
图1 抗生素抗性组[6]注:抗生素抗性组由所有的 ARGs组成,包括病原菌携带的抗性基因、抗生素生产者携带的抗性基因、隐秘的嵌入基因以及前体基因.出现在临床病原菌中的抗性基因,有一些与隐秘的抗性基因及抗生素生产者携带的抗性基因相似,因此这 3个集合有部分重叠.
3 抗生素抗性组学研究进展
大量的研究表明,临床上的抗生素抗性基因与环境中发现的抗性基因密切相关[18],环境 (包括非临床环境)是抗性基因的储库,而已知的抗性基因大部分来自临床病原体,对于非临床环境中的抗性基因则还未做充分研究[2].当前,随着分子生物学手段的发展和不断完善,它在方法学上为抗生素抗性组的研究提供了极大的可能性.
早期对抗性基因的研究主要是从分离培养的细菌中获得抗性基因,这种方法存在一定的局限性,自然环境中的大部分微生物至今仍无法培养[19],通过传统的分离培养细菌方法所获得的抗性基因只是抗性组的一部分.目前,应用较多的 PCR方法可以直接从环境样品中获得未培养细菌的抗性基因[20].Schmitt等人通过 PCR证明了土壤中四环素抗性基因的多样性[21].Chee-Sanford采用 PCR-DGGE和序列分析技术在养猪场附近的粪池中发现了 8种四环素抗性基因,分别为 tet(O)、tet(Q)、tet(w)、tet(M)、tetB(P)、tet(S)、tet(T)和 otr A[22].PCR技术的局限性在于它仅仅能得到与已知序列相似的基因,通常不能得到新的抗性基因[20].
宏基因组 (Metagenome)是 Handelsman等[23]于 1998年提出的新名词,是指生境中全部微小生物遗传物质的总和,既包含了可培养的又包含了未能培养的微生物基因.宏基因组学的方法是直接从环境样品中提取全部微生物的 DNA,构建宏基因组文库,并以功能基因筛选和测序分析为研究手段,研究环境样品所包含的全部微生物功能基因的多样性和进化关系.这种方法克服了分离培养和 PCR两种方法的局限性,能方便、快捷地获得抗性组以及新的抗性基因,极大地扩展了微生物资源的利用空间,能广泛地应用于抗性组的研究.
2004年,Riesenfeld等人[20]直接从土壤样品中提取 DNA,构建了 6个宏基因组文库,共包含 5.4 Gb的土壤DNA,筛选出 9个表达氨基糖苷类抗生素抗性的克隆子和 1个表达四环素抗性的克隆子,除了一个抗性基因之外,其余所有抗性基因所编码的氨基酸序列都与先前报道的序列明显不同 (相似度 <60%),这说明土壤是抗性基因的储库,通过宏基因组学的方法可以发现新的抗性基因,能客观反映环境中抗性基因的多样性,这是第一次关于运用宏基因组学的方法来研究抗性基因的报道.Sommer等人[13]采集了 2个健康人体的唾液和排泄物来研究人类口腔和肠道微生物的抗性基因.首先,构建 2个大小为 9.3×109bp的宏基因组文库,进行 13种抗生素的功能筛选,并对筛选得到的抗性克隆子进行测序,所得序列与已知病原菌抗性基因平均相似度为 60.7%;而同时进行的通过培养方法获得的抗性基因,其中有 95%与已知病原菌抗性基因的相似度超过 90%,有几乎一半的基因与已知抗性基因的相似度为 100%.Mori等人[8]构建了活性污泥的宏基因组文库,大小为 3.2 Gb,并筛选了博来霉素抗性基因,发现 2个博来霉素抗性基因 (4H7,7A10)具有很大差异,并且不同于博来霉素产生菌和临床上抗性菌株携带的抗性基因.Allen等人[5]对偏远的阿拉斯加的土壤进行了宏基因组学的分析,发现尽管没有人为活动的干扰,未被污染地区的土壤微生物群落也携带β-内酰胺抗生素抗性基因.功能筛选和日益增长的宏基因组数据库揭示了环境中的抗性基因水平远远超出预料[18],非临床环境是新的抗性基因的巨大储库.
4 研究展望
目前,对于抗性组的研究主要集中于土壤环境,其原因在于土壤为抗性基因的巨大储库.首先,大多数抗生素是由土壤放线菌产生的,抗生素生产者都携带具有自我保护作用的抗生素抗性基因,这些基因与临床抗性病原菌中发现的可移动遗传因子同源;其次,多数抗生素未被人或动物代谢而直接排出体外,携带抗性基因的抗性菌株也随之排出,其粪便首先进入土壤,之后随着抗性基因的水平转移,使得抗性基因在各种环境中广泛传播.
研究表明,抗性基因已广泛存在于整个微生物界,除了土壤抗生素抗性组,对于其他环境中抗性基因的研究也是必要的,例如,水产养殖环境、地下水系统、河流沉积物甚至未施用抗生素或未受人类影响的环境.因此应扩大抗性组的研究范围,探索环境系统中抗性基因的多样性和进化关系,以促进新一代抗菌药物的研发;甚至还可以根据抗性组来预测未来可能出现的抗生素抗性[24],以此来指导正确的治疗措施.
国内对于环境中ARGs的研究才刚刚起步[1],有关抗性组学的报道更未见到,还有很多的问题亟待解决 ,例如:
1)除了基因突变以及基因水平转移外,是否还存在其他尚未发现的能促使 ARGs在环境中持久存在并广泛传播的因素?
2)基因的水平转移使得环境细菌获得了抗生素抗性,那么是什么因素促使了基因的水平转移?是否可以通过控制这种因素来抑制基因的水平转移?
3)临床上发现的ARGs有一些与环境细菌携带的抗性基因极为相似,但两者的关系及环境中的抗性基因进入人类致病菌的机制尚不清楚.
4)ARGs在细菌细胞中除了编码抗生素抗性外,是否还有其他功能?
[1]罗义,周启星.抗生素抗性基因 (ARGs)—一种新型环境污染物[J].环境科学学报,2008,28(8):1499-1505.
[2]WR IGHT GD.The antibiotic resistome:the nexus of chemical and genetic diversity[J].Microbiology,2007,5:175-186.
[3]YOO M H,HUH M D,K IM E H,et a1.Characterization of chloramphenicol acctyltransferase gene bymultiplex polymerase chain reaction in multidrug—resistant strains isolated from aquatic environments[J].Aquaculture,2003,217:11-21.
[4]AGERSO Y,GUARDABASSI I. Identification of Tet39,a novel class of tetracycline resistance deter minant inAcinetobacter spp.of environmental and clinical origin[J].J Antimicrob Chemother,2005,55(4):566-569.
[5]ALLEN H K,MOE L A,RODBUMRER J,et al.Functionalmetagenomics reveals diverseβ-lactamases in a remote Alaskan soil[J].The IS ME Journal,2009,3:243-251.
[6]D’COSTA V M,MCGRANN K M,HUGHES D W,et al.Sampling the antibiotic resistome[J].Science,2006,311(5759):374-377.
[7]KAZ IM IERCZAK KA,SCOTT K P,KELLYD,et al.Tetracycline resistome of the organic pig gut[J].Appl EnvironMicrobiol,2009,75(6):1717-1722.
[8]MOR IT,M IZUTA S,SUENAGA H,et al.Metagenomic screening for bleomycin resistance genes[J].Appl EnvironMicrobiol,2008,74(21):6803-6805.
[9]周启星,罗义,王美娥.抗生素的环境残留、生态毒性及抗性基因污染[J].生态毒理学报,2007,2(3):243-251.
[10]蒋培余,潘劲草.细菌遗传元件水平转移与抗生素抗性研究进展[J].微生物学通报,2006,33(4):167-171.
[11]曹文斌,王文魁,薛静,等.细菌耐药机制及其控制对策[J].中国畜牧兽医,2005,32(11):52-54.
[12]SOMMERM O A,DANTAS G,CHURCH GM.Functional characterization of the antibiotic resistance reservoir in the human microflora[J].Science,2009,325(5944):1128-1131.
[13]MART INEZ J L.Environmental pollution by antibiotics and by antibiotic resistance deter minants[J].Environ Pollut,2009,157(11):2893-2902.
[14]BLUM S A E,LORENZ M G,WACKERNAGEL W.Mechanis m of retarded DNA degradation and prokaryotic origin of DNAases in nonsterile soils[J].SystApplMicrobiol,1997,20(4):513-521.
[15]CRECCH IO C,RUGGIERO P,CURCIM,et al.Binding ofDNA fromBacillus subtilison montmorillonite-humic acids-aluminum or iron hydroxypolymers:Effects on transformation and protection againstDNAase[J].Soil Sci Soc Am J,2005,69(3):834-884.
[16]H ILL K E,TOP EM.Gene transfer in soil systems usingmicrocos ms[J].FEMSMicrobiol Ecol,1998,25(4):319-329.
[17]PRUDEN A,PEIR,STORTEBOOM H,et al.Antibiotic resistance genes as emerging contaminants:Studies in Northern Colorado[J].Environ Sci Technol,2006,40(23):7445-7450.
[18]D’COSTA V M,GR IFFITHS E,WR IGHT G D.Expanding the soil antibiotic resistome:exploring environmental diversity[J].CurrOpinMicrobiol,2007,10(5):481-489.
[19]AMANN R I,LUDW IGW,SCHLEIFER K H.Phylogenetic identification and in situ detection of individualmicrobial cells without cultivation[J].Microbiol Rev,1995,59(1):143-169.
[20]R IESENFELD C S,GOODMAN R M,HANDELS MAN J.Uncultured soil bacteria are a reservoir of new antibiotic resistance genes[J].EnvironMicrobiol,2004,6(9):981-989.
[21]SCHM ITT H,STOOB K,HAMSCHER G,et al.Tetracyclines and tetracycline resistance in agricultural soils:microcosm and field studies[J].Microb Ecol,2006,51(3):267-276.
[22]CHEE-SANFORD J C,AM INOV R I,KRAPAC I J,et al.Occurrence and diversity of tetracycline resistance genes in lagoons and groundwater underlying two s wine production facilities[J].Appl EnvironMicrobiol,2001,67(4):1494-1502.
[23]HANDELS MAN J,RONDON M R,BRADY S F,et al.Molecular biological access to the chemistry of unknown soil microbes:a new frontier for natural products[J].Chem Biol,1998,5(10):245-249.
[24]MARTíNEZ J L,BAQUERO F,ANDERSSON D I.Predicting antibiotic resistance[J].Nat RevMicrobiol,2007,5:958-965.
Research Progresses of Antibiotic Resistom ics
XU Chun-yan1,2,SU Jian-qiang2,YAN Qing-pi1,MA Ying1
(1.The KeyLaboratory of Fujian’s Higher Institutions for Green Foods and the Science and Technology ofAquaculture,Fisheries College,JimeiUniversity,Xiamen 361021,China;2.The KeyLaboratory of Environment and Health in Cities,The Urban Environmental Institute in Chinese Academy of Sciences,Xiamen 361021,China)
Over the last decades,there has been growing concerns on antibiotic resistance. In the paper,the connotation,research significance and research progresses of the antibiotic resistomics were reviewed,and the application ofmetagenomic in the study of antibiotic resistome was discussed.The review will be helpful for the research in related fields.
antibiotic resistance;antibiotic resistance genes(ARGs);antibiotic resistomics;metagenomics
X 171 < class="emphasis_bold">文献标志码:A
A
1004-1729(2011)02-0188-05
2011-01-19
国家自然科学基金 (31070101);近海海洋环境科学国家重点实验室 (厦门大学)开放基金(MEL0701);集美大学创新团队基金 (2010A003)
徐春燕 (1986-),女,山东泰安人,集美大学水产学院 2008级硕士研究生.
马英,女,博士,副教授.E-mail:maying@jmu.edu.cn

